Command Palette
Search for a command to run...
デビッド・ベイカーのチームの最新の研究では、タンパク質配列生成モデルを使用して、非常に高い成功率で重複遺伝子設計を実現しています。
1977年、イギリスの生化学者フレデリック・サンガーは、ΦX174バクテリオファージのゲノムを分析した際に、初めて認識を覆す現象を発見しました。この5.4kbのDNA分子によってコード化されたタンパク質の全長が、その物理的容量の限界をはるかに超えていたのです。配列解析の結果、2 組の遺伝子が異なる読み取りフレームを介して同じ DNA 領域を共有します。この現象は重複遺伝子 (OLG) と呼ばれ、ウイルスの世界では非常に一般的です。たとえば、B 型肝炎ウイルスの 3.2kb ゲノムでは、50% 領域が重複する遺伝子の複数のペアでカバーされており、既知のウイルスの半数以上に少なくとも 1 つの OLG が含まれています。
この直感に反するゲノム設計は、ウイルスの生存の知恵を隠しています。ウイルスが宿主細胞内の限られた空間を奪い合うとき、OLG は「遺伝子スタッキング」戦略を使用して、1 つのヌクレオチドが同時に 2 つのコドンのエンコードに参加できるようにし、コンパクトな配列で機能の重ね合わせを実現します。サンガーチームの発見が関連研究の始まりとなった。その後の研究で、OLG によってコードされるタンパク質は多くの場合、高度な配列縮重を持ち、そのアミノ酸配列許容度により、2 つの機能タンパク質が同じ DNA 鎖上に共存できることが示されました。さらに重要なのは、明確な三次元構造を形成する必要があるタンパク質であっても、配列の配置によって異なる読み取りフレームでの折り畳みの互換性を実現できることです。
しかし、核心的な疑問は常に残ります。標準的な遺伝コードの下では、アミノ酸配列の縮重により、重複するフレームワーク内での任意の機能的タンパク質ペアの折り畳みをサポートできるのでしょうか?ヌクレオチドが二重コーディングを考慮しなければならない場合、タンパク質折り畳みの配列空間は厳しく制限されるのでしょうか?
ワシントン大学の David Baker 氏のチームは最近、高度な生成モデルを使用して合成 OLG 設計研究を実施し、エンジニアリングの観点からその実現可能性を検証しました。研究チームは、高度に秩序立った新規に設計されたタンパク質構造をコードするために、2 つのタンパク質ファミリーの重複配列を設計しました。コンピューターシミュレーションと実験検証の両方で非常に高い成功率が示されました。重複制約下では、代替リーディングフレームは明確な三次元折り畳みに対応できるだけでなく、その構造安定性と機能的完全性は重複しない配列に匹敵します。
関連する研究成果は、「タンパク質配列の深層生成モデルを用いた重複遺伝子の設計」というタイトルで、bioRxiv にプレプリントとして公開されています。

用紙のアドレス:
https://doi.org/10.1101/2025.05.06.652464
オープンソース プロジェクト「awesome-ai4s」は、100 を超える AI4S 論文の解釈をまとめ、大規模なデータ セットとツールを提供します。
https://github.com/hyperai/awesome-ai4s
データセット: 多次元データリソースと分析手法の統合
本研究では、遺伝コードの可塑性とタンパク質設計への応用を分析するために、多次元データリソースと分析手法を統合し、理論設計から実験検証までの完全な研究チェーンを構築します。
遺伝コードランダム化研究では、この研究では、アミノ酸の順列とコドンシャッフラー戦略に基づいて 1,000 個の代替コドンの組み合わせが生成されました。このデータセットは、明確なアルゴリズム設計を通じてサンプルの多様性と均一性を確保し、コドン再配置の機能的影響を評価するための統計ベンチマークを提供します。
同時に、本研究では代表的な二次構造標的タンパク質を3つ選択し、9つの組み合わせを構築することで、変数を制御するという前提の下で実験条件の標準化を実現し、遺伝子コードの変異とタンパク質構造機能の相関分析を効果的に結び付けました。
タンパク質ドメイン配列分析段階では、Pfam 37.0 データベースからシード配列を抽出し、長さ 100 アミノ酸のサブ領域をランダムにサンプリングし、マルコフ モデルを使用して k-mer 分布を保持した合成タンパク質配列を生成しました。この方法は、バイオインフォマティクススクリーニングと統計モデリングを組み合わせたもので、天然タンパク質の配列特性を保持するだけでなく、制御可能なランダム変数を導入することでコントロールサンプルも作成します。これは、その後の分析のために自然の特性と人工的に設計された特徴を組み合わせた革新的なデータセットを提供します。
タンパク質言語モデルの埋め込み解析では、研究者らはESM2、ESM3、ProstT5の隠れ層の特徴を抽出し、位置平均化後にUMAPアルゴリズムを通じて2次元空間に投影した。 n_neighbors = 15 などのパラメータを正確に設定することで、高次元シーケンス機能が直感的なトポロジカル マップに変換されます。シーケンスの類似性構造を維持しながら、モデル間の比較のための統一された視覚化フレームワークを提供します。これは、計算生物学とデータ視覚化の最先端の組み合わせを示しています。
実験検証段階では、研究者らは、重複する 192 個の遺伝子をクローン化して組み換え、384 個のフレームシフトしたタンパク質変異体を生成した。実験では主要なパラメータが厳密に制御され、37°C で 20 時間の培養により大腸菌発現システムの安定性が確保され、6M グアニジン塩酸塩勾配再生スキームにより封入体タンパク質の正しい折り畳みが確保されました。分子設計から精製、特性評価までのプロセス全体を定量的に制御することで、研究結果の再現性が向上するだけでなく、タンパク質工学のための標準化された実験パラダイムも提供されます。
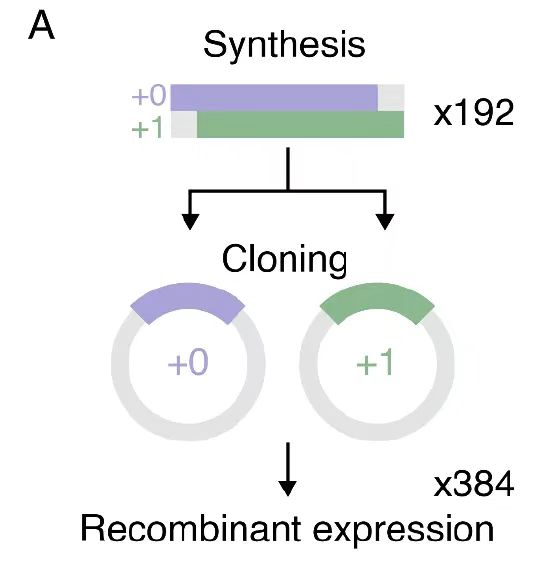
生成モデルに基づくOLG設計:マルチフレームワーク対応のシーケンス同期最適化手法
本研究では、重複遺伝子(OLG)設計におけるコーディングフレームの相互依存性によって引き起こされる配列空間制約の問題に効果的に対処し、2つのタンパク質配列の適応性の同時最適化を実現する計算アルゴリズムを開発しました。
アルゴリズム設計レベルでは、この研究では EvoDiff-MSA や ProteinMPNN などの生成モデルを統合しました。前者は MSA Transformer アーキテクチャに基づいており、自己回帰拡散ターゲット トレーニングを通じてターゲット タンパク質の多重配列アライメント (MSA) に基づいて設計シーケンスを生成できます。後者は、構造条件生成モデルとして、3次元構造が与えられた場合に対応するタンパク質配列を設計することができます。どちらのモデルも、位置ごとのマスキングと制約付きサンプリング戦略を使用して、さまざまなオフセットとフレーム配置をカバーする重複シーケンス ライブラリを生成しました。
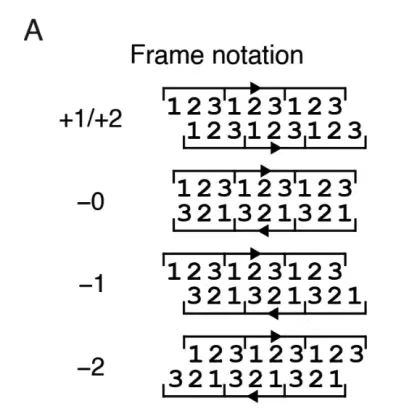
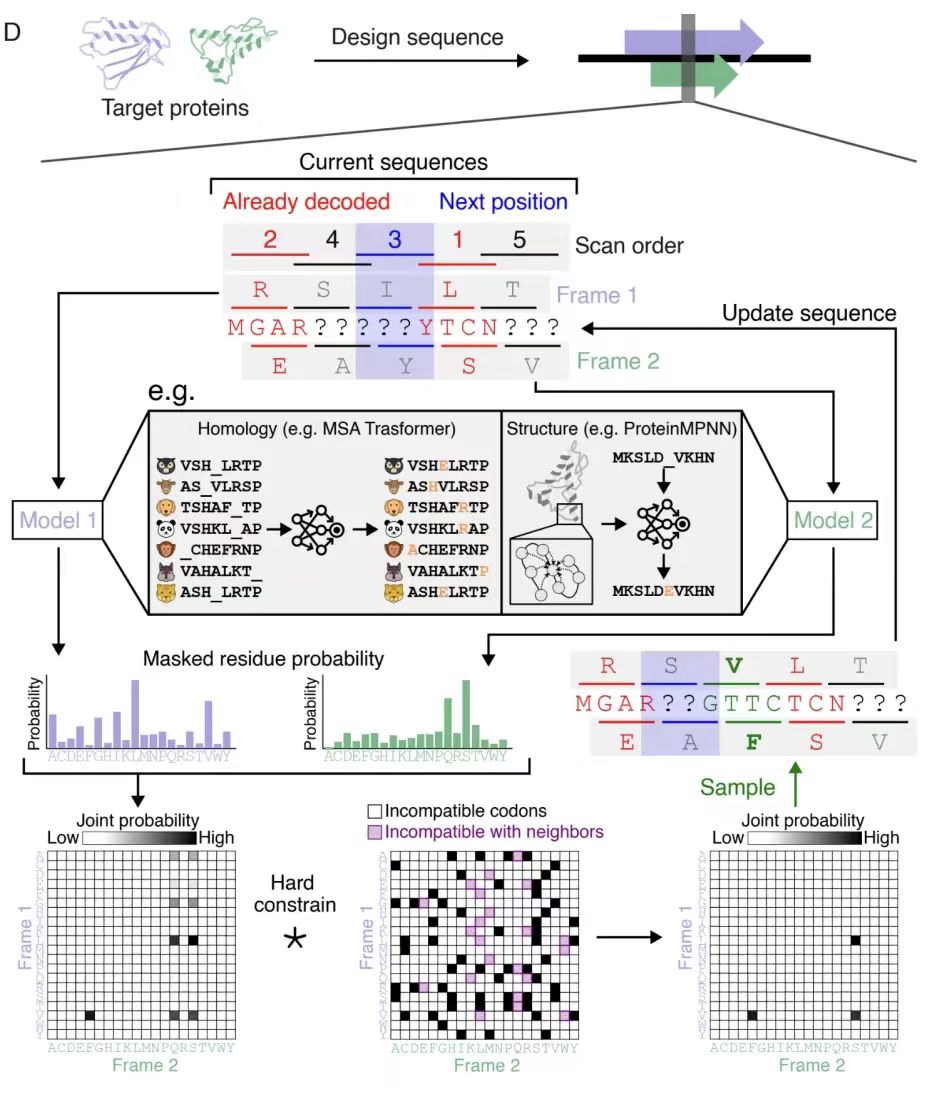
下の図 A に示すように、この研究では、5 つの可変読み取りフレーム (+1、+2、-0、-1、-2) の位相制約に対してフレームごとの反復サンプリング戦略を提案しました。
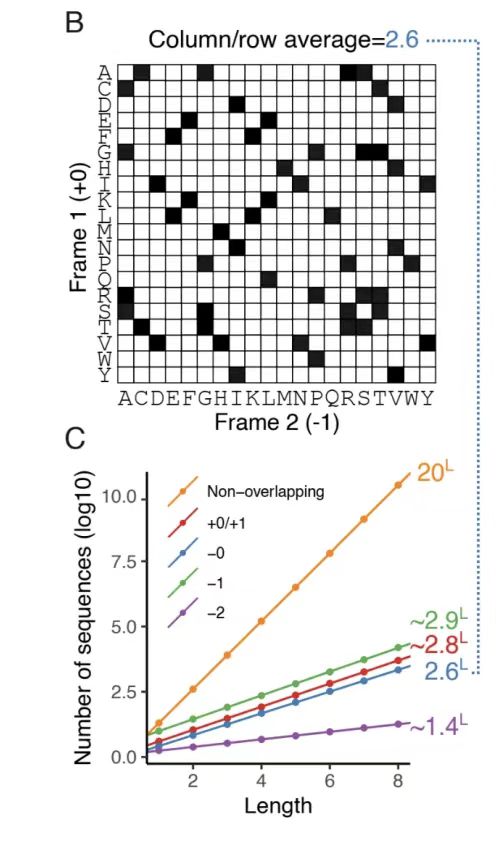
下の図 B に示すように、-0 フレームワークのアミノ酸互換性マトリックスを分析すると、参照フレームワークの 1 つの位置に平均 2.6 個の互換性のあるアミノ酸の選択肢があり、52ⁿ (n は配列の長さ) の潜在的な重複配列ペアが形成されることがわかりました。これは、遺伝コードの縮重によってもたらされる設計スペースを浮き彫りにしています。他のフレームワークの自由度は、下の図 C に示すように、モンテカルロ近似を使用して定量化されました。結果から、+1 フレームワークと -1 フレームワークの自由度はそれぞれ約 2.8 と 2.9 と高い一方、-2 フレームワークではコドン縮重の効率が低いため自由度が大幅に制限されていることがわかります (約 1.4)。
最後に、下の図 D に示すように、アルゴリズムはシーケンスの位置 (スキャン順序) を体系的にスキャンし、隣接するアミノ酸の制約と組み合わせて各スキャンの結合確率行列を動的に更新します。複数回の反復処理の後、生成された重複シーケンス ペアがフレームワークの互換性を満たすことが保証されます。この戦略は、位相オフセットを備えた複雑なフレームワークに拡張でき、スキャン順序をバイアスすることで設計品質を最適化し、生成モデルの反復デコードに重要な制約を提供します。
天然テンプレートの限界を超えて:任意のタンパク質ペアの合成OLGの効率的な生成
実験設計は、相同性に基づく OLG 設計評価、高度に秩序化されたタンパク質主鎖構造の重複実現可能性分析、OLG 配列の進化的アクセシビリティ研究、実験検証など、複数の方向をカバーしています。
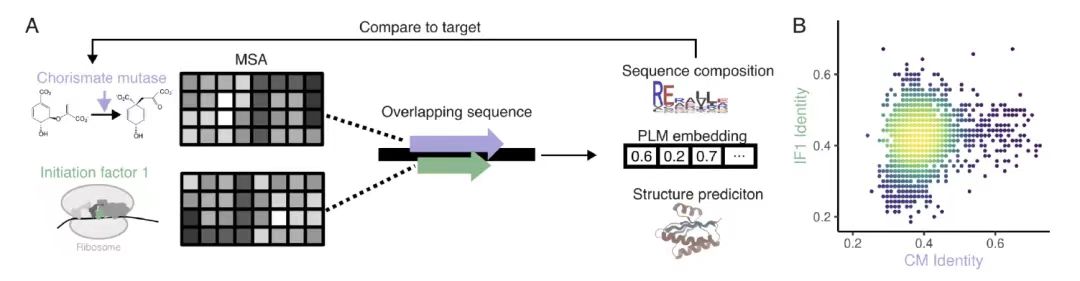
相同性に基づくOLG設計評価では、下の図 A に示すように、研究チームは細菌のシキミ酸ムターゼ (CM) と翻訳開始因子 1 (IF1) をターゲットとして選択し、EvoDiff-MSA 生成モデルを使用し、条件付きコンテキストとして多重配列アライメント (MSA) を使用して、位置ごとのマスキングと制約付きサンプリングを通じて 3,307 個の完全に重複する配列設計を生成しました。
下の図Bに示すように、設計された配列と天然の配列との相同性は38.9%(CM)と42.3%(IF1)のみであるが、しかし、タンパク質言語モデルの埋め込み解析により、2次元空間におけるその分布は自然配列と非常によく一致していることが示されました。これは、設計されたこれらの配列がターゲットタンパク質ファミリーの信頼できるメンバーであることを示しており、天然タンパク質ファミリーに対するアルゴリズムの設計能力を検証しています。
高度に秩序化されたタンパク質骨格構造の重なり合いの実現可能性を探る際、下の図 A に示すように、研究者らは ProteinMPNN 構造条件生成モデルを使用して、15 個の新規に生成された主鎖構造 (α、β、および混合折りたたみカテゴリをカバー) に対して 56,250 個の重複設計と 33,000 個の重複しない設計を生成しました。下の図Bに示すように、AlphaFold2の評価データは、重複設計の平均 pLDDT 値は 90.2 で、非重複設計の 92.0 に近かった。
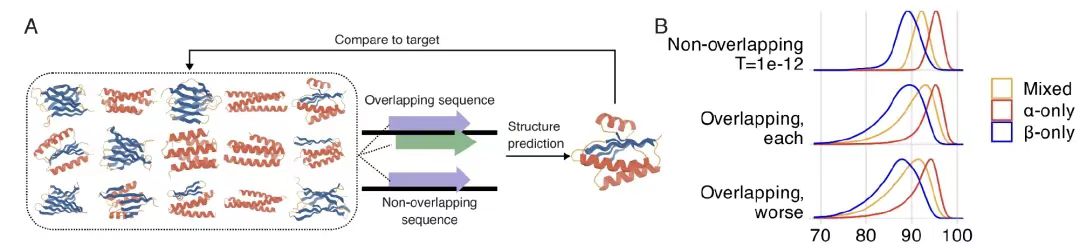
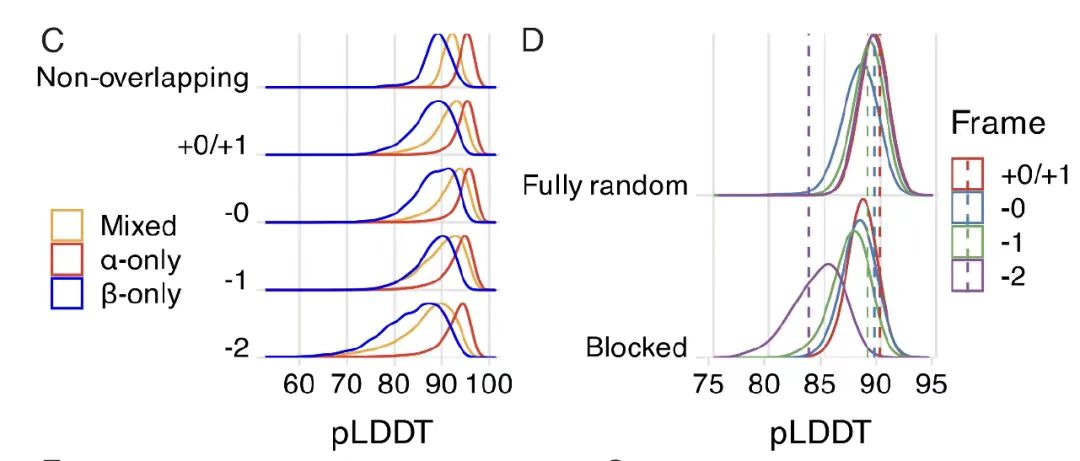
さらに分析を進めると、下の図 CD に示すように、コドンの縮重の効率が低いために -2 フレームのみパフォーマンスが悪かったことが判明しました。ランダム化遺伝コード解析により、天然遺伝コード (SGC) は OLG のエンコードにおいて大きな利点があり、-2 フレームを除いてパフォーマンスが良好で、高度に縮退したアミノ酸の構成が優先されることが示されました。SGC構造が重複配列の実現可能性に影響を及ぼすメカニズムが明らかになった。
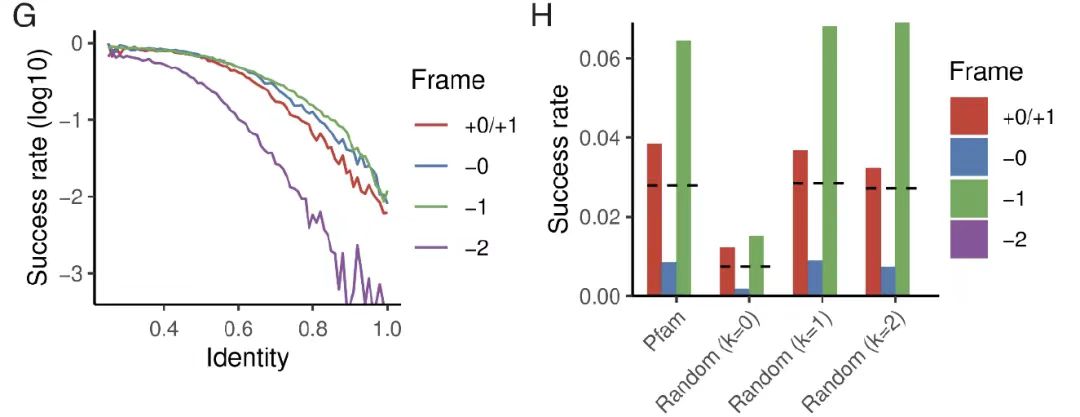
進化的アクセシビリティ研究では、研究チームは、一定数の変異を持つ種子タンパク質配列から出発しました。次の図GFに示すように、研究では、突然変異ゼロの極端な条件でも、約 1% 設計が高い構造安定性 (pLDDT>85、TM>0.7) を達成できることがわかりました。天然の Pfam 配列を親として使用すると、成功率は 3% まで増加し、この結果は一次構成偏差を保持したランダム配列と一致していました。これは、高度に最適化された天然タンパク質が、大きな配列変更なしに代替フレームワークで新しいタンパク質を収容できることを明確に示しており、進化レベルでの OLG の実現可能性を検証しています。
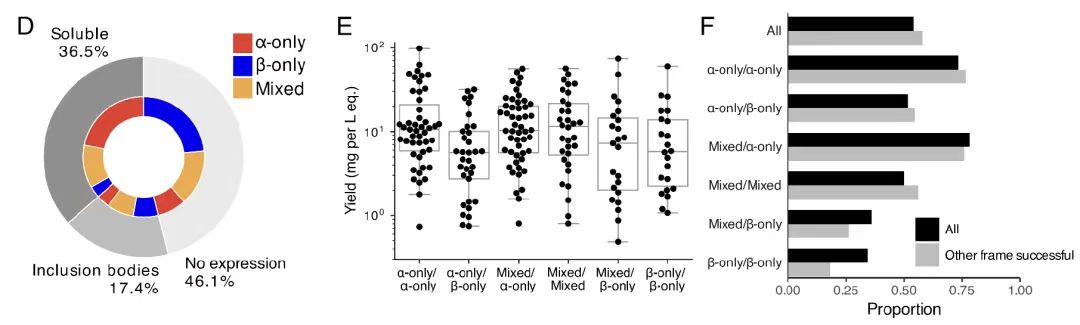
最後の実験検証部分では、研究チームは192個の重複配列に対して組み換え発現と構造特性評価を実施しました。結果は、図Bに示すように、54% の個々のタンパク質は正常に発現され、そのほとんどは予想された二次構造と高い熱安定性を有していました。
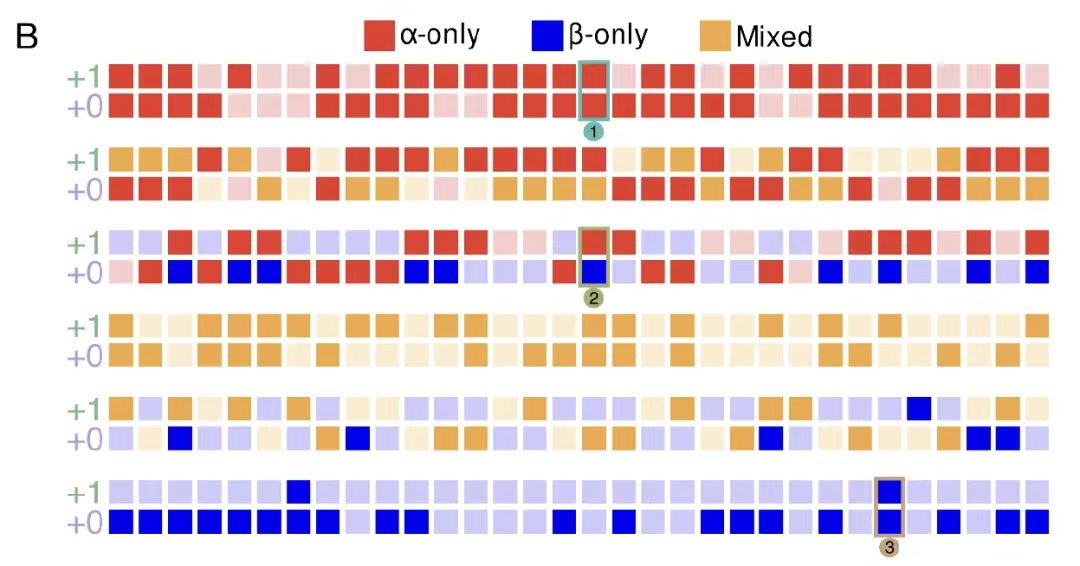
さらに、以下の図DFに示すように、成功率はタンパク質の二次構造の含有量によって異なり、αヘリックスタンパク質の成功率が最も高くなりました。さらに、31% の重複ペアが正常に精製され、一方のフレームワークの成功がもう一方のフレームワークの成功に影響を与えることはありませんでした。これらの結果は、OLG シーケンスの高い実現可能性と実験検証率をさらに裏付けるものであり、機能的かつ構造的に安定した重複タンパク質の設計におけるアルゴリズムの有効性を実証しています。
合成生物学分野のフロンティア探査、OLGエンジニアリングの応用が徐々に深化
合成生物学の分野では、世界各地の研究チームや企業が重複遺伝子 (OLG) の工学応用について深く研究しています。
例えば、清華大学の朱庭の研究グループはミラー生物システムの研究で大きな進歩を遂げ、ミラーPfu DNAポリメラーゼの完全な化学合成に成功しました。これにより、キロベース長のミラーDNAの組み立てが現実のものとなるだけでなく、ミラーDNAに基づく情報保存技術も開発されます。この技術は、ミラー遺伝子のコーディング戦略を使用して、OLG の双方向機能重ね合わせに新しいアイデアを提供します。ミラー DNA の二重らせん構造が天然の遺伝情報とミラーの遺伝情報の両方を保持すると、配列空間の利用が大幅に改善され、人工ゲノムのコンパクトな設計に重要な基盤が提供されます。
* 論文リンク:https://www.nature.com/articles/s41587-021-00969-6
さらに、マサチューセッツ工科大学のクリストファー・ボイト氏のチームは、遺伝子回路設計に基づいた合成生物学プラットフォームを開発しました。彼らは、原核生物の遺伝子クラスターの調節ロジックを再構築することで、代謝経路のモジュールアセンブリを成功裏に達成しました。この技術的な道筋は、OLG の設計哲学と密接に一致しています。複数の機能遺伝子が重複配列を通じてコンパクトな遺伝子モジュールを形成すると、ゲノムの冗長性を削減できるだけでなく、協調的な発現を通じてシステムの安定性も向上します。例えば、研究チームが設計した人工窒素固定遺伝子クラスターは、OLG戦略を採用して複数の主要酵素のコード配列を同じDNA領域に圧縮し、触媒効率を確保しながら宿主細胞の代謝負担を大幅に軽減しました。
* 論文リンク:https://www.nature.com/articles/s41467-022-33272-2
これらの研究は、自然進化における OLG の広範な存在を明らかにするだけでなく、工学的手段を通じてその生物物理学的実現可能性も検証していることは注目に値します。この記事で紹介されている研究では、David Baker 氏のチームがディープラーニング モデルを使用して合成 OLG を設計し、コンピューター シミュレーションで天然配列に匹敵する構造安定性を示しました。実験検証の高い成功率は、重複コーディングの生物学的適合性をさらに証明しています。基礎研究から応用変換までのこの完全な閉ループは、合成生物学の設計ロジックを再構築し、革新的な医薬品開発、精密診断、細胞治療など多くの分野で新たなブレークスルーをもたらすことが期待されています。
参考文献:
1.https://www.tsinghua.edu.cn/info/1181/86148.htm
2.https://tech.huanqiu.com/article/9CaKrnJUV0x
3.https://news.bioon.com/article/4161e88572ad.html








