Command Palette
Search for a command to run...
ICLR 2025口頭発表に選ばれた清華大学AIRの周浩氏のチームは、タンパク質ファミリーの進化を解明するためのタンパク質事前トレーニングの新しいパラダイムを提案した。

清華大学の AIR GenSI 研究グループと清華大学薬学部は共同で、タンパク質ファミリー固有の生成モデリングツールである ProfileBFN (Profile Bayesian Flow Network) を提案しました。 ProfileBFN は、多重配列アライメント (MSA) プロファイルの観点から離散ベイジアン フロー ネットワークを拡張し、効率的なタンパク質ファミリーの設計を実現します。実証的結果は、ProfileBFN は、多様で新しいファミリータンパク質を生成しながら、ファミリーの構造的特徴を正確に捉えることができます。

関連する結果は、「プロファイル ベイズ フローによるタンパク質ファミリー設計の制御」というタイトルで ICLR 2025 の口頭発表論文に選ばれました。同時に、チームのもう一つの成果である CrysBFN も ICLR 2025 Spotlight に選ばれました。研究論文のタイトルは「マテリアル生成のための周期的ベイズフロー」です。
前回のセッションでは、チームは幾何学的ベイジアンフローネットワーク GeoBFN を提案し、その関連成果が「ベイジアンフローネットワークによる 3D 分子の統合生成モデリング」というタイトルで ICLR 2024 口頭発表に選出されました。

論文リンク:
オープンソース プロジェクト「awesome-ai4s」は、200 を超える AI4S 論文の解釈をまとめ、膨大なデータ セットとツールを提供します。
https://github.com/hyperai/awesome-ai4s
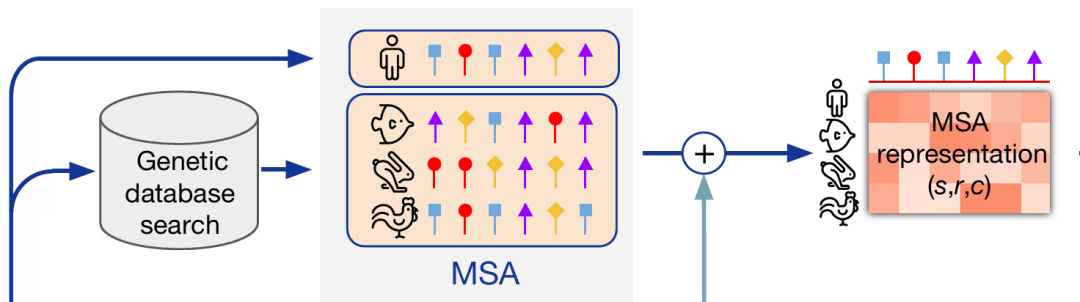
多重配列アライメント:タンパク質構造予測の基礎
多重配列アライメント (MSA) とは、3 つ以上の生物学的配列 (DNA、RNA、またはタンパク質) を整列させるプロセスを指します。多重配列アライメントは、機能的、構造的、または進化的関係により類似する領域を発見および識別するのに役立ち、生物学的高分子間の関係についてより包括的な視点を提供します。
近年、MSA 情報の利用はタンパク質設計の重要な部分となっています。 AlphaFold や ESM などのマイルストーン作品には、MSA 情報をエンコードする特別なモジュールがあります。
成功の順序は数多くあり、失敗の順序も数多くあります。
MSA は進化に関する情報の宝庫ですが、既存のモデルではそれを明らかにする能力を過大評価しているようです。技術の発展に伴い、深層生成モデル入力の MSA 深度は増加し続けていますが、その効果がボトルネックに遭遇し、MSA 情報を追加することの費用対効果に疑問が生じています。根本的な原因は、MSA の量と質の両方に重大な不確実性があることです。
研究者は、多重配列アライメントにおいて一定の類似性を満たす配列を相同配列と呼びます。量的に見ると、一部の「孤児」タンパク質では相同配列が 10 個以下であるのに対し、一部のタンパク質では 10,000 個を超える相同配列を検索できるため、大規模モデルでは大きな混乱が生じ、リソースの浪費や効率性への影響が生じます。
実際、自然の驚異は人間の想像を超えています。数十億年にわたる進化の中で、収束構造は自然選択の影響を反映し、突然変異は進化の新たな可能性をもたらします。特殊な環境におけるこれらの特殊な種は、進化樹の始まりにおける本来の外観情報を保持していることが多く、それがまさに共進化理論の演繹の基礎となります。相同配列をモデル入力として使用すると、この情報は大量の他の無関係な情報によって圧倒され、高確率の表現のみをモデル化できるようになります。これに対処するために、ProfileBFN は、相同配列の各クラスターを、数に依存しない統一された表現としてモデル化します。
優れた相同配列には、可能な限り多くの相同情報が含まれている必要があります。実験によれば、ほとんどの場合、情報エントロピーが最大の少数の相同配列を使用すると、数百の相同配列を使用する場合と同じ効果が得られます。いくつかの相同配列はわずか数個のアミノ酸が異なるだけであり、モデルに誤解を招くような冗長な情報を多く提供します。
プロフィール:次世代タンパク質台座モデルの礎
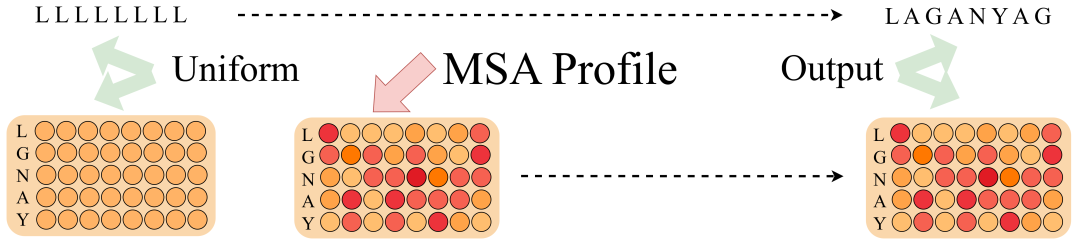
科学は発見に基づいています。ProfileBFN の革新性は、元の MSA に存在する大量の情報冗長性を発見したことにあります。 100 個の相同配列を情報エントロピー法に従ってソートすると、モデルは最初の 20 個だけをトレーニングに使用して同じ効果を達成できます。これを実行するには、単一のシーケンスと複数のシーケンス間のブリッジを確立する必要があり、そのためにプロファイルが表示されます。
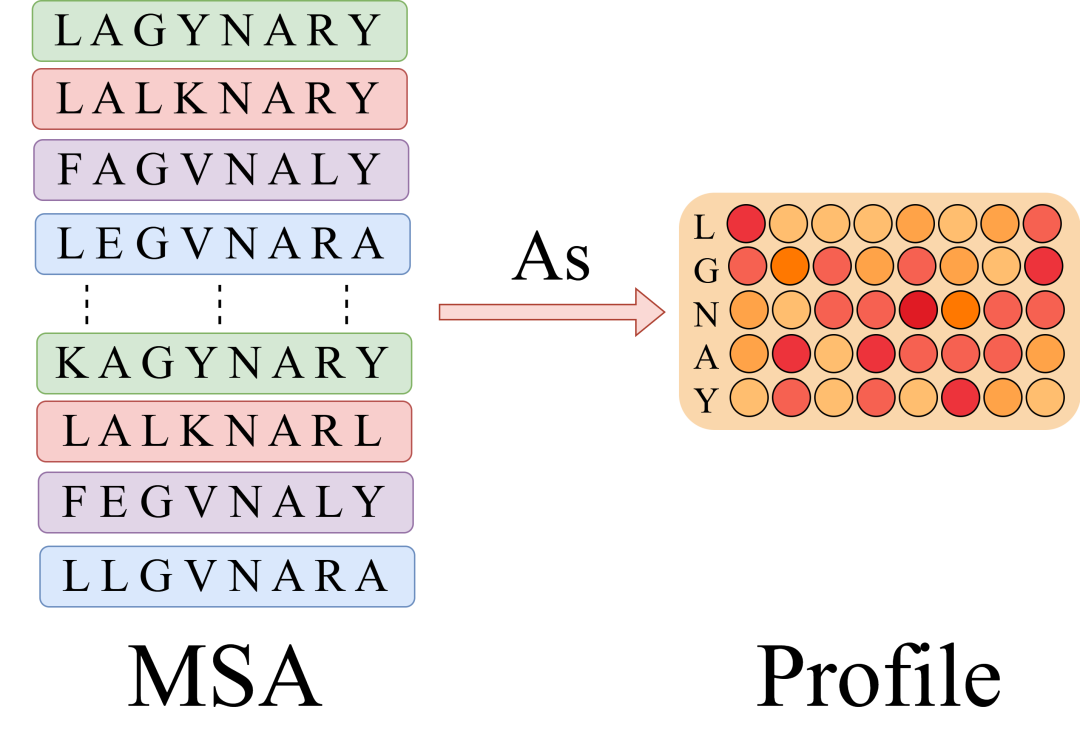
直感的に理解するために、プロファイルは、複数の配列アライメントにおけるアミノ酸の出現回数の列ごとの統計です。さらに、長さがそれぞれ 100 の相同配列が 1w 個ある場合、Profile はそれらを [10000,100] から [20,100] (共通アミノ酸 20 個) のリストに直接圧縮し、計算の複雑さを大幅に簡素化します。特に、各列に 1 が 1 つだけあるという点を除いて、単一のシーケンスも特別なプロファイルと見なすことができます。
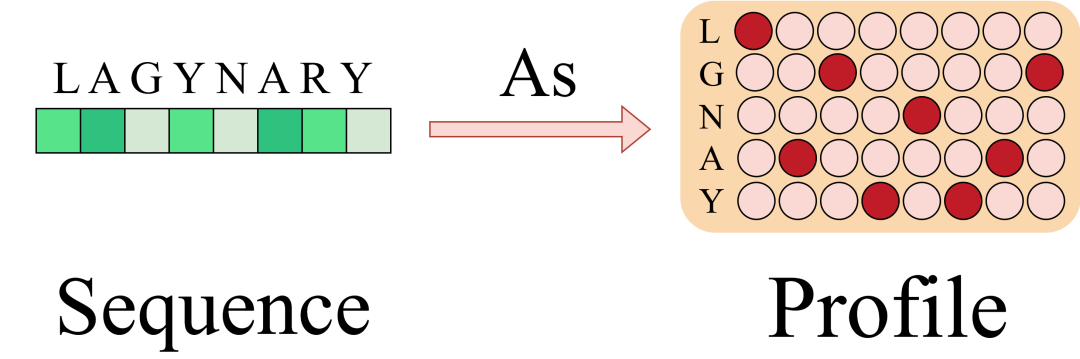
ProfileBFN では、MSA から Profile への圧縮によって、当初予想されていた重大な情報損失が発生しないだけでなく、モデルのパフォーマンスが大幅に向上することもわかりました。これは次のように理解できます。プロファイルを構築する大きな波の中で、それぞれの相同配列は、この位置に現れるアミノ酸の種類に投票し、小さな矛盾を隠し、全体的な傾向を強調します。
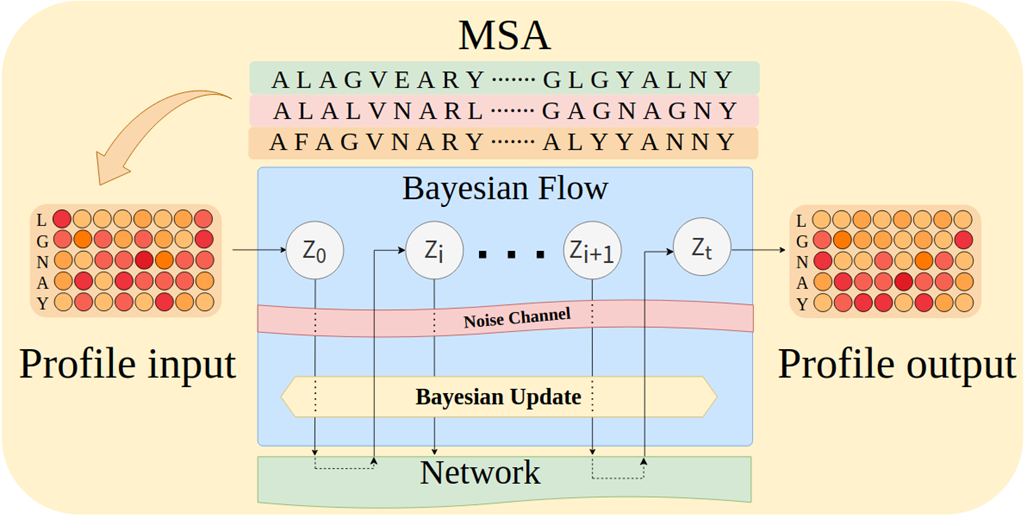
ProfileBFNの予想外の好業績
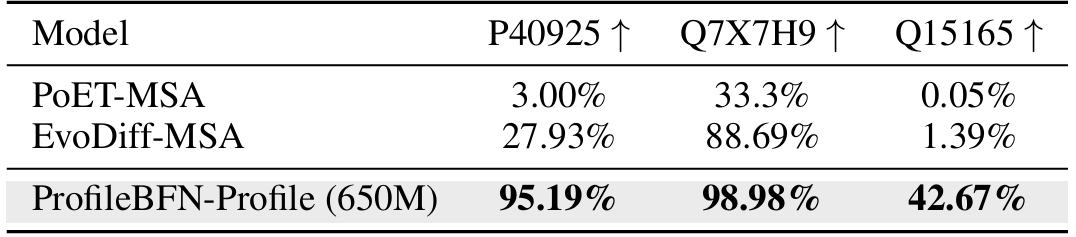
多重配列アライメントに基づく従来の方法と比較して、ProfileBFN は、10 倍少ないデータに依存し、タンパク質配列に関する 1.5 倍のコンテキスト情報を学習します。効果はすぐに現れます!
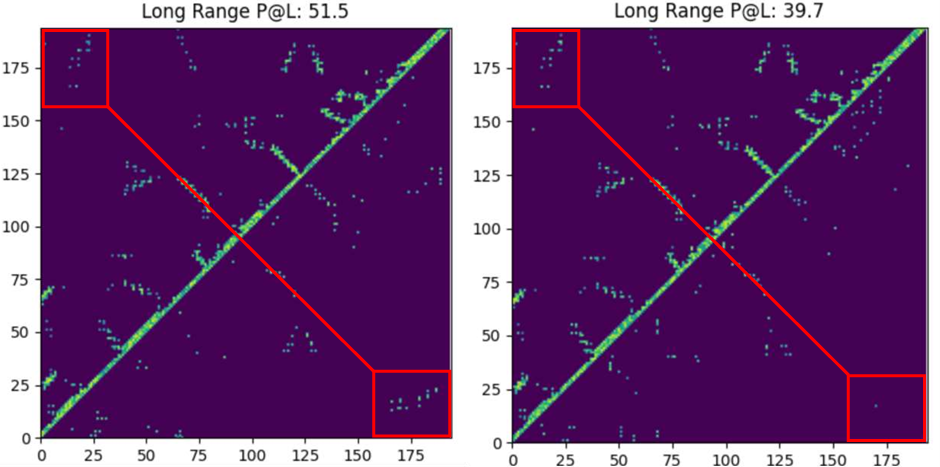
調査の結果、ProfileBFN はさまざまな下流タスクを促進する効果があることが確認されました。
* 酵素の分類:機能の忠実度を向上させ、スクリーニングコストを削減
* タンパク質表現学習:マルチタスク特徴抽出の支援
* タンパク質構造予測:相同性情報を強化し、モデリングの精度を向上
* 抗体産生:優れた移行効果、機能領域の正確な予測
酵素は触媒活性を持つタンパク質の特別なクラスであり、その機能の特異性は通常、EC 番号 (酵素委員会番号) によって説明されます。研究では、ProfileBFN によって生成された新しい酵素候補は、EC 数の点で野生型酵素と非常に一致していることがわかりました。これは、生成されたタンパク質が高度な機能的一貫性を維持していることを意味します。この機能により、実験スクリーニングの難易度が大幅に軽減され、新しい酵素設計の成功率が向上します。
ProfileBFN はタンパク質を生成すると同時に、モデル内に正確なタンパク質表現も構築します。研究者たちはこれらの表現を抽出し、タンパク質の熱安定性、タンパク質相互作用、タンパク質の細胞内局在などの複数のデータセットに基づいて微調整されました。結果は、ProfileBFN によって提供される表現が分類などの下流タスクにおけるモデル パフォーマンスを効果的に向上できることを示しました。これは、それが生成モデルであるだけでなく、強力な特徴学習ツールでもあることを示唆しています。
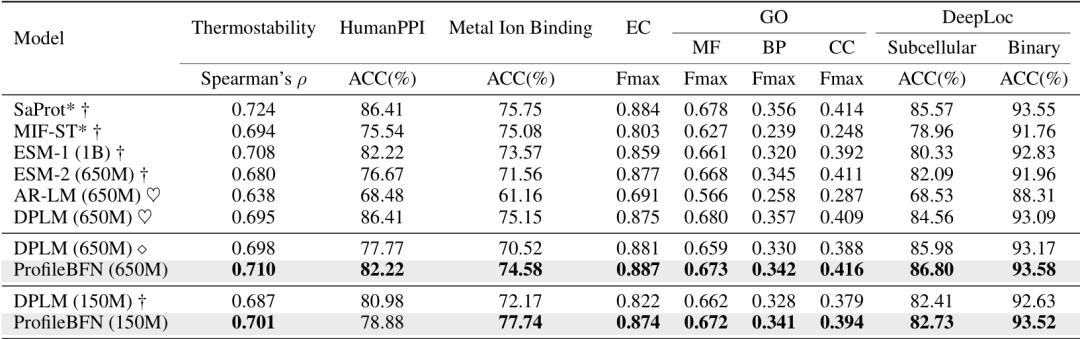
タンパク質構造の予測は構造生物学における重要な課題です。特に、孤立タンパク質(相同タンパク質が非常に少ないタンパク質)の場合、従来の方法の精度は大幅に制限されます。研究により、ProfileBFN は相同性情報の強化剤として使用できることが示されています。少量の MSA データで、より高品質の相同タンパク質が生成され、AlphaFold シリーズ モデルの予測精度が向上します。この機能により、ProfileBFN は構造生物学の分野で幅広い応用の見通しが得られます。
抗体は抗原に特異的に結合できる機能性タンパク質であり、免疫および病理学の研究において非常に重要です。抗体生成におけるProfileBFNの可能性を探るために、研究者らは、OAS(Observed Antibody Space)抗体配列データベースに基づいてモデルを微調整した。結果は、ProfileBFN が多様で高品質な抗体配列を生成するのに優れたパフォーマンスを発揮したことを示しました。
ProfileBFN の驚くべき効果は、この新しい研究が MSA 後の時代に生物学的配列を生成するためのパラダイムを提供するという事実から生まれます。
* MSAはトレーニングプロセスに入力として直接参加せず、追加のトレーニングオーバーヘッドを導入しません。
* 推論フェーズでは、単一シーケンスとMSAが均一にモデル化されます
* 相同配列はモデルの入力と出力の両方である
BFNは事前情報を完全に活用する
プロファイル情報は、元の相同配列よりもさらに重要なので、プロファイル情報をどのように使用すればよいのでしょうか? Bayesian Flow Network BFNはProfileにぴったりマッチします!これは次の 2 つの点に反映されています。
* BFNは分布から分布へのプロセスをモデル化し、入力はプロファイル表現であり、出力もプロファイル表現である。
* 最初から推論する代わりに、BFNは条件付き推論のためにプロファイル情報を事前に導入することができます。
自己回帰モデルや拡散モデルなどの従来のモデルでは、入力としてデータ (トークン) が必要であり、プロファイル情報を処理することでアルゴリズムの複雑さが増します。
BFN をモデル スケルトンとして使用することで、ProfileBFN はさらに次のことを実現できます。
* タスクの簡素化。相同情報の条件付き生成は、プロファイル情報の模倣になります。
* 効率が向上しました。サンプリング範囲が狭くなり、効率が上がる
ProfileBFNはウェットテストの救世主となることが期待されています
合成生物学などのタスクでは、長いサイクル、単一の評価指標、信頼性の欠如が研究者が遭遇する一般的な問題です。タンパク質ベースモデルとして、ProfileBFN は限られたリソースでより多くの相同情報を統合し、特定の事前情報を最大限に活用し、複数の指標に対する移行効果が優れているため、候補タンパク質の合成や指向性進化に最適な選択肢であることは間違いありません。
研究グループについて
清華大学知能産業研究所の生成的記号知能研究グループ(GenSI)の研究分野は、法学修士課程と科学のためのAIという2つの方向にまたがっています。これら 2 つの方向性が相互に促進し、AGI for Science (AI Scientist) の究極の使命を達成することが期待されます。
具体的な研究方向としては、新世代の大規模事前トレーニング技術、大規模強化学習(Large Scale RL)、深層生成モデル(Deep Generative Models)、およびそれらの科学データへの応用が含まれ、基本的な人工知能アルゴリズムと科学的問題の統合と革新に重点を置いています。現在、チームは、深層生成モデルの最先端理論とスケーラブルな構造ベースの生成モデル手法の探求に焦点を当て、LLMの推論能力の向上やAF3レベルの構造生成タスクを超えるなど、LLMとAI4Sciの分野における現実的で困難な科学的問題の解決に取り組んでいます。
チームへの連絡は以下のチャネルから可能です⬇️
* ホームページ:https://go.hyper.ai/7ye91
* メールアドレス: [email protected]
* 小紅書/知乎:GenSI
* ツイッター: @GenSI_official
* 微信: 15805171115








