Command Palette
Search for a command to run...
東京大学の研究チームは、事前の調整なしでバッチ効果を排除し、腫瘍微小環境の詳細な遺伝情報を明らかにするディープラーニングフレームワークSTAIGを開発しました。

生物組織は、特定の空間構成を通じて重要な機能を果たす複数の細胞タイプで構成された複雑なネットワークです。近年、10x Visium、Slide-seq、Stereo-seq、STARmap などの空間トランスクリプトミクス (ST) 技術の進歩により、生物学者は空間構造内で遺伝子データをマッピングできるようになり、さまざまな疾患に対するより深い洞察が得られるようになりました。
しかし、ST 技術は、均一な遺伝子発現と組織学的特徴を持つ空間領域を特定することに大きく依存しています。現在のところ、主な識別方法は、非空間クラスタリング法と空間クラスタリング法の2つがある。非空間クラスタリング手法では、遺伝子発現に基づいてクラスタリングのみが実行され、一貫性のないクラスタリング結果になることがよくあります。空間クラスタリング法では、グラフ畳み込みモデルを使用して遺伝子と空間情報を統合しますが、ST データをグラフ構造に変換するときに人工的に定義された距離基準に依存するため、バイアスが生じる可能性があります。同時に、組織学的画像を使用する方法も、染色品質の変化の影響を受けやすいという課題に直面しています。さらに、ほとんどの既存の方法のバッチ統合では、座標の手動調整や追加ツールへの依存など、手動による介入が依然として必要です。
これらの課題を克服するには、東京大学医科学研究所の研究チームは、STAIG(Spatial Transcriptomics Analysis with Image-Assisted Graph Comparative Learning)と呼ばれるディープラーニングフレームワークを提案しました。遺伝子発現、空間データ、組織学的画像を位置合わせなしで統合する機能。
STAIG は、事前トレーニングに大規模な組織学データセットに依存せずに、自己教師ありモデルを通じてヘマトキシリンおよびエオシン (H&E) 染色画像から特徴を抽出します。さらに、STAIG はトレーニング中にグラフ構造を動的に調整し、組織学的画像情報を使用して相同な陰性サンプルを選択的に除外することで、初期構築によって導入されたバイアスを軽減します。
最後に、STAIG はローカル比較を通じて遺伝子発現の共通点を識別し、手動での座標調整を必要とせずにエンドツーエンドのバッチ統合を可能にし、バッチ効果を効果的に削減します。研究者らは複数のデータセットでSTAIGを評価した。結果は、空間領域の識別において優れた性能を示し、腫瘍微小環境における詳細な空間情報と遺伝情報を明らかにし、複雑な生物学的システムの理解を促進することを示しました。
関連する結果は、「STAIG: ドメイン探索とアライメントフリー統合のための画像支援グラフ対照学習による空間トランスクリプトミクス分析」というタイトルで Nature Communications に掲載されました。
研究のハイライト:
* STAIGモデルは、事前調整なしで組織切片の統合を可能にし、バッチ効果を排除します。
* STAIGモデルは、組織学画像が含まれているかどうかに関係なく、さまざまなプラットフォームから取得されたデータに適用できます。
* 研究者らは、STAIGが空間領域を高精度で識別し、腫瘍微小環境に関する新たな知見を明らかにできることを実証し、空間的な生物学的複雑性を解析する幅広い可能性を示している。

用紙のアドレス:
https://www.nature.com/articles/s41467-025-56276-0
この研究で使用したデータセットのダウンロードアドレス:
https://go.hyper.ai/m5YC4
データセット: さまざまなプラットフォームからの ST データセットと組織学画像のコレクション
研究者らは、さまざまなプラットフォームから公開されている ST データセットと組織学画像をダウンロードしました。下の図の通りです。 ST データセットには、ヒト背外側前頭前野 (DLPFC) データセット、ヒト乳がんデータセット、マウス脳データセット、Slide-seqV2 データセット、STARmap データセットなどが含まれます。
データセットのダウンロードアドレス:
https://go.hyper.ai/m5YC4
* 10x Visium プラットフォームのヒト背外側前頭前皮質 (DLPFC) データセットには、3 人の個人からの 12 のスライスが含まれており、各スライスには 10μm と 300μm 間隔の 4 つのスライスがあり、各スライスのポイント数は 3,498 ~ 4,789 の範囲です。これらのスライスは、皮質層 L1~L6 および白質 (WM) として手動で注釈が付けられています。
* ヒト乳がんデータセットには 3,798 個のポイントが含まれています。
* マウス脳データセットには、それぞれ 2,695 個と 3,355 個のポイントを含む前部と後部の 2 つのスライスが含まれています。
* ゼブラフィッシュの黒色腫については、研究者らはセクション A とセクション B を分析しました。セクション A とセクション B にはそれぞれ 2,179 個と 2,677 個の斑点が含まれていました。
* アンサンブル実験では、DLPFC とマウス脳のデータセットが使用されました。マウス嗅球の Stereo-seq データセットには、解像度 14 μm の 19,109 個のポイントが含まれています。
* ラットの海馬(中心四分の一半径から 18,765 ポイント)とラットの嗅球(19,285 ポイント)を含む、10 μm 解像度の Slide-seqV2 データセット。
* STARmap データセットには 1,207 個のポイントが含まれています。
* MERFISH データセットの場合、ヒトの MTG には 3,970 個のスポットが含まれ、マウス 1 とマウス 2 の VIS 領域にはそれぞれ 5,995 個と 2,479 個のスポットが含まれます。
モデルアーキテクチャ: 空間トランスクリプトミクス解析のための画像支援グラフコントラスト学習
下の図は、グラフ対照学習と高性能な特徴抽出を組み合わせて遺伝子発現、空間座標、組織学的画像を統合するディープラーニング フレームワークである STAIG の全体的なフレームワークを示しています。 6 つのモジュールが含まれています:
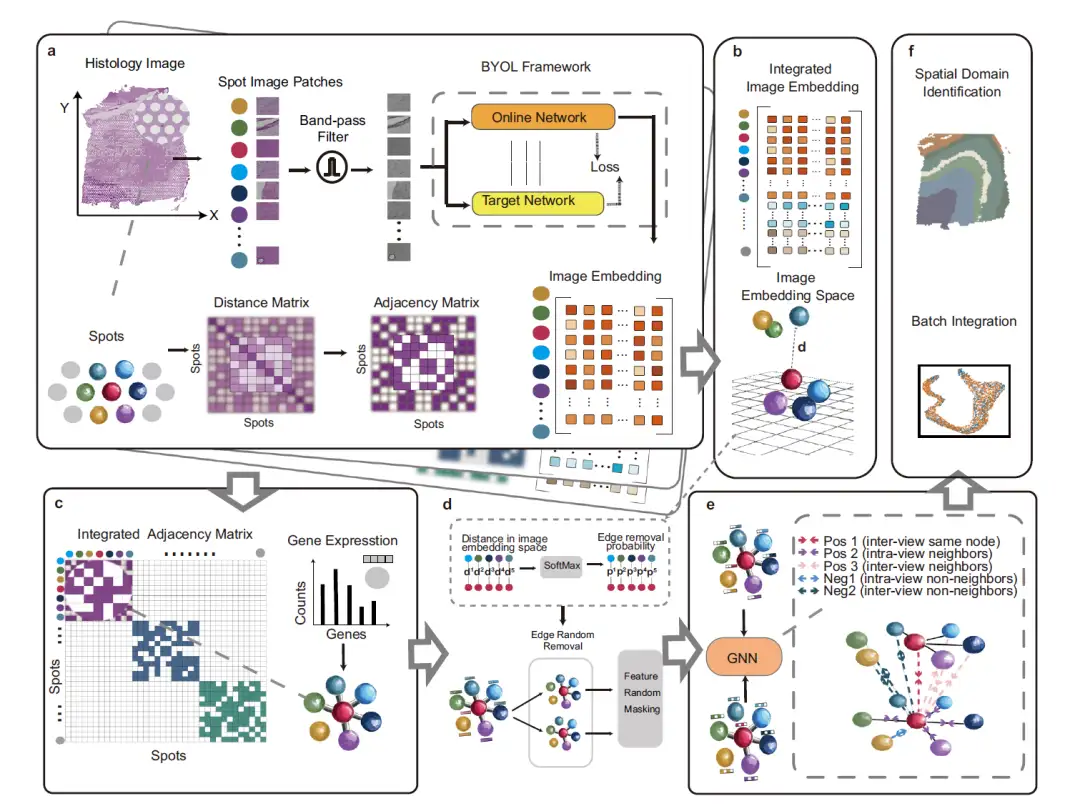
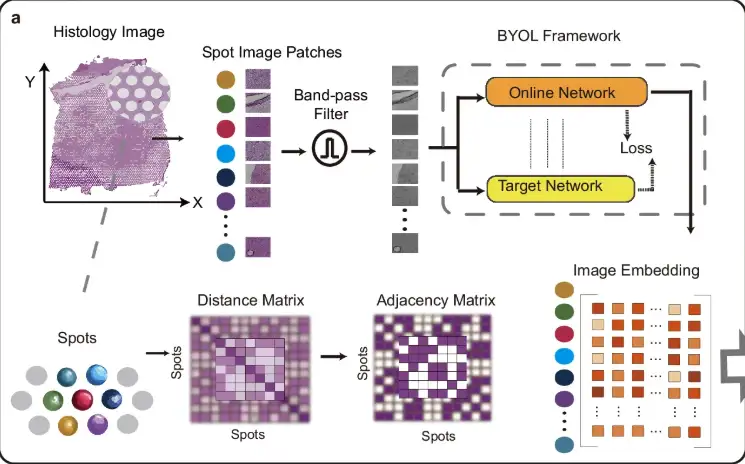
まず、次の図の a に示すように、ノイズや組織染色の不均一性の影響を軽減するために、STAIG はまず組織学的画像をデータ ポイントの空間位置に合わせて小さなパッチ (スポット画像パッチ) に分割し、次にバンドパス フィルターを使用して画像を最適化します。画像埋め込み機能は、Bootstrap Your Own Latent (BYOL) 自己教師モデルを通じて抽出され、データ ポイント間の空間距離に基づいて隣接行列が構築されます。
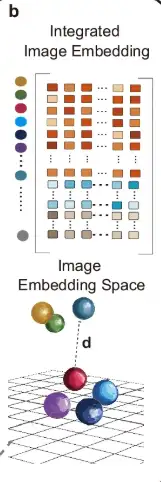
次の図の b 部分に示すように、異なる組織切片のデータを統合するために、STAIG は垂直スタッキング方式を使用して複数の組織切片の特徴を埋め込みます。
下図のc部分に示すように、各スライスの隣接行列を対角配置法で結合して統合隣接行列を形成し、これを用いて遺伝子発現データをノード情報とするグラフ構造を構築します。
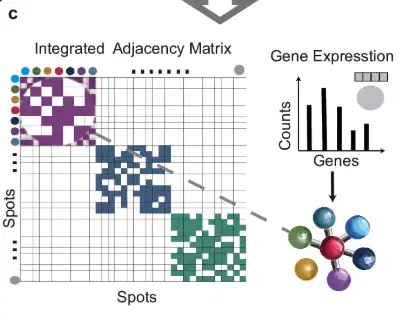
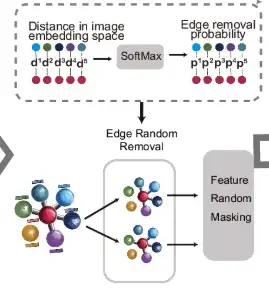
次の図の d 部分に示すように、エッジで接続された測定点については、画像埋め込み空間でその距離が計算され、SoftMax 関数を使用してこれらの距離がランダムエッジ除去の確率に変換されます。これに基づいて、元のグラフは 2 ラウンドのランダム エッジ削除 (エッジ ランダム削除) を経て、2 つの強化されたビューが生成されます。次に、これらのビュー内のノード機能がランダムにマスクされます。
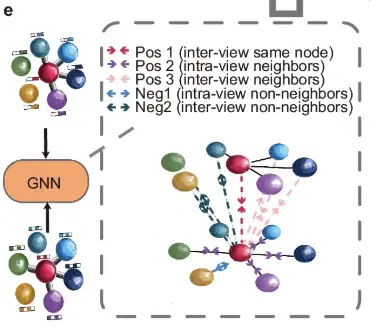
次に、図の e 部分に示すように、拡張ビューは共有グラフ ニューラル ネットワーク (GNN) によって処理され、両方のグラフ ビューで隣接ノードを近づけながら非隣接ノードを遠ざけることを目的とした隣接対照目的によって誘導されます。
最後に、図 f に示すように、トレーニングされた GNN は埋め込みを生成して空間領域を識別し、連続する組織セクション間のバッチ効果を最小限に抑えます。

研究結果:STAIGはさまざまな条件下で優れた性能を発揮
研究チームは、STAIG を他の最先端の ST 技術と比較するために、広範なベンチマーク評価を実施しました。結果は、STAIG がさまざまな条件下で優れたパフォーマンスを発揮することを示しています。
脳領域認識性能評価
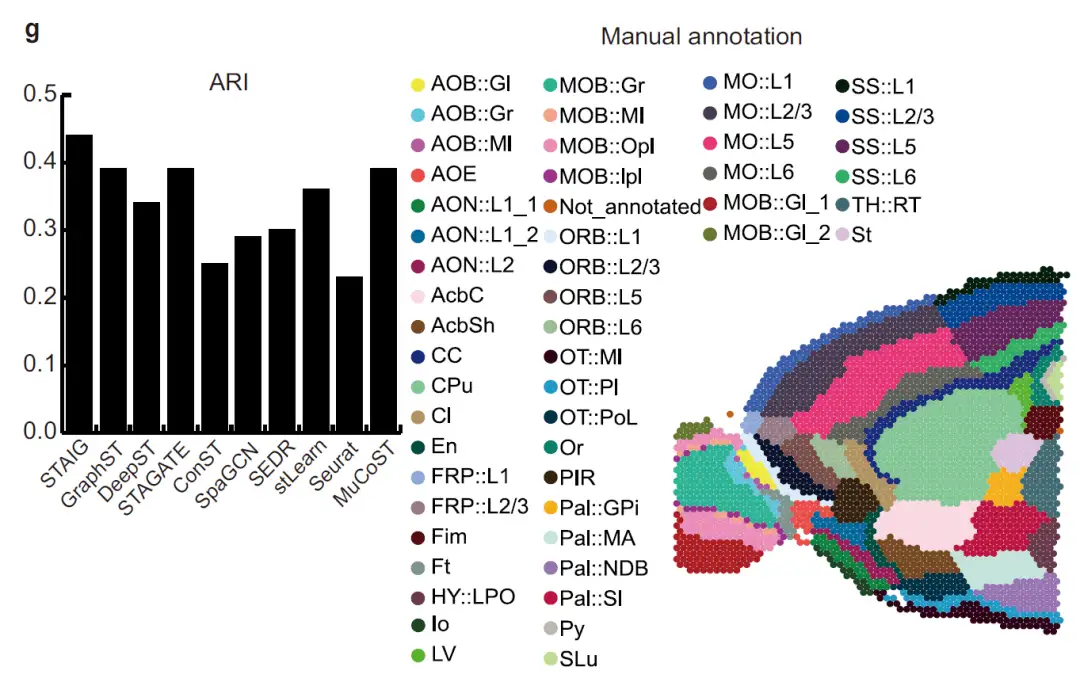
組織領域認識における STAIG のパフォーマンスを評価するために、研究者らは、Seurat、GraphST、DeepST、STAGATE、SpaGCN、SEDR、ConST、MuCoST、stLearn などの既存の方法と STAIG を比較しました。パフォーマンス評価指標には以下が含まれます。
* 調整ランド指数 (ARI) と正規化相互情報量 (NMI) (手動で注釈が付けられたデータセットの場合)。
* シルエット係数 (SC) と Davis-Bolding 指数 (DB) (他のデータセットの場合)。
① ヒト脳データセットのパフォーマンス
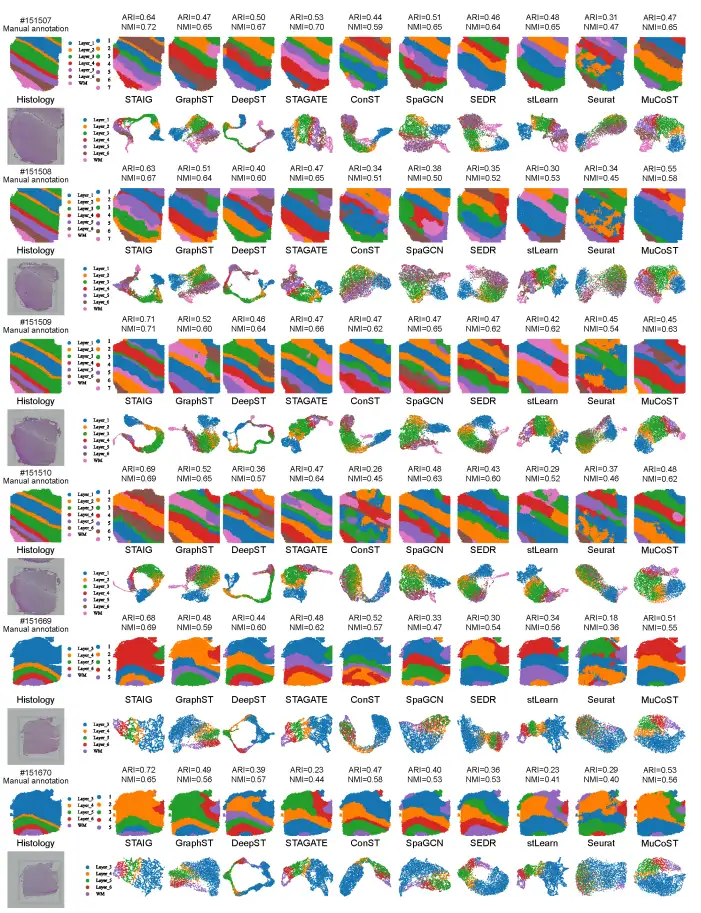
全体、STAIG は人間の脳のデータセットで最も優れたパフォーマンスを発揮します。次の図に示すように、最高の中央値 ARI (0.69) と NMI (0.71) が達成されました。
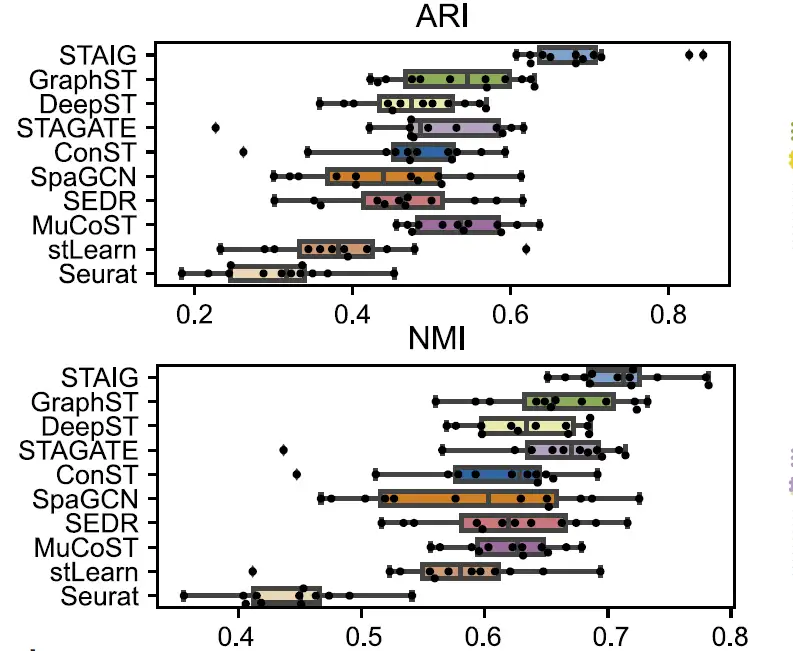
それに比べて、既存の方法のパフォーマンスは劣ります。stLearn はいくつかのポイントを誤って判断し、いくつかのレイヤーを見逃します。 GraphST の ARI は 0.64、NMI は 0.73 ですが、L4 層と L5 層の位置に大きな偏差があります。他の方法のARIは0.25~0.57の範囲であり、NMIは0.42~0.69の範囲であるが、これは主に層の割合の不正確な識別によるものである。
②マウス脳データセットのパフォーマンス
下の図に示すように、マウス後脳データセットでは、STAIG は小脳皮質と海馬を正常に特定し、さらにアモン角 (CA) と歯状回を区別しました。Allen マウス脳アトラス注釈と高い一致性があります。手動による注釈がないにもかかわらず、STAIG は最高の SC (0.31) と最低の DB (1.11) を達成し、優れたクラスタリング性能を示しました。
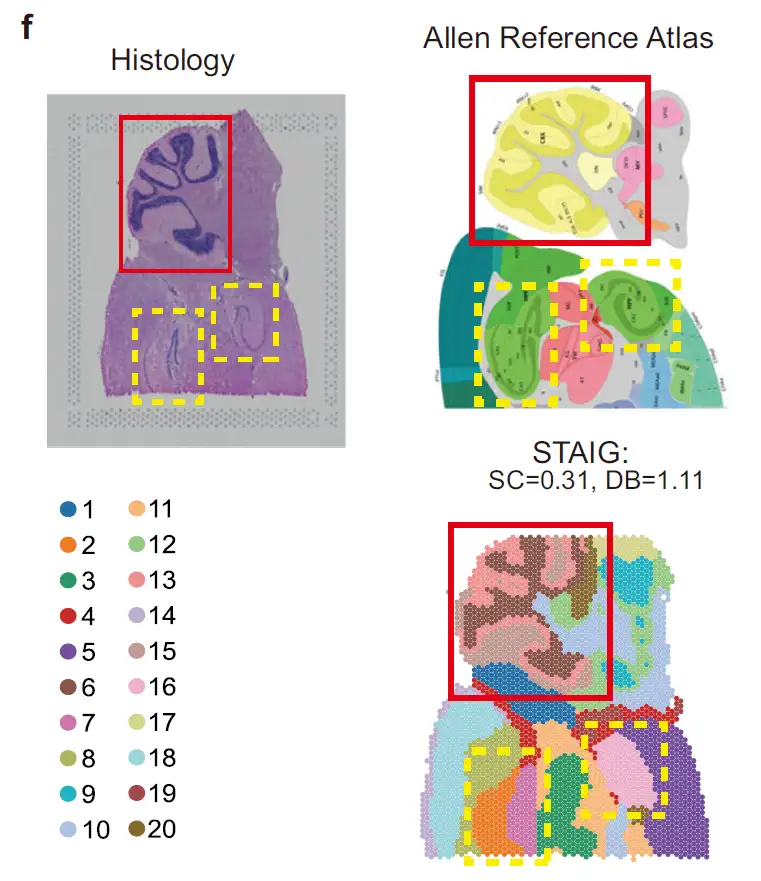
下の図に示すように、マウスの前脳データセットでは、STAIG は嗅球と背側外套を正確に分割しました。Longらの手動注釈を参照したところ、ARIは0.44、NMIは0.72となり、いずれも最高値となった。
画像特徴抽出の有効性
画像の特徴の影響を調査するために、研究者らは KNN アルゴリズムを使用して、STAIG によって抽出された画像の特徴と他の方法 (stLearn、DeepST、ConST) によって抽出された画像の特徴を比較しました。
①脳組織切片分析
スライス #151507 を例にとると、下の図に示すように、stLearn の画像特徴は染色強度によって深刻な影響を受け、実際の階層的注釈と一致しない結果になります。 DeepST と ConST はディープラーニングを使用していますが、脳組織の複雑なテクスチャ特徴を正確に捉えることができません。 STAIG の特徴抽出結果は手動で注釈を付けられたレベルと非常によく一致していますが、一部の境界はまだわずかにぼやけていますが、染色の違いによる影響はほとんど受けません。

②乳がん組織画像解析
研究者らは、下図に示すように、ヒト乳がんの H&E 染色画像を使用して画像特徴抽出機能をさらにテストしました。
結果は、stLearn の画像には腫瘍領域と正常領域が混在しており、識別が不十分であることを示しました。 ConST は画像をさまざまな領域に分割しているように見えましたが、拡大すると、領域の境界が手動の注釈から大きく外れました。 DeepST は有効な画像特徴を抽出できませんでした。STAIG は腫瘍領域を正確に特定します。空間クラスタリングの結果は高度な地域一貫性を維持し、セグメント化された領域は手動で注釈を付けた輪郭とほぼ完全に一致します。優れた画像特徴抽出能力を検証しました。

ヒト乳がんにおける腫瘍微小環境の定義 ST
ヒト乳がんデータセットの分析では、研究者らは、STAIG の結果が手動注釈と非常に一致しており、最高の ARI (0.64) と NMI (0.70) を達成したことを発見しました。STAIG が、特に手動注釈の Healthy_1 領域 (図 2a) に対して、わずかに異なるがより洗練された空間階層化を提案していることは注目に値します。STAIG は、この領域をサブクラスター 3 と 4 (図 2b) に細分化します。
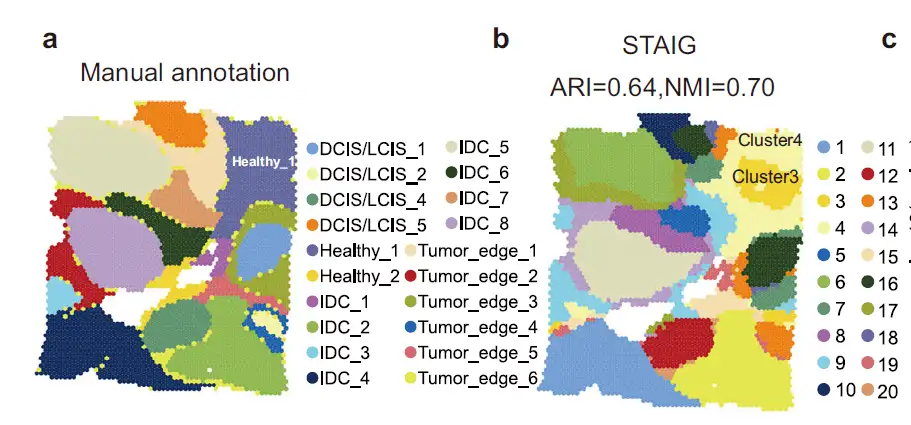
結論として、STAIG のマルチモーダル統合により、サブクラスター 3 が CAF 密度の高い腫瘍微小環境を形成し、CAF が豊富な領域の分子特性が明らかになりました。
ディープラーニングはSTの技術開発に強力なツールを提供する
ゲノミクスと ST 技術の急速な発展により、生物医学コミュニティは組織内の遺伝子発現の空間分布を調査できるようになり、生物の複雑な機能と構造を明らかにできるようになりました。 ST テクノロジーは、遺伝子発現に関する定量的な情報を提供するだけでなく、組織内の細胞の空間関係も保存するため、研究者は組織の微小環境、細胞相互作用、疾患発症の空間特性を研究することができます。しかし、ST データには通常、高次元性、強いノイズ、バッチ効果などの問題があるため、これらのデータをどのように効果的に統合して分析するかが現在の研究の中心的な課題となっています。
ディープラーニング技術、特にグラフニューラルネットワーク (GNN) と対照学習法の導入により、ST データの分析に強力なツールが提供されます。従来の分析方法では次元削減とクラスタリングに依存することが多いのに対し、ディープラーニング手法では、エンドツーエンドのトレーニングを通じて、マルチレベルの特徴を自動的に抽出し、データ表現を最適化できます。前述のように、GNN ベースの方法では空間隣接情報を使用してグラフ構造を構築できるため、モデルは遺伝子発現を捉えるだけでなく、細胞間の空間依存性を学習することもできます。対照学習の導入により、モデルの一般化能力がさらに強化され、注釈なしで主要な空間機能を学習できるようになります。
さらに、業界ではディープラーニング + ST テクノロジーの組み合わせでも大きな進歩を遂げています。
2024 年 11 月中国国立生物情報科学センターのヤン・ユンギ氏が率いるチームと中国科学院数学・システム科学研究所のチャン・シーホア氏が率いるチームは、STASCANと呼ばれるディープラーニングベースの空間トランスクリプトーム細胞注釈ツールを開発しました。遺伝子発現プロファイルと組織学的画像からの細胞特徴学習を統合することにより、組織切片の未知の領域における細胞タイプを予測し、キャプチャされた領域内の細胞に注釈を付けることで、空間的な細胞解像度を大幅に向上させます。さらに、STASCAN はさまざまな ST 技術からのさまざまなデータセットに適用可能であり、高解像度の細胞分布を解読し、強化された組織構造を解明する上で大きな利点を示します。
この結果は、「STASCAN は深層学習によって空間トランスクリプトミクスにおける高解像度の細胞分布マップを解読する」というタイトルで Genome Biology に掲載されました。
* 紙のアドレス:
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-024-03421-5
2025年1月23日米国のプリンストン大学の研究チームが、新たなディープラーニングアルゴリズムGASTON(Gradient Analysis of Spatial Transcriptomics Organization with Neural networks)を開発した。 GASTON は、教師なしのディープ ニューラル ネットワークと解釈可能なアルゴリズムを組み合わせることで、「isodepth」という革新的な概念を提案しました。これは地形図の高度に似ており、組織切片における遺伝子発現の空間トポロジー構造を定量化するために使用されます。 、
等深度とその勾配により、研究者は組織のさまざまな空間領域をセグメント化できるだけでなく、組織内の遺伝子発現の継続的な変化傾向と主要なマーカー遺伝子を特定することもできます。この研究では、マウスの脳、マウスの嗅球、大腸がんの腫瘍微小環境など、さまざまな生物学的サンプルにGASTONを適用して成功したことが実証されました。結果は、GASTONが組織構造を正確に分析し、細胞タイプの空間分布と変化パターンを明らかにし、他の方法では見落とされる多くの空間遺伝子発現パターンを発見できることを示しています。
関連する結果は、「解釈可能なディープラーニングによる空間遺伝子発現のトポグラフィーのマッピング」というタイトルで Nature Methods に掲載されました。
* 紙のアドレス:
https://www.nature.com/articles/s41592-024-02503-3
明らかに、ディープラーニングと ST テクノロジの組み合わせは、データ統合とノイズ低減の能力を向上させるだけでなく、空間生物学的情報の詳細なマイニングも促進します。将来的には、コンピューティング リソースの増加とアルゴリズムの最適化により、ディープラーニングは ST データ分析においてより重要な役割を果たし、精密医療と個別化治療をより強力にサポートするようになります。
参考文献:
1.https://static-content.springer.com/esm/art%3A10.1038%2Fs41467-025-56276-0/MediaObjects/41467_2025_56276_MOESM1_ESM.pdf
2.https://www.bjqykxy.com/kexueyanjiu/dongwuzhiwu/7361.html
3.https://news.qq.com/rain/a/20250128A057OQ00?suid=&media_id=
4.https://www.medsci.cn/article/show_








