Command Palette
Search for a command to run...
新しい癌治療法が開発されるかもしれません!デューク大学はPepPrCLIPを使用して「薬に頼れない」問題を解決
2021年、OpenAIは革新的なCLIP(Contrastive Language-Image Pre-training)モデルをリリースしました。CLIPは教師なし学習を通じて、追加の注釈情報を必要とせずに、画像とテキストの関係を効果的に理解して関連付けることができます。
数年後、生物医学科学者のグループがこれに触発されました。CLIP は画像と言語を一致させるので、同じアイデアをペプチドとタンパク質の一致にも使用できるのではないか?
対照的な言語と画像の事前トレーニングを通じてリアルな画像を生成するというOpenAIの画期的な研究を活用して、デューク大学生物医学工学部の研究チームは、これまで治療が不可能だった疾患の原因タンパク質に結合して破壊できる短いタンパク質(ペプチド)を設計できる PepPrCLIP(Peptide Prioritization by CLIP)パイプラインを構築しました。ターゲットの 3D 構造を使用してペプチドを生成する既存のプラットフォームである RFDiffusion と比較すると、PepPrCLIP はより高速で、ほぼ常にターゲット タンパク質に近いペプチドを作成します。研究者らはまた、PepPrCLIP によって選択された「ガイドペプチド」が、in vitro で阻害ペプチドとして使用された場合、または E3 ユビキチンリガーゼドメインと融合された場合に、強力で優れた標的結合および制御を実現できることを実験を通じてさらに検証しました。
関連する結果は、今年 1 月に「対照的な言語モデリングによる構造的に多様なターゲットに対するペプチド バインダーの新規設計」というタイトルで Science Advances に掲載されました。
用紙のアドレス:
https://www.science.org/doi/10.1126/sciadv.adr8638
関連データセットのダウンロードアドレス:
https://go.hyper.ai/AT5m9
オープンソース プロジェクト「awesome-ai4s」は、200 を超える AI4S 論文の解釈をまとめ、膨大なデータ セットとツールを提供します。
https://github.com/hyperai/awesome-ai4s
「薬に頼れない」問題を解決するための新たなアプローチ
病気を治療する一つの方法は、病気を引き起こすタンパク質を特異的に標的にして破壊できる薬を開発することです。時には、これらの重要なタンパク質は、丁寧に折られた折り鶴のように明確な構造を持っているため、従来の低分子治療薬が簡単に結合することができます。
しかし、80% を超える病原性タンパク質は、無秩序に絡み合った「絡み合った混乱」のようなもので、標準的な治療法ではその表面の結合部位を見つけて作用させることがほぼ不可能です。いわゆる「創薬不可能」という言葉は、従来の医薬品開発において、構造的および機能的特性のために医薬品のターゲットとなることが難しいタンパク質を表すためによく使用されます。
公開情報によると、薬物治療が困難なターゲットには、次のような特徴があることが多いです。
* 明確に定義されたリガンド結合ポケットを欠き、拡張された平坦な機能インターフェースを有する。
* 標的タンパク質を機能させるための特定のリガンドが不足している。
* ターゲットは疾患抑制剤であり、タンパク質の活性を活性化する薬剤が必要であり、薬剤開発がより困難になっています。
* 創薬不可能な標的は複雑な生理機能を持つことが多く、薬剤の設計と開発の難易度が増します。
* 医薬品開発戦略の限界。
これらの問題を回避するために、多くの研究者が、ペプチドを使用して病気の原因となるタンパク質に結合し、分解する方法を研究してきました。ペプチドはタンパク質の小型版であるため、結合に表面ポケットを必要とせず、代わりに、ペプチドはタンパク質内の異なるアミノ酸配列に結合することができます。
しかし、これらのアプローチにも限界があり、既存の「既製」の結合剤は不安定な、あるいは過度に絡み合ったタンパク質構造に結合するようには設計されていない。科学者たちは新しい結合タンパク質の開発に懸命に取り組んでいますが、これらのアプローチは依然として標的タンパク質の 3D 構造情報のマッピングに依存しており、これは無秩序な標的には利用できません。
この記事で紹介したデューク大学生物医学工学部の研究チームは、異なるアプローチを採用しました。病原性タンパク質の構造をマッピングするのではなく、大規模言語モデル (LLM) からヒントを得て、PepPrCLIP を構築しました。最初のコンポーネントである PepPr は、天然タンパク質配列の大規模なライブラリでトレーニングされた生成アルゴリズムを使用して、特定の機能を備えた新しい「ガイド」タンパク質を設計します。2 番目のコンポーネントである CLIP は、OpenAI によって最初に開発されたアルゴリズム フレームワークを使用して、これらのペプチドがターゲット タンパク質と一致するかどうかをテストおよびスクリーニングします。
CLIP ベースのペプチド優先順位付けプロセスの構築 - PepPrCLIP
PepPrCLIP はどのように構築されましたか?
簡単に言うと、研究者らはまず ESM-2 タンパク質言語モデル (pLM) を使用して、実際のペプチド バインダー シーケンスの埋め込みに対してガウス ノイズ摂動を実行し、自然な特性を持つ候補ペプチド シーケンスを生成しました。次に、これらの候補ペプチドは、CLIP ベースの対照学習アーキテクチャを通じて潜在空間でスクリーニングされ、相補的なペプチドとタンパク質のペアを共エンコードするモデルをトレーニングしました。最後に、構築された PepPrCLIP に生成識別フレームワークを統合して、ターゲット シーケンスに結合できる完全に新しいペプチド候補シーケンスを選別しました。
次の図は、PepPrCLIP モデルのトレーニングの具体的なプロセスを示しています。
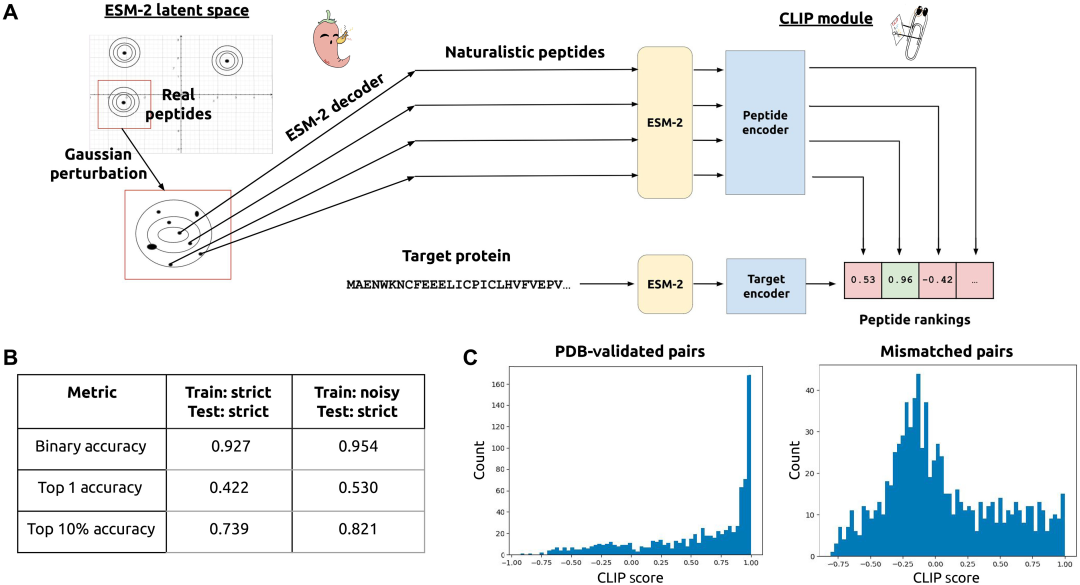
上の図に示すように、ESM-2 に埋め込まれた天然ペプチドをサンプリングしてガウス分布を生成し、その後、アミノ酸配列にデコードします。トレーニングされた CLIP モジュールは、対応するペプチドとタンパク質の埋め込みを共同でエンコードし、何千ものペプチドをスクリーニングし、ターゲットに対する特定の結合活性を評価します。具体的には、次のようになります。
* CLIPアーキテクチャとトレーニング
まず、入力シーケンスを凍結された ESM-2-650M モデルに埋め込んで入力埋め込みを生成します。次に、入力埋め込みをシーケンスの長さにわたって平均化して、ペプチドやタンパク質に適した埋め込みベクトルを取得します。h MLP レイヤーを適用し、埋め込みベクトルを修正線形ユニット (ReLU) 活性化関数を使用して処理して出力埋め込みを取得します。 CLIP スコアは、ペプチドとタンパク質のベクトル埋め込み間のドット積を実行することによって取得され、値は -1 から 1 の間になります。モデルは、ペプチドとタンパク質の結合ペアに高い CLIP スコアが得られるようにトレーニングされます。
* ペプチド候補配列の生成
候補ペプチドはトレーニング セット内のすべてのペプチドから生成され、各ペプチドは PyTorch の ESM-2-650M pLM を使用して埋め込まれます。特定のペプチド埋め込みについて、埋め込みのすべての次元の分散が計算されます。ソース ペプチド内の各残基について、標準正規分布からノイズがサンプリングされ、分散が乗算されて摂動が作成され、対応する残基の埋め込みに追加されます。推論時に、ソースペプチドはトレーニングセットからランダムにサンプリングされ、各ソースペプチドに対して、上記のノイズ法を使用して 1,000 個のペプチドが生成されました。最後に、これらのペプチド (約 100,000) が CLIP モデルに入力され、ユーザーが提供するターゲット シーケンスへの予測される結合に基づいてランク付けされます。
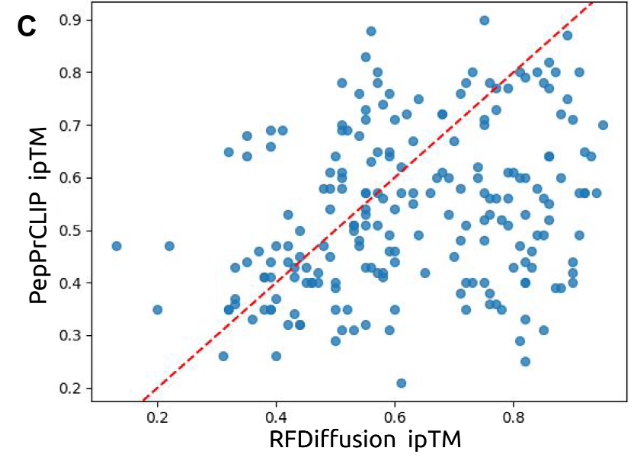
研究者らはコンピューターシミュレーションで、PepPrCLIP のパフォーマンスを RFDiffusion のパフォーマンスと比較しました。研究者らは、PepPrCLIP によって生成されたペプチドと RFDiffusion によって生成されたペプチドの ipTM スコアを比較し、下の図に示すように、33% を標的とするペプチドでは PepPrCLIP が RFDiffusion よりも優れていることを発見しました。さらに、PepPrCLIP は、シーケンス埋め込みのみを使用することで、生成と優先順位付けの速度を大幅に向上させ、1 分あたり約 1,000 個のペプチドを生成し、タンパク質ターゲットごとに 100,000 個のペプチドを約 1 分でランク付けすることができます。比較すると、RFDiffusion では、1 つのバインダーを設計するのに約 2 分かかります。この効率により、PepPrCLIP は構造情報の有無にかかわらず大規模なペプチドライブラリのスクリーニングに特に有利になります。
研究チームは、秩序だったタンパク質標的と無秩序なタンパク質標的に対する PepPrCLIP の効果をさらに評価するために、デューク大学医学部、コーネル大学、サンフォード・バーナム・プレビス医療発見研究所の研究チームとも協力し、プラットフォームを実験的にテストしました。
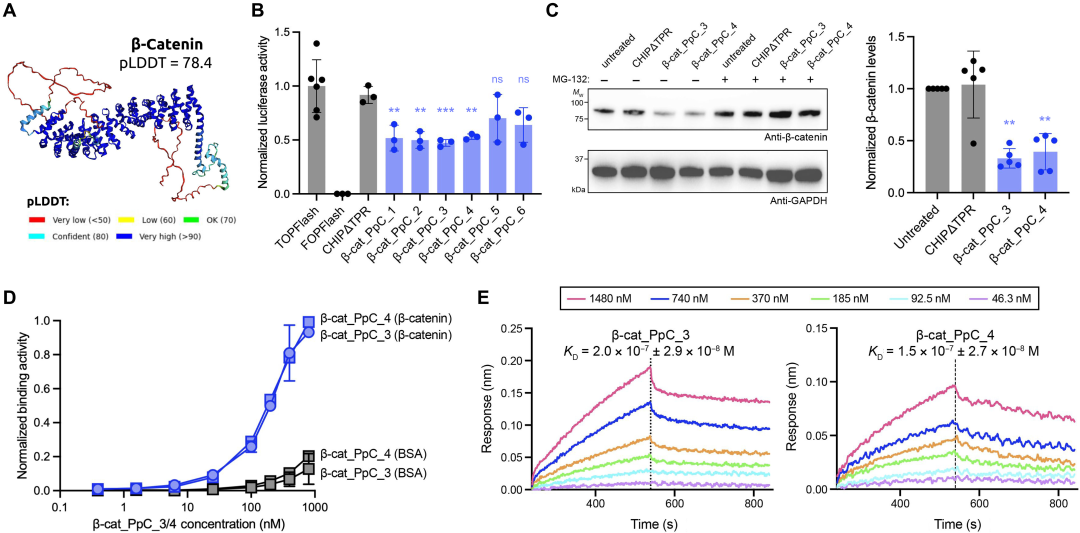
最初のテストで研究チームは、PepPrCLIP によって生成されたペプチドは、比較的単純で安定した酵素タンパク質である UltraID に効果的に結合し、その活性を阻害することができます。
次に、研究者らは PepPrCLIP を利用して、さまざまな種類の癌のシグナル伝達に関与する不規則な複雑なタンパク質である β-カテニンに結合できるペプチドを設計しました。下の図に示すように、研究チームは、CLIP によってタンパク質に結合できることが示された 6 つのペプチドを生成し、そのうち 4 つがターゲットに効果的に結合して分解できることを示しました。タンパク質を破壊することで、がん細胞のシグナル伝達を遅らせることができます。
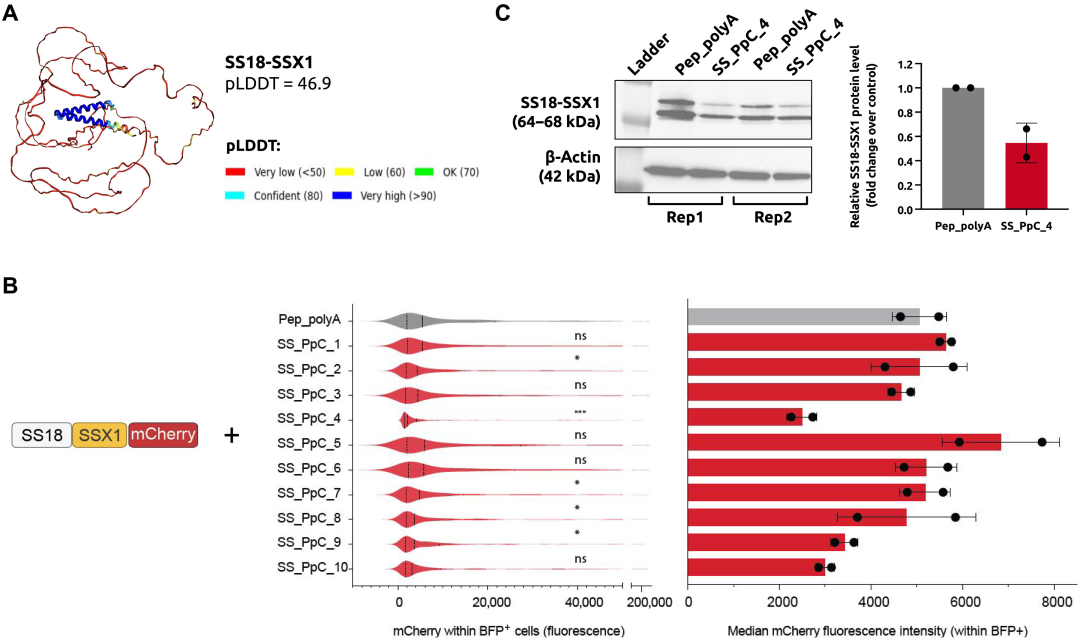
最も複雑なテストでは、研究チームは、滑膜肉腫に関連する高度に不規則なタンパク質に結合できるペプチドを設計しました。滑膜肉腫は、すべての軟部組織腫瘍の 5% ~ 10% を占める、まれな悪性癌です。滑膜肉腫は軟部組織で発生し、主に小児および若年成人に影響を及ぼします。この疾患は、SS18-SSX という独自の高度に不規則な発癌性融合タンパク質の存在が特徴です。
研究チームはペプチドを滑膜肉腫細胞に注入し、10種類のデザインをテストした。以下の結果に示すように、PepPrCLIP によって SS18-SSX1 に結合すると予測されたペプチドのうち、SS_PpC_4 は SS18-SSX1-mCherry 蛍光を大幅に減少させました。次に、研究者らは SS_PpC_4 の過剰発現が内因性 SS18-SSX1 融合タンパク質のレベルに与える影響もテストしました。SS_PpC_4 ペプチドの過剰発現によって SS18-SSX1 タンパク質レベルが大幅に減少したことは注目に値します (>40%)。
言い換えると、PepPrCLIP は、タンパク質に結合して分解できるペプチドを設計します。このタンパク質を破壊できれば、研究者はこれまで治療薬がなかったがんに対する治療法を開発する機会を得ることができ、多くの刺激的な臨床的可能性が開けることになる。
AIは「治療不可能な」病気を克服するための新たなツールを提供する
2023年9月にネイチャー誌に掲載された最新のレビューでは、「創薬不可能な」タンパク質を標的とした創薬の最新の進歩とその臨床応用が包括的に紹介されています。類似した創薬不可能な特性を持つさまざまな分子が、以下のカテゴリーに分類されました。
① 小型 GTPase: KRAS、HRAS、NRAS などの RAS ファミリータンパク質など。表面に標的化可能なポケットがないため、薬物治療が不可能と考えられています。
②ホスファターゼ:各ホスファターゼは構造に多くの類似性があるため、選択性が低く、副作用が避けられず、創薬の進歩を大きく妨げています。
③ 転写因子(TF):さまざまなヒト疾患は、多くの生物学的プロセスに関与する転写因子の調節異常と関連しており、そのほとんどは、構造の異質性と処理可能な結合部位の欠如のために、従来の小分子では標的とすることができません。
④ エピジェネティックターゲット:エピジェネティックターゲットは遺伝子発現パターンの調節に重要な役割を果たし、さまざまな生物学的プロセスや疾患に影響を及ぼします。
⑤ その他のタンパク質:タンパク質間相互作用(PPI)とそのネットワークは、生物学的プロセスと細胞周期の調節において非常に重要です。平坦な相互作用表面を持つ一部のPPIは、他のPPIよりも標的にするのが難しく、ある程度「薬物治療が不可能」になります。
現在、いわゆる「創薬不可能」な標的に直面して、学術界は数十の革新的な方法を開発してきました。創薬不可能なタンパク質のメカニズムに応じて、フラグメントベースの創薬(FBDD)、コンピューター支援薬物設計(CADD)、仮想スクリーニング(VS)、DNAエンコードライブラリ(DEL)などの最先端技術を採用し、体系的な薬物設計戦略を形成しています。今日では、人工知能技術の発展とタンパク質大規模言語モデルの台頭により、この問題を克服するための新しいツールが提供されています。近年、産業界と学術界の両方で重要な進歩がありました。
業界、2023年12月、生成AI抗体発見のリーダーであるAbsci Corporationは、腫瘍標的に対するAI設計抗体の開発でAstraZenecaと提携すると発表しました。この提携により、Absci の統合医薬品創出プラットフォームと AstraZeneca の腫瘍学の専門知識が統合され、潜在的な新たな癌治療候補の発見が加速されます。 Absci の統合医薬品創製プラットフォームは、何百万ものタンパク質間相互作用を測定することで独自のデータを生成し、そのデータを使用して Absci 独自の AI モデルをトレーニングし、その後の反復で新しい AI モデルを使用して設計された抗体を検証します。このプラットフォームは、データ収集、AI 駆動型設計、実験室検証を約 6 週間で完了することで創薬を加速し、これまで創薬不可能と考えられていた標的に対する医薬品の開発を含め、創薬標的の範囲を拡大する可能性があります。
学界、2025年1月、AI製薬業界のリーダーであるInsilico Medicineとカナダのトロント大学が共同で実施した研究では、量子コンピューティングモデルと従来のコンピューティングモデル、生成型人工知能を組み合わせ、膨大なデータセットのトレーニング、生成、スクリーニングを通じて幅広い化学的可能性を探り、「創薬不可能な」がんドライバータンパク質KRASを標的とした新しい分子を発見しました。
KRAS 変異は癌における最も一般的な変異の 1 つであり、ヒトの腫瘍の約 4 分の 1 で発生します。KRAS 変異は制御不能な細胞増殖を引き起こし、癌を引き起こす可能性があります。本研究では、潜在的な新しいKRAS阻害剤を生成するために、研究者らは量子変分生成モデル(QCBM)と長短期記憶ネットワーク(LSTM)を組み合わせた量子古典ハイブリッドフレームワークモデルを提案し、量子コンピューティングと古典コンピューティング手法を組み合わせて新しい分子を設計しました。この研究は、セントジュード小児研究病院を含む複数の研究機関からも支援を受けています。関連する研究結果は、「量子コンピューティング強化アルゴリズムが潜在的なKRAS阻害剤を発見」というタイトルでネイチャーバイオテクノロジー誌に掲載されました。

関連技術の飛躍的進歩により、人類は新たな想像空間と病気克服の無限の可能性を切り開きました。