Command Palette
Search for a command to run...
LeCun 氏の提案により、カリフォルニア大学バークレー校らは、配列と全原子タンパク質構造を同時に生成するマルチモーダルなタンパク質生成法 PLAID を提案しました。

過去何年にもわたって、科学者たちは「生命の暗号」の謎をよりよく解明するために、タンパク質の構造と組成を研究し続けてきました。タンパク質の機能は、側鎖と主鎖の原子の正体と位置、およびそれらの生物物理学的特性を含むその構造によって決まります。これらは総称して全原子構造と呼ばれます。ただし、側鎖原子をどこに配置するかを決定するには、まずその配列を知る必要があります。したがって、全原子構造の生成は、配列と構造の同時生成を必要とする多峰性の問題とみなすことができます。
ただし、既存のタンパク質の構造と配列の生成方法は、通常、配列と構造を独立したモードとして扱い、構造生成方法は通常、骨格原子のみを生成します。通常、全原子設計用に設計された方法は、構造の予測とアンチフォールディングに外部モデルの助けを必要とします。などのステップを交互に実行します。
これらの課題に対処するために、カリフォルニア大学バークレー校 (UC Berkeley)、Microsoft Research、および Genentech の研究チームは、より豊富なデータ モダリティからデータを生成できるマルチモーダルなタンパク質生成手法である PLAID (タンパク質潜在誘導拡散) を提案しました (たとえば、 、シーケンス)は、マルチモーダル生成を達成するために、より稀なモード(結晶構造など)にマッピングされます。この方法の有効性を検証するために、研究者らは、遺伝子オントロジーの 2,219 の機能と、Tree of Life に広がる 3,617 の生物について実験を実施しました。トレーニング中に構造入力は使用されませんが、生成されたサンプルは強力な構造品質と一貫性を示します。
関連する研究のタイトルは「Generating All-Atom Protein Structure from Sequence-Only Training Data」で、トップカンファレンスICLR 2025に提出されている。 「AI ゴッドファーザー」の楊立坤氏も結果をソーシャル プラットフォームに転送しました。
PLAID プロジェクトのオープンソース アドレス:
http://github.com/amyxlu/plaid
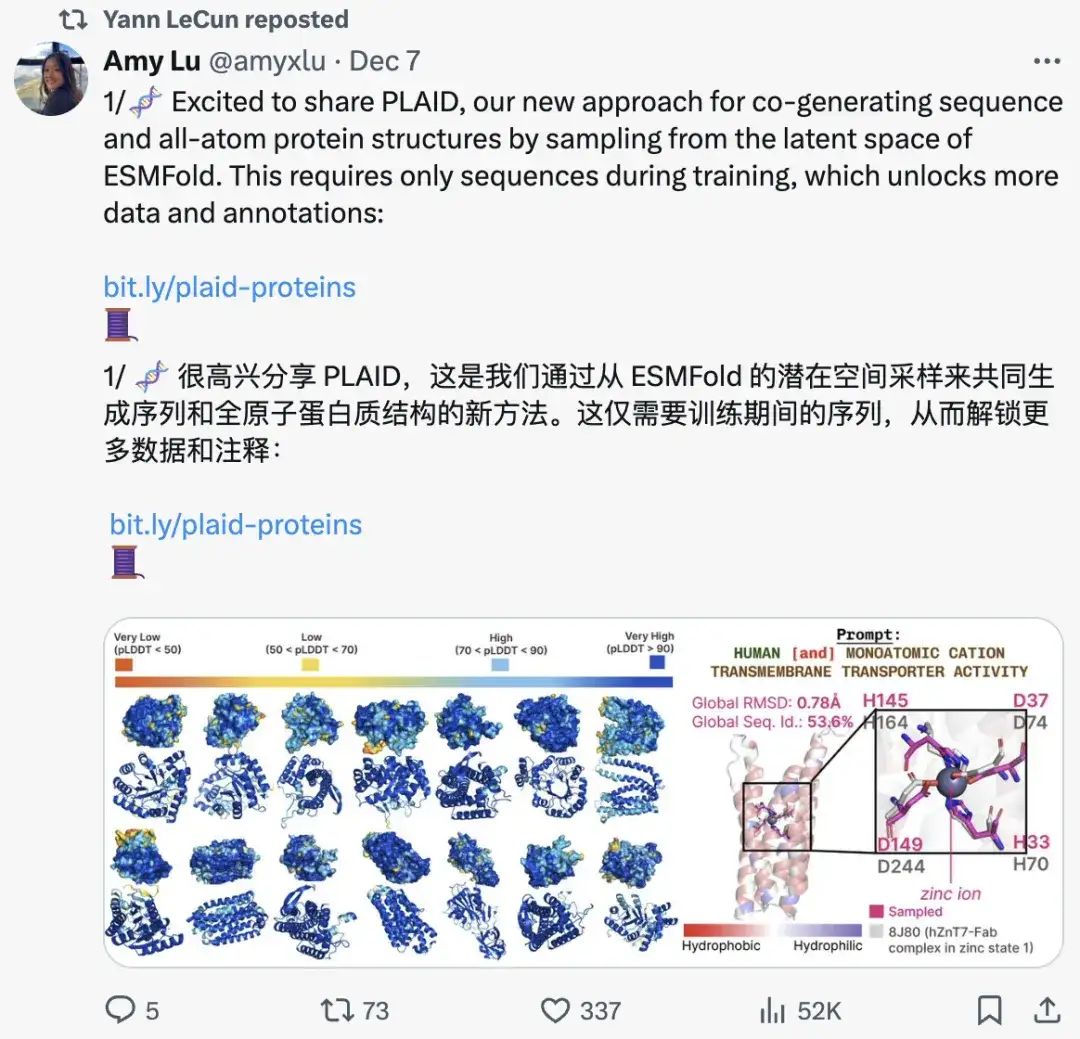
研究のハイライト:
* 研究者らは、大規模タンパク質言語モデル ESMFold と全原子構造生成に焦点を当て、トレーニング プロセス中に配列入力のみを必要とし、配列と全原子タンパク質構造を同時に生成できる制御拡散モデルを提案しています。
* このアプローチは、トレーニング データではなく、事前にトレーニングされた重みにエンコードされた構造情報を活用し、制御可能な生成のためのシーケンス アノテーションの可用性を高めます。
※論文ではESMFoldモデルを使用していますが、この手法はあらゆる予測モデルに適用できます。
用紙のアドレス:
https://www.biorxiv.org/content/10.1101/2024.12.02.626353v1
オープンソース プロジェクト「awesome-ai4s」は、100 を超える AI4S 論文の解釈をまとめ、大規模なデータ セットとツールを提供します。
https://github.com/hyperai/awesome-ai4s
研究のハイライトの概要
データセット
研究者らは、2023年9月にリリースされたPfamデータベースを使用した。このデータベースには、57,595,205の配列と20,795のファミリーが含まれている。 PLAID は UniRef や BFD (約 20 億配列) などの大規模な配列データベースと完全に互換性がありますが、この研究では Pfam を使用することを選択しました。その理由は、その配列ドメインにはより多くの構造ラベルと機能ラベルが含まれており、生成されたサンプルのコンピューターシミュレーションが容易になるためです。評価がより便利です。 。さらに、研究者らは検証のために約 15% データを保存しました。
Pfam ドメイン由来生物の UniRef コードは、Pfam FTP サーバーによって提供される Pfam-A.fasta ファイルから取得できます。研究者らは、データセット内のすべての固有の生物を分析し、合計 3,617 種類の異なる生物を発見し、これらの生物に対して実験を行って PLAID 法の有効性を検証しました。
モデルアーキテクチャ
PLAID は、予測モデルの潜在空間での拡散によるタンパク質のマルチモーダルで制御可能な生成のための新しいパラダイムです。この方法の概要を次の図に示します。簡単に 4 つのステップに分かれています。
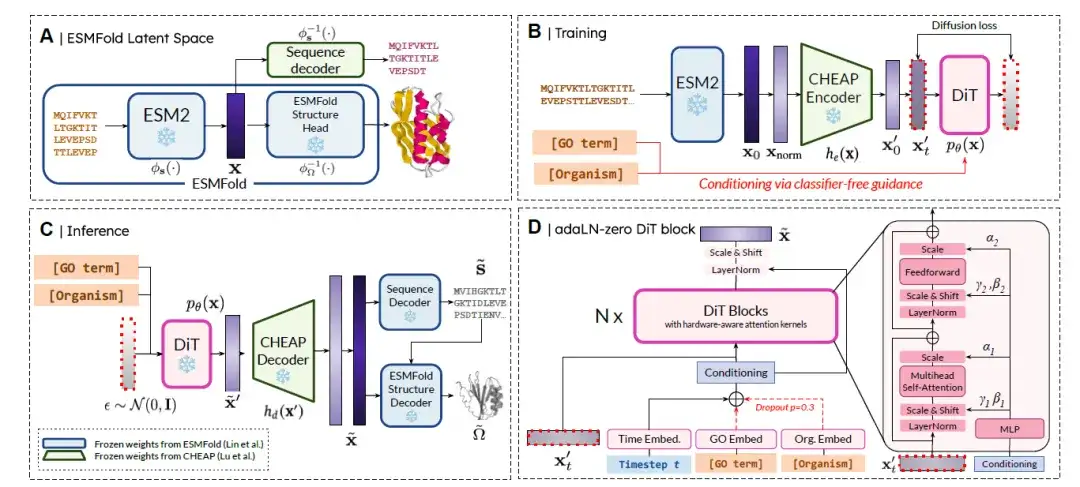
(A) ESMFold 潜在空間:潜在空間 p(x) は、シーケンスと構造の結合埋め込みを表します。
(B) 潜在的普及訓練(訓練):目標は、拡散公式に従って、pθ(x) から学習してサンプリングすることです。学習効率を向上させるために、研究者は CHEAP エンコーダ he(・) を使用して圧縮埋め込み x' = he(x) を取得し、拡散目標が pθ(he(x)) からのサンプリングになるようにします。
(C) 推論:推論中にシーケンスと構造の両方を取得するために、研究者らはトレーニングされたモデルを使用して ˜x' 〜 pθ(x') をサンプリングし、その後 CHEAP デコーダーを使用して解凍して ˜x = hd(˜x') を取得しました。この埋め込みは、CHEAP でトレーニングされた凍結配列デコーダーによって、対応するアミノ酸配列にデコードされます。残基同一性シーケンスと ~x は入力として使用され、ESMFold でトレーニングされた凍結構造デコーダーに渡されて、全原子構造が取得されます。
(D) DiT ブロック アーキテクチャ:研究者らは、拡散トランスフォーマー (DiT) アーキテクチャと adaLN-zero DiT ブロックを組み合わせて条件情報を融合しました。機能 (つまり、GO 用語) と生物クラスのラベルは、分類子を使用しないガイダンスを使用して埋め込まれました。
研究結果
研究者らは、さまざまな長さのタンパク質の構造品質と多様性の分析を実施しました。その結果を以下の図に示します。ネイティブタンパク質と PLAID で生成されたサンプルは、長さ全体にわたって一貫したメトリクスを維持します。ProteinGenerator と Protpardelle では特定の長さでモード崩壊が発生しましたが、Multiflow ではより長いシーケンスで多様性の低下が見られました。
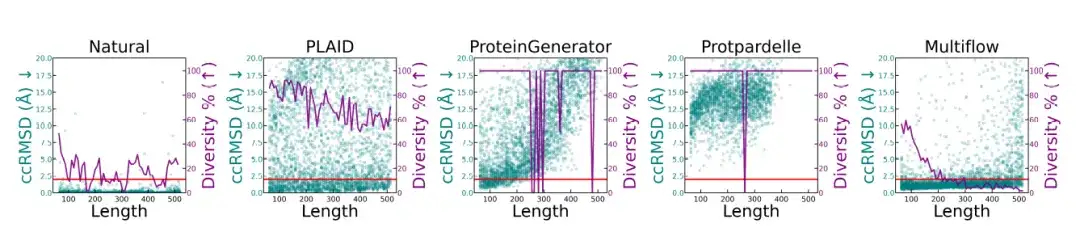
* この図はネイティブタンパク質とさまざまな生成方法を比較しており、さまざまな長さ (64 ~ 512 残基) でのタンパク質の構造品質 (ccRMSD、シアンの点) と多様性 (紫色の線、サンプル全体における固有の構造クラスターの割合) を示しています) 。 2Å の赤い線は設計のしきい値を表します)
さらに、ベースライン手法と比較して、PLAID によって生成される二次構造の多様性は、天然タンパク質の分布によりよく似ています。以下の図に示すように、ProteinGenerator、Protpardelle、Multiflow は二次構造分布に偏りを示しており、既存のタンパク質構造生成モデルは、β シート含有量の高いサンプルを生成するのに苦労することがよくあります。
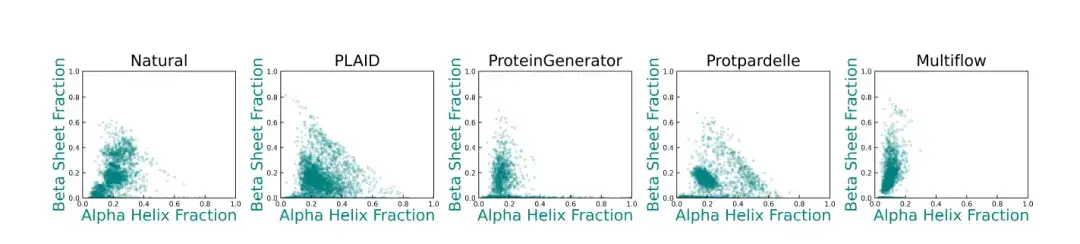
* この図は、ネイティブタンパク質およびさまざまな製造方法におけるタンパク質構造のαヘリックスおよびβシート含有量の分布を示しています。各点は構造を表し、その座標はαヘリックス残基の割合「x軸」とβシート残基の割合「y軸」を表します。
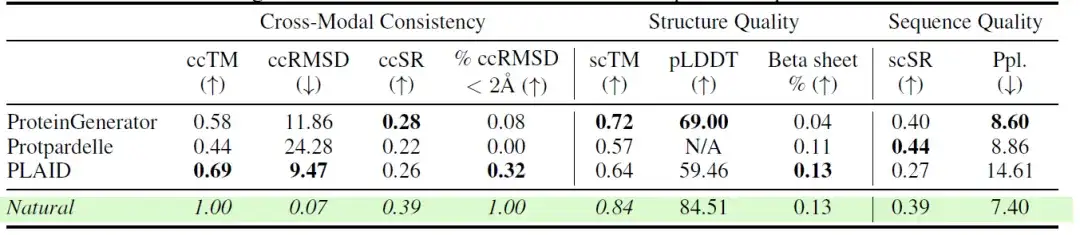
研究者らはまた、全原子タンパク質生成タスクにおけるさまざまな一貫性および品質メトリクスにわたって、さまざまなモデルのパフォーマンスを比較しました。結果は次のとおりです。PLAID で生成されたサンプルは、配列と構造の間で高度なクロスモーダル一貫性を示します。
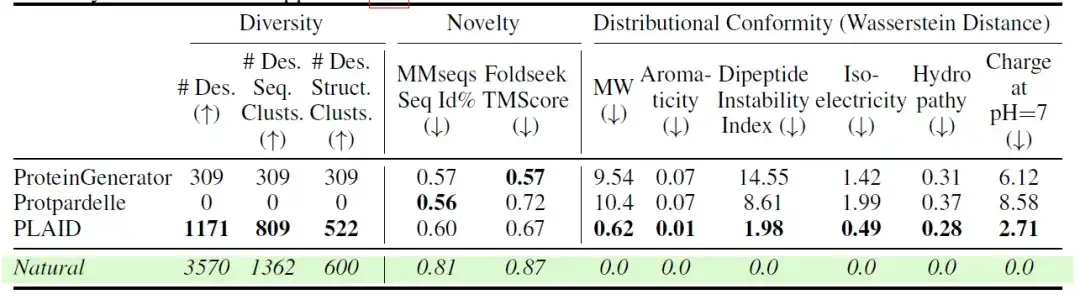
研究者らは、さまざまなモデルの多様性、新規性、自然性もさらに評価しました。結果は次のとおりです。全原子モデルの中で、PLAID は配列空間と構造空間の両方で最も多くのユニークで設計可能なサンプルを生成しました。
PLAID は、ESMFold に限定されず、多くのダウンストリーム機能に簡単に拡張でき、あらゆる予測モデルにも適用できることを強調する価値があります。
AI がタンパク質研究に新たな道を開く
生物分野での拡散トランスの使用が増加
この論文では、モデル構築プロセス中に研究者が拡散トランスフォーマー (DiT) を使用してノイズ除去タスクを実行したと述べました。
DiT の基本原理は、Transformer アーキテクチャを拡散モデルに適用することです。拡散モデルは通常、ノイズを段階的に追加することで元のデータを破壊しますが、その後、モデル学習を通じて復元されます。 DiT は、Transformer ブロック (適応層正規化、クロスアテンションなど) を拡散モデルに導入することにより、モデルの生成機能を強化します。
近年、DiT は画像とビデオの生成分野で大きな進歩を遂げています。Sora などの最先端の生成モデルの主要なアーキテクチャは DiT です。生物医学の分野では、拡散トランスフォーマーは、研究者が潜在的な薬物分子を迅速にスクリーニングし、その生物学的活性を予測するのに役立ち、生命科学研究を提供します。強力なツール。タンパク質のノイズ除去を例にとると、DiT は複雑な配列と構造の関係を捉えることができます。つまり、Transformer のグローバルなセルフアテンション メカニズムを通じて、タンパク質の配列と構造の間の複雑な相互作用関係を効果的にモデル化し、次に、次の逆プロセスを使用できます。タイム ステップにより、ノイズが除去された潜在ベクトルが予測され、ノイズからタンパク質の構造と配列が徐々に復元されます。
この論文に特化して、DiT は、特にタンパク質構造予測モデルが核酸と低分子リガンド複合体を統合し始めるにつれて、混合入力モダリティの処理を微調整するためのより柔軟なオプションを提供します。さらに、このアプローチでは、Transformer のトレーニング インフラストラクチャをより有効に活用できます。
研究者らは初期の実験で、三角形のセルフアテンションを使用するよりも、利用可能なメモリを大規模な DiT モデルに割り当てる方が効果的であることも発見しました。 xFormers によって実装された最適化アルゴリズムを使用してモデルをトレーニングすると、推論フェーズのベンチマーク テストで 55.8% の速度向上と 15.6% の GPU メモリ使用量の削減を達成しました。
機械学習によりカスタマイズされたタンパク質が「夢の実現」に
前述のカリフォルニア大学バークレー校関連の研究は、タンパク質のカスタマイズにおいて新たな重要な一歩を踏み出したと言えます。タンパク質は通常、生命の構成要素と考えられる 20 種類の異なるアミノ酸で構成されていることがわかっています。非常に複雑な構造のため、タンパク質の三次元構造を予測し、人間が使用できる新しいタンパク質を設計することは、数十年前にはまだ科学者にとって「夢のような話」でしたが、近年、機械学習の急速な進歩により、設計するという夢が実現しました。カスタマイズされたタンパク質が徐々に可能になります。
有名なAlphaFoldに加えて、いくつかの研究の進歩も注目に値します—
2024 年 11 月、米国エネルギー省のアルゴンヌ国立研究所チームは、MProt-DPO と呼ばれる革新的なコンピューティング フレームワークの開発に成功しました。このフレームワークは人工知能技術と世界トップクラスのスーパーコンピューターを組み合わせたもので、タンパク質設計の新時代を切り開きます。実際の事例を取り上げて、科学者たちはMProt-DPOを通じて、特定の条件下で化学反応を効率的に触媒できる新しいタイプの酵素を設計しました。以前の設計方法と比較して、新しい酵素の反応効率は 30% 近く向上しており、実験の進行が速まるだけでなく、産業応用の可能性が広がります。さらに、MProt-DPO の応用の成功により、抗ウイルスタンパク質を設計するための新しいアイデアも開かれます。関連する研究結果は、「MProt-DPO: Breaking the ExaFLOPS Barrier for Multimodal Protein Design Workflows with Direct Preference Optimization」というタイトルで IEEE Computer Society に発表されました。
用紙のアドレス:
https://www.computer.org/csdl/proceedings-article/sc/2024/529100a074/21HUV88n1F6
タンパク質ポケットは、特定の分子に結合するのに適したタンパク質上の点です。タンパク質ポケットの設計は、タンパク質のカスタマイズのプロセスにおいて重要な手法の 1 つです。 2024 年 12 月、中国科学技術大学とその協力者は深層生成アルゴリズム PocketGen を設計しました。タンパク質ポケットの配列と構造は、タンパク質フレームワークと結合小分子に基づいて生成できます。実験の結果、PocketGen モデルの親和性や構造的合理性などの指標が従来の手法を超え、計算効率も大幅に向上することが示されました。関連する研究結果は、「PocketGen によるタンパク質ポケットの効率的な生成」というタイトルで Nature Machine Intelligence に掲載されました。
用紙のアドレス:
https://www.nature.com/articles/s42256-024-00920-9

将来的には、タンパク質の分野で人工知能がさらに応用されることで、人々はタンパク質の空間構造の秘密についてさらに深く理解できるようになると思います。
参考文献:
1.https://www.biorxiv.org/content/10.1101/2024.12.02.626353v1
2.https://mp.weixin.qq.com/s/_5_L7bvl-vHtls8gBbfSmQ
3.https://mp.weixin.qq.com/s/sfrm2rj_8kH0JA2vu4NmTw
4.http://www.news.cn/globe/20241014/f7137840e56340f081f9eb819d87ba40/c.html
5.http://www.bfse.cas.cn/yjjz/202412/t20241212_5042432.html
6.https://www.sohu.com/a/826241274_12








