Command Palette
Search for a command to run...
AlphaFold 3 の超完全な解体、上海交通大学の Zhong Bozitao 氏: すべての生体分子の構造を原子精度で予測するためのデータの極端な使用、しかしそれは完璧ではない

すべての生体分子の構造と相互作用を「原子精度」で予測できる AlphaFold 3 は、リリースされるやいなや業界で広範な議論を引き起こしました。 8月13日、上海交通大学AI for Bioengineeringサマースクールイベントで、Zhong Bo Zitao 博士は、「AlphaFold 3: 原理、応用、展望」と題して、自身の学習経験を体系的に整理し、科学研究コミュニティからの多くの関連研究結果を広範囲にまとめ、AlphaFold 3 についての考えを深く共有しました。洞察力、HyperAI は、当初の意図に反することなく、スピーチの核となる内容をまとめました。以下はスピーチの書き起こしです。

今日はタンパク質の構造予測に焦点を当てて、AlphaFold 3 について説明します。 現在のトップタンパク質、そしてさらに広範な生体分子の構造予測ツールとして、AlphaFold 3 のステータスは自明です。
タンパク質の合成はDNAの転写から始まり、遺伝情報がRNAに伝達され、さらにRNAが翻訳されてタンパク質となり、さらに二次構造、三次構造、四次構造に折り畳まれます。ほとんどのタンパク質は独自の立体構造に折り畳まれ、その構造に必要な情報はアミノ酸配列にコード化されています。これはよく言われることですが、配列が構造を決定し、構造が機能を決定します。タンパク質構造の予測は、生物学的機能を理解するために重要です。
AlphaFold 3 の画期的な進歩: 革新的なモデル アーキテクチャとデータ利用の向上
AlphaFold 3 と AlphaFold 2 のモデル アーキテクチャを比較する
過去には、AlphaFold 2 はタンパク質構造予測において他のアルゴリズムを直接「打ち負かしました」。そのコア アーキテクチャは、以下の図に示すように、3 つの主要な部分に要約できます。 最初の部分、青いボックス内の MSA & テンプレート モジュール。その機能は、複数の配列アラインメント (MSA) およびテンプレート構造情報を収集および統合することです。モデルデータに入力します。 2 番目の部分 (緑色のボックスの Evoformer モジュール) は、マルチシーケンス組織における共進化情報を理解する機能を持ち、収集した情報を精製および処理することで、それを 3 番目の部分 (紫色のボックス) の構造モジュール モジュールに渡します。箱。
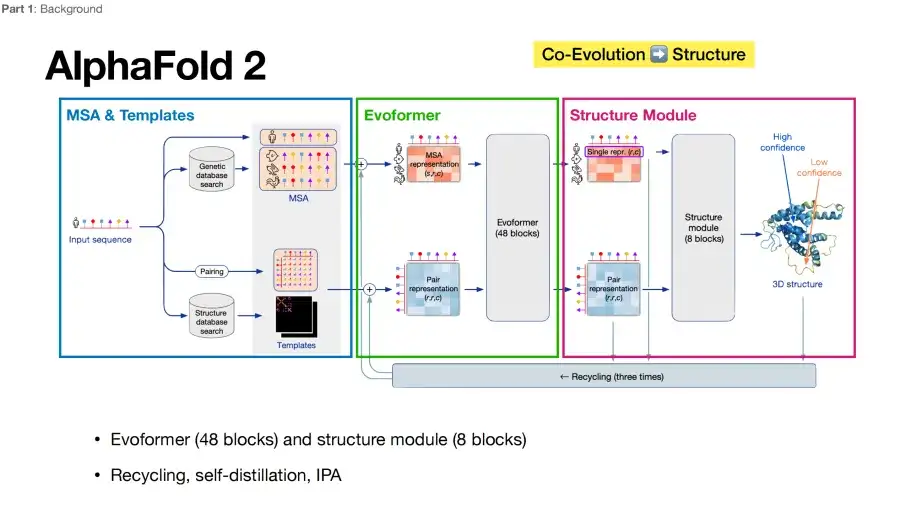
ディープラーニングの観点から見ると、Evoformer はエンコーダーの役割を果たしますが、この点では Structure Module はデコーダーに相当します。AlphaFold 2 が高く評価されているのは、シーケンス入力から構造出力に直接マッピングするエンドツーエンドの最適化機能が主な理由です。
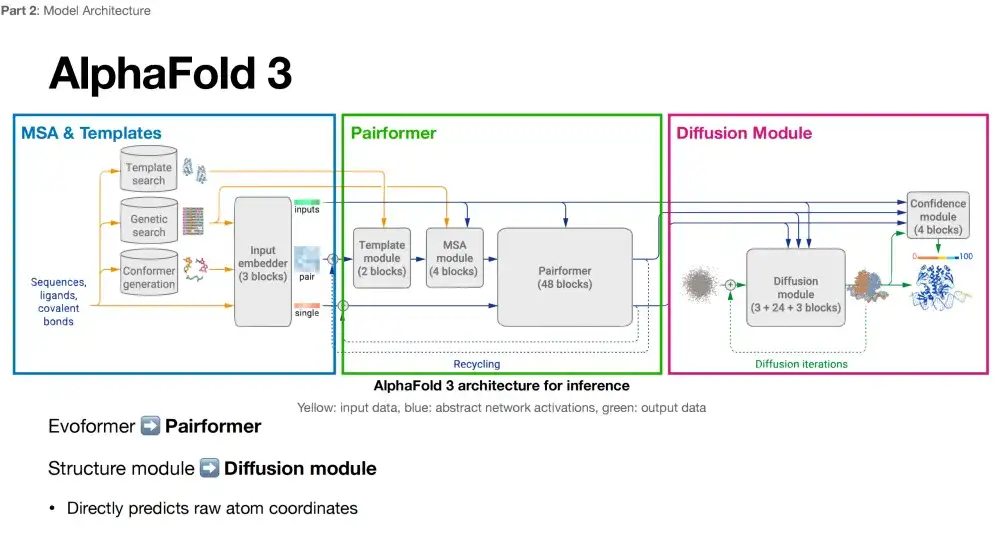
AlphaFold 3 のモデル アーキテクチャは想像されているほど変わっていないと一般的に考えられています。モデル フレームワークも 3 つの主要な部分で構成されています。AlphaFold 2 との各部分の比較は次のとおりです。
パート 1: 似たものにする
以下の図に示すように、AlphaFold 3 と AlphaFold 2 のアーキテクチャ図を比較すると、AlphaFold 3 の最初の部分 (青いボックス内) には依然として MSA とテンプレートが含まれており、追加の Conformer 生成ステップが導入されています。
パート 2: MSA シーケンスへの依存性の低減
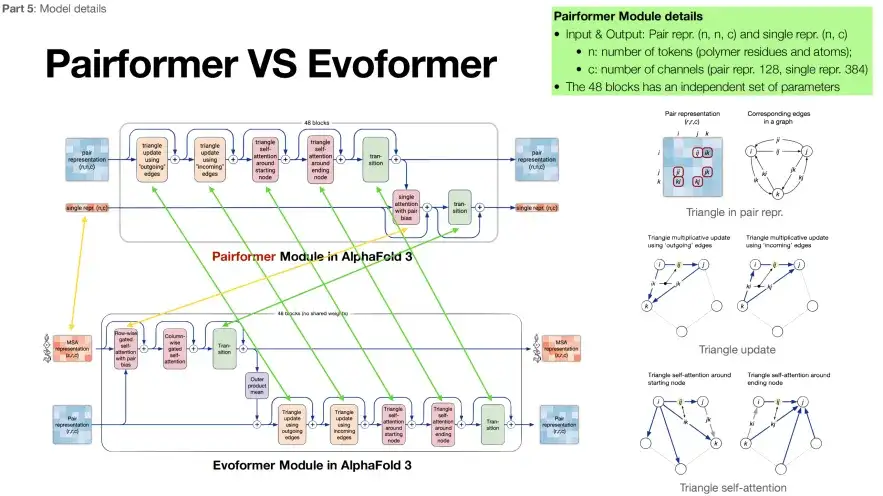
AlphaFold 3 の 2 番目の部分 (緑色のボックス内) は、Pairformer と名付けられており、その構造は基本的に Evoformer と非常に似ていますが、MSA モジュールの数が 4 つに減っています。以下の図に示すように、緑色の矢印は 2 つのモジュールの同じ内容を示し、黄色の矢印は相違点を示していることがわかります。AlphaFold 3 は、標的タンパク質配列に重点を置き、MSA 配列にはあまり依存しません。
さらに、AlphaFold 3 が複数のタスクで優れたパフォーマンスを発揮できる理由は、多重配列アラインメント (MSA) への依存度を低減しているためであると考えられます。以下の図に示すように、右側は、AlphaFold 2 のパフォーマンスに対する MSA の影響を示しています。MSA の数が増加するにつれて、特定のしきい値 (ピンクの線) を超えると、AlphaFold 2 のパフォーマンスの向上は横ばいになります。以下の図の中央部分によれば、AlphaFold 2 と比較して、AlphaFold 3 に対する MSA の影響が弱くなっています (曲線の変動はほとんどありません)。
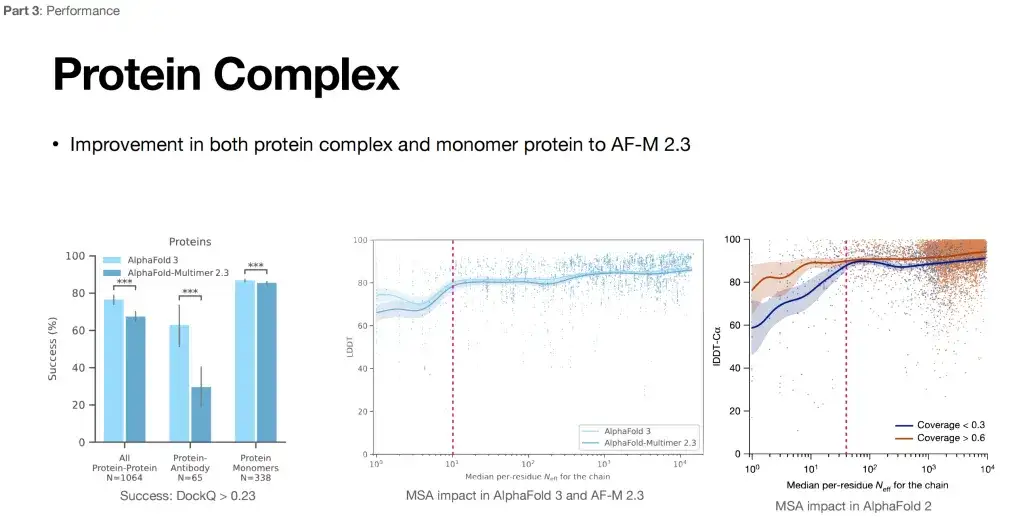
さらに、抗体の成熟には、生体内での超突然変異のプロセスが必要となることが多く、MSA 情報はその構造を予測するのにあまり役立ちません。この観点から、このアプリケーションは、タンパク質とその複合体のペアとなる MSA 情報を見つけることも困難です。 AlphaFold 3 の範囲 この拡張により、MSA への依存度が低減される可能性があります。
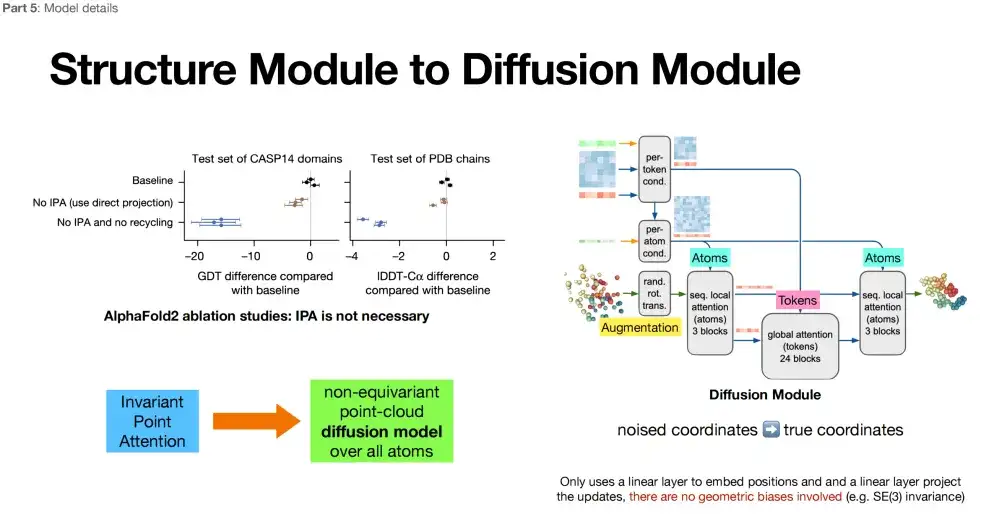
パート 3: 全原子構造生成 + 立体回転不変性の除去
AlphaFold 3 の 3 番目の部分 (紫色のボックス内) は、同じく構造モジュール カテゴリに属する拡散モデルを使用します。違いは、拡散モデルが構造モジュールで繰り返される最適化を拡散モデルと呼ばれる新しいメカニズムに置き換えることです。 。
*拡散モデル: モデルにノイズを追加し (順方向)、モデルにノイズを除去させ (逆方向)、逆のプロセスを学習させ、同様のデータ分布を生成します。
以下の図に示すように、第 3 部では、AlphaFold 3 は全原子レベルの構造生成を実現します。分子の基本構成要素である原子には、より豊富な物理情報が含まれている可能性があります。これは、AlphaFold 3 がタンパク質の構造を予測する際に、より深い物理法則を捉えることができる可能性があることを意味します。さらに、AlphaFold 3 では、AlphaFold 2 で強調されていた 3 次元回転不変性が放棄されました。AlphaFold 2 でこの機能の追加アーキテクチャを削除した後、研究者らは、モデル (拡散モジュール) の設計がより自由になったことを発見しました。
AlphaFold 3 によりデータ使用率が向上
タンパク質データ リソースは限られていますが、AlphaFold 3 はデータ セットを大きくするだけでなく、データ利用率も向上します。具体的には、AlphaFold 2 の 100 万レベルのデータセットと比較して、AlphaFold 3 は 10 億レベルのデータセットに直接近づき、トレーニングセットが増加しています。さらに、そのトレーニング セットには、PDB 内のデータに加えて、他の大量のデータも組み込まれています。たとえば、AlphaFold 2 によってより正確に予測された構造データがトレーニング セットの拡張として選択されます。特定のトレーニング セットを次の図に示します。

AlphaFold 3 はアプリケーション範囲で大幅な飛躍を達成
AlphaFold 3 の最大の変化は、アプリケーションの範囲において質的な飛躍を達成したことです。これまでの AlphaFold 2 はアミノ酸構造の予測に重点を置いていましたが、AlphaFold 3 では原子レベルの構造を直接予測できるようになり、その機能拡張は具体的には次の 4 つの側面に反映されています。
* リガンド (Ligand)、つまりタンパク質内の小分子の結合部位を正確に予測できる。
* タンパク質の複雑な構造を予測する能力。
* タンパク質および核酸の翻訳後修飾構造を予測する能力。
* DNAとRNAの構造、DNA/RNAとタンパク質の複合体の構造を予測することができます。
AlphaFold 3 はリガンドドッキングの分野を変える
その中で、AlphaFold 3 が科学分野に与えた最大の影響は、リガンド ドッキング タスクの改善です。以下の図に示すように、ベンチマーク テスト PostBusters Benchmark では、4 つの異なるリガンド ドッキング タスクにおけるさまざまな深層学習アルゴリズムの成功率が評価されています。AlphaFold 3 は、未知の事前知識を前提として最高の成功を達成できることがわかります。ポケットと構造のレートは 76.4% です。
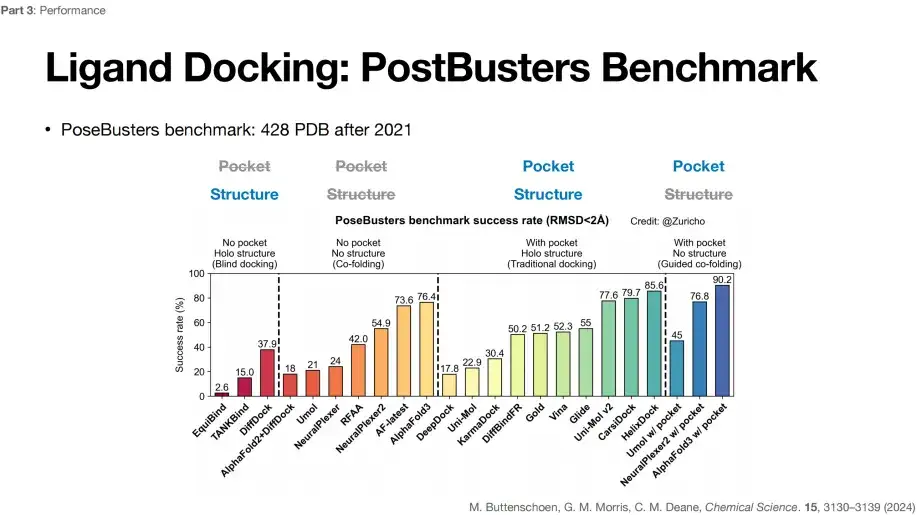
PostBusters Benchmark が 2021 年以降の 428 の PDB データを選択
ミッション成功の基準: 予測された小分子のドッキング位置と実際のドッキング位置との偏差が 2 Å 未満である
上の図に示すように、未知のポケット位置と既知のタンパク質構造 (ポケットなし、ホロ構造) の最初のタイプのブラインド ドッキング タスクでは、DiffDock は 37.9% という最高の成功率を達成できます。
2 番目のタイプの共フォールディング タスク (小分子とタンパク質構造の両方が折りたたまれます) では、ポケットの位置とタンパク質構造が不明 (ポケットなし、構造なし) で、AlphaFold 2 + DiffDock の組み合わせ予測の成功率は 18% に低下しました。さらに、AlphaFold 3 は 76.41 という最高の TP3T 成功率を達成しました。これは、AlphaFold 3 が正確に予測するだけでなく、ポケットや構造に関する事前の知識に依存する必要がないことを示しています。
低分子のポケットの位置とタンパク質の構造(ポケットあり、ホロ構造)が既知である、つまりポケットが露出している従来のドッキングタスクの 3 番目のタイプでは、Gold は 51.2% の成功率を達成し、Vina は成功率を達成しました。は 52.3% の成功率を示し、Glide は 55% に増加し、他の深層学習アルゴリズムも比較的良好なレベルに達する可能性があり、成功率がポケットの影響を受けることを示しています。
既知のポケット位置と未知のタンパク質構造 (ポケットあり、構造なし) の 4 番目のタイプのガイド付き共フォールディング タスクでは、AlphaFold 3 モデルの成功率が 76.4% から 90.2% に大幅に向上しました。ポケット情報が分かることでミッション成功率が向上します。ただし、ポケットの定義については現在議論があるため、したがって、リガンドドッキングタスクにおける AlphaFold 3 の具体的な改善を知りたい場合は、比較的安定している 2 番目のタイプのタスクの成功率のみを考慮する必要があります。
以下の図に示すように、モデル間でポケットの定義には大きな違いがあります。 Gold のポケットは 25 Å の球体 (図の左上の青い部分) ですが、Vina モデルはポケットの表現として 25 Å の立方体を使用し、DeepDock のポケット サイズは 10 Å、Uni-Mol のポケット サイズは 8 Å です。
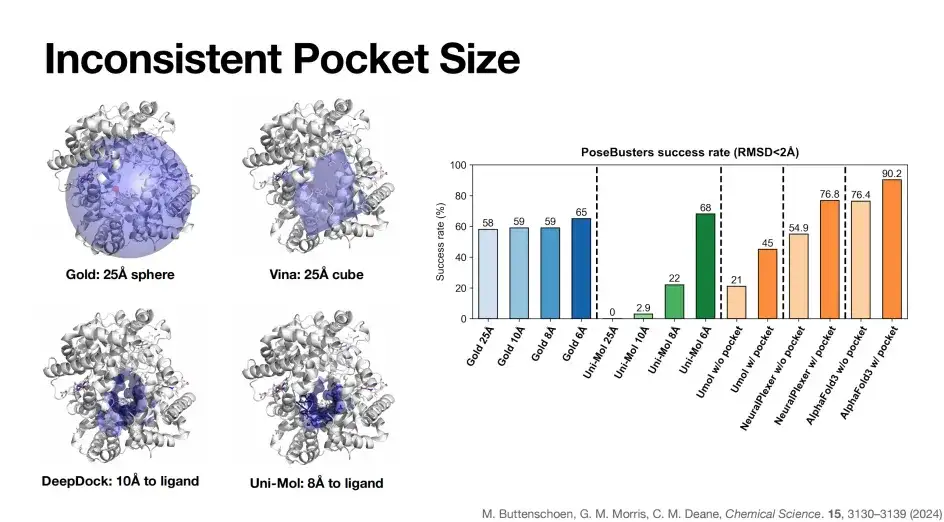
上図の右側に示すように、Gold モデルのポケット サイズが 25 Å から 6 Å まで徐々に減少すると、Gold の物理ベースのアルゴリズムの特性により、PoseBusters ベンチマークの成功率は比較的安定しています。逆に、深層学習アルゴリズム Uni-Mol はポケットを 6 Å まで徐々に縮小し、成功率は 68% まで増加し、25 Å 以下ではゼロに低下します。これは、一部の深層学習ドッキング アルゴリズムのポケットへの依存を反映しています。
同様に、前述したように、ポケット情報の導入後、AlphaFold 3 のドッキング成功率は 76.4% から 90.2% へと大幅に増加しました。要約すると、ポケット情報はモデル予測の成功率を向上させる上で重要な役割を果たします。ただし、理想的には、AlphaFold 3 など、ポケットや構造情報なしで高精度を達成できるモデルが最適です。
AlphaFold 3 は抗体と抗原の構造予測を実現します
AlphaFold 3 のもう 1 つの用途は、抗体と抗原の構造予測です。下図の左側は、AlphaFold 3 の抗体および抗原構造予測の性能評価です。低い評価基準 (DockQ>0.23) で 1 回だけ実行した場合、AlphaFold 3 の成功率は 40% (水色の線) よりも低くなりますが、1,000 回の試行後に予測成功率は 60% まで改善できます。
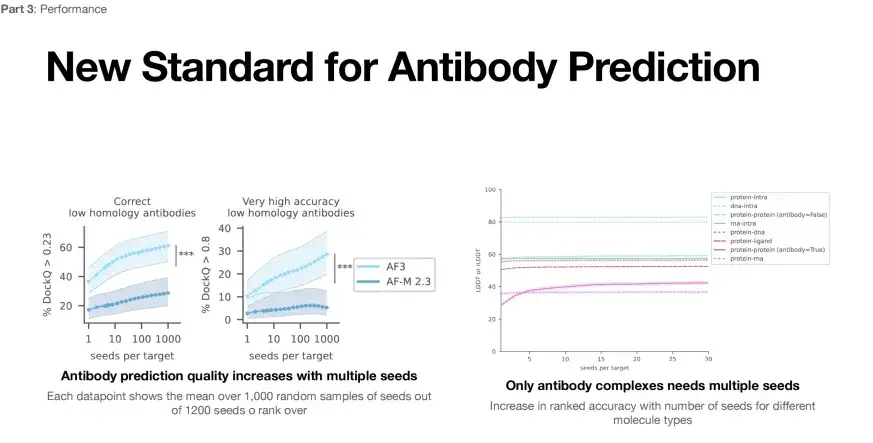
* 左: 抗体構造予測。各データ ポイントは、1,200 シードからランダムに選択された 1,000 シードの平均スコアを表します。
※右図:評価指標DockQが0.23より大きい場合は構造精度が検証されていないと考えられ、DockQが0.8を超える場合は構造予測の精度が高いと考えられます。
さらに、より厳格な基準 (DockQ > 0.8) で測定すると、1 回の実行の成功率は 10% に達する可能性がありますが、実行回数を 1,000 回に増やすと、成功率は 30% まで増加します。これは、AlphaFold 3 (ターゲットごとのシード) の実行数を増やすことで、抗体抗原構造予測の成功率を向上できることを示しています。
ただし、上図の右側に示すように、AlphaFold 3 はタンパク質間複合体の構造を予測する場合、実行回数を増やすことによってのみ成功率を向上させることができます。これは、AlphaFold 3 の適用可能性には予測も必要であることを示しています。他のタイプの複雑な構造のさらなる最適化。
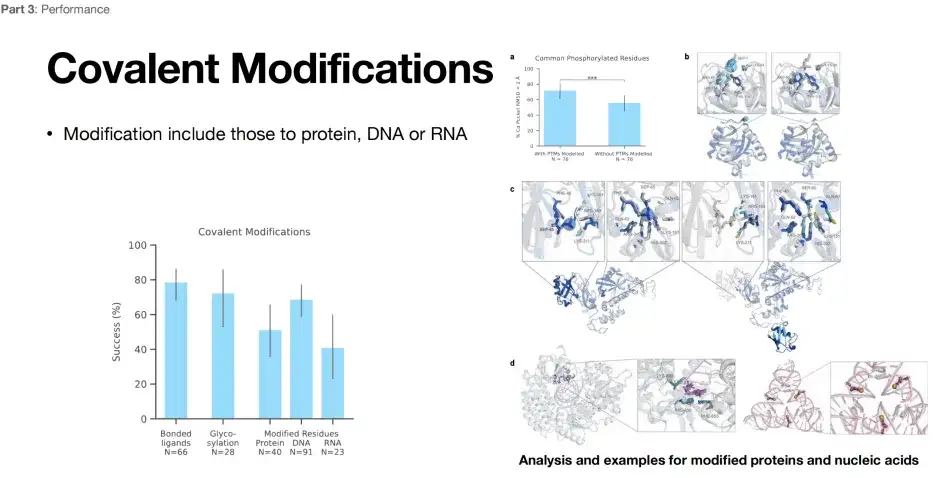
AlphaFold 3 は共有結合修飾予測を実装します
以下の図に示すように、AlphaFold 3 は修飾予測の点でも優れた構造予測機能を示します。成功率は、80%、60%、40% 程度に達します。 AlphaFold 3 は、共有結合修飾に携わる研究者にとって間違いなく強力なツールです。
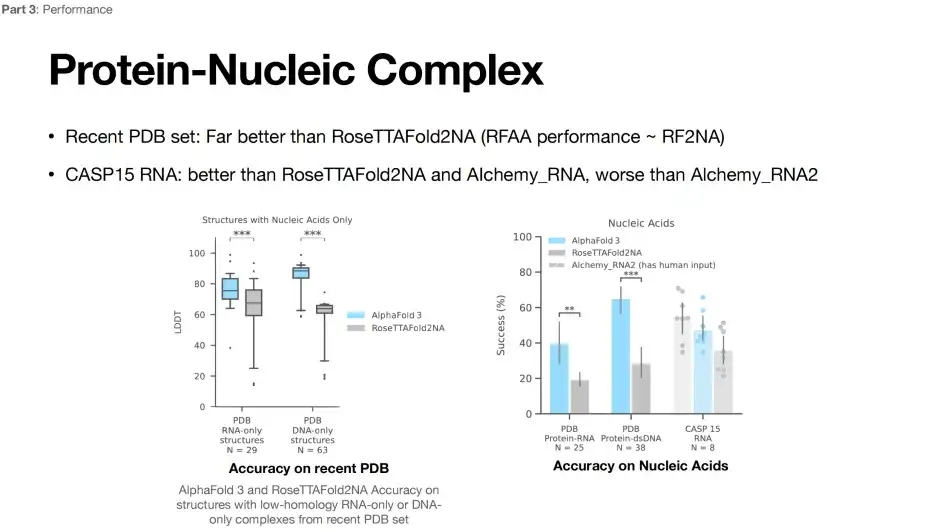
RNA構造予測におけるAlphaFold 3の限界
現在、RNA の構造予測は依然として困難です。以下の図に示すように、RoseTTAFoId2NA モデルと比較して、AlphaFold 3 の予測パフォーマンスは大幅に向上しています。ただし、CASP15 RNA の構造を予測する場合、AlphaFold 3 の精度は Alchemy_RNA2 (人間の入力あり) モデルの精度よりも低くなります。
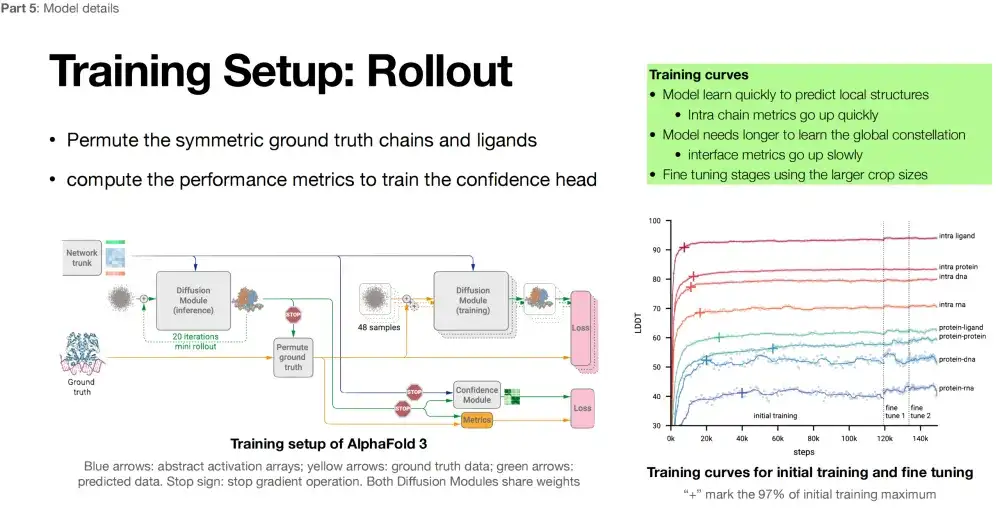
さまざまなタスクにおける AlphaFold 3 の長所と短所を比較する
AlphaFold 3 のトレーニング曲線を分析することにより、さまざまなタスクにおけるモデルのパフォーマンスを明らかにできます。LDDT インデックスが高いほど優れています。以下の図に示すように、このモデルはリガンド内 (内部リガンド) 構造を予測する場合に最高のパフォーマンスを発揮します。また、DNA (内部 DNA) 内予測の場合にも高い精度を示します。 DNA の安定した二重らせん構造のおかげで、このモデルは良好ですが、対照的に、このモデルはイントラ RNA (内部 RNA) ではあまり機能しません。
複合体予測の分野に目を向けると、モデルはタンパク質-リガンド複合体構造予測で最も優れたパフォーマンスを示し、次にタンパク質-DNA複合体予測ではモデルのパフォーマンスが低下し、タンパク質-RNA複合体予測のパフォーマンスが最も悪くなっています。この結果は、RNA の構造データが不足しており、その構造が動的で柔軟であること、これが構造生物学の分野で現在直面している問題の 1 つである、RNA 構造の予測の難しさを反映しています。
さらに、研究者が構造予測に AlphaFold 3 を使用したとき、予測結果の信頼性は、PAE テーブルを通じて評価することもできます。

AlphaFold 3 は完璧ではありません
AlphaFold 3 は完璧ではありません。たとえば、間違ったキラリティーが検出されてしまうことがあります。動作中に異常が発生した場合は、複数回実行して結果の安定性を確認することをお勧めします。第二に、AlphaFold 3 にはタンパク質の動的予測にも限界があります。これは、構造データの欠如とタンパク質の多次元立体構造情報を把握できないことが原因である可能性があります。
※左手と右手が鏡像で重ねられないのと同じように、物体が鏡像と異なることを「キラル」といいます。
さらに、AlphaFold 3 には、幻覚という生成モデルに共通する問題もあります。下図のタンパク質構造予測結果に示すように、タンパク質構造の左側の灰色の部分しか解析できず、残りの部分は電子密度不足により折りたたまれた状態になっている可能性があります。中央の図は、AlphaFold 2 がタンパク質を予測した結果です。青色の領域は折りたたまれた状態であると考えられ、その他の「リボン」部分は折り畳まれていない状態であると考えられます。予測された構造は比較的合理的です。右側は、AlphaFold 3 の予測結果です。この構造は、折り畳まれる可能性のあるすべての領域を折り畳む傾向がありますが、実際の状況に基づくと、上記の領域のほとんどは実際には折り畳まれません。したがって、AlphaFold 3 錯視は、タンパク質が折り畳まれていない状態を維持するのではなく、折り畳まれた状態にあると予測する傾向があります。

AlphaFold 3の幻覚問題に対処するために、研究者らは直接的で効果的な方法を選択しました。AlphaFold 2 によって予測された結果は比較的合理的であるため、AlphaFold 2 によって予測された結果は、モデルのトレーニング効果を高めるために AlphaFold 3 のトレーニング データセットに組み込まれました。ただし、この方法には制限があります。AlphaFold 2 自体の予測が間違っている場合、モデルをさらに最適化するために他のデータ ソースを導入できない限り、AlphaFold 3 の予測品質に影響を与える可能性があります。
さらに、256x OLA が AlphaFold 3 への入力として送信されると、以下の図に示すように、予測結果は二重層のような構造を示します。この構造は予想外または典型的ではありません。
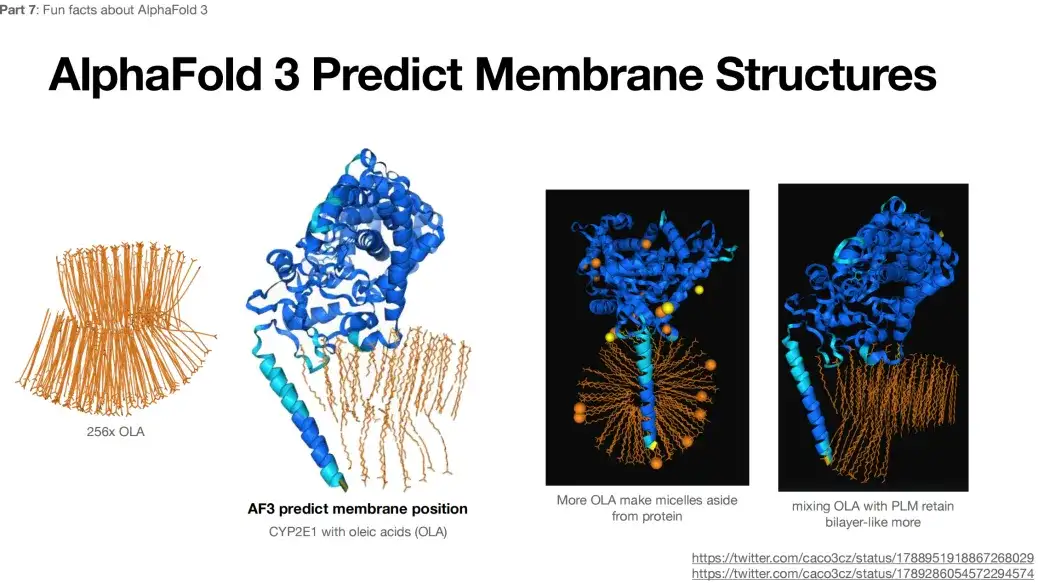
さらに、AlphaFold 3 による RNA と DNA の構造の予測は正確ではありません。下の図に示すように、RNA 構造を予測する場合には、G:G、G:A などの信じられないほどの相補的なペアも存在します。
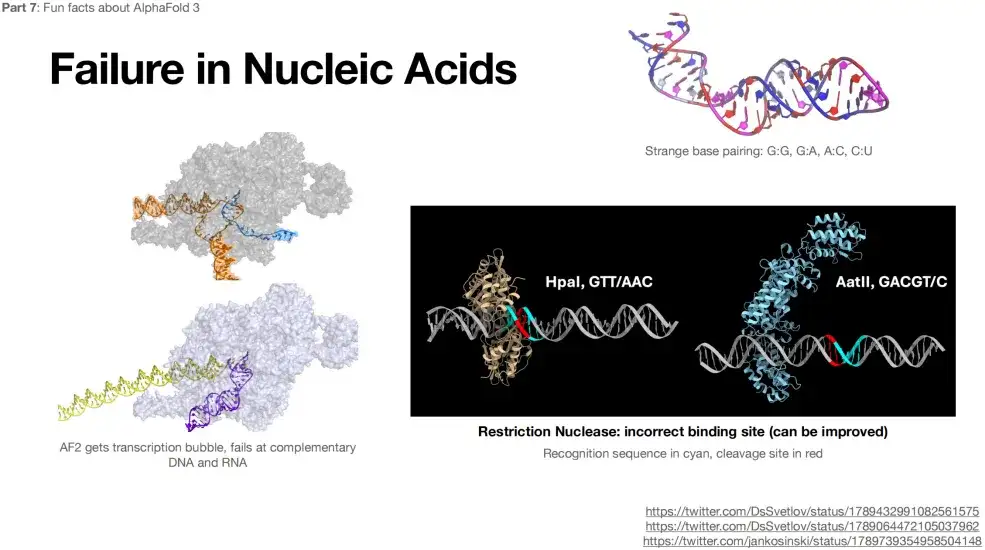
AlphaFold 3 の使用制限
データの機密性が高くないことを前提として、Google が提供する Web サイトから AlphaFold 3 にアクセスできます。ただし、以下の図に示すように、AlphaFold 3 は、タンパク質修飾に関して、現在、特定の 3 種類の修飾のみをサポートしており、合計 23 種類の DNA 修飾のみをサポートしています。 RNA 修飾は 15 種のみをサポートし、金属イオンは 10 種類の金属のみをサポートし、リガンドは 14 個の小分子に限定されます。
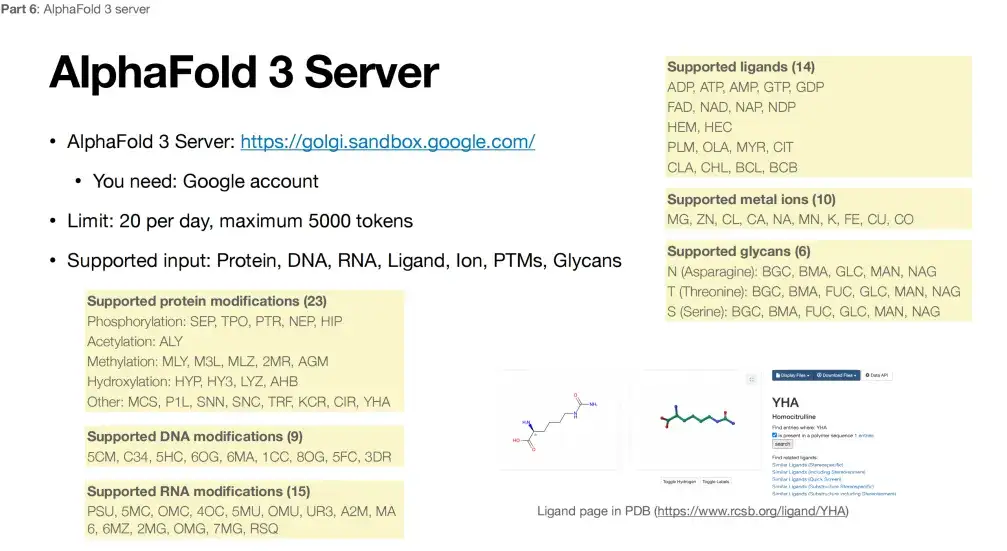
したがって、上記の特定の制限を考慮すると、おそらく真のオープンソースになるまで、AlphaFold 3 はほとんどの調査と応答を処理できない可能性があります。
要約すると、AlphaFold 3 は既存の AI モデルを超えて予測範囲を拡大するという点で大きな成果を上げましたが、特定のタスク、特に微細構造予測におけるパフォーマンスはまだ改善する必要があります。したがって、AlphaFold 3 は大幅な進歩を遂げましたが、特定の複雑な問題を完全に解決するには、引き続き研究と努力が必要です。
鍾伯子韬について

Zhong Bozitao は現在、上海交通大学で人工知能の博士課程の学生です。彼の主な研究対象は、ハイスループットのタンパク質の構造と機能の予測、タンパク質の立体構造生成などです。 2019 年以来、深海プロテオームと代謝経路の関係に関するハイスループットの AlphaFold 構造予測と解析の結果を 20 本以上発表し、国際遺伝学誌で金賞を受賞しました。エンジニアリングマシンコンペティション(iGEM)において3回、研究員を何度も務めた。
Google Scholar:
https://scholar.google.com/cita








