Command Palette
Search for a command to run...
ACL2024 メインカンファレンスに選ばれました InstructProtein: 知識命令を使用したタンパク質言語と人間の言語の調整

タンパク質は細胞の生存の基盤として、人体を含むあらゆる生物に存在し、組織や器官を構成する足場や主要物質であり、生命に不可欠な化学反応において中心的な役割を果たしています。
タンパク質構造の複雑さと多様性に直面して、タンパク質構造を解析する従来の実験方法は時間と労力がかかります。タンパク質大規模言語モデル (PLM) は、アミノ酸配列を入力として使用し、予測できる歴史的な瞬間に登場しました。タンパク質の機能や、まったく新しいタンパク質の設計さえも可能です。しかし、PLM はアミノ酸配列の理解には優れていますが、人間の言語を理解することはできません。
同様に、自然言語の処理に優れている ChatGPT や Claude-2 などの大規模言語モデル (LLM) が、タンパク質配列の機能を記述したり、特定の特性を持つタンパク質を生成したりするように求められた場合も、それを行うことができません。その理由は、現在のプロテインとテキストのペアのデータセットには 2 つの大きな欠点があります。1 つ目は、明確な指示信号が欠如していることです。2 つ目は、データのアノテーションが不均衡であることです。要約すると、LLM に関する現在の研究には未解決のギャップがあり、それは、人間の言語とタンパク質の言語を迅速に切り替えることができないということです。
このような問題を解決するために、浙江大学のChen Huajun氏とZhang Qiang氏のチームは、知識命令を使用してタンパク質の言語と人間の言語を整合させるInstructProteinモデルを提案した。これは、タンパク質言語と人間の言語の間の双方向の生成能力を調査し、2 つの言語間のギャップを効果的に橋渡しし、生物学的配列を大規模な言語モデルに統合する能力を実証します。
この研究のタイトルは「InstructProtein: 知識指導による人間とタンパク質の言語の調整」です。ACL2024本カンファレンスで承認される。
研究のハイライト:
* InstructProtein は、知識の指示を通じて人間の言語とタンパク質の言語を整合させる研究です
* タンパク質の言語と人間の言語の間の双方向の生成能力を調査し、2 つの言語間のギャップを効果的に埋める
* 多数の双方向プロテインテキスト生成タスクの実験により、InstructProtein が既存の最先端の LLM よりも優れていることが示されています

用紙のアドレス:
https://arxiv.org/abs/2310.03269
オープンソース プロジェクト「awesome-ai4s」は、100 を超える AI4S 論文の解釈をまとめ、大規模なデータ セットとツールを提供します。
https://github.com/hyperai/awesome-ai4s
データセット: 包括的な科学データセット
モデルの事前トレーニング段階のコーパスには、UniRef100 のタンパク質配列と PubMed 抄録の文が含まれています。このデータに基づいて、研究者らは 280 万件のデータを含む指導データセットを生成しました。
モデルの微調整段階では、UniProt/Swiss-Prot が提供するアノテーションを使用してタンパク質のナレッジ グラフが構築されます。これには、タンパク質のスーパーファミリー、ファミリー、ドメイン、保存部位、活性部位、結合部位、位置、機能、および関与する生物学的プロセス、知識因果モデリングのデータは InterPro および Gene Ontology データベースから取得されます。
モデルの評価段階では、研究者らはタンパク質の機能アノテーションにおけるモデルの能力を評価するためにジーンオントロジー(GO)データセットを選択し、次に金属イオン結合(MIB)予測におけるモデルの能力を評価するためにHuらのデータセットを選択した。
モデル アーキテクチャ: タンパク質知識指示データ セットを構築することにより、事前トレーニングされたモデルを微調整する
LLM にタンパク質の言語を理解する能力を与えるために、InstructProtein は 2 段階のトレーニング方法を採用しています。最初にタンパク質と自然言語コーパスに関する事前トレーニングを行い、次に確立されたタンパク質知識の指示データセットを通じて微調整します。
トレーニング前の段階
この研究では、多言語の事前トレーニング段階で、生物学に関連する大規模なテキスト データベースを使用して、モデルの言語理解と生物学分野における知識の背景を強化します。多言語主義とは、自然言語 (英語の要約など) および生物学的配列言語 (タンパク質配列など) を処理する能力を指します。
モデル微調整ステージ
モデルの微調整段階では、本研究では「知識教示」と呼ばれるデータセット構築手法を提案する。ナレッジ グラフ (KG) と大規模な言語モデルを活用して連携して、バランスの取れた多様な命令データ セットを構築します。この方法は、大規模な言語モデルがタンパク質言語を理解する能力に依存しないため、モデルのバイアスや錯覚によってもたらされる誤った情報を回避できます。具体的な構築プロセスは、次の図に示すように、主に 3 つの段階に分かれています。
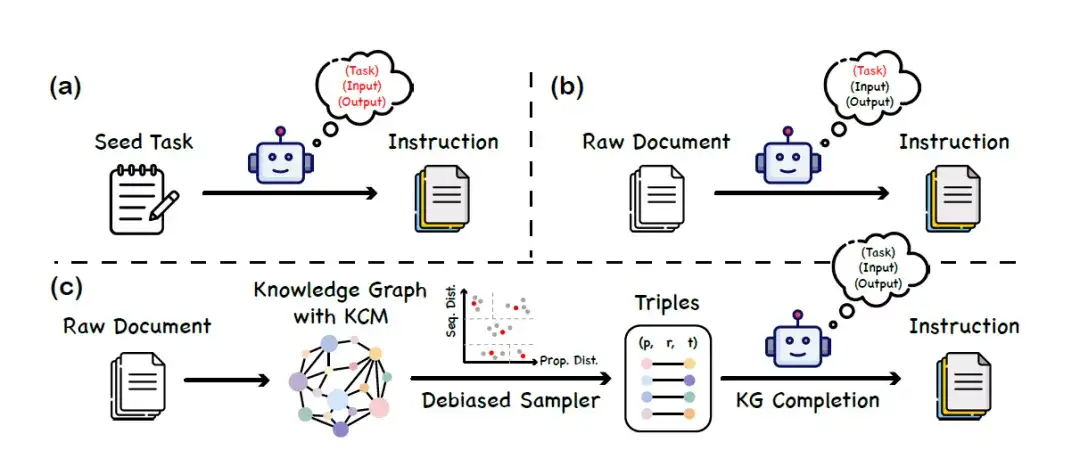
a. 一連のシード タスクを指定して、LLM に新しい命令データを生成するように指示します。
b. LLM を使用して、元のドキュメント (生のドキュメント) の内容に対応する指示データを生成します。
c. ナレッジグラフ(KG)に基づく命令生成フレームワーク
* ナレッジグラフの構築:研究者は、UniProtKB をデータ ソースとして使用して、タンパク質ナレッジ グラフを構築します。研究者らは、連鎖思考のアイデアを活用して、タンパク質のアノテーションにも論理的な連鎖が存在することに気づきました。例えば、タンパク質が関与する生物学的プロセスは、その分子機能および細胞内局在化と密接に関連しており、分子機能自体はタンパク質ドメインの影響を受ける。
このタンパク質知識の因果関係を表すには、研究者らは、知識因果モデリング (KCM) と呼ばれる新しい概念を導入しました。具体的には、知識因果モデルは、有向非循環グラフに編成された複数の相互接続されたトリプルで構成され、エッジの方向が因果関係を表します。この図は、微視的レベル (構造などのタンパク質配列の特徴をカバー) から巨視的レベル (生物学的機能をカバー) までにわたるトライアドを整理しています。以下の図は、KCM を含むトリプレットが与えられた場合、タスクを完了するためにナレッジ グラフと組み合わせた大規模な言語モデルを使用して、事実に基づいた論理的で多様な命令を生成するプロセスを示しています。
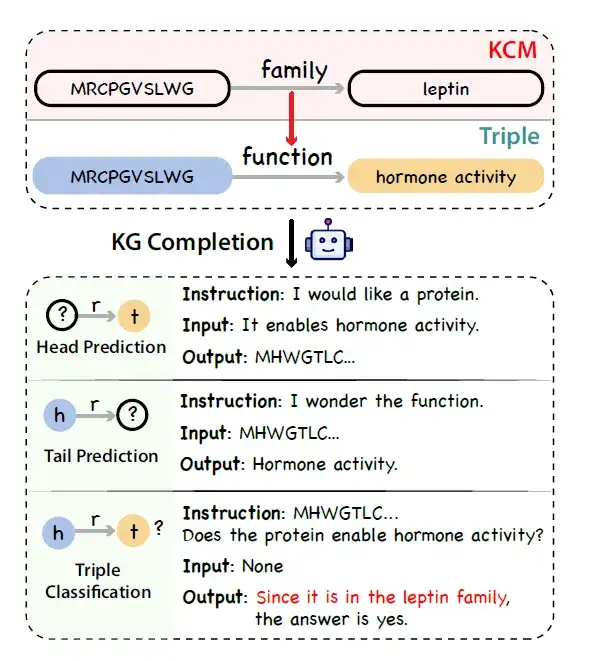
* ナレッジグラフのトリプルサンプリング:ナレッジ グラフにおける注釈の不均衡問題を考慮して、研究者らは、均一サンプリングの代替戦略として、均一サンプリングに代わる偏りのないサンプリング戦略を提案しました。具体的には、まず配列と特性の類似性に基づいてタンパク質をグループ化し、次に各グループ内のトリプルを均一に抽出します。
* コマンドデータの生成:研究者らは、タスクを完了するためにナレッジ グラフをシミュレートし、汎用 LLM (ChatGPT など) を使用して、KCM を備えたナレッジ グラフ トリプルを命令データに変換しました。
このようにして、タンパク質の言語を理解するために事前に設定されたモデルに依存することなく、タンパク質の機能と位置に関する豊富でバランスの取れた命令データセットを効果的に作成できます。その後のタンパク質機能の研究と応用のために、より信頼性の高いデータ サポートを提供します。
事前トレーニングと微調整を組み合わせることで、InstructProtein と呼ばれる結果のモデルは、タンパク質配列に関係するさまざまな予測タスクやアノテーション タスクをより適切に実行できるようになります。たとえば、タンパク質の機能を正確に予測したり、特定の細胞内の位置を特定したりすることは、タンパク質工学、創薬、およびより広範な生物医学研究にとって重要な意味を持ちます。
研究結果: InstructProtein は既存の最先端 LLM を上回るパフォーマンスを発揮
この研究では、タンパク質配列の理解と設計における InstructProtein の能力を包括的に評価しています。
タンパク質配列の理解
研究者らは、次の 3 つの分類タスクに関する InstructProtein モデルのパフォーマンスを評価しました。タンパク質の位置予測、タンパク質の機能予測、タンパク質の金属イオン結合能予測。これらのタスクは自然言語の読解問題と同様に設計されており、各データにはタンパク質配列と質問が含まれており、モデルはすべての評価がゼロショット設定で実行されます。
評価結果を以下の表に示します。すべてのベースライン モデルと比較して、InstructProtein はすべてのタスクで新しい最先端のパフォーマンスを達成します。
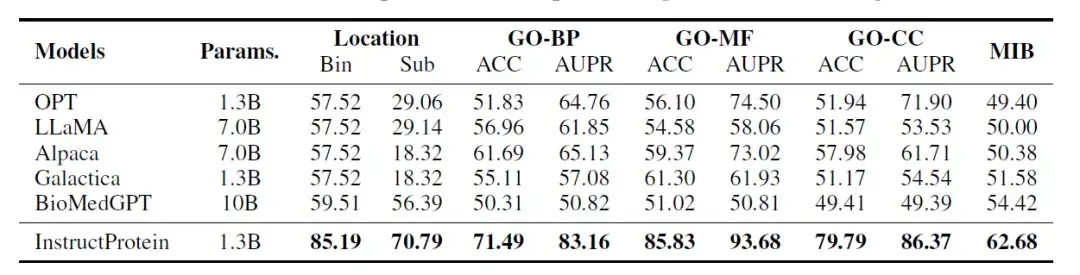
さらに、2 つの重要な発見は注目に値します。まず、InstructProtein は、自然言語トレーニング コーパス (OPT、LLaMA、Alpaca) から派生した LLM よりも大幅に優れています。これは次のことを示していますタンパク質と自然言語が共存するコーパスを使用したトレーニングは、LLMにとって有益であり、タンパク質の言語理解能力を向上させます。
第 2 に、Galaxy と BioMedGPT は両方とも自然言語とタンパク質のアラインメントのためのコーパスとして UniProtKB を利用していますが、InstructProtein は常にそれらを上回っています。その結果が検証されたのですが、研究内の高品質な指導データにより、ゼロショット設定でのパフォーマンスが向上します。
さらに、タンパク質の細胞内局在化 (bin) タスクでは、LLM (OPT、LLaMA、Alpaca、および Galactica) に重大な偏りがあるため、すべてのタンパク質が同じグループに分類され、精度は 57.52% となりました。
タンパク質配列設計
タンパク質の設計に関して、研究者らは「コマンドタンパク質ペアリング」タスクを設計した。タンパク質とその記述が与えられると、モデルは対応する記述と9つの非対応する記述から最も適切なものを選択する必要がある。
次の表に示すように:命令とタンパク質のペアリング タスクでは、InstructProtein がすべてのベースライン モデルを大幅に上回りました。
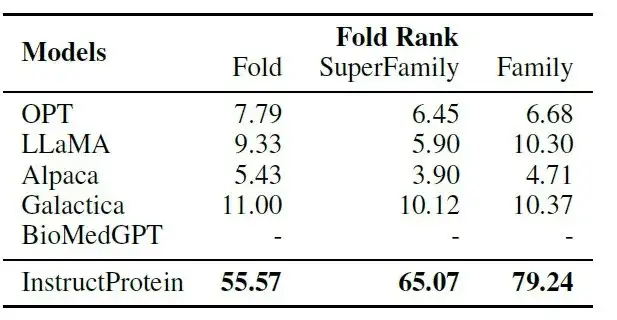
その中で、BioMedGPT はタンパク質をテキストに変換することに焦点を当てており、タンパク質の設計機能が欠けています。Galactica は、ナラティブタンパク質コーパスに基づいてトレーニングされているため、命令をタンパク質に揃えるゼロショット設定ではパフォーマンスが制限されています。これらの結果は、タンパク質生産の指示に従う能力における InstructProtein モデルの優位性を裏付けています。
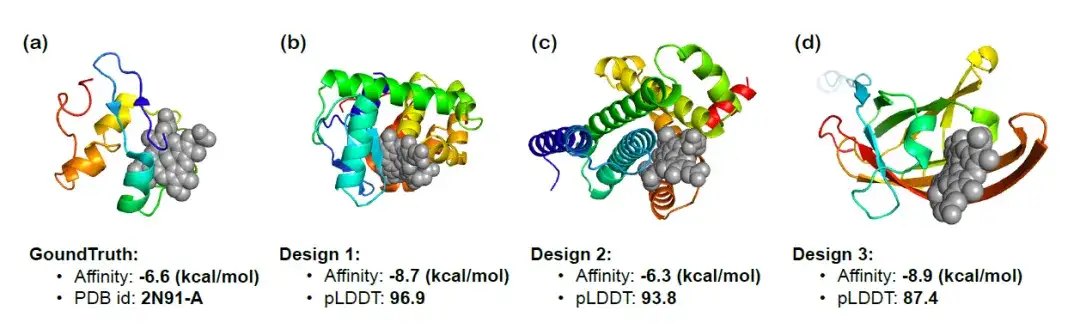
機能的に関連する指示に従ってタンパク質を設計する InstructProtein の能力をさらに検証するために、研究者らは InstructProtein を使用して特定の化合物に結合できるヘム結合タンパク質を設計し、生成された 3 つのタンパク質の 3D 構造を視覚化しました。以下の図は、ドッキング結果、結合親和性の予測 (結合親和性、低いほど良い)、および pLDDT スコア (絶対値が高いほど良い) を示しています。得られたタンパク質が顕著な結合親和性を示すことが観察できます。ヘム結合タンパク質の設計における InstructProtein の有効性が確認されました。
大型タンパク質モデルの探索はまだ始まったばかりです
近年、大規模な言語モデルが自然言語処理の分野に革命をもたらしました。これらのモデルは、言語翻訳、情報取得、コード生成など、日常生活のさまざまな側面で広く使用されています。ただし、これらの言語モデルは、自然言語やコード言語の処理では良好に機能しますが、生物学的シーケンス (タンパク質シーケンスなど) を処理することはできません。この文脈において、タンパク質大規模言語モデルの出現は適切なタイミングで起こります。
タンパク質大規模言語モデルは、アミノ酸配列、タンパク質の折り畳みパターン、その他のタンパク質関連の生物学的データなどのタンパク質関連データに基づいて特別にトレーニングされています。したがって、タンパク質の構造、機能、相互作用を正確に予測する能力があります。タンパク質言語モデルは、生物学における AI テクノロジーの最先端の応用例であり、タンパク質の配列パターンと構造を学習することで、タンパク質の機能と形状を予測することができ、新薬の開発や病気の治療に非常に重要です。そして基礎的な生物学的研究。
2023年4月、Science誌に掲載された研究では、メタAIチームの研究者が進化情報から導き出される大規模言語モデルを使用して、単一配列タンパク質を予測するための配列-構造予測子ESMFoldを開発したことが示された。その精度はAlphaFold2を上回っている。また、相同配列を持つタンパク質の予測精度は AlphaFold2 に近く、速度は一桁向上しています。このモデルは 6 億を超えるメタゲノムタンパク質を予測し、天然タンパク質の幅広さと多様性を実証しています。
2023 年 7 月、Baitu Biotech と清華大学は共同で、パラメーター量が最大 1,000 億 (100B) の xTrimo タンパク質一般言語モデル (xTrimoPGLM) と呼ばれるモデルを提案しました。理解タスクに関しては、xTrimoPGLM は複数のタンパク質理解タスクにおいて他の高度なベースライン モデルよりも大幅に優れており、生成タスクに関しては、xTrimoPGLM は天然のタンパク質構造に似た新しいタンパク質配列を生成できます。

論文リンク:
https://www.biorxiv.org/content/10.1101/2023.07.05.547496v3
2024 年 7 月清華大学知能産業研究所の准研究員である周昊氏は、北京大学、南京大学、水夢分子チームと協力して、マルチスケールタンパク質言語モデルESM-AA(ESM All Atom)を提案した。残基拡張やマルチスケール位置エンコーディングなどのトレーニング メカニズムを設計することにより、原子スケールの情報を処理する能力が拡張されました。ターゲットとリガンドの結合などのタスクにおける ESM-AA のパフォーマンスは大幅に向上し、ESM-2 などの現在の SOTA タンパク質言語モデルを上回り、Uni-Mol などの現在の SOTA 分子表現学習モデルも上回っています。関連する研究は、機械学習に関するトップカンファレンスである ICML で「ESM All-Atom: 統一分子モデリングのためのマルチスケールタンパク質言語モデル」と題して発表されました。

用紙のアドレス:
https://icml.cc/virtual/2024/poster/35119
タンパク質の大規模言語モデルに関連する研究では大きな進歩が見られましたが、タンパク質配列の空間的複雑性を完全に把握するにはまだ初期段階にあることを強調する価値があります。例えば、上記の InstructProtein モデルには数値タスクを処理するという課題があります。これは、3D 構造の確立、安定性評価、機能評価などの定量的な分析が必要なタンパク質モデリングの分野で特に重要です。未来、関連する研究は、定量的な説明を含むより広い範囲の指示に拡張され、定量的な出力を提供するモデルの能力を強化し、それによってタンパク質言語と人間の言語の統合を促進し、さまざまなアプリケーションシナリオでの有用性を拡大します。
参考文献:
1.https://arxiv.org/abs/2310.03269
2.https://mp.weixin.qq.com/s/UPsf9y9dcq_brLDYhIvz-w
3.https://hic.zju.edu.cn/ibct/2024/0228/c58187a2881806/page.htm








