Command Palette
Search for a command to run...
ACL2024代表に選出!タンパク質データとテキスト情報のクロスモーダル解釈を実現するために、中国科学技術大学のWang Xiang チームはタンパク質テキスト生成フレームワーク ProtT3 を提案しました。

タンパク質の動的構造の秘密を探ることは、新薬の開発を促進するための重要なステップであるだけでなく、生命プロセスを理解するための重要な基礎でもあります。しかし、タンパク質は複雑であるため、その深い構造情報を直接捕捉して分析することは困難であり、複雑な生物学的データを直感的でわかりやすい表現に変換する方法は、科学研究の分野において常に大きな問題となっています。
言語モデル (LM) の急速な発展により、革新的なアイデアが生まれました。言語モデルは大量のデータからテキスト情報を学習して抽出できるため、タンパク質データからタンパク質情報を「読み取る」ことを学習し、動的タンパク質構造情報を人間が理解しやすいテキストの物語に直接変換できるでしょうか?
大きな発展の可能性を秘めたこのアイデアは、実際の応用においては多くの課題に直面しています。たとえば、言語モデルはタンパク質配列のテキスト コーパスで事前にトレーニングされていますが、タンパク質の構造を理解するのは困難です。人間は「言葉」に関しては無力なようです。対照的に、タンパク質言語モデル (PLM) はタンパク質配列コーパスで事前トレーニングされており、優れたタンパク質の理解と生成能力を備えています。しかし、テキスト処理機能の欠如という制限も同様に重要です。
PLMとLMの利点を組み合わせて、タンパク質の構造を深く理解できるだけでなく、テキスト情報をシームレスに接続できる新しいモデルアーキテクチャを構築できれば、医薬品の研究開発、タンパク質の特性予測、分子設計、および分子設計に大きな影響を与えるでしょう。他の分野。しかし、タンパク質の構造と人間の言語のテキストは異なるデータ モダリティに属しており、障壁を突破して統合するのは簡単ではありません。
この点について、中国科学技術大学のWang Xiang氏は、シンガポール国立大学のLiu Zhiyuan氏のチームおよび北海道大学の研究チームとともに、新しいタンパク質テキストモデリングフレームワークProtT3を提案した。このフレームワークは、クロスモーダル プロジェクターを介して、モードの違いを持つ PLM と LM を組み合わせます。PLM はタンパク質の理解に使用され、LM はテキスト処理に使用されます。効率的な微調整を実現するために、研究者らは LoRA を LM に統合し、タンパク質からテキストへの生成プロセスを効果的に制御しました。
さらに、研究者らは、タンパク質キャプション、タンパク質 QA、およびタンパク質テキスト検索を含むタンパク質テキスト モデリング タスクの定量的評価タスクも確立しました。これらの 3 種類のタスクで ProtT3 は優れたパフォーマンスを達成しました。
「ProtT3: Protein-to-Text Generation for Text-based Protein Understanding」と題されたこの研究は、トップカンファレンスACL 2024に選ばれました。
研究のハイライト:
* ProtT3 フレームワークはテキストとタンパク質の間のモーダル ギャップを橋渡しし、タンパク質配列解析の精度を向上させます。
* プロテイン サブタイトル タスクでは、Swiss-Prot および ProteinKG25 データセット上の ProtT3 の BLEU-2 スコアは、ベースラインより 10 ポイント以上高くなっています。
* タンパク質の質問と回答タスクでは、PDB-QA データセットに対する ProtT3 の正確なマッチングのパフォーマンスが 2.5% 改善されました。
* タンパク質テキスト検索タスクでは、Swiss-Prot および ProteinKG25 データセット上の ProtT3 の検索精度はベースラインより 14% 以上高い
用紙のアドレス:
https://arxiv.org/abs/2405.12564
データセットのダウンロードアドレス:
https://go.hyper.ai/j0wvp
オープンソース プロジェクト「awesome-ai4s」は、100 を超える AI4S 論文の解釈をまとめ、大規模なデータ セットとツールを提供します。
https://github.com/hyperai/awesome-ai4s
タンパク質研究のための 3 つの主要なデータセットの構築と最適化
研究者らは、Swiss-Prot、ProteinKG25、PDB-QA の 3 つのデータセットを選択しました。
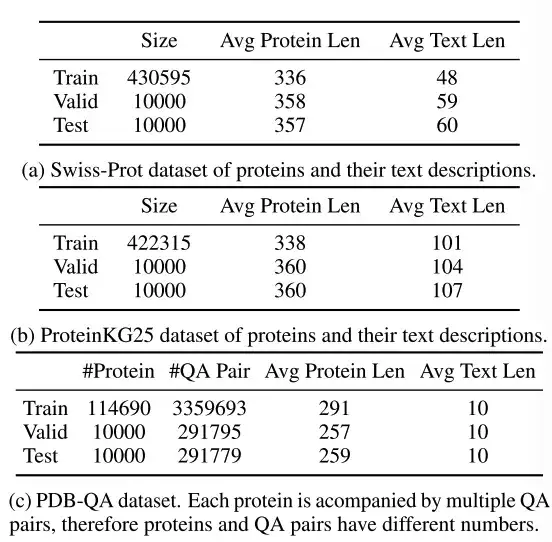
上の表に示すように、Swiss-Prot は、テキスト注釈付きのタンパク質配列データベースです。研究者らは情報漏洩を防ぐためにデータセットを処理し、テキスト注釈からタンパク質名を除外した。生成されたテキストの説明は、タンパク質の機能、位置、ファミリーの注釈を結び付けます。
ProteinKG25 は、Gene Ontology データベースから派生したナレッジ グラフです。研究者らはまず、同じタンパク質のトリプルを集約し、次にタンパク質情報を事前定義されたテキスト テンプレートに入力することで、トリプルをフリー テキストに変換しました。
PDB-QA は、RCSB PDB2 から派生したタンパク質のシングルラウンド質問応答データセットです。タンパク質の構造、特性、補足情報に関する 30 の質問テンプレートが含まれています。以下の表に示すように、詳細な評価を行うために、研究者は回答の形式 (文字列または数値) と内容の焦点 (構造/プロパティまたは補足情報) に基づいて質問を 4 つのカテゴリに分類しました。

ProtT3: 革新的なタンパク質からテキストへの生成モデル アーキテクチャ
下の図aに示すように、ProtT3 は、タンパク質言語モデル (PLM)、クロスモーダル プロジェクター (Cross-ModalProjector)、言語モデル (LM)、および LoRA モジュールで構成されます。プロテインからテキストへの生成プロセスを効率的に制御します。
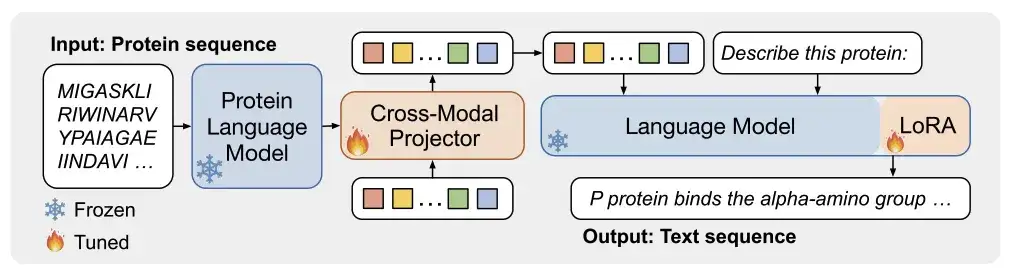
その中で、研究者が選択したタンパク質言語モデルは、タンパク質の理解に使用される ESM-2150M です。選択されたクロスモーダル プロジェクターは、PLM と言語モデル LM の間のモーダルな違いを埋めるために使用されます。次に、タンパク質を LM のテキスト空間にマッピングします。テキスト処理のために選択された言語モデルは、効率的な微調整を実現するために LoRA も言語モデルに組み込みました。 。
以下の図 b に示すように、ProtT3 は、タンパク質テキストの効率的なモデリングを強化するために 2 つのトレーニング ステージを採用しています。それらは、タンパク質テキスト検索トレーニングとタンパク質からテキストへの生成トレーニングです。
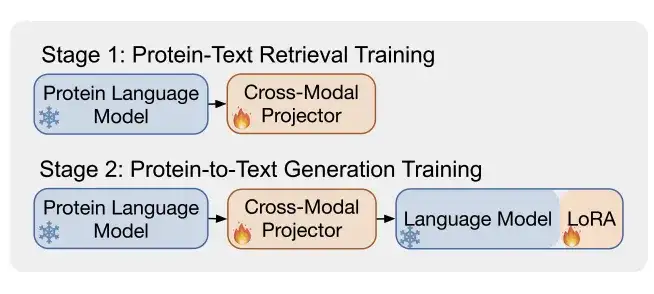
* フェーズ 1: タンパク質テキスト検索トレーニング
以下の図 a に示すように、クロスモーダル プロジェクター Q-Former は、タンパク質のエンコード用のプロテイン トランスフォーマーとテキスト処理用のテキスト トランスフォーマーの 2 つのトランスフォーマーで構成されています。 2 つのトランスフォーマーは自己注意を共有して、タンパク質とテキストの間の相互作用を実現します。
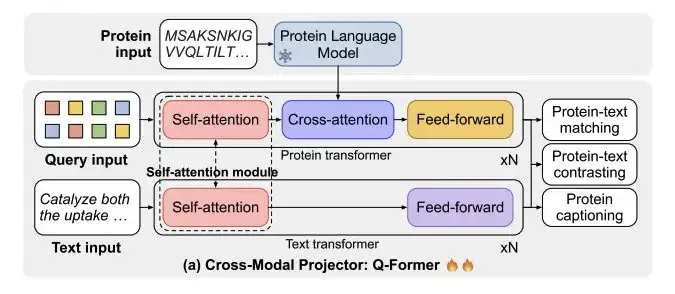
研究者らは、タンパク質テキスト検索のために、Swiss-Prot と ProteinKG25 を組み合わせたデータセットで ProtT3 をトレーニングしました。それぞれ、タンパク質とテキストの対比、タンパク質とテキストのマッチング (PTM)、タンパク質 キャプション (PCap) の 3 つのタスクが含まれます。
* フェーズ 2: プロテインからテキストへの生成トレーニング
研究者らはクロスモーダルプロジェクターを言語モデル(LM)に接続し、タンパク質情報を通じてテキスト生成プロセスを制御するためにタンパク質表現ZをLMに供給した。その中で、研究者らは、線形レイヤーを使用して Z を言語モデル入力の同じ次元に投影し、生成されたデータセットごとに ProtT3 を個別にトレーニングし、生成プロセスをさらに制御するためにタンパク質表現の後に異なるテキスト プロンプトを追加しました。
さらに研究者らは、タンパク質からテキストへの生成タスクで 3 つのデータセットに対して個別の微調整を実行するために LoRA を導入しました。
タンパク質分野のオールラウンダーが、3 つの主要なタスクで ProtT3 のパフォーマンスを評価します
ProtT3 のパフォーマンスを評価するには、研究者らは、タンパク質のキャプション作成、タンパク質の QA、およびタンパク質のテキスト検索という 3 種類のタスクについてテストを実施しました。
ProtT3 はタンパク質の真の記述に近く、精度が高い
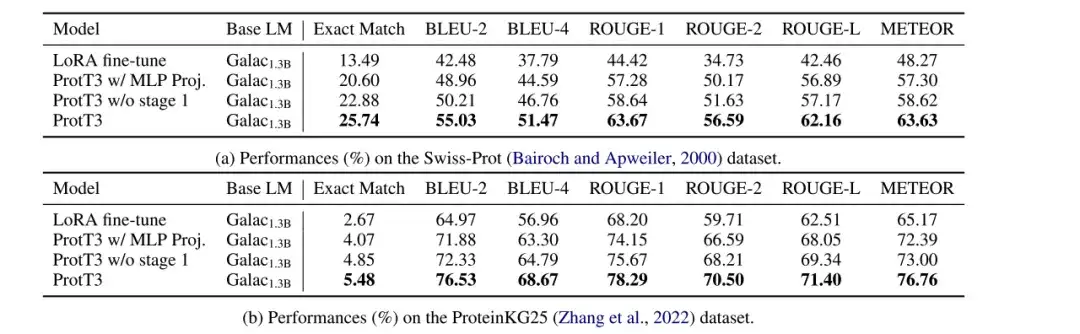
研究者らは、Swiss-Prot および ProteinKG25 データセットでのタンパク質キャプション タスクにおける LoRA 微調整 Gaoptica1.3B、MLP Proj. 付き ProtT3、ステージ 1 なしの ProtT3、ProtT3 モデルのパフォーマンスを評価し、BLEU、ROUGE、METEOR を使用しました。評価指標として。
* ProtT3 w/ MLP Proj.: ProtT3 のクロスモーダル プロジェクターの代わりに MLP を使用する ProtT3 のバリアント
* ステージ 1 なしの ProtT3: ProtT3 のトレーニング ステージ 1 をスキップした ProtT3 のバリアント
以下の図に示すように、LoRA ファインチューニング Gaoptica1.3B と比較すると、ProtT3 は BLEU-2 スコアを 10 ポイント以上改善します。タンパク質言語モデルを導入することの重要性と、タンパク質入力の理解における ProtT3 の有効性を視覚的に示します。さらに、ProtT3 は、さまざまな指標で 2 つのバリアントよりも優れたパフォーマンスを示しており、Q-Former プロジェクターとトレーニング フェーズ 1 を使用する利点を示しています。
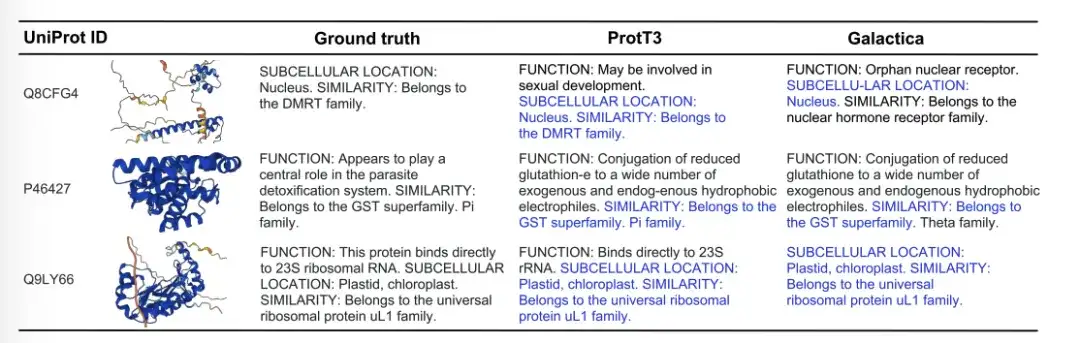
以下の図は、Ground truth、ProtT3、および Gaoptica の 3 つのプロテイン サブタイトル生成例を示しています。 Q8CFG4 の例では、ProtT3 のアノテーション コンテンツにより DMRT ファミリーがより正確に識別されましたが、Gaoptica では識別されませんでした。 P46427 の例では、どちらのモデルもタンパク質の機能を特定できませんでしたが、ProtT3 はタンパク質ファミリーをより正確に予測しました。 Q9LY66 の例では、両方のモデルが細胞内の位置とタンパク質ファミリーを予測することに成功しました。 ProtT3 は、タンパク質の機能を予測する上でさらに一歩前進し、真の説明に近づきます。
ベースラインモデル14%より精度が高く、ProtT3タンパク質テキスト検索能力が優れています
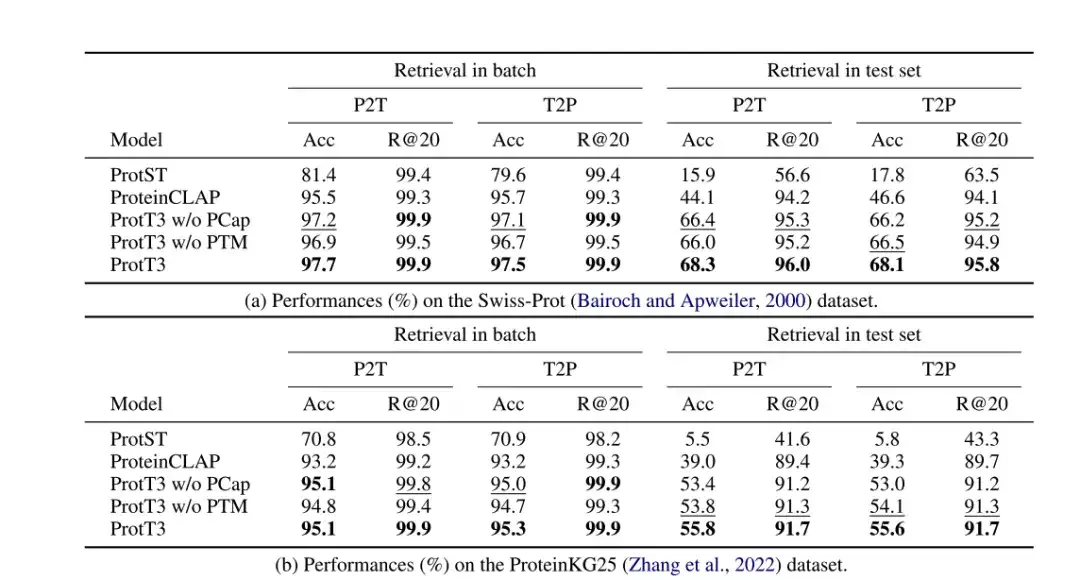
研究者らは、評価指標として精度と Recall@20 を使用し、ベースライン モデルとして ProtST と ProteinCLAP を使用して、Swiss-Prot および ProteinKG25 データセットに対するタンパク質テキスト検索における ProtT3 のパフォーマンスを評価しました。
以下の表に示すように、ProtT3の精度はベースラインモデルより14%以上高く、これは、ProtT3 がタンパク質を対応するテキストの説明と一致させる能力が優れていることを示しています。また、タンパク質テキストマッチング (PTM) により、ProtT3 の精度が 1% ~ 2% 向上します。これは、PTM により、タンパク質とテキスト情報が Q-Former の初期層で相互作用することが可能になり、それによってよりきめの細かいタンパク質とテキストの類似性測定が達成されるためです。Protein Caption (PCap) は ProtT3 の検索精度を約 2% 向上させます。これは、PCap がテキスト入力に最も関連するタンパク質情報を抽出するためにクエリ トークンを奨励し、タンパク質とテキストの位置合わせを容易にするためです。
* PTM なしの ProtT3: ProtT3 の PTM ステージをスキップします。
* PCap なしの ProtT3: ProtT3 の PCap ステージをスキップします。
ProtT3 はタンパク質の構造と特性を予測でき、より優れた質問と回答機能を備えています。
研究者らは、PDB-QA データセット上で ProtT3 のタンパク質質問応答パフォーマンスを評価し、評価指標として完全一致を選択し、ベースライン モデルとして LoRA 微調整された Gaoptica1.3B を使用しました (LoRA ft)。
以下の図に示すように、ProtT3 の完全一致パフォーマンスはベースラインより 2.5% 高く、タンパク質の構造と特性の予測においては常にベースラインよりも優れており、これは ProtT3 がタンパク質とテキストの問題を理解する優れたマルチモーダルな能力を備えていることを証明しています。
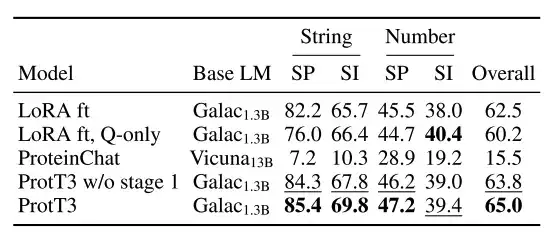
以下に示すように、以下の 3 つのタンパク質の質問と回答の例では、ProtT3 と Gaoptica の両方が、タンパク質の特性/構造に関する最初の 2 つの質問に正解しましたが、数値回答が必要な 3 番目の質問で不合格となり、ProteinChat は 3 つの質問で失敗しました。すべての質問に答えられませんでした。
タンパク質の言語を解き放つ、LLM のライフサイエンスにおける最先端の探求
タンパク質からテキストへの生成の分野における研究者らの探求により、人間は複雑な生物学的現象をわかりやすい方法で解明できるようになる可能性があります。上記の研究における言語モデルは、タンパク質の「潜在空間」についての深い理解を示すだけでなく、生物医学的タスクと自然言語処理の間の架け橋としても機能し、創薬やタンパク質の機能予測などの研究に新たな道を切り開きます。 。さらに遠く、数十億以上のパラメータを持つこれらの大規模な言語モデルがより複雑な言語構造を処理するために使用されれば、複数のレベルでの生命科学の将来の探求が改善されることが期待されます。
例えば、浙江大学のZhang Qiang氏とChen Huajun氏のチームはかつて、革新的な大規模言語モデルInstructProteinを提案した。このモデルは、双方向の人間の言語とタンパク質の言語を生成する機能を備えています。(i) タンパク質配列を入力として受け取り、そのテキストの機能記述を予測します。(ii) 自然言語を使用してタンパク質配列の生成を促します。

具体的には、研究者らはLLMをタンパク質と自然言語コーパスで事前トレーニングし、その後、2つの異なる言語の調整を容易にするために教師あり命令チューニングを採用した。 InstructProtein は、大規模な双方向のタンパク質テキスト生成タスクに優れており、テキストベースのタンパク質の機能予測と配列設計において先駆的な一歩を踏み出し、タンパク質と人間の言語理解の間のギャップを効果的に橋渡しします。
この論文は「InstructProtein: Knowledge struction による人間とタンパク質の言語の調整」と題され、ACL 2024 に選ばれました。
※論文原文:https://arxiv.org/pdf/2310.03269
また、シドニー工科大学のチームも浙江大学の研究チームと協力して、大規模言語モデル ProtChatGPT を共同で立ち上げました。このモデルはタンパク質の構造を学習して理解するため、ユーザーがタンパク質関連の質問をアップロードして対話型の会話に参加できるようになり、最終的には包括的な回答が得られます。
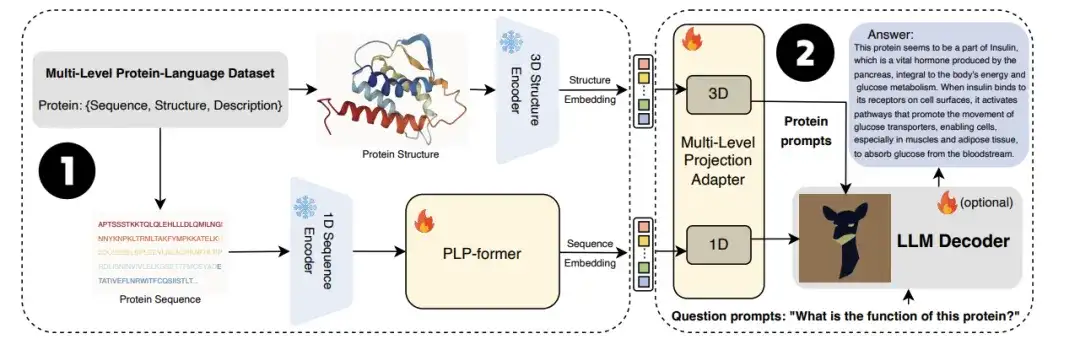
具体的には、タンパク質はまずタンパク質エンコーダーとタンパク質言語事前トレーニング トランスフォーマー (PLP フォーマー) を通過してタンパク質エンベディングを生成し、次にこれらのエンベディングがプロジェクション アダプターを通じて LLM に投影されます。最後に、LLM はユーザーの質問と投影された埋め込みを組み合わせて、有益な回答を生成します。実験では、ProtChatGPT がタンパク質とそれに対応する質問に対して専門的な応答を生成し、タンパク質研究の徹底的な探索と応用拡大に新たな活力を注入できることが示されています。
※論文原文:https://arxiv.org/abs/2402.09649
将来、大規模な言語モデルが大量かつ豊富なデータを使用して、人間の認知の限界をはるかに超えるタンパク質の潜在的なパターンや深い構造を推測できるようになると、テクノロジーが進歩し続けるにつれて、その可能性が大きく解き放たれることになると私たちは期待しています。言語モデルはタンパク質研究をより明るい未来に導くでしょう。








