Command Palette
Search for a command to run...
ICMLに選ばれました! MITチームがAlphaFoldに基づいて新たなブレークスルーを達成、タンパク質の動的な多様性を明らかに
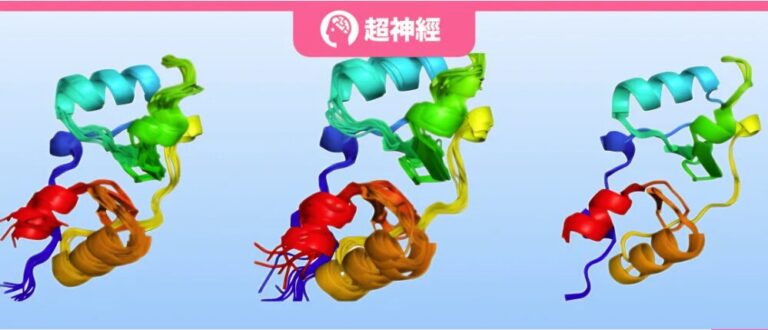
生物の重要な部分であるタンパク質は、集団運動や無秩序な変動のさまざまな構造の組み合わせに基づいてさまざまな状態をとり、複雑な三次元構造をとり、たとえばタンパク質の構造変化はトランスポーター、チャネル、酵素の機能に影響を与えます。は重要であり、特性のバランスの取れた組み合わせは、分子相互作用の強度と選択性を制御するのに役立ちます。
近年、AlphaFold などの深層学習手法はタンパク質の単一状態モデリングで大きな成功を収めていますが、構造の不均一性を説明することはできません。したがって、構造生物学者にとっては、単一の構造を正確に予測しながら、潜在的な構造の組み合わせを明らかにする方法、それは早急に解決する必要がある問題です。
最近、MIT の研究チームは、AlphaFold と ESMFold の新しいサンプリング方法を組み合わせて、フロー マッチング技術を通じてタンパク質の立体構造空間を観察および理解するための新しい視点を提供しました。
この調査では、フロー マッチングのバリアントである AlphaFlow と ESMFlow のパフォーマンスを 2 つの異なるシナリオで実証しています。このモデルは最終的に PDB で微調整され、ATLAS データセットでさらにトレーニングされました。どちらも優れたパフォーマンスを示し、構造の柔軟性の予測と原子位置分布モデリングにおいて従来の MSA ベースラインを上回っただけでなく、高次のグループ観察の再現においても優れたパフォーマンスを示しました。大きな進歩も見られました。
関連研究は「AlphaFold Meets Flow Matching for Generating Protein Ensembles」というタイトルで、AI分野のトップ学会であるICML 2024に選出されました。

用紙のアドレス:
https://openreview.net/forum?id=rs8Sh2UASt
オープンソース プロジェクト「awesome-ai4s」は、100 を超える AI4S 論文の解釈をまとめ、大規模なデータ セットとツールを提供します。
https://github.com/hyperai/awesome-ai4s
データセット: 実験結果の公平性を確保するために、PDB と ATLAS という 2 つの主要なデータセットに基づいています。
AlphaFold は PDB の構造に基づいてエンドツーエンドの方法で開発およびトレーニングされるのに対し、ESMFold はタンパク質言語モデル (PLM) からの埋め込みを入力として使用することが知られています。したがって、この研究では主に PDB データセットと MD データセットを使用します。
まず、PDB から構造的に不均一なタンパク質のテスト セットを構築するために、この研究では SIFTS アノテーション データベースと、PDB 鎖から UniProt 参照配列へのその残基レベルのマッピングを使用して、堆積された各鎖をフラグメントと関連付けました。次にこの研究では、Jaccard の類似性閾値 0.75 に基づいてクラスターのすべてのフラグメントを完全に結合し、結果として得られる各クラスターを固有のタンパク質として扱いました。これにより、75,000 個のタンパク質が得られました。
さらに、この研究では以下の情報も収集されました。
* AlphaFold はトレーニング期限前にチェーンを提出しませんでしたが、期限後に 2 ~ 30 チェーンのタンパク質をデポジットしました。
*長さが 256 ~ 768 残基のタンパク質。
* 鎖凝集の閾値が対称 IDDT-Cα で 0.85 で完全に結合している場合、少なくとも 2 つの構造クラスターを持つタンパク質。
これにより、2,843 個の鎖で表される 563 個のタンパク質が得られました。研究者らは、500 鎖で表される 100 個のタンパク質をサンプリングしてテスト セットを作成しました。
次に、研究者らは MD データセットに基づいて ATLAS データセットを構築しました。後者は、ECOD ドメイン分類に基づいて選択された 1,390 個のタンパク質で構成されます。各タンパク質について、データセットは 100 ns 長の 3 回の反復シミュレーションを提供し、それぞれに 10,000 フレームが含まれています。これらの軌跡のトレーニングと検証を実行するために、研究ではまず、提供されたシーケンスと ColabFold MMSeqs2 パイプラインを使用して、1,390 個の ATLAS エントリすべての MSA を生成しました。
その後、研究者らは、2018年5月1日と2019年5月1日をそれぞれトレーニング期限と検証期限として使用して、トレーニングパイプラインから300の立体配座をランダムに選択し、最終的に1265/39/82セットのトレーニングセット、検証セット、およびテストセットを取得しました。 。

モデル構築: AlphaFold をノイズ除去モデルとして使用し、タンパク質コレクションのフロー マッチングを実行します。
AlphaFold と同じ精度と一般化機能を備えた分布モデルを再開発するというかなりの課題を考慮して、この研究では生成モデルにおける最近の概念的な進歩を活用しています。AlphaFold を生成モデルとして再利用するのはほぼ簡単です。

これまで、テキストから画像への典型的な拡散モデル アーキテクチャは、ほとんどすべて、テキスト プロンプト s を条件とした画像 x の条件付き分布 p(x | s) を採用していました。これらのモデルの中核となるのは、ノイズの多い画像をテキスト キューとともに受信し、きれいな画像を予測するノイズ除去ニューラル ネットワークです。
これらの条件に基づいて、このようなモデルは通常、単純な平均二乗誤差 (MSE) 目標を使用してトレーニングされます。同様に、回帰のような損失関数 (AlphaFold や ESMFold など) でトレーニングされたタンパク質構造予測子は、ノイズの多い構造入力を追加するだけでノイズ除去モデルに変換できます。これらのアーキテクチャ調整を通じて、この研究ではさらに、AlphaFold と ESMFold を反復ノイズ除去ベースの生成モデリング フレームワークに挿入できます。
この研究では、フロー マッチング生成フレームワークの設計は、条件付き確率パス pt(x | x1) とそれに対応するベクトル フィールド ut(x | x1) を選択することと同等であると考えています。したがって、この研究では、q(x0) からノイズ x0 をサンプリングし、それをデータ点 x1 で線形補間することによって条件付き確率パスを定義し、それによって高度にパラメーター化されたニューラル ネットワーク x1(x, t; θ) を定義します。したがって、AlphaFold アーキテクチャがノイズ除去モデルとして使用されます。
フローマッチングをタンパク質構造に適用するために、この研究では、そのβ炭素(グリシンの場合はα炭素)の3D座標x ∈ R^N×3によって構造も記述しています。これにより、ニューラル ネットワークへの入力が常にポリマー状で物理的に健全な 3 次元構造になることも保証されます。
フロー マッチング フレームワークにはノイズ プロセスの定義と逆転が含まれるため、タンパク質構造の調和拡散と多くの類似点があり、どちらも同じ事前分布に収束します。ただし、より一般的な枠組みとしては、ストリーム マッチングには、次の 2 つの主な利点があります。
初め、調和拡散は、無限の時間制約下でのみ事前分布に収束し、収束速度はデータの次元、つまりタンパク質のサイズに依存します。これにより、比較的小さいサイズの作物のみをトレーニングする場合、推論時間の分布が変化します。
第二に、フロー マッチングは、PDB 内の非常に一般的な欠落残基を単純に省略することで処理する簡単な方法を提供します。対照的に、調和拡散では原子の位置間に依存性が生じるため、欠落している残基のデータ補完が必要になります。

最後に、この研究では、プロセス マッチング フレームワークに基づいて、PDB 上の AlphaFold と ESMFold のすべての重みを微調整しました。使用された AlphaFold と ESMFold のトレーニング期限は、それぞれ 2018 年 5 月 1 日と 2020 年 5 月 1 日でした。トレーニングのこのフェーズの終わりに、研究の結果、AlphaFold と ESMFold のフロー マッチング バリアントが生成されました。これらを AlphaFLOW と ESMFLOW と呼びます。
MD アンサンブルから学習する能力を評価するために、この研究では、全原子 MD シミュレーションを含む ATLAS データセット上の 2 つのモデルをさらに微調整しました。それぞれ 43,000 と 27,000 の追加の例でトレーニングした後、この研究では、MD 固有のモデル バリアント、AlphaFLOW-MD および ESMFLOW-MD を取得しました。
実験結果: 従来の手法を上回る性能を示し、構造生物学の分野で幅広い応用が期待できます。
研究者らはまず、PDB に登録されたタンパク質の多様な立体構造を組み立てる AlphaFLOW と ESMFLOW の能力を評価しました。
この目的を達成するために、研究では、AlphaFold トレーニング期限 (2018 年 5 月 1 日) の後に寄託された 100 個のタンパク質を含むテスト セットを構築し、複数の鎖と構造的不均一性の証拠、ターゲティング精度、再現率と多様性の 3 つの主要な指標を評価しました。

結果は、AlphaFLOW は、精度を犠牲にして予測の多様性を高めるという点で MSA のサブサンプリングに似ていることを示しています。ただし、MSA のサブサンプリングと比較して、AlphaFLOW のバリアントはパレート フロントを大幅に追跡します。
精度と再現率の観点からは、AlphaFLOW は、MSA サブサンプリングと非常によく似た動作を示します。少し驚くべきことに、どちらの方法も、ベースライン AlphaFold と比較して全体的な再現率を大幅に改善しません。
全体として、ESMFold と ESMFLOW の精度は、AlphaFold ファミリーのメソッドと比較して比較的低くなります。ただし、ESMFLOW は、ベースライン ESMFold と比較して大量の多様性を注入できます。精度をほとんど犠牲にすることなく再現率を向上させます。
さらに、この研究の RMWD 分析は、AlphaFlow が平均原子位置の予測において AlphaFold よりわずかに優れており、モデリングの分散において MSA サブサンプリングよりも大幅に優れていることを示しています。

この研究では、ATLAS データベース内の 82 のタンパク質テスト セットの代理 MD セットを生成する AlphaFLOW と ESMFLOW の能力をさらに評価しました。この研究では、各方法を使用して個別にサンプリングし、一連の評価を通じてサンプルとMD集団の類似性を調べました。
結果は次のことを示していますAlphaFLOW-MD は、MSA サブサンプリングのパフォーマンスをはるかに上回る、類似性の大幅な向上を実現します。

MD は真であるとみなされるため、収束までの実行にはコストがかかります。したがって、この調査では、GPU 時間などの同等の限られたコンピューティング予算の下で、AlphaFLOW がより良い結果を提供できるかどうかをさらに分析します。この目的を達成するために、この研究では、AlphaFLOW から抽出されるサンプルの数を減らし (250 から 4 に大幅に減少)、MD 軌道の長さを短縮しました (100ns から 160ps に調整)。
結果は、AlphaFLOW アンサンブルの品質は同じままですが、MD 軌跡が同じ品質レベルに達するか、それを超えるまでに時間がかかることを示しています。
タンパク質汎用事前学習モデルは3本の柱で立ち、構造生物学の分野は活力に満ちています
ここ数年、タンパク質とAIは絶えず衝突して新たな火種を生み出してきた。現在、一般的なプロテインプレトレーニングは3つの柱からなる新たな状況を形成しています。それは、DeepMind Alphafold シリーズ、David Baker の RoseTTAFold シリーズ、および Meta ESM シリーズです。これら 3 つの主要なモデルに基づいて、関連する科学研究の結果が爆発的に増え始めています。 2024 年上半期だけでも、多くの研究結果が Nature や Science などのトップジャーナルに掲載されました。
2024 年 3 月、ノースカロライナ大学医学部、カリフォルニア大学サンフランシスコ校、スタンフォード大学、ハーバード大学の研究者らは、次のことを裏付ける研究結果を Science 誌に発表しました。AlphaFold2 が予測した構造は、将来の創薬を導く可能性があります。研究チームは、AlphaFold2が数十億の化合物をスクリーニングし、ライブラリとタンパク質の構造を照合して潜在的な新薬を見つけることにより、構造生物学、タンパク質設計、相互作用、標的予測、機能予測、生物学的メカニズムにおいて重要な有用性を示すことを発見した。
2024 年 5 月、Google DeepMind チームは、Nature で AlphaFold 3 をリリースしました。これにより、この技術はタンパク質の折り畳みを超えて拡張され、タンパク質、DNA、RNA、リガンドなどの生命分子の構造と相互作用が前例のない精度で正確に予測されます。これはつまり、AlphaFold 3 は医薬品設計とゲノム研究をさらに加速します。人工知能細胞生物学の新時代を切り開きます。
AlphaFold 3 のリリースにより、Alphafold シリーズはついに全原子ベースを構築しました。同様に、RoseTTAFold シリーズも今年上半期に RoseTTAFold All-Atom のリリースに成功し、タンパク質の共有結合修飾と複数の核酸鎖と低分子のアセンブリを合理的に予測する機能を実現しました。
Alphafold3 と RoseTTAFold All-Atom の助けを借りて、研究者は想像力の力を解き放ちます。たとえば、2024 年 6 月、国際研究チームは、AlphaFold 3 と RoseTTAFold All-Atom を組み合わせた戦略を使用して、より効率的に薬剤を疾患細胞に直接送達し、症状を改善できる新しいタンパク質足場の設計に成功する方法を示す論文を Nature Biotechnology に発表しました。治療効果と副作用の軽減。この発見は、精密医療の分野における AI の応用における確かな一歩を示しています。
残念ながら、2023 年 8 月に Meta は ESMFold チームを解散し、AI の商用化の推進に注力してきました。しかし、ESMシリーズの研究は止まっていません。たとえば、このモデルはタンパク質言語モデリングの分野で重要な進歩を遂げ、マルチスケール情報を統合する統合モデリング ソリューションを提供します。注目すべき点は、アミノ酸情報と原子情報の両方を処理できる最初のタンパク質事前トレーニング済み言語モデルであることです。
このことからわかることは、Alphafoldシリーズ、RoseTTAFoldシリーズ、ESMシリーズが歩みを進める新時代に、AI とタンパク質研究はより密接に統合され、タンパク質の構造と機能の理解を加速するだけでなく、疾患治療、医薬品開発、バイオテクノロジー応用に革命的な変化をもたらすでしょう。 AI技術がもたらす飛躍的な発展により、構造生物学の分野はさらにダイナミックになり、生物医学の分野でも新たな章がゆっくりと展開されています。








