Command Palette
Search for a command to run...
上海交通大学のYu Xiang氏の研究グループは、複数のタイプのRNA修飾を特定し、計算コストを大幅に削減するための移植可能な深層学習モデルをリリースした。

2021年、中国科学院の高富氏の大声により、mRNAワクチンは一夜にして有名になり、新型コロナウイルスが猛威を振るう中、人々の希望となった。今日、その特別な過去は歴史になりましたが、mRNA ワクチンの背後にある RNA 修飾は依然として急速に開発されています。
いわゆる RNA 修飾は、RNA のさまざまな転写後プロセシングおよび代謝経路に広く関与する重要なタイプの転写後制御です。
RNA 修飾は、真核生物の成長と発達において重要な生物学的機能を果たしているため、注目に値します。たとえば、最近の研究では、哺乳動物の胚性幹細胞におけるN⁶-メチルアデノシン(m⁶A)の不安定化効果がさまざまな疾患に関連しており、5-メチルシトシン(m⁵C)が高温に対するイネの耐性に関連していることが判明しています。
しかし、RNAには多くの種類の修飾があり、天然のRNAではこれまでに160種類以上の修飾が見つかっています。これまで、オックスフォード ナノポア テクノロジーズ (ONT) が開発したナノポア ダイレクト RNA シークエンシング (DRS) 技術とディープラーニング手法を組み合わせることで、単一塩基の修飾識別を実現できました。しかし、この方法では、単一サンプル内で複数の修飾タイプを同時に検出することは困難です。
上記の問題に対して、上海交通大学生命理工学院常任准教授のYu Xiang氏の研究グループと、上海辰山植物園のYang Jun/Wang Honxiaチームは、論文を発表した。 『Nature Communications』のタイトル「転移学習により複数種類の研究論文の識別が可能になる」「ナノポアダイレクトRNAシーケンスを使用したRNA修飾」、転移可能な深層学習モデル TandemMod は、DRS における複数のタイプの RNA 修飾を識別するために開発されました。
研究のハイライト:
* 同じパフォーマンスを確保しながら、トレーニング セットのデータ量やモデルのトレーニング時間などの計算コストを大幅に削減します
* TandemMod は、動物、植物、微生物におけるさまざまな種類の RNA 修飾部位の同定やエピトランスクリプトーム研究に重要な技術サポートを提供します
*TandemMod は、RNA ワクチンなどの人工的に改変された RNA の検出にも使用できます。

用紙のアドレス:
https://www.nature.com/articles/s41467-024-48437-4
オープンソース プロジェクト「awesome-ai4s」は、100 を超える AI4S 論文の解釈をまとめ、大規模なデータ セットとツールを提供します。
https://github.com/hyperai/awesome-ai4s
データセット: 複数のデータセットを使用したターゲットを絞ったトレーニング
TandemMod モデルのパフォーマンスをトレーニングおよび評価するために、研究チームは複数のデータセットを使用して実験を実施しました。
初め、研究チームは、Nookaew研究所が生成したin vitro転写データセットELIGOSを使用しました。6 つの修飾塩基 (m¹A、m⁶A、m⁵C、hm⁵C、m⁷G、Ψ) の 5 つの塩基レベルの特徴 (平均、中央値、標準偏差、シグナル長および塩基の品質) を計算し、未修飾塩基と比較しました。
次に、研究チームは、真核生物の mRNA で最も一般的な 2 つの修飾、m⁵C と m⁶A に基づいて TandemMod のパフォーマンスを研究することを選択しました。研究者らは、Curlcake データセットで TandemMod m⁵C モデルをトレーニングしました。データセットは、考えられるすべての 5-mer を含む in vitro 転写配列から得られ、トレーニング セットとテスト セットに 4:1 の比率で分割されます。
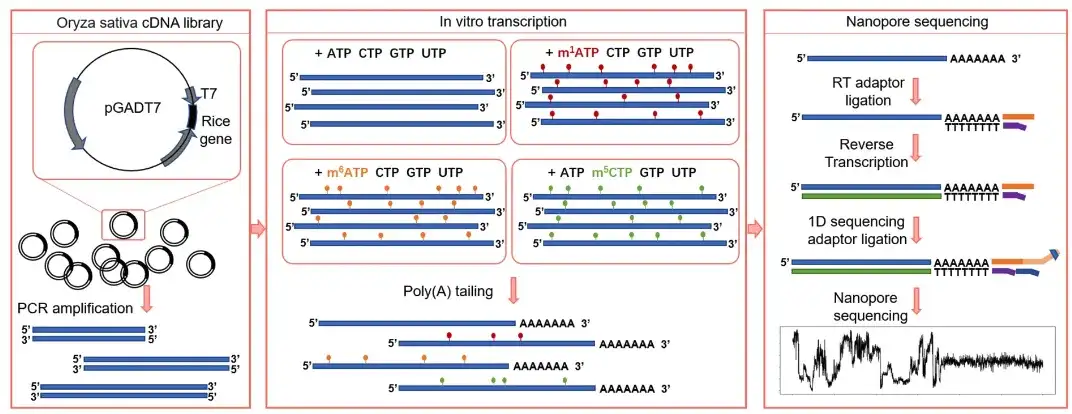
同時に、in vitro合成配列から転写されたRNAでは天然配列の全範囲をカバーできないという問題を解決するために、研究チームはT7プロモーターを含むイネcDNAライブラリーのin vitro転写を実施し、T7プロモーターを含む数千の転写物を取得した。その後、DRS を通じて 4 つのトレーニング セット (m1A、m6A、m5C、および未修飾塩基) を構築しました。これは、in vitro エピトランスクリプトーム データセット (IVET) と呼ばれます。
モデル アーキテクチャ: 深層学習フレームワーク
これをもとに研究チームは、5塩基ごとに割り当てられた電気信号とその統計的特徴を入力として、複数のRNA修飾タイプを同時に検出できる転移学習モデルTandemModを学習させた。
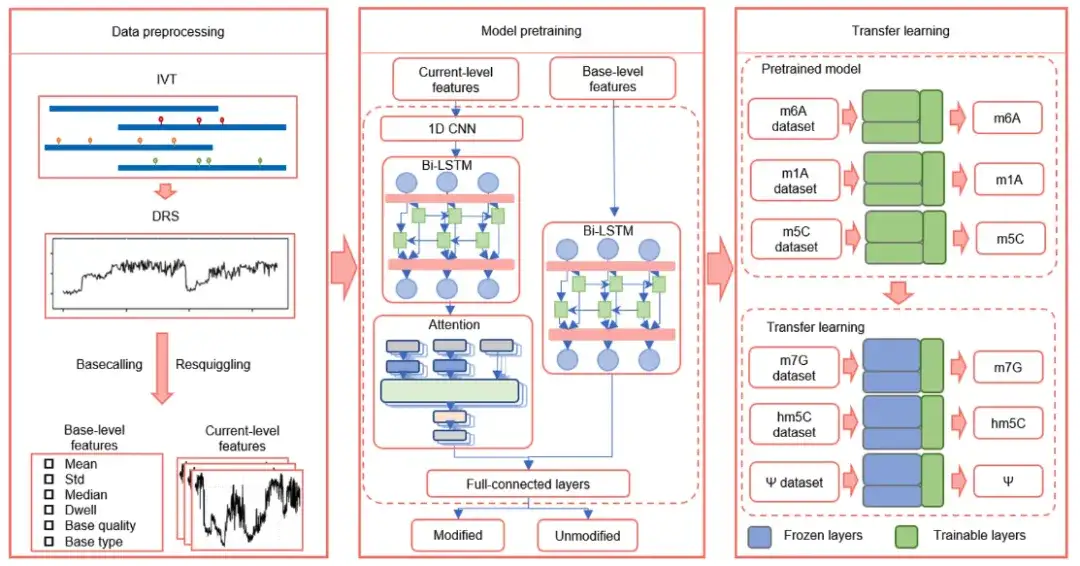
上の図に示すように、TandemMod は、データの前処理、モデルの事前トレーニング、転移学習で構成されます。
その中で、モデルの事前トレーニングは 4 つの主要なコンポーネントで構成されます。
* 1 次元畳み込みニューラル ネットワーク (1D-CNN)、元の電流強度信号の局所的特徴を抽出するために使用されます。
* 双方向長期短期メモリ モジュール (Bi-LSTM)。隣接する信号間の長期相関を捕捉し、より長いプロセスにおけるコンテキストの理解を向上させるために使用されます。
* アテンション メカニズム (アテンション) は、さまざまな時点での各特徴の重要性を重み付けし、重要な信号を捕捉するモデルの能力を向上させるために使用されます。
* 完全に接続されたレイヤーを持つ分類子は、すべての特徴の結合情報に基づいて予測を行う責任を負います。
さらに、DRS データに転移学習を適用して複数種類の RNA 修飾を検出できるかどうかを検証するために、研究者らは、IVET m5C データセットで TandemMod をトレーニングし、事前トレーニングされたモデルを取得しました。TandemMod モデルでは、最上位層は特徴抽出器として機能し、最下位層は分類器として機能します。研究者らは、分類エラーを最小限に抑えるために、事前トレーニングされたモデルの最上位層をフリーズし、ELIGOS トレーニング セット (hm5C、m7G、Ψ、および I) で最下位層を再トレーニングしました。

2サイクル後、全モデルで高精度を達成、hm⁵C、m⁷G、Ψ、I の ROC-AUC はそれぞれ 0.98、0.95、0.96、0.97 に達しました。上記の a、b、c、d に示すとおりです。
実験結果: TandemMod はトレーニング セットのデータ量とモデルのトレーニング時間を大幅に削減します。
実験段階で、研究チームは TandemMod モデルを従来の機械学習アルゴリズムと比較し、そのパフォーマンスを評価しました。比較対象は XGBoost、サポート ベクター マシン (SVM)、および k 最近傍 (KNN) でした。 Curlcake テストデータセット m⁶A 認識の場合、TandemMod は、精度 0.90 で他のアルゴリズムを上回ります。同様に、m⁵C の識別に関して、TandemMod は 0.95 の精度を達成しました。この比較は、DRS データを使用した修飾の識別における TandemMod の有効性を強調しています。
また、TandemMod は、in vivo での修飾率レベルが異なるサンプルの同定において、tombo や xPore よりも優れた優位性を示しました。これは、TandemMod がネガティブ コントロール サンプルを必要とせずに、さまざまな修飾率のサンプルを正確に予測できることを示しています。
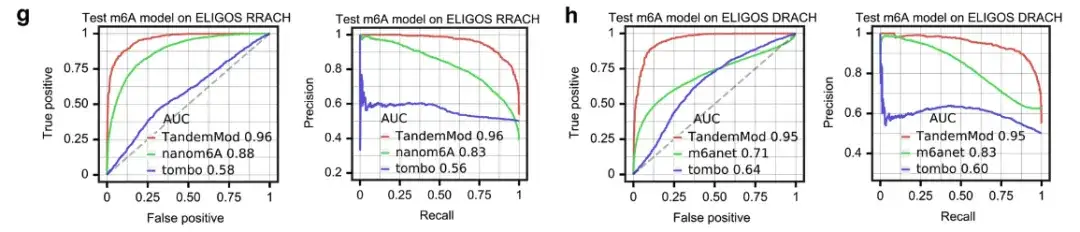
さらに、研究チームは、上図に示すように、TandemMod m⁶A モデルを tombo、nanom6A、m6Anet と比較しました。
ELIGOS 発疹 (RA または G、HA、または C または U) モチーフでは、TandemMod、nanom6A、および tombo の ROC-AUC はそれぞれ 0.96、0.88、および 0.52 でした。 ELIGOS DRACH (DA、G、または U) モチーフでは、TandemMod、m6Anet、および tombo の ROC-AUC はそれぞれ 0.95、0.71、および 0.64 でした。
これらの結果は次のことを示していますin vitro DRS データセットを使用してトレーニングされた TandemMod は、既存のツールの中で最も正確な読み取りレベルの予測を提供します。
研究チームは、m⁶A検出におけるTandemMod m⁵Cモデルの転移学習の分類パフォーマンス、必要なトレーニングデータ、コンピューティングリソースの利用状況を検証し、標準インスタンスのTandemMod m⁶Aモデルと比較しました。その結果、転移学習により、同じパフォーマンスを確保しながら、トレーニング セットのデータ量やモデルのトレーニング時間などのコストを大幅に削減できることがわかりました。
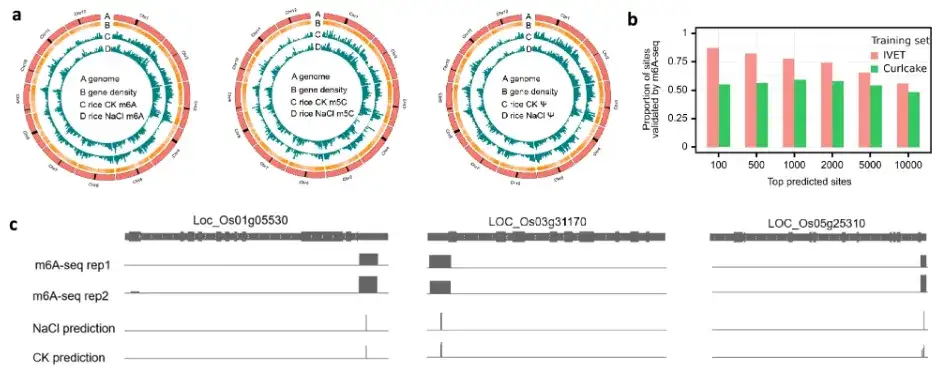
最後に、研究チームは、TandemMod モデルが DRS データ配列決定のために新種に一般化できる能力をテストし、ヒト細胞株 (2 つの修飾酵素ノックアウト サンプルと 5 つの野生型サンプル) を使用して TandemMod の信頼性をさらに検証しました。同時に、研究チームはTandemModを用いて高塩ストレス下のイネ苗におけるm⁶A、m⁵C、Ψの見かけの修飾マップを作成し、mRNAにおけるm⁶Aとm⁵Cの共修飾とその修飾率の変化を明らかにしました。塩分の多い環境。上の写真に示すように。
RNA修飾が生命探求への新たな扉を開く
いつの時代も、人々の人生の探求は決して止まることはありません。 RNA 世界仮説が提唱されて以来、生命の起源としての RNA という議論は、間違いなく現在最も説得力のある答えの 1 つとなっています。 1960 年に最初の RNA 修飾が発見されて以来、これは科学研究コミュニティにおいて長らく最優先事項であり、最近の研究でも依然として高い注目を集めています。
この論文の Yu Xiang の研究グループと Yang Jun/Wang Honxia チーム、および記事で言及されている ONT 会社に加えて、RNA 修飾の研究を行っているチームや企業は他にもあります。

たとえば、2021年、西安交通リバプール大学のMeng Jia教授のチームは、「広く発生する12のRNA修飾の統合予測と解釈のための注意ベースのマルチラベルニューラルネットワーク」というタイトルの論文を「Nature Communications」誌に発表した。 。
用紙のアドレス:https://www.nature.com/articles/s41467-021-24313-3
この記事では、アテンション メカニズムに基づくマルチラベル深層学習フレームワークに基づくモデル MultiRM について言及しています。12 の広範なトランスクリプトーム部位を同時に予測できるだけでなく、予測プロセスにおける重要な配列が抽出および分析され、さまざまなタイプの RNA 修飾間の強い相関関係が明らかになり、より包括的な分析と配列ベースの RNA 修飾メカニズムの理解に貢献します。

偶然にも、2021年の『Nature Biotechnology』に掲載された「xPoreを使用したナノポアダイレクトRNAシーケンスによる差次的RNA修飾の同定」というタイトルの論文では、研究チームは、xPore を使用して、Direct RNA-seq データから RNA 修飾を高精度で特定し、単一のハイスループット実験からの差次的修飾と発現を分析しました。
用紙のアドレス:https://www.nature.com/articles/s41587-021-00949-w
これらの研究は、RNA の世界への扉をさらに開くのに役立ち、「人生の真の意味」をさらに探求できるようになります。現在のさまざまな研究の進歩には、まだ克服しなければならないボトルネックがたくさんありますが、「先駆者」たちの絶え間ない挑戦によって、RNA研究の扉はすでに開かれています。
参考文献:
1. https://news.sjtu.edu.cn/jdzh/2








