Command Palette
Search for a command to run...
Cellサブマガジンにご参加ください!清華大学の張強峰氏の研究グループは SPACE アルゴリズムを開発し、その組織モジュール発見機能は同様のツールよりも優れています。
多細胞生物の細胞は同じゲノムを共有していますが、内部の遺伝子制御ネットワークや、周囲の微環境の性における隣接する細胞との外部シグナル交換の違いにより、形態、遺伝子発現、および機能において大きな多様性を示します。細胞型情報と組織内の空間的位置を相関させるために、空間トランスクリプトミクス (ST) テクノロジーが登場しました。この技術は、高解像度のトランスクリプトームデータを取得できるだけでなく、生命、個体発生、進化の構造を再理解するために重要な、異なる細胞サブタイプや転写状態の空間分布や位置関係を決定する位置情報にも対応します。人生の定義と病気の定義は重要な役割を果たします。
近年、空間トランスクリプトミクス技術の継続的な発展により、研究者は組織内の細胞の空間位置情報を保持しながら、単一細胞解像度で細胞の遺伝子発現プロファイルを取得できるようになりました。この空間情報を効果的に使用して空間細胞サブタイプを特定し、組織モジュールを発見する方法は、空間トランスクリプトーム データ解析の中核的なタスクとなっています。
現在、空間トランスクリプトーム データ解析は次の 2 つの困難に直面しています。 まず、空間的な細胞の種類の同定について、多くの研究では細胞の遺伝子発現プロファイルのみが使用され、細胞の空間的位置情報が無視されています。近年の研究では、もともと均一であると考えられていた細胞型が、組織内の位置に基づいて複数のサブタイプにさらに細分される可能性があることが示されています。第二に、組織モジュールのヘアスタイルについては、組織を構成するさまざまな細胞の遺伝子発現サインが非常に不均一である可能性があるため、これまでの解析方法では、単一細胞解像度の空間トランスクリプトームデータにおける細胞型の不均一性を十分に活用することができませんでした。
これに基づいて、清華大学生命科学部 張強峰准教授の研究グループ/構造生物学先端イノベーションセンター/清華大学・北京大学共同生命科学センター最近、「細胞間相互作用を認識した細胞埋め込みによる単一細胞解像度の空間トランスクリプトミクスデータにおける組織モジュールの発見」というタイトルの研究論文が Cell Systems 誌のオンライン版に掲載されました。
この研究では、グラフ オートエンコーダーの深層学習フレームワークに基づいた人工知能アルゴリズム SPACE (「相互作用を意識した」セル埋め込みによる空間トランスクリプトミクス データ分析) を開発しました。単一細胞解像度で空間トランスクリプトーム データから空間細胞タイプを識別し、組織モジュールを発見できることは、大規模な空間トランスクリプトーム研究に使用できます。
研究のハイライト:
* 単一細胞解像度で空間トランスクリプトームデータから空間細胞の種類を識別し、組織モジュールを発見できる空間トランスクリプトームデータ人工知能解析ツール SPACE を開発しました。
* SPACE は、特に複数の細胞型を含む複雑な組織において、細胞型の識別と組織モジュールの発見において他のツールよりも大幅に優れています。
* SPACE は、空間的に近い細胞間の相互作用が細胞の種類や組織モジュールの生物学的機能にどのような影響を与えるかを理解するための大規模な空間トランスクリプトーム研究に使用できます。
用紙のアドレス:
https://www.cell.com/cell-systems/fulltext/S2405-4712(24)00124-8
オープンソース プロジェクト「awesome-ai4s」は、100 を超える AI4S 論文の解釈をまとめ、大規模なデータ セットとツールを提供します。
https://github.com/hyperai/awesome-ai4s
データセット: 複数のデータセットで SPACE の機能を検証
SPACE の機能を検証するために、研究では複数のデータ セットが使用され、次のようにまとめられました。
データセットのダウンロードアドレス:
https://go.hyper.ai/CBJfX
MERFISH マウス PMC データセット
MERFISH マウス PMC データセットの場合、対数変換された正規化された細胞遺伝子マトリックスが Brain Image Library から取得され、「その他」とラベル付けされた細胞またはメイン サンプル領域の外側に位置する細胞が除去されました。
データセットのリンク:
STARmap マウス PLA データセット
STARmap マウス PLA データセットの場合、正規化された細胞遺伝子マトリックスが元の論文から提供され、対数変換されました。
データセットのリンク:
https://drive.google.com/file/d/1DDCowUuZ7PPFUSZsjvSqntWkYJMjf1Na/view?usp=sharing
MERFISH マウス AB データセット
MERFISH マウス AB データセットの場合、遺伝子数行列は CELL x GENE ライブラリから取得されました。細胞あたりの総数は 10,000 に正規化され、正規化された細胞遺伝子行列が対数変換されました。
データセットのリンク:
https://cellxgene.cziscience.com/collections/31937775-06024e52-a799-b6acdd2ba2e
MERFISHマウスWBデータセット
MERFISH マウス WB データセットの場合、対数変換された正規化された細胞遺伝子行列が GitHub リポジトリから取得されました。
データセットのリンク:
https://github.com/AllenInstitute/abc_atlas_access
Xenium ヒト BC データセット
Xenium ヒト BC データセットの場合、遺伝子数行列は 10x genomics Web サイトから取得しました。細胞あたりの総数は 10,000 に正規化され、正規化された細胞遺伝子行列が対数変換されました。
データセットのリンク:
https://www.10xgenomics.com/products/xenium-in-situ/preview-dataset-human-breast
CosMx ヒト NSCLC データセット
CosMx ヒト NSCLC データセットの場合、対数変換された正規化された細胞遺伝子行列は、nanoString Web サイトから取得されました。
データセットのリンク:
https://nanostring.com/products/cosmx-spatial-molecular-imager/ffpe-dataset/nsclc-ffpe-dataset
Visium 人間の脳データセット
Visium 人間の脳データ セットの場合、遺伝子数行列は Bioconductor パッケージ spatialLIBD を使用して取得されました。 Python パッケージ SCANPY (v1.9.1) の scanpy.pp.highly_variable_genes() 関数 (flavor = “seurat_v3”) を使用して、Visium 人間の脳データセットの各サンプルで上位 3,000 個の非常に変動性の高い遺伝子が特定されました。次に、細胞あたりの総数を 10,000 に正規化し、正規化された細胞遺伝子行列を対数変換しました。
データセットのリンク:
https://bioconductor.org/packages/release/data/experiment/html/spatialLIBD.html
モデルアーキテクチャ: 細胞間相互作用の知覚に基づく細胞埋め込みのモデル
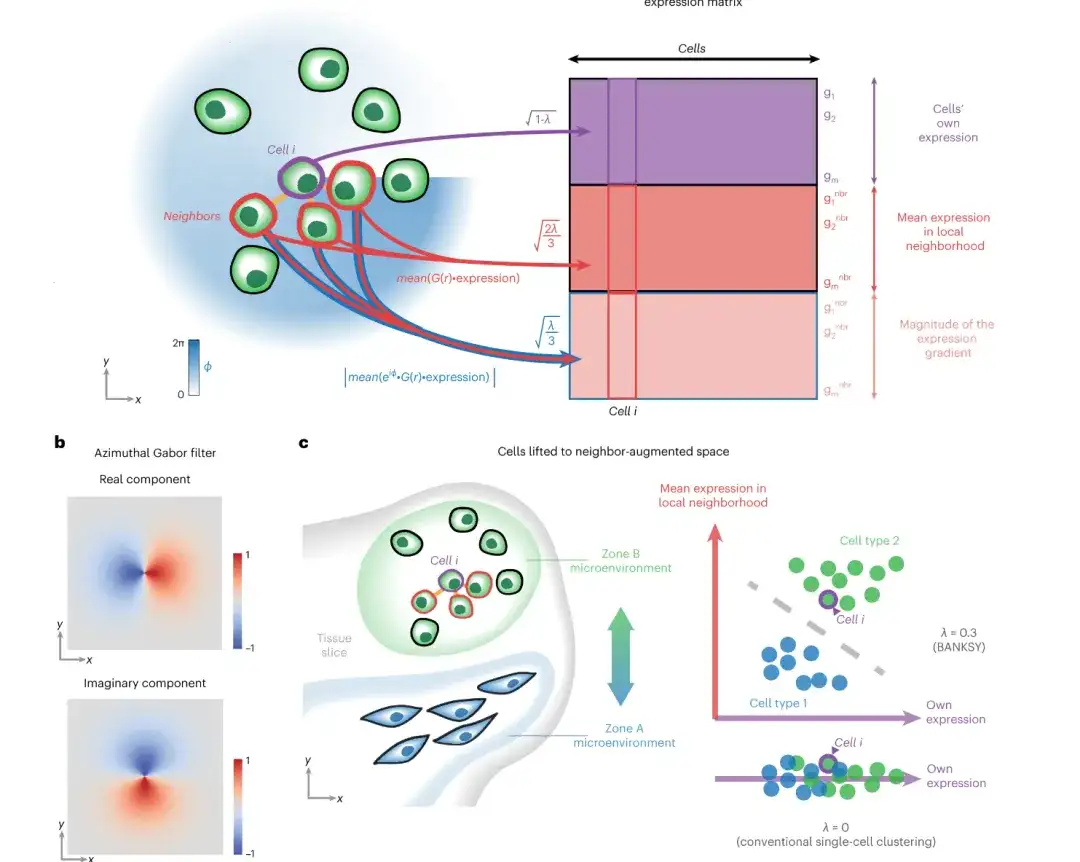
SPACE は、グラフ オートエンコーダー フレームワークを使用して、各細胞自身の遺伝子発現情報と、空間トランスクリプトーム データ内の空間的に隣接する細胞との相互作用情報を記述する低次元の細胞埋め込みを学習します (そのため、「細胞埋め込み」という用語は、「細胞間相互作用を意識した細胞」です)埋め込み。この細胞の埋め込みに基づいて、SPACE はクラスタリング アルゴリズムを使用して空間細胞のサブタイプを特定し、組織モジュールを発見します。
建築的な観点から見ると、SPACE モデルは、エンコーダー (3 層グラフ アテンション ネットワーク)、隣接グラフ デコーダー、遺伝子発現デコーダーの 3 つの部分で構成されます。以下の図は、モデルの全体的なフレームワークを示しています。
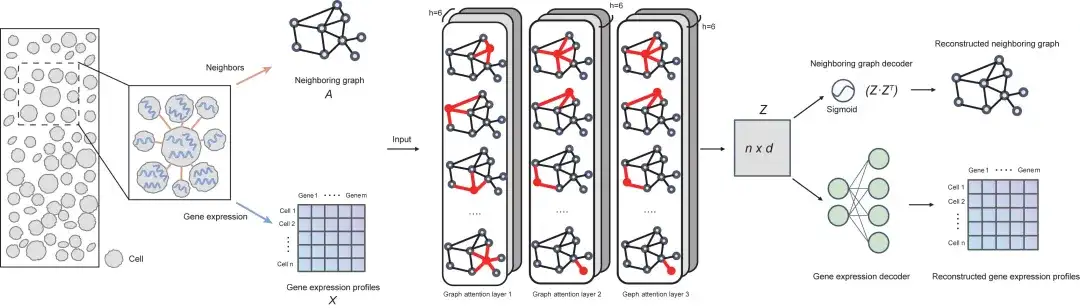
まず、SPACE は、空間的近接性に基づいて各セルをその k 個の最近接セルと接続することによって隣接グラフを構築します。次に、SPACE は 3 層のグラフ アテンション ネットワーク (GAT) をエンコーダとして使用して、隣接グラフに入力された遺伝子発現プロファイルを変換します。低次元のセル表現として、これらの表現は 2 つの独立したデコーダ ネットワークを通じて使用され、各セルの遺伝子発現プロファイルと隣接グラフが再構築されます。
GAE モデルをトレーニングするために、SPACE は自己教師あり学習を採用し、遺伝子発現プロファイルと隣接グラフの全体的な再構成損失を最小限に抑えることを目指しています。学習された細胞表現は、さまざまなクラスタリング アルゴリズムを使用した細胞タイプの識別や組織モジュールの発見に使用できます。
以前に開発された深層学習ツールは、グラフ畳み込みネットワーク (GCN) (SpaGCN、SpaceFlow、GraphST、SEDR など) またはグラフ アテンション オートエンコーダー (STAGATE など) を使用して、遺伝子発現の分析によって集約される「近傍認識」埋め込みを生成していました。組織モジュールを発見するための細胞とその隣接細胞のプロファイル。 SPACE は、次の 3 つの主な点でこれらのツールと異なります。
まず、SPACE では、同じ低次元細胞表現から (2 つの独立したデコーダーを介して) 遺伝子発現プロファイルと隣接グラフを再構築する必要があります。この設計により、SPACE は、分析された細胞とそれぞれの隣接細胞の遺伝子発現プロファイルと空間的相互作用を記憶することができます。対照的に、他の方法は、隣接グラフを入力として受け取りますが、グラフを再構築しません。この違いを強調するために、この研究では、SPACE によって生成されるセル埋め込みを「セル間相互作用を意識したセル埋め込み」と呼んでいます。
第二に、SPACE は、遺伝子発現プロファイルと隣接グラフ再構成損失の相対的な重みを決定するために使用される知覚ドメイン スケールを定義します。この調整可能な比率により、SPACE は特定の研究ニーズに合わせて学習を調整し、分析された各細胞の遺伝子発現プロファイルや空間的に隣接する細胞の相互作用を強調することができます。
第三に、SPACE は、GAT エンコーダのアテンション メカニズムも使用して、近傍情報の集約プロセス中に各近傍の重みを適応的に学習します。この方法では、遺伝子発現プロファイルの再構築プロセスにおいて、異なる近傍のそれぞれの寄与が自動的に考慮されます。
研究結果: SPACE は、細胞型の識別と組織モジュールの発見において他の同様のツールよりも優れています。
SPACE は、複数の空間トランスクリプトーム データセットを使用してテストされ、SPACE によって発見された細胞コミュニティは、空間分布特性において手動で注釈が付けられた組織構造と類似していることが実証されました。
空間的に有益な細胞タイプを識別する SPACE の能力の評価
研究では最初に、細胞型を識別するSPACEの能力を調査するために、MERFISHで記述されたマウス一次運動野(PMC)のSTデータセット(スライス153から開始)を使用した。結果は次のようになります。SPACEによって同定された細胞型は、元の研究で報告された細胞型とよく一致していた。さらに、下の図に示すように、SPACE は、アストロ サイトや希突起膠細胞などの特定の細胞タイプに対して高解像度の細胞タイプ アノテーションを提供します。
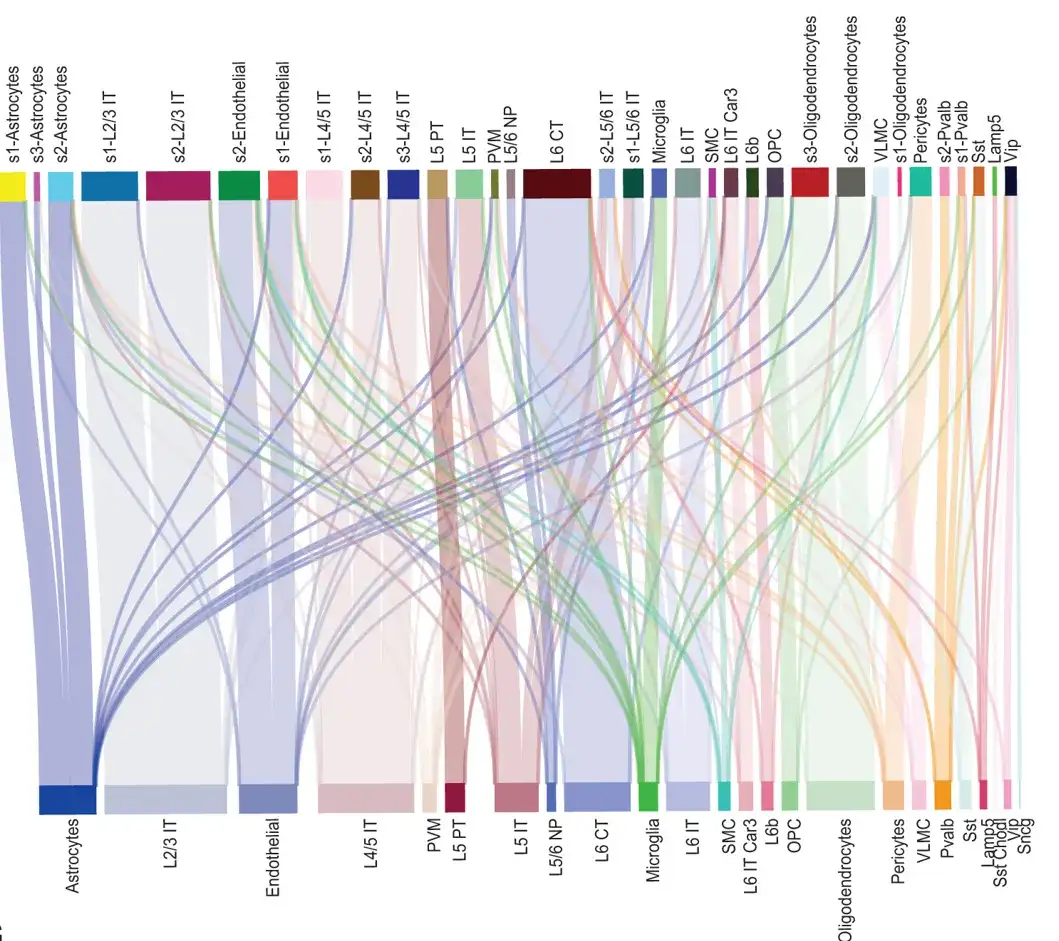
サンキー図は、MERFISH マウス PMC データセットのスライス 153 内のすべての細胞の空間情報における、関連する細胞タイプと元の細胞タイプの間の対応を示します。
次に研究者らは、同定されたアストロサイト(皮質のグリア細胞)と希突起膠細胞(中枢神経系のミエリン細胞)のサブタイプに焦点を当てた。アストロサイトはかつて均一な細胞タイプであると考えられていましたが、最近の ST 研究では、アストロサイトが異なる脳領域で異なる機能を持っていることが報告されています。
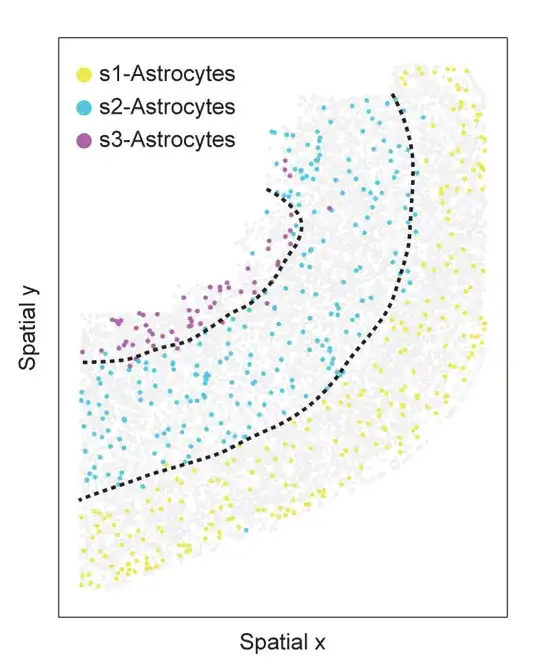
MERFISH マウス PMC データセットのスライス 153 の星状細胞サブタイプに関連する空間情報。細胞は星状細胞のサブタイプごとに色付けされており、明るい灰色の点は他の細胞を示します。破線は上部、深部、白質を示します
実験では、SPACE は PMC スライス 153 で 3 つの異なるサブタイプを発見しました。上の図に示すように、各サブタイプは異なる皮質層に空間的に分布しています。アストロサイトと同様に、SPACE は希突起膠細胞も、異なる空間分布パターンを持つ 3 つの空間情報を提供するサブタイプに分類します。

研究者らはまた、別の ST テクノロジーである STARmap によって生成されたマウス胎盤 (PLA) データセットに SPACE を適用しました。結果は、上の図に示すように、SPACE が細胞を 16 種類の細胞に注釈付けし、元の研究の細胞種類とよく一致していることを示しました。 SPACE は、グリコーゲン栄養膜細胞の 2 つのサブタイプを同定しました。最初の研究ではどちらも「巨大栄養膜細胞 2」細胞と呼ばれていました。 2 つのサブタイプは胎盤の異なる領域に位置し、相互作用する独自の隣接細胞タイプを持っています。
要約すると、異なる ST 手法と組織に基づく 2 つの独立したデータセットの分析により、次の結論が裏付けられます。SPACE は、ST データセット内の空間情報に基づいて、生物学的に異なる空間情報を持つ細胞の種類を識別できます。
細胞型識別における SPACE のパフォーマンスの評価
研究者らは、SPACEを、遺伝子発現に加えて空間情報を考慮に入れる、空間トランスクリプトームデータから細胞型を特定するために現在使用されている2つのツールであるBANKSYおよびFICTと比較した。研究者らは分析に、遺伝子発現のみを考慮しているものの、細胞型の識別に広く使用されているツールであるSCANPYも含めた。
比較のために、研究者らは前述の MERFISH マウス PMC データセットと STARmap マウス PLA データセットを使用しました。以下の図に示すように、SPACE は空間的に情報を提供するさまざまなアストロ サイトと希突起膠細胞のサブタイプを識別できますが、SCANPY も FICT も、皮質層で分解された空間分布パターンでアストロ サイトと希突起膠細胞を定義することはできません。
STARmap マウス PLA データセットの場合、SPACE と BANKSY は 2 つの糖栄養芽層サブタイプの同定に成功しましたが、SCANPY と FICT は糖栄養芽層のサブタイプを同定できませんでした。これは、2 つの糖栄養芽層サブタイプが原因である可能性があります。細胞サブタイプ間では周囲の細胞タイプに明らかな違いがあります。
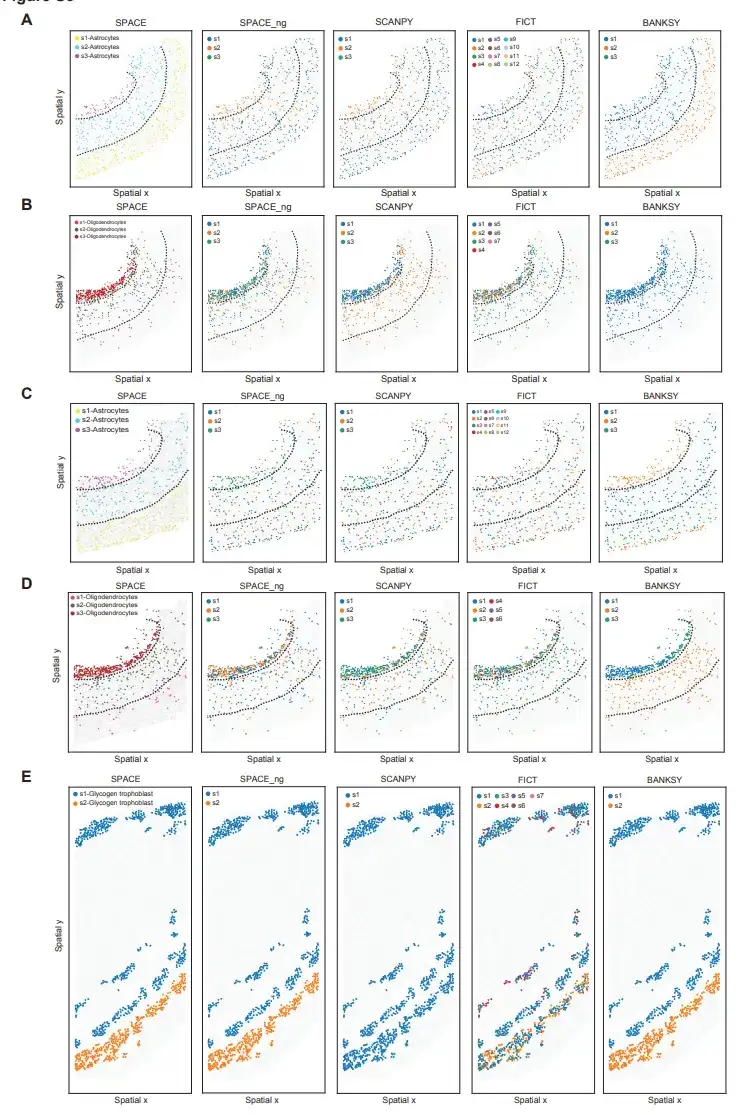
これらの結果を総合すると、次のことがわかります。SPACE は、ST データから空間的に有益な細胞タイプを区別するために現在利用可能なツールよりも優れた性能を発揮します。
SPACE は、組織モジュールの検出において最先端のツールを上回るパフォーマンスを発揮します
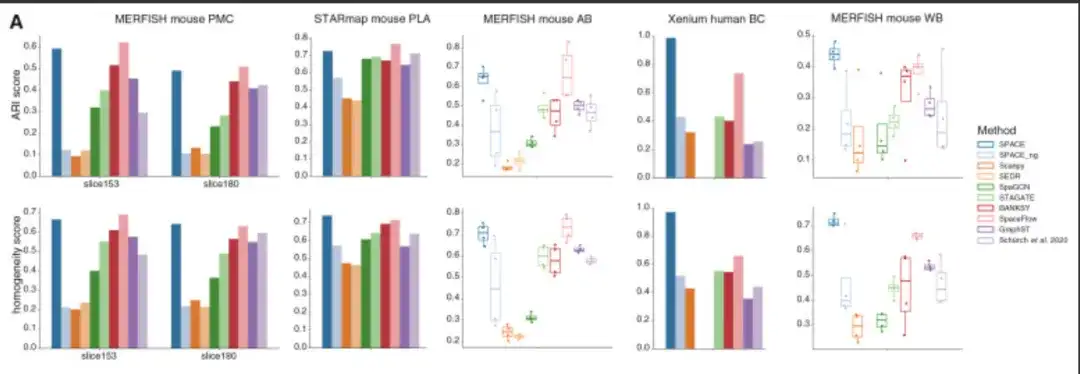
空間トランスクリプトーム研究における重要なタスクは、特定の組織内の組織モジュールを発見することです。この点における SPACE の機能を評価するために、研究者らは SPACE を SEDR、SpaGCN、STAGATE、BANKSY、SpaceFlow、GraphST、Schürch らの手法、および SCANPY および SPACE_ng と比較し、前述の 2 つの ST データセット (MERFISH マウス PMC) を使用しました。データ セットおよび STARmap マウス PLA データ セット)、および MERFISH を含む注釈付き組織モジュールを含む 3 つの追加データ セットマウス老化脳 (AB) データセット、MERFISH マウス全脳 (WB) データセット、および Xenium ヒト乳がん (BC) データセットは、異なる組織および異なる条件下で取得された ST データを表します。
全体、SPACE は、5 つのデータセットのうち 2 つで競合ツールのパフォーマンスを大幅に上回り、他の 3 つのデータセットでは (それぞれの最高のツールと比較して) 最高のパフォーマンスを発揮したツールとほぼ同等のパフォーマンスを示しました。以下に示すように:
空間トランスクリプトームデータ解析の課題を克服する
空間トランスクリプトーム技術は、近年のバイオインフォマティクス分野における画期的な進歩の 1 つであり、2020 年にネイチャー手法によるテクノロジー オブ ザ イヤーに選ばれました。本技術は、多数の細胞の空間的位置と細胞内のトランスクリプトーム数を同時に測定することで、個々の細胞間の位置関係を測定することが難しい単一細胞シークエンス技術の欠点を補い、細胞を理解するための新たなデータを提供します。複数の細胞間の相互作用 基礎 - 空間トランスクリプトームデータの基本的な分析方法の開発は、バイオインフォマティクスの分野における現在の最前線の問題の 1 つです。
細胞の空間位置情報とその分子シグネチャの結合により、新しいマルチモーダルな高スループット データ リソースが生成され、効率的なデータ分析と情報マイニング方法の開発に多くの課題が生じていますが、人工知能はこれらの課題に対する解決策を提供します。 。

2022年7月、上海交通大学電子情報電気工学院オートメーション学科のシェン・ホンビン教授とユアン・イェ准教授の研究チームは、「グラフニューラルネットワークによる空間トランスクリプトミクスデータの細胞クラスタリング」というタイトルの論文を発表した。 Nature サブジャーナル Nature Computational Science に掲載。グラフ ニューラル ネットワークを使用した空間トランスクリプトーム データの細胞クラスタリングに関する研究論文。
論文リンク:https://www.nature.com/articles/s43588-022-00266-5
この論文では、グラフ畳み込みニューラル ネットワークに基づいた空間トランスクリプトーム細胞クラスタリング手法 (Cell Clustering for Spatial Transcriptomics、CCST) を提案しています。これは、空間トランスクリプトーム データを処理するための新しいソリューションを提供し、遺伝子発現の空間分布のモデル化、細胞動態の分析、主要な細胞サブタイプの発見など、生命科学および医学におけるマルチレベルの基本問題の研究に適用できる可能性があります。相互作用とその分子機構など
2023年4月ジョンズ・ホプキンス大学の研究チームはスペースマーカーを開発しました。これは、ST データの潜在空間解析を使用して細胞間相互作用における分子変化を推測できるバイオインフォマティクス アルゴリズムです。研究者らはこのアプローチを使用して、転移、浸潤性病変および前駆病変、および免疫療法からの Visium 空間トランスクリプトーム データにおける腫瘍と免疫の相互作用における分子変化を推測しました。
この研究は、「潜在空間を介した腫瘍微小環境内の分子相互作用の空間的景観の解明」というタイトルで Cell Systems に掲載されました。
今年4月、「BANKSYはスケーラブルな空間オミックスデータ解析のための細胞タイピングと組織ドメインセグメンテーションを統合する」と題された研究レポートが国際誌Nature Geneticsに掲載された。シンガポールの A*STAR 研究所およびその他の機関の科学者は、研究を通じて BBANKSY (Building Aggregates with a Neighborhood Kernel and Spatial Yardstick) と呼ばれるアルゴリズムを報告しました。革新的な空間オミクス データ解析ツールとして、このアルゴリズムの主な機能は、タイプと組織ドメインに従って空間オミクス データ内の細胞を効果的に分類することです。
論文リンク:https://www.nature.com/articles/s41588-024-01664-3
明らかに、将来的には、人工知能技術のサポートにより、空間トランスクリプトーム技術により、組織内のさまざまな細胞型の空間分布、さまざまな細胞群間の相互作用がより明らかになり、重要なさまざまな組織領域の遺伝子発現マップが描画されるようになるでしょう。病気やがんのメカニズムには、広範囲にわたる応用価値があります。
参考文献:
1.https://www.cell.com/cell-systems/fulltext/S2405-4712(24)00124-8#secsectitle0030
2.https://www.tsinghua.edu.cn/info/1175/112190.htm
3.https://news.bioon.com/article/367a820e60b9.html
4.https://www.sohu.com/a/677912398_12








