Command Palette
Search for a command to run...
CVPR 最優秀学生論文! 1,000 万枚の画像と 450,000 以上の種にわたる非常に大規模なデータセットを備えたマルチモーダル モデル BioCLIP は、ゼロサンプル学習を実現します。

従来の学術分野におけるジャーナル出版の重視とは異なり、コンピュータの世界、特に機械学習、コンピュータビジョン、人工知能などの分野では、トップカンファレンスが王様であり、無数の「注目の研究方向性」や「革新的な手法」が発表されます。ここからの流れ。
コンピューター ビジョン、さらには人工知能の分野において学術的に最も影響力のある 3 つの会議の 1 つとして、今年のコンピューター ビジョンとパターン認識に関する国際会議 (CVPR) は、会議の規模と受理された論文の数の点で新記録を樹立しました。

CVPR からの最新の公式発表によると、CVPR 2024 は、カンファレンス史上最大かつ最も多くの参加者が集まるイベントとなりました。6月19日現在、会場参加者数は120,000人を超えています。

さらに、CVPR は、コンピュータ ビジョンの主要なイベントとして、毎年、現在のビジョン分野における最新の研究を受け入れています。今年は 11,532 件の有効な論文が提出され、CVPR 2023 と比較して 2,719 件の論文が採択されました。採択された論文数は 20.6% 増加しましたが、採択率は 2.2% 減少しました。これらのデータは、CVPR 2024 の人気、競争の激しさ、受賞論文のゴールドコンテンツが増加していることを示しています。

北京時間6月20日早朝、CVPR 2024はこのカンファレンスの最優秀論文およびその他の賞を正式に発表した。統計によると、今回は合計10論文が受賞した。その中には、最優秀論文が 2 件、学生優秀論文が 2 件、最優秀論文ノミネートが 2 件、学生優秀論文ノミネートが 4 件あります。

で、「BIoCLIP: A Vision Foundation Model for the Tree of Life」が最優秀学生論文に選ばれました。この点に関して、MITコンピュータサイエンス・人工知能研究所のサラ・ビアリー助教授は、論文の筆頭著者であるサミュエル・スティーブンス氏とそのチームを「当然の」受賞者であると評価し、壇上ですぐに感謝の意を表明した。 。
HyperAIは、「BIoCLIP: A Vision Foundation Model for the Tree of Life」をデータセット、モデルアーキテクチャ、モデルパフォーマンスなどの側面から包括的に解釈し、サム・スティーブンスのその他の業績も要約して編集します。
論文ダウンロードアドレス:
https://arxiv.org/pdf/2311.18803
最大かつ最も多様な生物学的画像データセットを作成する
現在、最大の機械学習生物画像データセットは iNat21 で、これには 10,000 種をカバーする 270 万枚の画像が含まれています。 iNat21 の分類範囲は ImageNet-1k などの一般的なドメイン データ セットと比較して大幅に改善されていますが、生物学に関しては 10,000 種というのはまだ稀です。国際自然保護連合(IUCN)は、2022年に既知の種が200万種を超え、鳥類と爬虫類だけでもそれぞれ1万種以上を報告した。
生物学的画像データセットにおける種カテゴリ制限の問題に対応して、研究者らは 1,000 万枚の画像のデータセット、TreeOfLife-10M を構築しました。45 万種以上の種にまたがり、データセットのサイズと種の多様性において革命的な進歩を遂げました。
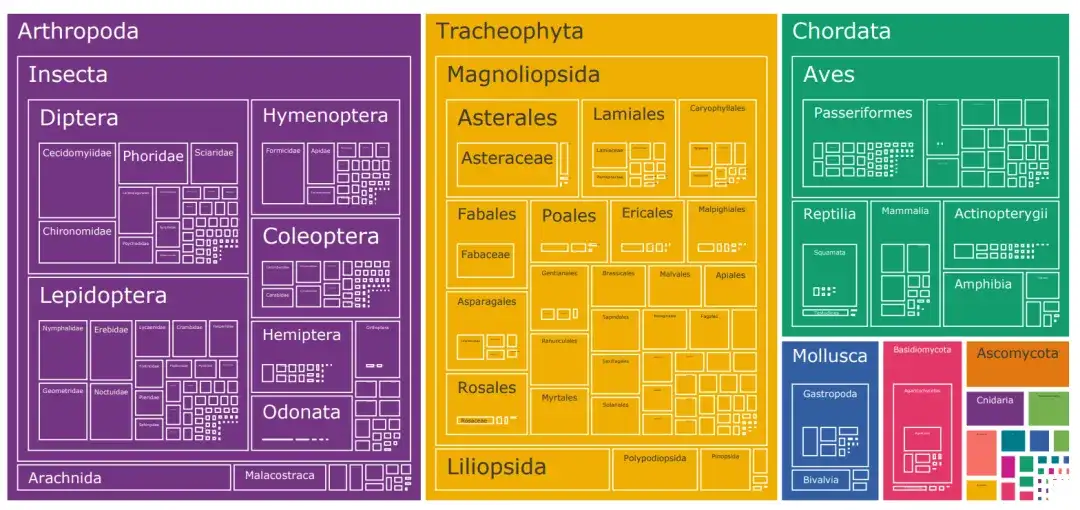
このデータセットは、iNaturalist、BIOSCAN-1M、Encyclopedia of Life (EOL) からの生物学的画像を組み合わせたものです。
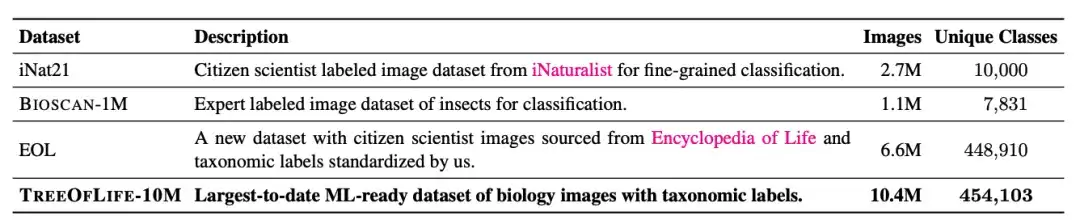
iNat21 でカバーされる 10,000 種のカテゴリーに加えて、研究者らは EOL から 660 万枚の画像をダウンロードし、TreeOfLife-10M を拡張してさらに 440,000 分類群をカバーできるようにしました。同時に、基本モデルが昆虫の非常にきめ細かい視覚表現を学習できるようにするために、研究者らは、494 の異なる科と 7,831 種の分類をカバーする 100 万枚の実験室昆虫画像を含む最新のデータセットである BIOSCAN-1M も組み込みました。
TreeOfLife-10Mのダウンロードアドレス:
https://go.hyper.ai/Gliol
マルチモーダル モデル BioCLIP: CLIP に基づいた汎化機能の向上
一般的なタスクと比較して、生物学的コンピューター ビジョンのラベル空間は、分類ラベルの数が膨大であるだけでなく、階層的な分類システムで相互に接続されています。これは間違いなく、高い種の範囲をトレーニングするのに良い方法です。基本モデルには大きな課題があります。
研究者らは、数百年にわたる生物学の研究で蓄積された経験をもとに、基本モデルが注釈空間の構造をうまくエンコードできれば、たとえ特定の生物種が見えていなくても、モデルが対応する属を識別できる可能性があると考えている。この階層表現は、新しい分類群の少数ショットまたはゼロショット学習を達成するのに役立ちます。
これに基づいて、研究者らは OpenAI が開発したマルチモーダル モデル アーキテクチャ CLIP を選択しました。そして、CLIP のマルチモーダル対比学習ターゲットを使用して、TREEOFLIFE-10M での事前トレーニングを継続します。
具体的には、CLIP は 2 つの単一モーダル埋め込みモデル (ビジュアル エンコーダーとテキスト エンコーダー) をトレーニングして、ポジティブ ペア間の特徴類似性を最大化し、ネガティブ ペア間の特徴類似性を最小化します。ここで、ポジティブ ペアはトレーニング データからのものであり、負のペアは、バッチ内の他のすべての可能なペアです。

さらに、CLIP の重要な利点は、そのテキスト エンコーダが自由形式のテキストを受け入れ、生物学の分野における多様なクラス名形式の問題に正確に対処できることです。この研究のテキスト形式に関して、研究者は主に次のことを考慮しました。
* 分類名:標準的な 7 レベルの生物学的分類は、高位から低位まで、界、門、綱、目、科、属、種です。種ごとに、根から葉までのすべてのラベルを 1 つの文字列 (分類名) に連結することによって、分類が「平坦化」されます。
※学名:属と種から構成されます。
※通称(通称。):分類名はラテン語であることが多く、これは一般的な画像テキストの事前トレーニング データセットでは珍しいことです。代わりに、「クロハシカササギ」などの一般的な名前の方が一般的です。一般名と分類群の間のマッピング関係は 1 対 1 ではない場合があり、種が複数の一般名を持つ場合や、同じ一般名が複数の種を参照する場合があることに注意してください。
実際のアプリケーションでは、推論時の柔軟性を向上させるために、アノテーション入力は 1 種類のみになる場合があります。研究者らは、混合テキストタイプのトレーニング戦略を提案しました。つまり、各トレーニング ステップで、各入力画像が、利用可能なすべてのテキスト タイプからランダムに抽出されたテキストとペアになります。実験によれば、このトレーニング戦略は分類名の一般化の利点を維持するだけでなく、推論時の柔軟性も向上します。
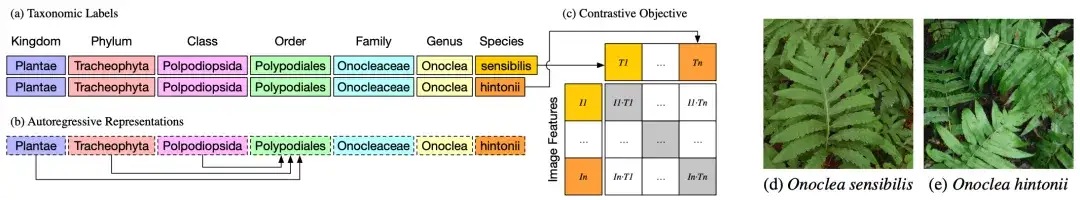
上の図 a に示すように、2 つの異なる植物、Onoclea sensibilis (d) と Onocleahintonii (e) の分類または分類ラベルは、種を除いて同一です。
上の図 b に示すように、テキスト エンコーダーは、分類学の階層表現を自然にエンコードできる自己回帰言語モデルです。このモデルでは、順序表現 (順序表現) の Polypodiales は、王国、門、階級の情報を吸収することにより、より高次の順序にのみ依存できます。トークン。これらの分類ラベルの階層表現は、標準的な対比事前トレーニング ターゲットに入力され、画像表現 (d) および (e) と照合されます。
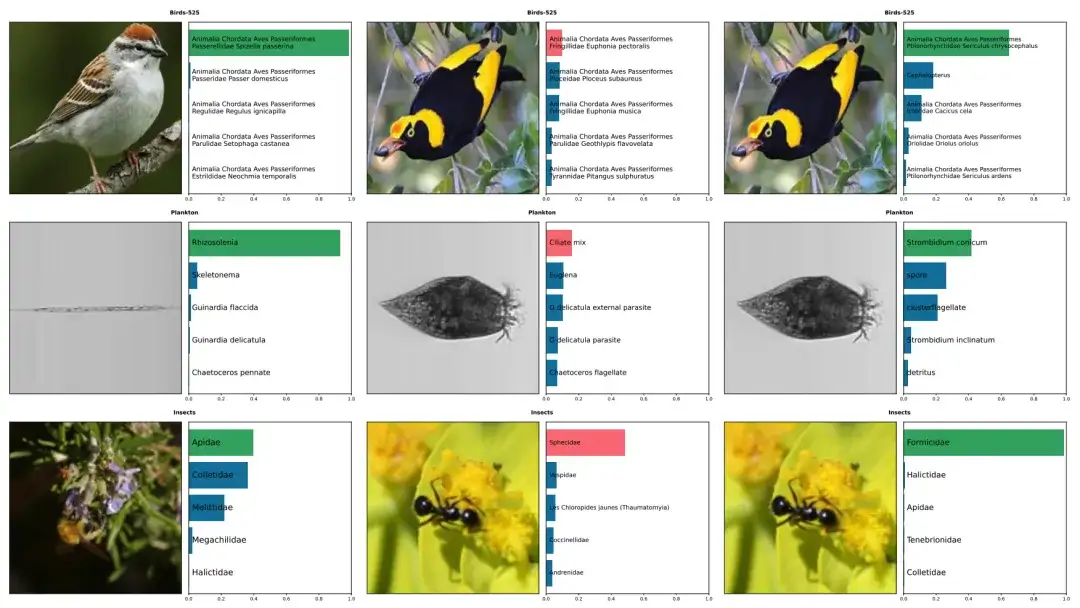
上の図は、鳥、プランクトン、昆虫など 5 種の BioCLIP と CLIP による予測の例を示しています。正しいものは緑色でマークされ、不正確なものは赤色でマークされています。左の列には、BioCLIP からの正しい予測がリストされています。中央と右は、CLIP では誤ってアノテーションが付けられたが、BioCLIP では正しくアノテーションが付けられた画像です。
BioCLIP はゼロショットタスクや数ショットタスクで優れたパフォーマンスを発揮します
研究者らは、BioCLIP を一般的な視覚モデルと比較しました。結果は次のとおりですBioCLIP は、ゼロショットタスクと数ショットタスクの両方で優れたパフォーマンスを発揮し、CLIP や オープンクリップ、ゼロショットタスクと少数ショットタスクの平均絶対改善(平均絶対改善)は、それぞれ17%と16%です。さらに、固有の分析により、BioCLIP が生命の木と一致するよりきめの細かい階層表現を学習することが示され、その優れた汎化能力が説明されます。
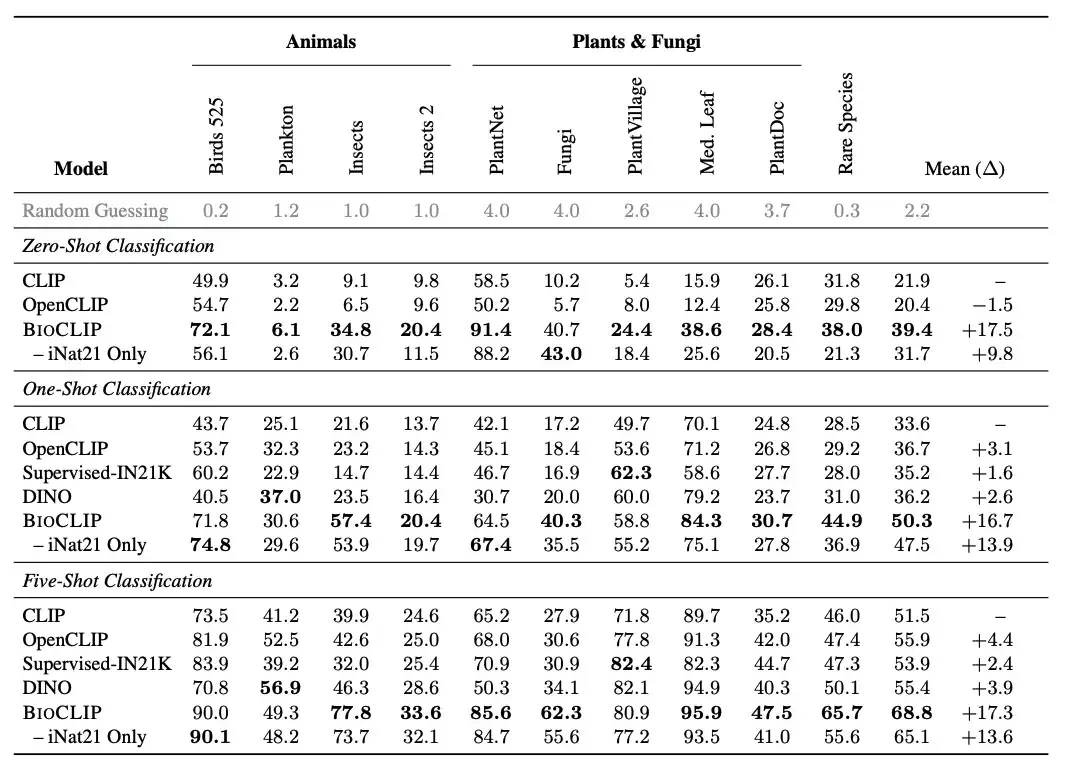
具体的には、研究者らは、IUCNレッドリストで準絶滅危惧種、絶滅危惧種、絶滅危惧種、絶滅危惧種に分類されている約2万5千種を収集する新しい評価タスク「希少種」を導入した。研究者らは、EOL データセット内の少なくとも 30 枚の画像を持つそのような種 400 種を選択し、それらを TreeOfLife-10M から削除しました。新しい希少種のテストセットを作成し、種ごとに 30 枚の画像があります。
比較結果は、上の図に示されています。BioCLIP は、特に目に見えない分類において、ベースライン CLIP モデルと iNat21 でトレーニングされた CLIP モデルを大幅に上回っています (希少種の列を参照)。
実りある結果、最高の BioCLIP の背後にある科学的研究を探る
「BioCLlP: A Vision Foundation Model for the Tree of Life」は、オハイオ州立大学、Microsoft Research、カリフォルニア大学アーバイン校、レンセラー工科大学によって共同リリースされました。この論文の筆頭著者であるサミュエル・スティーブンス博士と責任著者であるジャマン・ウー氏は、どちらもオハイオ州立大学の出身です。
サミュエル・スティーブンスは自身のウェブサイトで自分自身を「自分自身をあまり真剣に考えていない人」と控えめに述べていますが、彼の科学研究の成果と近年のたゆまぬ努力から判断すると、彼は明らかに科学研究に真剣に取り組んでいる人です。
Samuel Stevens 氏は 2017 年からコンピューターの方向に取り組んでいるとされています。マルチモーダル モデル BioCLlP は、彼が 2023 年 12 月に発表した研究成果で、2024 年 2 月に CVPR 2024 に承認されました。
実際、BioCLIP などのコンピューター ビジョンに関する研究は、彼の研究方向の 1 つにすぎません。彼は幅広い興味を持っており、暗号通貨やさまざまな LLM プロジェクトの AI の分野で一連の研究を行ってきました。
たとえば、「MMMU: A Massive Multi-discipline Multimodal Understanding and Reasoning Benchmark for Expert AGI」に参加しました。新しいベンチマーク MMMU (Massive Multi-task Language Understanding) を提案しました。業界で最も影響力のある大規模モデル評価ベンチマークの 1 つである MMMU は、高度な認識と推論のために特定の分野 (科学、健康と医学、人文科学など) の知識を活用することに焦点を当てており、マルチモーダル モデルが実行できる必要があります。専門家が直面する同じタスク。
研究者らは、これを使用して 14 個のオープンソース LMM と独自の GPT-4V (ision) を評価したところ、先進的な GPT-4V でさえ 56% の精度しか達成できなかったことがわかり、このモデルにはまだ改善の余地がたくさんあることがわかりました。この点に関して、研究者らは、このベンチマークがコミュニティに、専門家レベルの汎用人工知能を実現するための次世代のマルチモーダル基本モデルの構築を促すことになるとの期待を表明した。
MMMU: https://mmmu-benchmark.github.io
もちろん、科学研究に対する彼の情熱とオープンさも彼の成功の重要な要素でした。昨日、BioCLlP が最優秀学生論文に選ばれたというニュースが出たばかりで、サミュエル・スティーブンス博士はすぐにソーシャル プラットフォームを通じて外の世界に自分の意見を表明しました。「動物のコンピューター ビジョン、マルチモーダル基本モデル、または AI について話したいのであれば。科学については、個人的にメッセージを送ってください。」

サミュエル・スティーブンス博士は科学研究の道を前進するだけでなく、遅れている人々への支援も決して忘れないことは注目に値します。彼の個人的なウェブページでは、初心者向けのアドバイスも紹介しています。「機械学習と人工知能を始めたいなら、Coursera の機械学習コースと Andrej Karpathy の Neural Networks: Zero to Hero から始めてみてはいかがでしょうか。この 2 つのコースはいずれも品質が非常に高く、提供されるはずです」他の無料リソースと比べて非常に価値があります。」
参考文献:
1. https://samuelstevens.me/#news
最後にオススメのアクティビティをご紹介します!
QRコードを読み取ってテクノロジーサロン「Meet AI Compiler」第5回オフライン集会に参加申し込み↓









