Command Palette
Search for a command to run...
AlphaFoldの韓国版?深層学習モデル AlphaPPIMd: タンパク質間複合体の構造アンサンブル探索用

生命の舞台において、タンパク質は欠かせない役割を果たしています。これらは生物内で最も活性な分子であり、細胞の構築、修復、エネルギー変換、シグナル伝達、および数え切れないほどの重要な生物学的機能に関与しています。同時に、タンパク質の構造はその機能と密接に関係しており、その機能はタンパク質、ペプチド、ヌクレオチド、およびさまざまな低分子との複雑な相互作用によって達成されます。このタンパク質間相互作用 (PPI) は、細胞シグナル伝達から免疫応答、細胞周期制御に至るまで、細胞内の多くの生物学的プロセスの中核であり、すべて PPI が関与します。
しかし、タンパク質の三次元構造とその相互作用特性に対する人々の現在の理解はまだ不完全です。 X線結晶構造解析やクライオEMなどの伝統的な実験手法、高解像度のタンパク質構造情報を提供できますが、時間とコストがかかります。また、動的なプロセスと低濃度タンパク質の解決には課題があります。これは、タンパク質の機能と相互作用メカニズムに対する人々の深い理解を大幅に制限し、ひいては創薬とタンパク質工学の開発に影響を与えます。
これに応えて、延世大学の王建民博士と彼の共同研究者は、深層学習と生成 AI を組み合わせて、Transformer ベースの生成ニューラル ネットワーク学習を使用して、タンパク質間複合体の構造セットを探索します。タンパク質-タンパク質複合体の立体構造および動的機構に影響を与える重要な残基は、複数の分子動力学 (MD) 軌跡から学習され、タンパク質-タンパク質結合の機構に関する洞察が得られました。

用紙のアドレス:
https://doi.org/10.1101/2024.02.24.581708
AlphaPPIMd モデル: 自己注意メカニズムを核とした分子動力学シミュレーションに基づく
研究チームは、barnase-barstar 複素軌道セットをデータセットとして使用しました。まず、バルナーゼ・バルスター複合体の結晶構造をタンパク質データバンク(PDB)からダウンロードし、リガンドと結晶水を除去することで、初期複合体構造としてA鎖とD鎖を抽出した。次に研究者らは、AmberTools の tleap モジュールを介して欠落した水素原子を追加し、Na+ イオンと Cl- イオンを追加してそれらを中和し、12 Å TIP3P 水分子周期境界ボックス内で溶媒和を実行しました。最後に、システムのトポロジーと座標ファイルは、AmberTools の tleap モジュールと AMBER ff14SB 力場を使用してコンパイルされました。
その後、研究チームは分子動力学シミュレーションシステムを使用して、ランジュバン積分器による典型的な NVT シミュレーションを 500 ステップ実行し、エネルギーを最小限に抑えました。次に、300KでのNPTシミュレーションの10,000ステップを通じて平衡状態にさらに到達し、粒子ネットワークEwaldアルゴリズムを使用して、直接空間相互作用のカットオフ値を1nmに設定し、シミュレーション時間ステップで長距離静電相互作用を計算しました。 SHAKE アルゴリズムも、水素原子に関係するすべての結合の長さを制限するように設定され、6 つの独立した 100ns 分子動力学シミュレーションが実行されました。すべてのシミュレーションは OpenMM 7.7 を使用して実行されました。
分子動力学シミュレーションが完了したら、研究チームは、従来の分子動力学では解析が難しいタンパク質の立体構造状態を捕捉するための深層生成モデルを使用して、Transformer に基づいた AlphaPPIMd モデルを構築しました。 AlphaPPImd フレームワークの中核は、タンパク質間複合体の立体構造に影響を与える主要なアミノ酸残基ペアを MD 軌道から捕捉できるセルフアテンション メカニズムです。
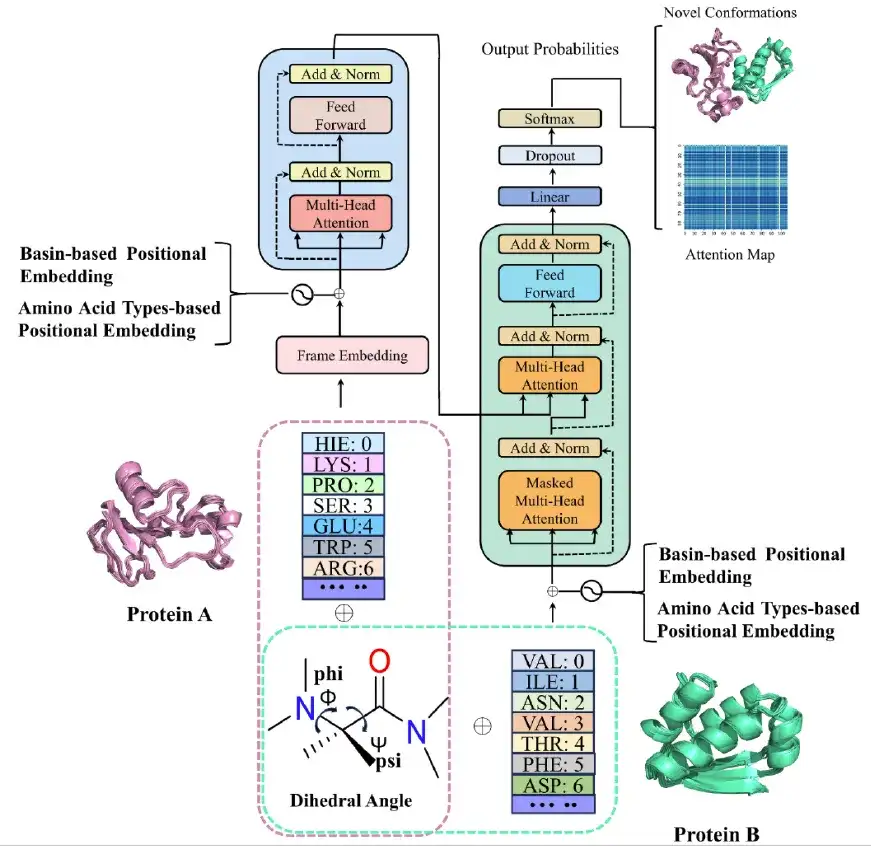
初め、AlphaPPImd フレームワークは、タンパク質間複合体の MD トラジェクトリを前処理して、2 つの鎖の配列長、配列組成、およびアミノ酸残基の種類を取得し、トラジェクトリ内の選択された残基の Φ および Ψ 角度を計算して、異なる立体構造を表します。州。 (上の図のピンクと緑の点線のボックスに示されているように)
第二に、研究者らは、マルチヘッド自己注意メカニズム、注意スコア(注意スコア)、および特徴最適化モジュールを含む埋め込みモジュールを介して、タンパク質-タンパク質複合体MD軌道の各フレームをAlphaPPImdのエンコーダモジュールに入力しました。 AlphaPPImd のデコーダーは、タンパク質複合体中の残基のさまざまなタイプと位置の立体構造への寄与を学習および捕捉するために使用されます。
やっと、予測モジュールは次のフレームの基底状態を繰り返し生成し、Modeller は拡張された基底状態エンコード軌跡に基づいてタンパク質-タンパク質複合体の立体構造モデルを再構築できます。
AlphaPPImd デコーダ モジュールのマルチヘッド セルフ アテンション層は、特定の残基ペア間の相互作用を学習します。アテンション関数は、クエリ (Q) とキーと値のペア (KV) 出力間のマッピングと考えることができます。 AlphaPPImd は、タンパク質複合体残基の埋め込みを Q、グローバルタンパク質複合体特徴を K および V として受け取り、Q および K を使用して注意の重みを計算します。計算式は次のとおりです。
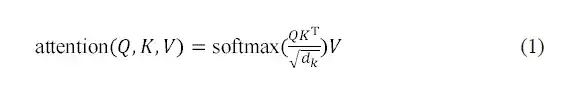
一方、この研究では、バルナーゼ-バースター複合体の6つの独立した100ns MD軌道を、それぞれ1,000フレームで構成される300のプリミティブに分割しました。 MD 軌跡はタンパク質原子のみを保持するように前処理されました。各 MD 実行では、タンパク質間複合体の限られた物理スナップショットのセットが提供されます。軌跡内の各フレームは、Φ、Ψ でエンコードされた基底状態として表されます。したがって、タンパク質-タンパク質複合体のねじれた状態は、テキスト表現に次元的に縮小されます。ダイナミクスの主なマイナーな特徴は維持されます。
研究の結論: 平均トレーニング精度は 0.995 と高く、これはより多くのタンパク質複合体に一般化できます。
バルナーゼ-バースター複合体は、合計 197 残基の 2 つの異なる鎖から構成されます (バルナーゼ鎖: 108 残基、バースター鎖: 89 残基)。この調査では、KMeans アルゴリズムを使用して、ポイントを 0 (下図の紫色)、1 (下図の紺色)、2 (下図の緑色)、3 (下図の緑色) のラベルが付いた 4 つのクラスターに分割します。 ) (黄色)、基底状態によってエンコードされたねじり状態からバルナーゼ-バースター複合体の全原子モデルを再構築するために、各クラスターの質量中心が記録および保存されます。
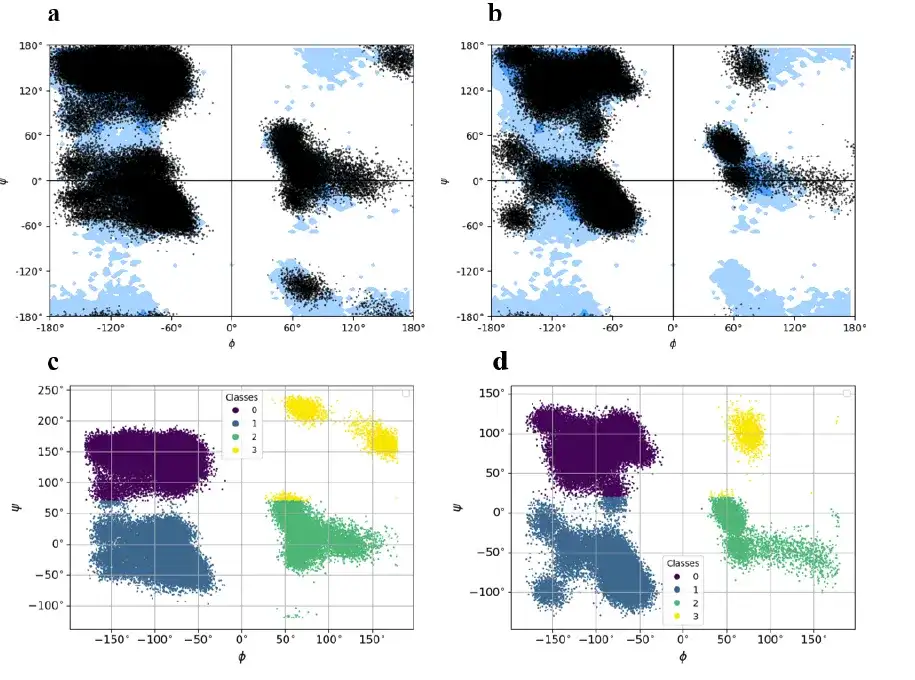
この研究では、各フレームの軌跡を文字ベクトルに変換します。各文字ベクトルは、4 つのクラスターに対応する 4 つのシンボルで構成されます。最後に、この研究では、barnase-barstar 複合体の MD 軌跡データセット内の 300 個のプリミティブすべてに対して同様の表現プロセスを実行しました。
要約すれば、バルナーゼ-バルスター複合体は、2 つの鎖にコードされている残基の基底状態が著しく異なるヘテロ二量体です。これは、バルナーゼ-バースター複合体は、新しい基底状態をコードするフレームワークの生成と個々のタンパク質の立体構造モデルの再構築において大きく異なることを意味します。
研究によると、AlphaPPImd モデルの平均トレーニング精度は 0.995、平均検証精度は 0.999 です。AlphaPPImd はすぐに安定したパフォーマンスを達成しましたが、Transformer モデルをさらに改善し、モデルによって学習された MD 立体構造分布を強化するために、この研究では複数の MD 軌跡をデータセットとして使用しました。たとえば、この研究では、入力としてテスト セットの軌跡からフレームをランダムに選択し、トレーニングされた AlphaPPImd フレームワークを使用して 100 個の基底状態エンコード フレームを生成します。
結果は次のことを示していますモデルは立体構造を正常にサンプリングして展開することができます。そして、Φ と Ψ の二面体制約を正しく実行できます。
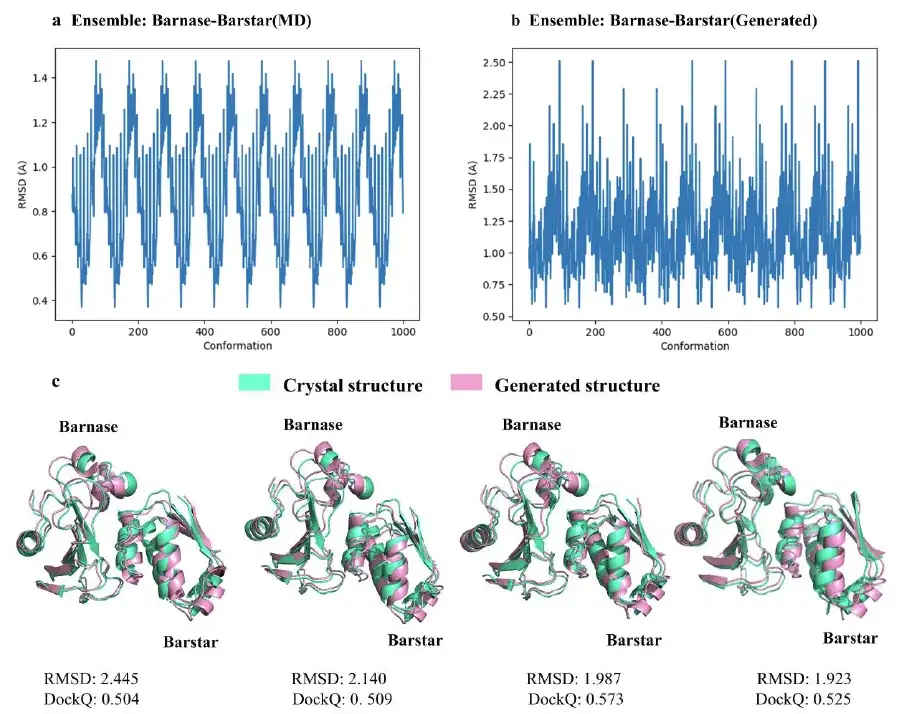
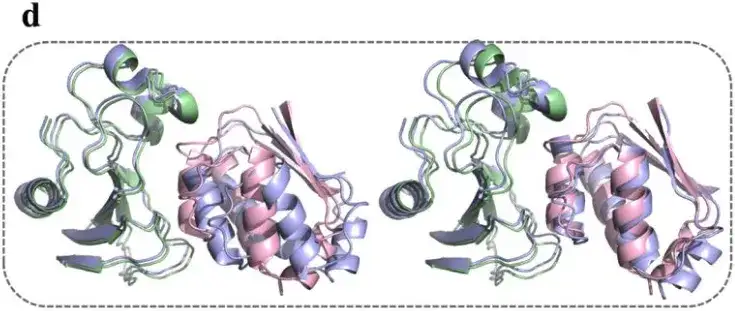
この研究では、AlphaPPImd モデルによって生成された 1,000 個のバルナーゼ-バースター複合構造から、RMSD が 2 Å に近い 4 つの代表的な構造も選択されました。研究結果は次のことを示していますAlphaPPImd によって生成されたタンパク質複合体構造モデルは、参照結晶構造に近く、より高い精度 (RMSD < 2Å) とより優れた許容性 (DockQ ≥ 0.23)。
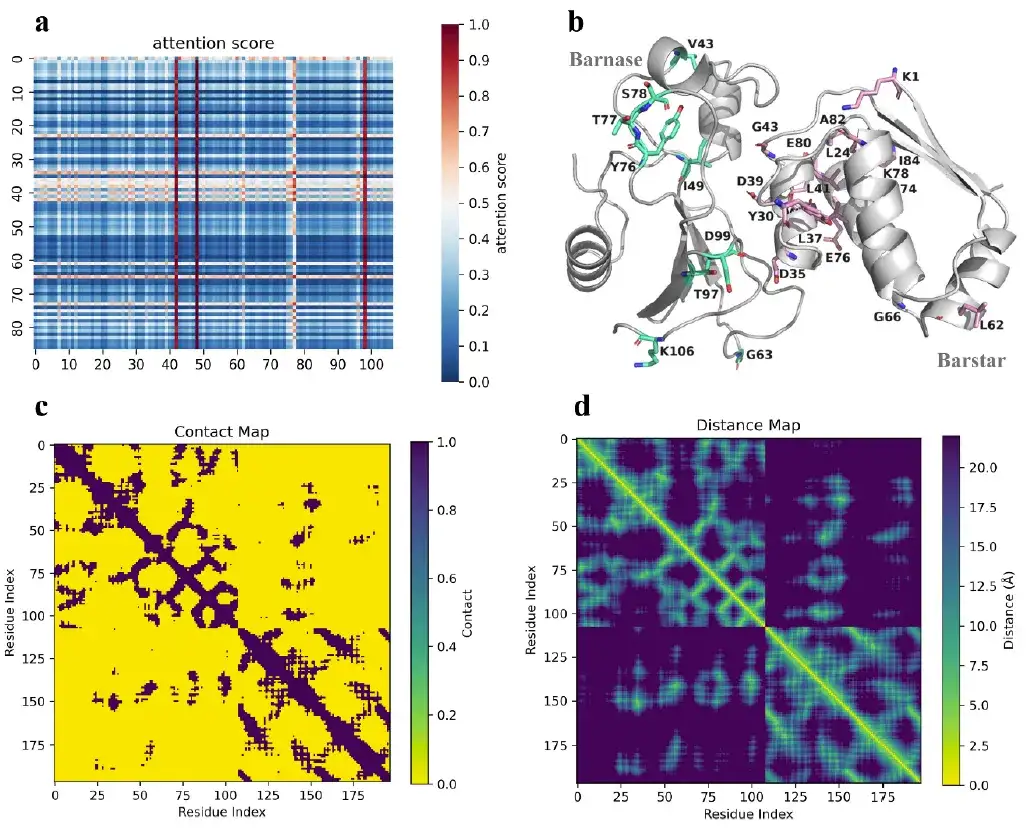
さらに、AlphaPPImd のアテンション メカニズムは、主要な残基間のアテンション ウェイトを捕捉し、タンパク質間の結合に関する機構的な洞察を提供します。
研究により、AlphaPPImd モデルによって捕捉された主要な残基は主にタンパク質相互作用、ループ、ヘリックスの界面に位置していることが示されています。深層生成モデルは、バルナーゼ-バルスター複合体のダイナミクスと立体構造に影響を与える重要な残基をバルナーゼ-バルスター複合体の MD 軌道から捕捉します。MD 結果を補足するために使用できます。同時に、AlphaPPImd モデルによって捕捉された主要な残基は主に Mdm2-p53 相互作用界面に位置しており、これはこのモデルが他のタンパク質間複合体にも一般化できることも証明しています。
AIタンパク質予測:から アルファフォールド 百の学派が争う
AlphaGo が有名になった後の 2016 年には、DeepMind チームはタンパク質のフォールディング問題の研究を開始しました。
2018年末の第13回CASP(タンパク質構造予測の重要評価)では、AlphaFoldは43種類のタンパク質のうち25種類のタンパク質の構造を正確に予測し、参加者98社中第1位にランクされました。 2020年にはAlphaFold 2が発売され、タンパク質モノマー構造の高精度予測を実現した。 2021 年 10 月に、DeepMind は AlphaFold-Multimer と呼ばれるアップデートをリリースしました。これは AlphaFold 2 に基づいて拡張され、複数のタンパク質の複合体をモデル化できます。 2024 年 5 月 8 日、AlphaFold 3 はタンパク質から広範囲の生体分子に至る予測範囲をもたらし、再び世界を驚かせました。
AlphaFold 2 の発売の早い段階で、中国科学院の学者 Shi Yigong 氏はメディアに次のように語った。「私の意見では、これは科学分野に対する人工知能の最大の貢献であり、また、これは21世紀に人類が成し遂げた最も重要な科学的進歩である。第一に、これは自然界を理解するという人類の科学的探求における非常に注目すべき歴史的成果である。」
AlphaFold の宝石が最前線に立つことで、タンパク質設計分野における AI によって引き起こされる産業革命が静かに到来しています。
2023年世界初の大規模AIタンパク質生産モデルNewOrigin(中国名「ダーウィン」)が世界製造会議で正式発表された。レポートによると、NewOrigin の大規模モデルは条件付き生成メカニズムに基づいており、AI、分子動力学、量子コンピューティング、湿式実験などの多次元フィードバック メカニズムを使用して、タンパク質配列、タンパク質機能、タンパク質知識表現、その他のモーダルタンパク質を生成します。実際の産業アプリケーションのニーズを満たすために、親和性、安定性、アクティビティ、表現などの多次元タスクを高精度で実行します。
2022年、ワシントン大学医学部の生物学者らは、主要な発見を紹介する2本の論文をサイエンス誌に発表した。研究者らは次のように述べています。機械学習を使用すると、タンパク質分子を数秒で作成できます。以前は、この期間は数か月続きました。自然界に存在しないタンパク質の作成は、ワクチン開発、がん治療のための研究の加速、炭素回収ツールの開発、持続可能な生体材料の開発などに役立ちます。
AIによるタンパク質の構造予測により、タンパク質の理解と理解が深まり、ひいては生命の理解が可能になることは疑いの余地がありません。しかし、将来的には、科学者はタンパク質をオンデマンドで変更したり、自然界に存在しないタンパク質をゼロから設計したりするなど、医療分野の実際的な問題を解決するために AI を使用してタンパク質を予測する必要があります。 。まだまだ道のりは長いですが、AI が生命科学の探求にさらなる驚きをもたらすことを期待しています。








