Command Palette
Search for a command to run...
復旦脳科学研究所の新たな成果: セマンティック セグメンテーションを利用して、空間トランスクリプトーム意味論的アノテーション ツール Pianno が開発されました

2020年にNature Methods社のTechnology of the Yearに選ばれて以来、「空間トランスクリプトミクス」は、今日のライフサイエンス分野で最も注目されている革新的な技術の一つとなっています。簡単に言うと、この技術は組織の空間情報やトランスクリプトームデータを取得し、組織内の遺伝子発現パターンを時間的・空間的次元から正確に解析したり、細胞群の空間的位置関係などの生物学的特徴を疾患研究や増殖・解析に活用することができます。これは、種や種の進化などの分野の研究において非常に価値があります。
空間トランスクリプトミクスは学術研究の分野で引き続き人気があり、10x Visium、Slide-seq、Stereo-seq などの空間トランスクリプトミクス技術も登場しており、これらの最新の成果と進歩により、組織内の遺伝子に関する人間の理解が完全に変わりつつあります。表現パターン。ただし、組織内の特定の物理座標の遺伝子発現プロファイルを取得するだけでは、生物学的システムの複雑さを完全に理解することはできません。その本質を理解するには、組織内の各空間点の生物学的同一性を特定する必要があります。
現在、空間点のクラスターを特定し、マーカー遺伝子を使用してそれらの生物学的アイデンティティを解釈するために、機械学習ベースの方法が広く使用されています。しかし、これらの方法は、クラスター内の既知の構造への明示的な接続を確立する機能がないため、多くの場合制限されます。さらに、既知の構造の特定を支援するために手動による注釈がよく使用されますが、この方法は研究者の専門知識と主観的な判断によって制限されることが多く、大規模な分析には適用できません。
上記の課題に応えて、復旦大学脳科学研究所のZhu Ying氏のチームは最近、「Pianno: a probabilistic Framework automating semantic annotation for spatial transcriptomics」と題する研究結果を「Nature Communications」誌に発表した。研究チームは、コンピュータビジョンにおける「セマンティックセグメンテーション」の考え方を参考にして、「空間トランスクリプトーム意味論的アノテーション」の概念を提案し、空間トランスクリプトーム意味論的アノテーションツール「Pianno」を開発しました。組織内の空間点の構造または細胞タイプを自動的に定義する機能により、複数の次元からの情報が結合され、複雑な生物学的システムの解釈が強化されます。
研究のハイライト:
* Pianno には、さまざまな空間トランスクリプトミクス技術によって生成されたデータを処理する独自の自動ラベリング モードがあります。
* Pianno は、最先端の空間クラスタリング手法と比較して優れたパフォーマンスを示し、空間トランスクリプトーム データに新たな視点を提供します

用紙のアドレス:
https://doi.org/10.1038/s41467-024-47152-4
データセット: オープンデータ、厳密な計算
この研究で使用されるデータセットは主に、ヒト背外側前頭前野データセット dlPFC、成体マウス大脳半球冠状スライスデータセット Stereo-seq、マウス海馬前頭前野データセットなど、さまざまな空間技術プラットフォームからの公開データセットです。処理データセット Slide-seqV2、ヒト膵管腺がんデータセット ST、ヒト乳がんデータセット Visium、マウス一次視覚野データセット 複数のヒト皮質領域のscRNA-seq、snRNA-seqデータセット、マウス嗅球のDAPI染色画像など
研究では、ノイズリダクション、スムージング、シャープ化などの画像処理技術が生物本来の特性を損なわないようにするため、研究チームは、生のカウントに基づいてベイジアン分類器を構築し、初期のアノテーションを微調整しました。同時に、研究チームは高次マルコフランダム場(MRF)事前モデルを適用した。空間トランスクリプトミクスの文脈では、遺伝子発現と各部位の空間的位置を一緒に考慮する必要があるため、研究チームは空間ポアソン点過程(sPPP)モデルも採用しました。
Pianno: 空間トランスクリプトームの自動セマンティック アノテーションのための革新的な新しいツール
研究チームはベイジアンフレームワークに基づいた新しいツールPiannoを提案しました。このツールは、マルコフランダム場 (MRF) と空間ポアソン点プロセス (sPPP) を組み合わせ、空間点の位置情報を考慮しながら RNA-seq カウントデータ分布をモデル化する sPPP の機能を最大限に活用し、事前定義された A リストを使用できます。空間トランスクリプトーム データの各点の生物学的アイデンティティに自動的に注釈を付けるマーカー遺伝子。
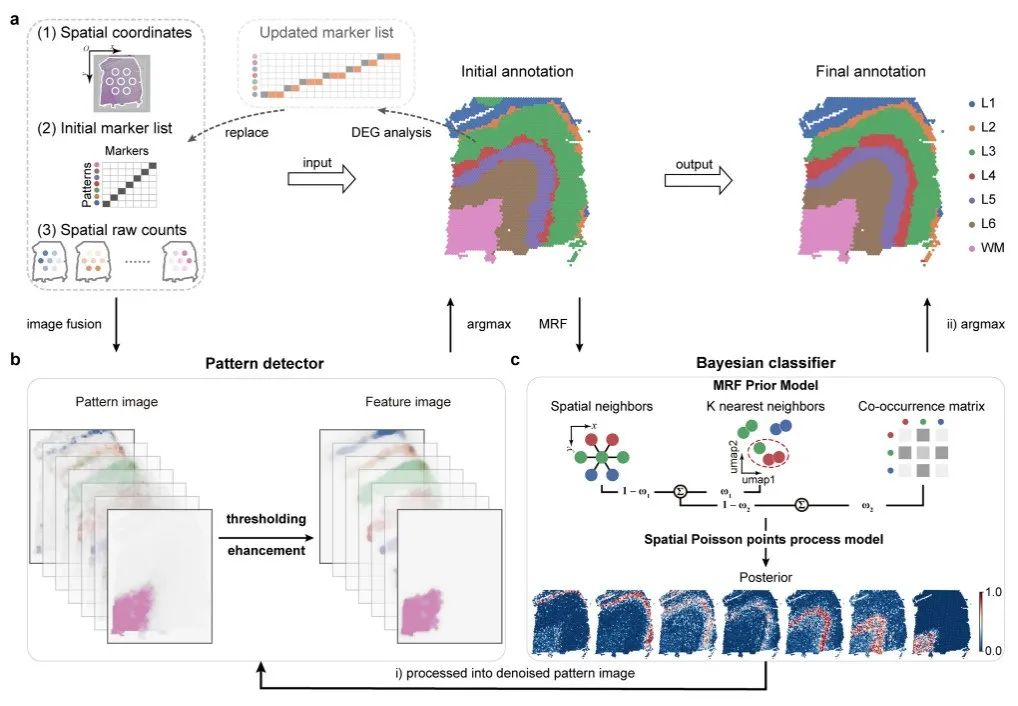
Pianno によって入力された空間トランスクリプトーム データは、空間座標、初期マーカー リスト、および空間 raw カウントで構成されます。各パターンは少なくとも 1 つの既知のトークンを提供します。
アノテーション プロセスは、最初のセグメンテーション ステップとリファインメント ステップで構成されます。
最初のセグメンテーションステップでは、各遺伝子の空間発現をグレースケール画像に変換しました。ターゲット パターンごとに、そのパターンに関連付けられたマーカー遺伝子のグレースケール画像を集約してパターン画像を作成し、各パターンの追加の候補マーカー遺伝子を特定して、初期マーカー リストを更新します。更新されたマーカー リストは、最初にアノテーションが付けられた構造における固有の発現パターンを考慮して、後続の改良ステップに統合されます。
改良ステップでは、ベイジアン分類器を構築して、各空間点が異なるパターンに属する事後確率を評価し、事後確率に基づいてアノテーションを更新します。
Pianno には、注釈を更新するための 2 つの方法が用意されています。
* セマンティック アノテーションの連続パターンの場合は、確率分布をパターン画像として使用し、それをパターン検出器 (パターン検出器) に返してアノテーションを更新することをお勧めします。 * 散乱した画像パターンまたは鮮明な画像パターンの場合は、詳細を保持できるため、確率値に応じてラベルを付けます。
一般的に、Pianno は、最初の単一マーカー遺伝子を使用して追加のマーカー遺伝子を識別するヒューリスティックなアプローチを使用することで、既知のマーカー番号の入力を最小限に抑えながら、アノテーション プロセスを簡素化します。
研究結果:優れた性能と高い応用性
本研究において研究チームは、Piannoの性能、精度、適応性などを検証し、既存の手法と比較することでPiannoの能力をさらに実証しました。
解剖学的構造のアノテーション用のクラスタリングベースのツールとの比較において、研究チームは、dlPFC データセットからの 12 個のサンプルを使用して Pianno のパフォーマンスを評価し、マーカーに基づくが空間情報を持たない別のアノテーション手法である CellAssign と比較しました。さらに、教師なしクラスタリング手法であるライデン アルゴリズムと 5 つの空間クラスタリング手法 (SpaGCN、SEDR、BayesSpace、DeepST、STAGATE) も評価プロセス中に考慮されました。
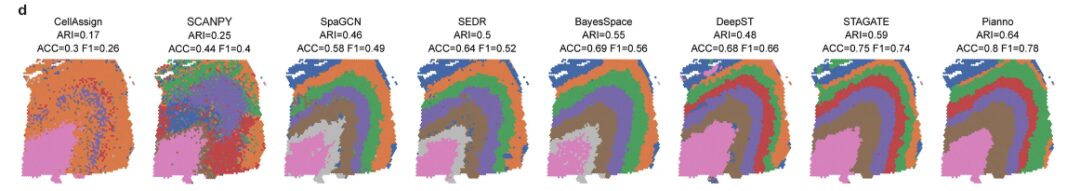
評価の結果、Pianno のパフォーマンスは、経験豊富な研究者による形態学的特徴とマーカーに基づく手動の注釈と最高の一貫性を実現します。12 サンプル中 11 サンプルで、他のテスト方法を上回りました。
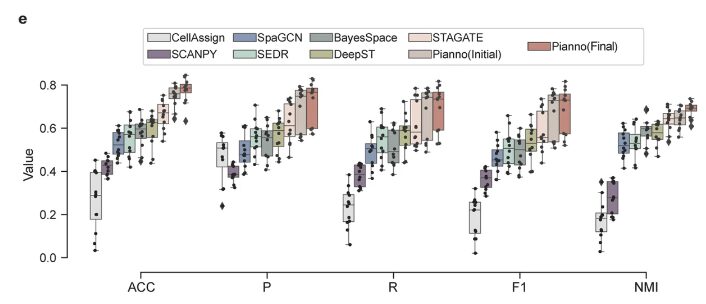
さらに、研究チームは、精度(ACC)、マクロ平均精度(P)、マクロ平均再現率(R)、マクロ平均F1スコア(F1)、正規化相互情報量(NMI)などの他の分類指標も使用しました。上の図 e に示すように、Pianno の優れたパフォーマンスをさらに包括的に評価します。ピアノ関連の指標はいずれも比較的高い水準にある。
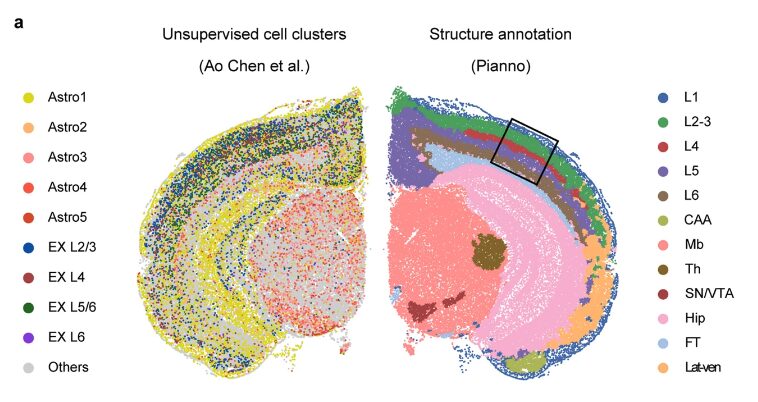
次にチームは、細胞型の空間分布を予測するピアノの能力を評価しました。このラウンドの検証では、研究チームは成体マウスの半脳冠状切片のステレオseqデータセットを使用し、その結果を、細胞セグメンテーション後の教師なしクラスタリングや、空間的および空間的データに基づく3つの空間デコンボリューションツールなど、さまざまな戦略を通じて推定された細胞型分布と比較しました。単一細胞トランスクリプトーム統合。
この研究では、興奮性ニューロンのサブタイプの分布に関するピアノノの予測がタングラムと RCTD に匹敵するパターンを示し、各層の既知の位置でタングラムと RCTD と高度な一貫性を示したことがわかりました。一般的に言えば、この結果は、特に教師なし手法が課題に直面する場合に、空間データセット内の複雑な細胞型分布を予測する際の Pianno の堅牢性と精度を示しています。
研究チームはその後、さまざまなプラットフォームからの空間トランスクリプトームデータのさまざまな形状構造に注釈を付ける際の Pianno のパフォーマンスをさらに評価し、STAGATE と比較しました。
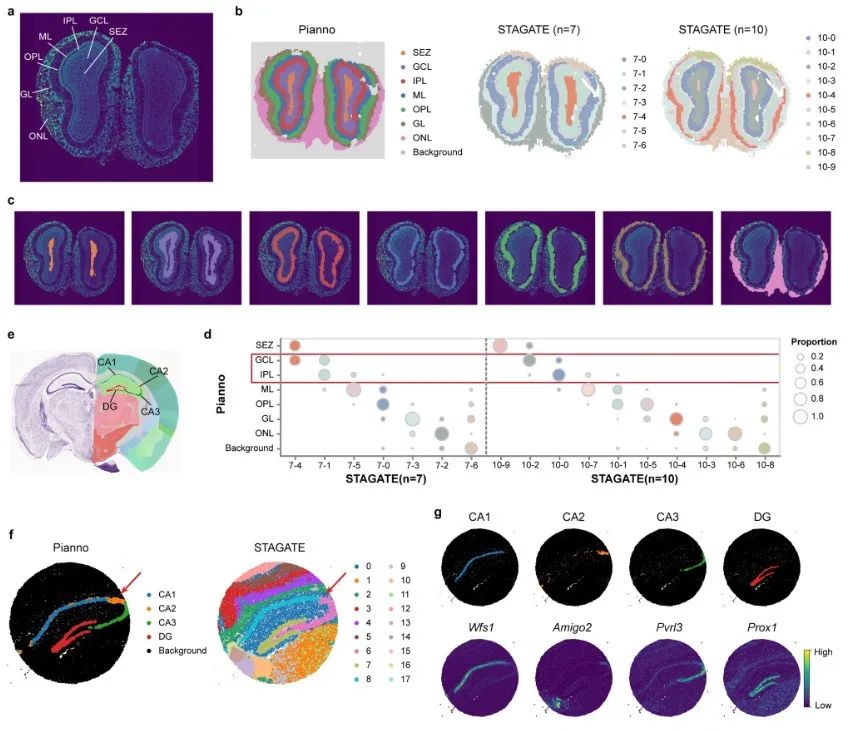
研究チームは、Pianno を使用して、マウス嗅球のステレオ seq データセットの解剖学的構造に注釈を付けました。このデータセットには、組織カバー領域と背景領域をカバーする 10,747 個の空間点が含まれています。
Pianno は、背景の削除と構造の注釈の両方を数分で実行できます。対照的に、クラスターの数が構造の数に設定されている場合、STAGATE はすべての解剖学的構造に対応するクラスターを識別できません。
研究チームはまた、腫瘍微小環境の不均一性が高いため、複雑で分散した構造組織に注釈を付ける際の Pianno のパフォーマンスも評価しました。このラウンドの試験では、2 つのヒト膵管腺癌サンプルと 2 つの乳癌サンプルの微小環境を分析しました。
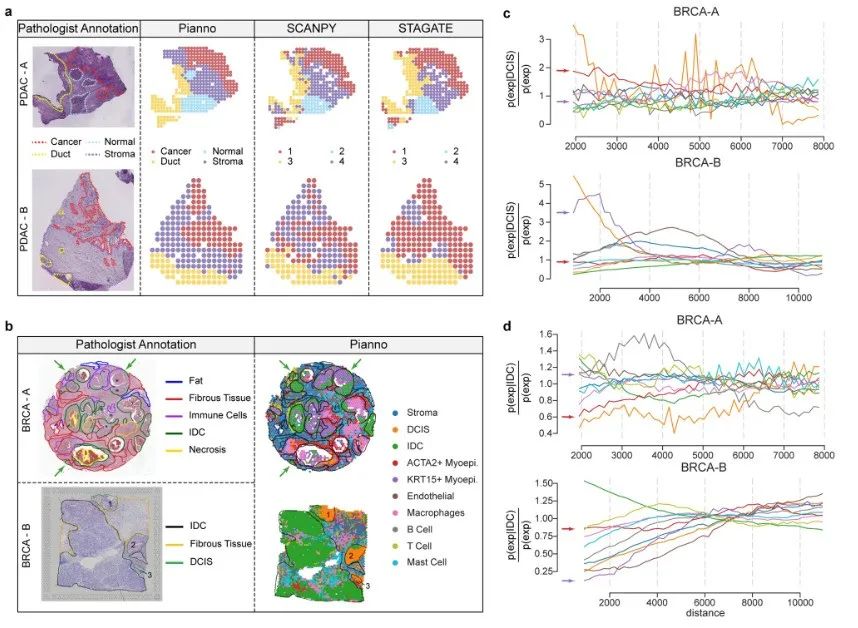
全体、Pianno は、専門の病理学者による手動アノテーションと一定レベルの一貫性を示し、特に不均一な腫瘍微小環境における不規則で複雑な構造のアノテーションにおける大きな可能性を実証しました。これは、病理学者が腫瘍生物学の複雑さを理解する上で貴重な支援を提供し、個別化された治療戦略を提供するための新しいアイデアにつながる可能性があります。
人工知能と複雑な生物学の組み合わせには大きな可能性があります
復旦大学脳科学研究所の報告によると、この研究プロジェクトは、科学技術イノベーション2030~国家重点研究開発プログラム「生物と情報の融合(BTとITの融合)」の重点プロジェクトに選ばれた。 「脳科学と脳にインスピレーションを得た研究」主要プロジェクト、および自然科学財団、上海主要科学技術プロジェクト、張江研究所による国の資金提供。
復丹大学脳科学研究所は2006年4月に設立されたとされています。復丹大学の全学的な神経科学研究機関であり、教育部の第2期教育で構築された重要な科学技術イノベーションプラットフォームの1つです。 「985 プロジェクト」。これは医療神経生物学と密接に関係しています。州立重要研究所は「2 つの機能を備えた」建設プロジェクトです。
復旦大学脳科学研究所は設立以来、実りある成果を上げてきました。同研究所は、国際的および国内的な主要なニーズに繰り返し直面し、主要な科学研究プロジェクトに着手し、重要な研究結果を生み出してきました。公式ウェブサイトによると、同研究所の研究者らは、科学技術省の「973計画」、「863計画」、科学技術イノベーション2030「脳科学と科学」を含む一連の主要な科学研究プロジェクトを主宰し、参加してきた。 「脳からインスピレーションを得た研究」、国家重点研究開発計画、国家重点科学技術プロジェクト、「主要新薬創出」など
実際、復旦大学脳科学研究所以外にも、多くの研究室や企業が空間トランスクリプトーム技術に注目し始めている。
例えば、中国科学院数学システム科学研究所の Zhang Shihua 氏のチームは、STA シリーズのツールを開発しました。 2022 年、チームは、さまざまな空間トランスクリプトーム技術とさまざまな生体組織を使用して生体組織の空間下部構造を識別するように適合された人工知能ツールである STAGATE をリリースしました。 2023 年に入ってから、チームは空間トランスクリプトーム技術に関する多くの成果を発表しました。 * 新しい統合分析ツール STAligner は、さまざまな技術、さまざまな開発時点、およびさまざまな疾患状態からの生体組織のマルチスライス空間トランスクリプトーム データに対して確立されました。 ※STAMarkerは、深層学習顕著性マップに基づく空間領域固有の可変遺伝子同定手法であり、空間領域の同定と対応する空間変数遺伝子の同定を同時に実現しており、空間トランスクリプトームデータのきめ細かな解析に効果的な手法を提供することが期待されています。 * 中国科学院北京ゲノミクス研究所(国立バイオインフォマティクスセンター)のYung Yungui氏とCai Jun氏のチームと協力して、地中海プラナリアの再生過程における三次元空間トランスクリプトームマップSTAPRを作成し、体系的に複数のプラナリアを同定した再生のための重要な調節因子。

華中師範大学数学統計学部の張小飛教授の研究グループは、ENGEP と呼ばれる計算方法を開発しました。k 最近傍重み付け回帰およびアンサンブル学習戦略を使用すると、空間トランスクリプトームにおける未測定の遺伝子の発現を正確に予測できます。さらに、ENGEP は空間的に未測定の遺伝子の発現パターンを正確に予測することもでき、これは空間トランスクリプトーム データを強化する上で非常に重要です。
空間転写、さらには生物学の分野における AI の強化は、研究効率を向上させるだけでなく、科学研究の困難な問題を解決するための新しいアイデアを提供することは疑いの余地がありません。論文のディスカッションセクションで指摘されているように、Pianno によってもたらされる価値は、既存の労働集約的な手動アノテーションに取って代わり、自動化された方法で効率的で正確かつ低コストのフォームを提供し、空間トランスクリプトミクスに変化をもたらす可能性があります。生物学の新たな発展も促進します。
参考文献:
1. https://news.fudan.edu.cn/2024/0407/c2474a139894/page.htm
2. https://bfse.cas.cn/sxyqyjc/kyjz/202311/t20231110_4985132.html
3. https://kjc.ccnu.edu.cn/info/1009/3744.htm








