Command Palette
Search for a command to run...
特徴選択戦略: 乳がんバイオマーカーを検出するための新しい手段を見つける

内容の概要:microRNA (低分子リボ核酸) は、短い一本鎖のノンコーディング RNA 転写物の一種です。これらの分子はさまざまな悪性腫瘍内で制御不能に増殖するため、近年、がん診断の信頼できるバイオマーカーとして多くの研究によって特定されています。さまざまな病理学的分析において、発現差分析は重要なバイオマーカーを検出するための効果的な方法とみなされますが、イタリアのナポリ大学フェデリコ 2 世の研究者は、より効果的な検出を可能にする機械学習に基づく特徴選択 (特徴選択) 戦略を提案しました。発見された 20 個のマイクロ RNA は、乳がんの診断バイオマーカーとして推奨されています。
キーワード:機能選択マイクロRNA乳がん
この記事は、HyperAI Super Neural WeChat パブリック プラットフォームで初めて公開されました~
国家保健委員会が発表した2022年版「乳がん治療ガイドライン」によると、乳がんは女性によく見られる悪性腫瘍の一つであり、女性の悪性腫瘍の中で最も発生率が高い。世界保健機関の統計によると、2020 年に世界中で合計 230 万人の女性が乳がんと診断されました。治療方法の継続的な改善により、早期乳がんの 5 年生存率は 90% 以上に達する可能性があります。したがって、乳がんの正確な早期診断は特に重要です。
マイクロRNAの多くの重要な生物学的役割に加えて、マイクロRNAの発現変化はさまざまながんにも関連しているため、信頼できる診断バイオマーカーとして使用できます。イタリアのナポリ大学フェデリコ 2 世の研究者らは、機械学習に基づいて、特徴選択戦略を使用し、3 つの方法の安定性と分類パフォーマンスを分析します。乳がんに特異的な診断バイオマーカーのパネルが取得され、乳がん疾患の発症と進化における推定上の重要な遺伝子も発見されました。
現在、この研究成果は「Proceedings of the 18th Conference on Computational Intelligence Methods in Bioinformatics and Biostatistics (CIBB 2023)」に、「Robust Feature Selection Strategy detects a pane of microRNAs as putative Diagnostic biomarkers in Breast" Cancer」と題して掲載されている。

研究結果はCIBB 2023で発表されました
用紙のアドレス:
https://www.researchgate.net/publication/372083934
実験概要
この研究では、研究者らは 3 つの特徴選択方法 (ゲイン レート、ランダム フォレスト、サポート ベクター マシンの再帰的特徴除去) の助けを借りて、診断分子の組み合わせをより効率的に抽出できることを発見し、20 個のマイクロ RNA を含むパネルを明らかにしました。そのうち、hsa-mir-337、hsa-mir-378c、および hsa-mir-483 は、乳がん診断のための現在のバイオマーカーの中で医学界から広く注目されていません。この方法は、一般的に使用される差次的発現方法と比較して、健康なサンプルと腫瘍サンプルを区別でき、分類性能が優れており、過小評価されやすい、あるいは無視されやすい特徴を識別するのが容易です。
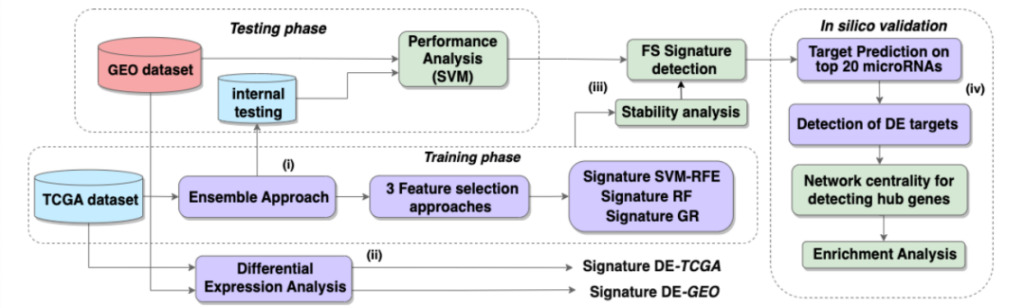
図 1: パイプラインの概要
ワークフローには 4 つの主要なステップが含まれています。
(私) トレーニング TCGA サブセットの Ensemble-FS 計算
(ii) TCGA/GEO データセットの発現差解析
(iii) 差分発現解析の分類性能と特徴選択結果を比較し、特徴選択手法の安定性を評価します
(iv) コンピューター シミュレーションを使用して、選択したシグネチャーの上位 20 個の microRNA を検証し、ハブ遺伝子ターゲットを検出します。
実験の詳細
データセット
実験的なデータ ソースには 2 つのチャネルが含まれています。米国 GDC 公式 Web サイトおよび Gene Expression Omnibus (GEO) データ リポジトリ (GSE97811) の TCGA-BRCA プロジェクト。
実験チームは、GDC TCGA-BRCA プロジェクトから合計 1,881 個の microRNA-Seq データを収集し、それを 8:2 の比率でトレーニング セットとテスト セットに分割しました。データは、300 個の固形原発腫瘍サンプル (T) と 101 個の正常隣接腫瘍 (NAT) サンプルに関するもので、すべて乳管および小葉組織に属します。特徴選択方法を適用する前に、これらのデータには分散安定化正規化が適用されています。
同時に、研究者らは 2,565 個のマイクロ RNA を含むマイクロアレイ データセットを GEO データベース (GSE97811) からダウンロードしました。今回の実験の検証セットとして。データセットには 16 個の正常サンプルと 45 個の腫瘍サンプルが含まれており、データ補完が実行されます。
GEO データ (この実験の検証セット) には成熟した microRNA 発現が含まれるため、TCGA データ (この実験のトレーニング セットとテスト セット) には初期の形式 (前駆体形式) が含まれており、データを統合するために研究者らは GEO を使用しました。データ 反対鎖の平均カウント値よりも高い平均カウント値を持つ代替の成熟マイクロ RNA のみがサンプルから選択され、同時にマイクロ RNA の名前も対応する初期形式の名前に変換されました。
このようなプロセスを経て、GEO データ (検証セット) の次元は 1,361 個の microRNA に削減され、合計 20,404 個の遺伝子を含む対応する TCGA RNA-Seq データも収集されました。
1. 特徴選択方法とEnsembleプログラムの適用
研究者らは、差次的発現解析法と比較するために 3 つの特徴選択法を選択しました。彼らです ゲイン比、ランダム フォレスト、および SVM-RFE (サポート ベクター マシン再帰的機能除去)。3 つの方法を microRNA-Seq 発現 TCGA データの 500 サブセットに適用して、正常サンプルと腫瘍サンプルを区別できる堅牢な特徴パネルを特定しました。観察された結果は 8:2 の比率に従ってトレーニング セットとテスト セットに分割され、データは Data Perturbation Ensemble 手順に準拠するようにリサンプリングを通じてブートストラップによって処理されました。各計算では、「重要度スコア」の降順にソートされた 500 個の microRNA ベクトルが返されます。
|備考:重要度スコアは、アルゴリズムによって計算された分類における各特徴の影響を表します。
重要度スコアが高いほど、機能に割り当てられるランクは低くなります。次に研究者らは、集計手順を使用して各特徴選択方法のコンセンサス シグネチャを導き出し、最終的にマイクロ RNA の各グループが上位 200 のスコアリング特徴を保持しました。
2. 安定性試験
Kuncheva Index (KI) と重複する遺伝子/特徴の割合 (POG) を使用して特徴選択法の一貫性を評価し、Stot 統計 (KI のペア測定) を使用してすべての方法間の安定性を決定しました。これらの統計は、署名の長さが徐々に増加するにつれて計算されます。特徴の数は 2 から始まり 200 で終わり、再計算されるたびに 2 単位ずつ増加します。

Stot統計手法の公式
3. 発現差解析と DE シグネチャ
TCGA データセット (microRNA-Seq および RNA-Seq を含む) に対して、生カウント (生カウント) から開始して正確検定 (Exact Test) を使用して差次的発現解析を実行し、FDR <= 0.01 および Log2FC を保持します。 DE 機能のしきい値。DE-microRNA のシグネチャを取得するために、Log2FC 値を絶対値に変換し、microRNA (最初の 200 個の特徴を保持) を abs (Log2FC) の降順にソートしました。
GEO 検証セットは、差分分析と表現に Limma を使用します。このデータ セットの DE 署名を取得するためのパラメーターと手順は、TCGA データ セットと一致しています。
4. 分類パフォーマンス分析
各署名が健康な人とがん患者を区別できるかどうかを判断するために、研究者らは、テストサブセット (TCGA) と検証セット (GEO) の 4 つのシグネチャ (特徴選択パネルと差次的発現パネルを含む) について予測分析を実施しました。
最後に、平均精度 (ACC)、K 統計量 (KK)、およびマシューズ相関係数 (MCC) が各フォールドと各シグネチャの複数の長さの平均に対して計算されます。
5.SVM-RFE マイクロRNAシグネチャーターゲット検出
マイクロRNAの潜在的な遺伝子標的を特定するには、研究者らは次の操作を実行しました。
1. 上位 20 個の SVM-RFE microRNA は、腫瘍サンプルにおける上方制御または下方制御状態に従って分類されました。
2. RNA-Seq データに対して発現差解析を実行して、発現差のある遺伝子 (FDR <= 0.05) を検出します。
3. スピアマン相関分析を使用して、マイクロ RNA 発現と発現差のある遺伝子を比較しました。ダウン マイクロ RNA と負の相関を示したアップ遺伝子と、アップ マイクロ RNA と負の相関を示したダウン遺伝子 (rho <= -0.5) のみが保持されました。
4. 検証されたすべてのマイクロ RNA 遺伝子ターゲットが収集され、DE との相関も示したもののみが保持されました。
6. ネットワークの中心性とハブ遺伝子の同定
選択された調節不全遺伝子の相関行列 (Spearman)、そしてそれを使用してグラフ構造の遺伝子ネットワークを構築します。クラインバーグのハブ中心性スコア > 75、rho > 0.8、または rho < -0.6 を持つハブ遺伝子が保持されました。 REACTOME データベースから最も濃縮された経路を探索するために、ハブ遺伝子に対して遺伝子濃縮分析 (ORA) が実行されました。 FDR 調整後の pValue 値のしきい値は 0.005 に設定されます。
実験結果
実験では、3 つの特徴選択方法を適用した後、重要度スコアの降順に並べられた 500 個のマイクロ RNA シグネチャが返され、集約後に 3 つのコンセンサス パネルが得られたことが示されています。上位 3 つのマイクロ RNA (hsa-mir-139、hsa-mir-96、および hsa-mir-145) がすべてのパネルに表示され、腫瘍サンプルと健康なサンプルを区別する際のこれらの分子の役割が重要であることは注目に値します。
|結論1:SVM-RFEが最も安定性が高い
コンセンサスパネルでのKIとPOGの計算から、SVM-RFE 方法は最も安定しており、署名の長さが 20 特徴に達すると最も顕著になります。同様に、Stot 指数の結果からも、SVE-RFE 法が最も安定性が高いことがわかります。
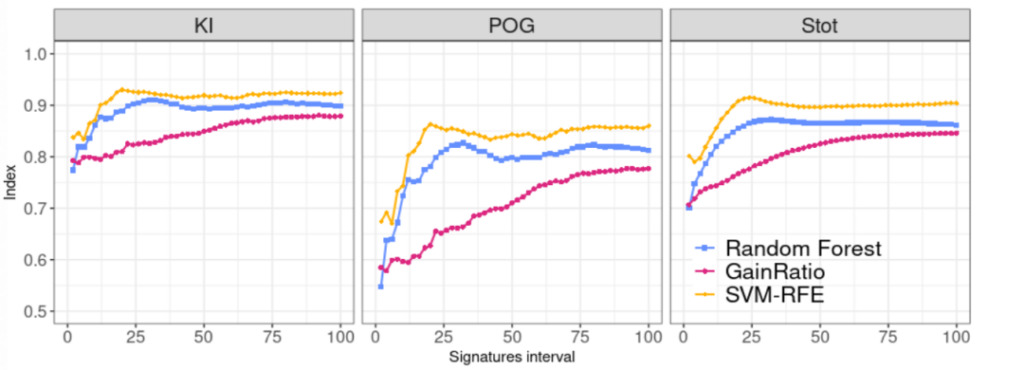
図 2: 3 つの特徴選択方法の安定性指数の比較
青:ランダムフォレスト
ピンク:ゲイン比
黄色:SVM-RFE (サポート ベクター マシンの再帰的機能の削除)
|結論 2: SVM-RFE 署名は、分類において差分表現署名よりも優れたパフォーマンスを発揮します。
テスト セット (TCGA) であっても検証セット (GEO) であっても、すべての個々のパネルの分類パフォーマンスを分析した結果、SVM-RFE で取得されたシグネチャが最も高い予測能力を備えていることが示されました。
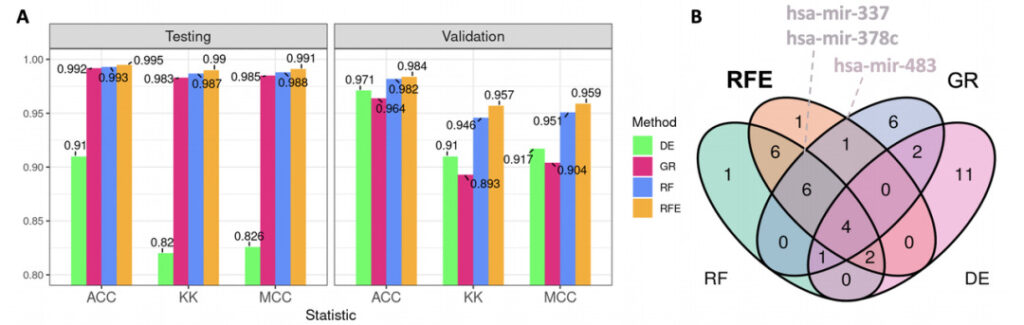
図 3: 上位 20 の microRNA 分類パフォーマンスとベン図
答え:棒グラフは、テスト サブセットと外部検証 GEO データセットで計算された平均統計を示します。
ACC:正確さ
KK:K統計量
クライアント センター:マシューズの相関係数
緑:DE(発現差解析、本実験における制御方法)
ピンク:GR(ゲインレート)
青:RF(ランダムフォレスト)
オレンジ色:RFE (SVM-RFE、サポート ベクター マシンの再帰的機能の削除)
B:各シグネチャの上位 20 のマイクロ RNA のベン図。SVM-RFE パネルの上位 20 にあるいくつかの興味深いマイクロ RNA、hsa-mir-337、hsa-mir-378c、および hsa-mir-483 を示しています。は 3 つの特徴選択方法すべてに含まれていますが、乳がんに関する現在の研究では、それが診断の根拠として信頼できるかどうかはまだ完全には判断されていません。
|結論 3: ネットワーク分析により、疾患の進化における潜在的な重要な遺伝子が明らかになりました
実験では、CDC25、TPX2、KIF18Bがさまざまな種類のがんやトリプルネガティブ乳がん患者の幹細胞で高度に発現しており、TGFBR2の下方制御ががんの進行と関連していることが示されている。
マイクロRNA:乳がんの早期スクリーニングのためのもう一つの理想的な候補
従来の乳がんのスクリーニング方法は依然として X 線画像診断と組織生検に基づいており、完全ながんゲノムについてより深くより包括的な理解を確立することはできません。この方法は侵襲性が高く、費用がかかり、副作用が発生しやすいだけでなく、偽陽性または偽陰性の結果が得られることが多く、早期乳がんスクリーニングの精度と患者エクスペリエンスが向上します。乳がんの負担に対処するためには、依然として新しい戦略を開発する必要があります。
1993 年の最初の発見以来、microRNA はがんに対する人類の理解を深め続け、乳がんの信頼できる診断バイオマーカーとして大きな可能性を示してきました。
マイクロRNAは、長さが約19〜25ntの小さな非コードRNAであり、さまざまな標的遺伝子を制御できます。さまざまな生物学的および病理学的プロセスの制御に関与し、がんの形成と進行を含め、臨床乳がんスクリーニングの主流の診断方法である現在の X 線画像診断と組織生検の限界を補うことが期待されています。
しかし、マイクロRNAの臨床応用はまだ十分に開発されておらず、マイクロRNAの使用に対する安全性評価システムも確立されていません。マイクロRNAががんの診断の主流となるには、まだ時間がかかるかもしれない。
参考記事:
[1]https://www.who.int/zh/news-room/fact-sheets/detail/breast-cancer
[2]https://guide.medlive.cn/guideline/25596
[3]https://www.abcam.cn/kits/micrornas-as-biomarkers-in-cancer-1
[4]https://caivd-org.cn/webfile/file/20220508/20220508153691029102.pdf
[5]https://www.sohu.com/a/318088245_100120288
この記事は、HyperAI Super Neural WeChat パブリック プラットフォームで初めて公開されました~








