Command Palette
Search for a command to run...
Meta AI Et al. Ont Proposé Un Nouveau Cadre De Caractérisation De Fusion Dynamique Des Protéines, FusionProt, Qui Permet Un Échange d'informations Itératif Et Atteint Des Performances SOTA Dans Plusieurs tâches.
Les protéines sont les exécutantes des fonctions vitales, et leurs mystères résident dans deux dimensions :L'une est la séquence unidimensionnelle (1D) formée par des acides aminés connectés bout à bout, et l'autre est la structure tridimensionnelle (3D) formée par le pliage et l'enroulement de la séquence.Les modèles précédents se spécialisaient généralement dans l'un ou l'autre de ces domaines, soit en maîtrisant le « langage des séquences » comme les modèles de langage protéique (PLM) comme ProteinBERT et ESM, soit en distinguant la « morphologie structurale » comme les technologies de représentation tridimensionnelle des protéines comme GearNet. Même lorsque les modèles tentent de combiner les deux, ils adoptent souvent une approche simplifiée et segmentée, permettant apparemment à chaque spécialiste de travailler de manière indépendante plutôt que collaborative.
Dans ce contexte, les équipes de recherche du Technion-Israel Institute of Technology et de Meta AI ont proposé conjointement le cadre d'apprentissage de la représentation des protéines FusionProt.Il vise à apprendre simultanément une représentation unifiée de la séquence unidimensionnelle et de la structure tridimensionnelle des protéines.Cette recherche introduit de manière innovante un jeton de fusion apprenable comme passerelle adaptative entre le modèle de langage protéique (PLM) et le graphe de structure 3D, permettant un échange d'informations itératif entre les deux. FusionProt a atteint des performances de pointe dans diverses tâches biologiques liées aux protéines.
La recherche connexe a été publiée sur bioRxiv sous le titre « FusionProt : fusion de séquences et d'informations structurelles pour l'apprentissage unifié de la représentation des protéines ».
Points saillants de la recherche :
* Le cadre FusionProt dépasse les limites du traitement de fragmentation de structure précédent en intégrant efficacement les modalités unidimensionnelles et tridimensionnelles, et améliore la précision de la capture des fonctions des protéines et des propriétés d'interaction.
* Une nouvelle architecture de fusion intermodale qui utilise des jetons de fusion apprenables pour permettre l'échange d'informations itératif entre le modèle de langage protéique (PLM) et les graphiques de structure 3D des protéines.
* FusionProt atteint des performances de pointe dans de multiples tâches protéiques et démontre le potentiel du modèle pour l'application dans des scénarios biologiques du monde réel à travers des études de cas.
Adresse du document :
Suivez le compte officiel et répondez « FusionProt » pour obtenir le PDF complet
Autres articles sur les frontières de l'IA :
https://hyper.ai/papers
Construire systématiquement des ensembles de données à l'aide de bases de données protéiques publiques
Dans cette étude, l'équipe de recherche a pleinement utilisé les bases de données protéiques publiques et a assuré l'efficacité de FusionProt dans de multiples tâches de compréhension des protéines grâce à la construction systématique d'ensembles de données, à une stratégie stricte de partitionnement des données et à un cadre d'évaluation multitâche.
Au cours de la phase de pré-formation, l’étude a utilisé la base de données sur la structure des protéines (AlphaFold DB) comme source de données principale.La base de données contient 805 000 structures protéiques tridimensionnelles de haute qualité prédites par AlphaFold2. Les chercheurs ont sélectionné cet ensemble de données principalement pour les raisons suivantes : premièrement, AlphaFold2 est actuellement reconnu comme un modèle de pointe en matière de prédiction de la structure des protéines, et ses prédictions sont extrêmement fiables, réduisant ainsi la dépendance à la qualité et à la disponibilité de données structurales expérimentales externes ; deuxièmement, l'utilisation d'une structure prédite unifiée garantit la cohérence entre les sources de données, facilitant ainsi une comparaison équitable avec des travaux de pointe antérieurs tels que SaProt et ESM-GearNet.
L'équipe de recherche a ensuite évalué systématiquement les performances du modèle à travers trois tâches en aval faisant autorité. Ces tâches ont utilisé des ensembles de données de DeepFRI, qui fournit un partitionnement de données faisant autorité et utilise Fmax comme indicateur d'évaluation unifié, mesurant de manière exhaustive les performances du modèle en matière d'annotation des fonctions enzymatiques et d'inférence d'ontologies génétiques. La tâche de prédiction de la stabilité mutationnelle (MSP) a utilisé le même ensemble de données et le même protocole d'évaluation qu'ESM-GearNet, utilisant AUROC comme indicateur d'évaluation pour évaluer la capacité du modèle à prédire les effets des mutations sur la stabilité des complexes protéiques.
Mécanisme d'échange d'informations itératif piloté par un « jeton de fusion »
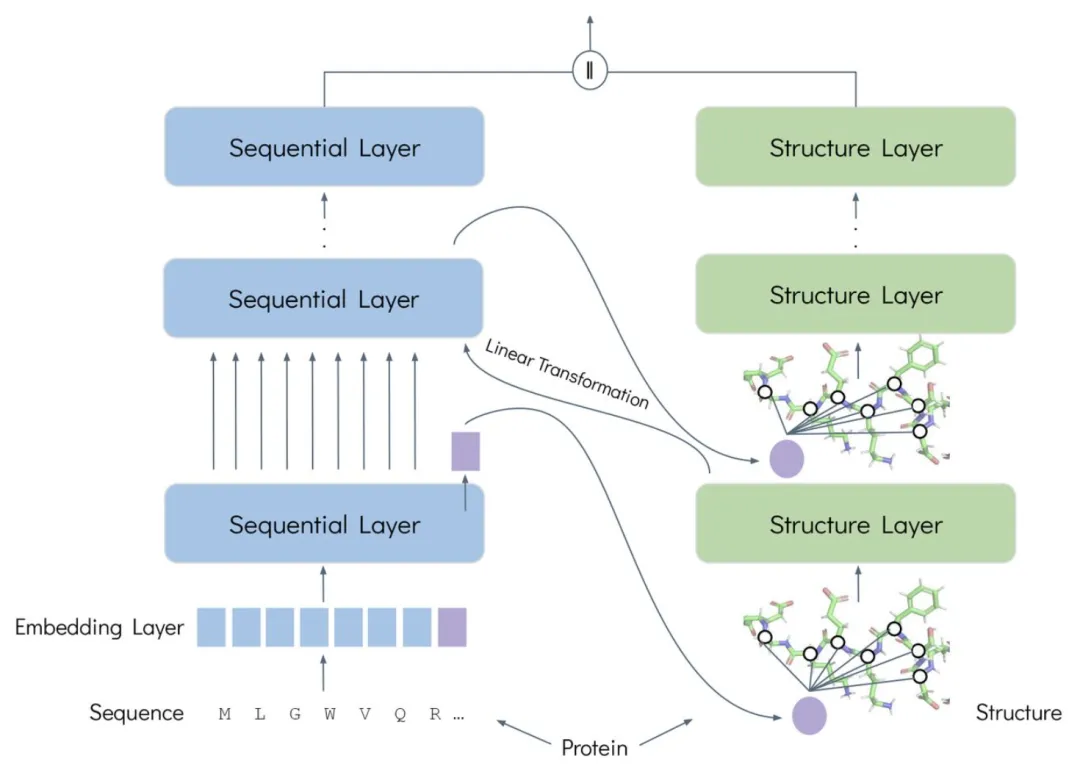
La conception de FusionProt s'articule autour d'une idée centrale :Grâce à des jetons de fusion apprenables, il agit comme un pont pour une interaction intermodale bidirectionnelle et itérative entre la séquence et la structure des protéines.Cela permet une fusion profonde et une représentation unifiée des deux types d’informations.
Tout d'abord, le framework repose sur l'architecture de codage parallèle bimodale « séquence-structure ». Au niveau séquence, ESM-2 est utilisé comme modèle de langage protéique pour coder la séquence d'acides aminés ; au niveau structurel, l'encodeur GearNet sert de modèle structurel pour modéliser le diagramme de structure tridimensionnel de la protéine. Le jeton de fusion apprenable alterne dynamiquement entre les deux modalités pendant le processus d'apprentissage, permettant l'échange itératif et la fusion d'informations. Au niveau séquence, il est connecté à la séquence protéique, et l'acide aminé interroge le jeton de fusion unique pertinent pour extraire et intégrer des informations précieuses. Au niveau structurel, il est ajouté au graphe tridimensionnel de la protéine, fusionné et connecté en tant que nœud. La couche structurelle est traitée par un réseau neuronal à transmission de messages, permettant au jeton de fusion d'intégrer des informations structurelles spatiales globales.
Deuxièmement, la force motrice principale de ce cadre réside dans l’algorithme de fusion itérative.Le processus consiste à intégrer les jetons de fusion à la séquence mise à jour, puis à les transmettre à la couche structure et à les introduire dans le réseau de graphes de structure sous forme de nouveaux nœuds. Les jetons de fusion mis à jour sont ensuite renvoyés à la couche séquence pour la prochaine interaction. Ce processus itératif aligne et ajuste les différents espaces modaux grâce à des transformations linéaires apprenables. Grâce à ce processus itératif, les représentations du modèle sont combinées pour former une représentation protéique unifiée et riche.
Enfin, FusionProt utilise l’apprentissage contrastif multivue comme objectif de pré-formation.Une vue diversifiée est construite en sélectionnant aléatoirement des sous-séquences consécutives et en masquant les arêtes du graphe de 15%. La fonction de perte InfoNCE est ensuite utilisée pour aligner les représentations dans l'espace latent, préservant ainsi la similarité des sous-composants protéiques apparentés lorsqu'ils sont mappés à l'espace latent de faible dimension. Pour la mise en œuvre, l'équipe de recherche a effectué un pré-entraînement sur la base de données AlphaFold DB mentionnée ci-dessus. Lors du pré-entraînement, FusionProt a utilisé un taux d'apprentissage de 2e-4, un lot global de 256 protéines et a effectué 50 cycles d'entraînement. La séquence d'entrée a été tronquée à 1 024 jetons pour prendre en compte les longues séquences protéiques. De plus, un réglage fin a été effectué en ajoutant des prédictions de tête de classification spécifiques à la tâche et en évaluant les mêmes hyperparamètres que le dernier modèle SaProt. Toutes les expériences ont été réalisées sur 4 GPU NVIDIA A100 80 Go, avec une seule session de pré-entraînement d'une durée d'environ 48 heures.
Surpassant largement le SOTA existant, le mécanisme de fusion a des effets significatifs
L’étude a été largement testée dans plusieurs tâches en aval.Les résultats montrent que le framework FusionProt atteint des performances SOTA dans plusieurs benchmarks.Les résultats expérimentaux sont présentés dans la figure ci-dessous.
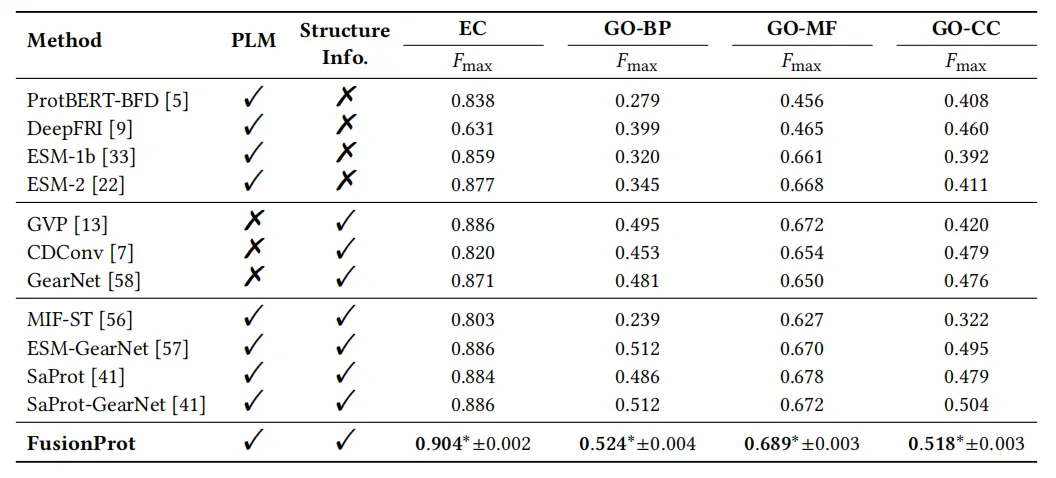
Lors de l'évaluation de la prédiction du nombre d'EC, l'équipe de recherche a comparé les performances de FusionProt à celles de 11 modèles de référence. Les résultats ont montré que FusionProt a atteint le Fmax le plus élevé (0,904), surpassant ainsi significativement les modèles reposant uniquement sur la séquence (tels que ProtBERT-BFD, 0,838, ESM-2, 0,877), et GearNet (0,871) qui utilise uniquement des informations structurelles. Parallèlement, comparé à d'autres méthodes qui tentent d'exploiter ces deux types d'informations (telles que MIF-ST, ESM-GearNet, etc.), il conserve son avance. Ce résultat montre que, comparé à la simple utilisation d'une modalité comme contexte d'une autre, le mécanisme de fusion itérative de FusionProt permet de mieux conserver les informations structurelles tridimensionnelles clés, capturant ainsi avec plus de précision les subtiles différences structurelles dont dépend l'activité catalytique.
Dans l'évaluation de la prédiction du terme GO, FusionProt a obtenu les meilleurs résultats dans les trois sous-tâches du processus biologique, de la fonction moléculaire et du composant cellulaire, démontrant une fois de plus l'efficacité des jetons de fusion apprenables dans la modélisation conjointe de la séquence et de la structure.
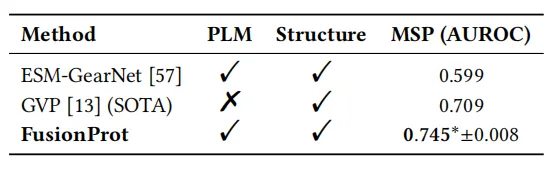
L'équipe de recherche a également évalué la prédiction de la stabilité mutationnelle. Les résultats expérimentaux ont montré que FusionProt a obtenu l'AUROC le plus élevé de toutes les méthodes évaluées, avec une significativité statistique (p < 0,05). Cette performance a été significativement améliorée de 5,11 TP3T par rapport à la méthode de pointe actuelle, GVP, soulignant l'efficacité de son mécanisme de fusion itératif pour intégrer les dépendances séquence-structure à longue portée. De plus, FusionProt permet une interaction intermodale bidirectionnelle grâce à des jetons de fusion apprenables, rendant la représentation des protéines plus expressive et biologiquement fondée.
Afin d'évaluer l'efficacité des conceptions clés de FusionProt, l'équipe de recherche a mené d'autres expériences d'ablation. L'équipe a testé le système à différentes fréquences d'injection de fusion et a constaté que les performances optimales étaient obtenues lorsque les marqueurs de fusion effectuaient plusieurs cycles d'interactions entre les encodeurs de séquence et de structure à une fréquence standard ; en revanche, une réduction de la fréquence d'interaction affaiblissait considérablement les performances.Cela montre qu’un échange fréquent d’informations est crucial pour saisir les dépendances intermodales.
Enfin, dans une analyse de cas biologique, FusionProt a réussi à prédire le nombre EC de la sous-unité ω de l'ARN polymérase, un résultat difficile à traiter avec les méthodes traditionnelles. Ce résultat a été totalement inopérant dans des modèles comme ESM-2, prouvant une fois de plus que la représentation acquise peut capturer des relations « structure-fonction » complexes et présente un vaste potentiel d'application pour le développement de médicaments et l'analyse de la fonction des protéines.
La fusion intermodale est devenue une tendance évidente
FusionProt a ouvert une nouvelle voie pour l'apprentissage de la représentation des protéines, démontrant que le « langage » et la « morphologie » des protéines devraient communiquer entre eux, et non entre eux. Avec les progrès constants de l'intelligence artificielle dans les sciences de la vie, la fusion intermodale est devenue une tendance claire.
L'Université Westlake a proposé le concept d'un vocabulaire sensible à la structure et a combiné des jetons de résidus d'acides aminés avec des jetons de structure pour former un modèle universel de langage protéique à grande échelle, SaProt, sur un ensemble de données d'environ 40 millions de séquences et de structures protéiques. Ce modèle a largement surpassé les modèles de référence établis dans dix tâches importantes en aval. La recherche connexe, intitulée « SaProt : Modélisation du langage protéique avec un vocabulaire sensible à la structure », a été sélectionnée pour l'ICLR 2024.
Adresse du document :
https://openreview.net/forum?id=6MRm3G4NiU
Une étude conjointe intitulée « Modèle de langage protéique à structure alignée », publiée par l'Université de Montréal et Mila, propose un modèle de langage protéique à structure alignée qui intègre les informations structurelles grâce à l'apprentissage contrastif. En optimisant les jetons structuraux prédits par le modèle, celui-ci améliore significativement les performances des tâches de prédiction de contact protéique.
Adresse du document :
https://arxiv.org/abs/2505.16896
Obtenez des articles de haute qualité et des articles d'interprétation approfondis dans le domaine de l'IA4S de 2023 à 2024 en un seul clic⬇️









