Command Palette
Search for a command to run...
ACL 2025 : l'Université d'Oxford Et d'autres Proposent GraphRAG Médical, Établissant Un Nouveau Record De Précision Des Réponses Aux Questions Et Obtenant Des Résultats SOTA Sur 11 Ensembles De Données

Le système de connaissances du domaine médical repose sur des milliers d'années de découvertes et d'accumulation, couvrant une vaste quantité de principes, de concepts et de normes pratiques. Adapter efficacement ces connaissances à la fenêtre contextuelle limitée des grands modèles de langage actuels présente des obstacles techniques insurmontables. Bien que le réglage fin supervisé (SFT) offre une alternative, en raison du caractère fermé de la plupart des modèles commerciaux, cette approche est non seulement coûteuse, mais aussi extrêmement peu pratique en pratique. De plus, le domaine médical impose des exigences extrêmement élevées en matière de précision terminologique et de rigueur factuelle. Pour les utilisateurs non professionnels, vérifier l'exactitude des grands modèles pour des réponses médicales est en soi une tâche extrêmement complexe. Par conséquent, la question de savoir comment permettre aux grands modèles d'utiliser de vastes ensembles de données externes pour un raisonnement complexe dans les applications médicales et de générer des réponses précises et crédibles, étayées par des sources vérifiables, est devenue un enjeu central de la recherche actuelle dans ce domaine.
L’émergence de la technologie de génération améliorée par récupération (RAG) offre une nouvelle approche pour résoudre les problèmes ci-dessus.Il peut répondre aux requêtes des utilisateurs à l'aide d'ensembles de données spécifiques ou privés sans formation supplémentaire du modèle.Cependant, les RAG traditionnels restent insuffisants pour synthétiser de nouvelles connaissances et gérer des tâches nécessitant une compréhension globale d'un large éventail de documents. GraphRAG, récemment proposé, surpasse largement les RAG classiques en matière de raisonnement complexe. Il exploite les LLM pour construire un graphe de connaissances à partir de documents bruts, puis extraire les connaissances issues de ce graphe afin d'améliorer les réponses. Cependant, la construction du graphe par GraphRAG manque de conception spécifique pour garantir l'authentification et la crédibilité des réponses, et son processus de construction de communauté hiérarchique est coûteux en raison de sa nature polyvalente, ce qui rend son application directe et efficace au domaine médical difficile.
Pour remédier à cette situation, une équipe conjointe de l'Université d'Oxford, de l'Université Carnegie Mellon et de l'Université d'Édimbourg a proposé une méthode RAG basée sur des graphiques spécifiquement destinée au domaine médical : Medical GraphRAG (MedGraphRAG).Cette méthode améliore efficacement les performances du LLM dans le domaine médical en générant des réponses fondées sur des preuves et des explications officielles de la terminologie médicale, ce qui non seulement améliore la crédibilité des réponses, mais améliore également considérablement la qualité globale.
Les résultats de recherche associés, intitulés « Medical Graph RAG : Towards Safe Medical Large Language Model via Graph Retrieval-Augmented Generation », ont été sélectionnés pour ACL 2025.
Points saillants de la recherche :
* Cette étude a proposé pour la première fois le cadre Tukey RAG spécifiquement pour une utilisation dans le domaine médical.
* Cette étude a développé une méthode unique de construction de triple graphe et de récupération U pour permettre au Large Language Model (LLM) d'utiliser efficacement l'ensemble des données RAG et de générer des réponses fondées sur des preuves.
* MedGraphRAG surpasse les autres méthodes de récupération et les modèles de langage spécifiques au domaine médical affinés sur plusieurs critères de réponse aux questions médicales.
Adresse du document :
Autres articles sur les frontières de l'IA :
Recherche basée sur trois types de données
Les données utilisées dans cette étude sont divisées en trois catégories, avec des caractéristiques de chaque type adaptées à son rôle dans l'étude :
* Données RAG
Étant donné que les utilisateurs peuvent utiliser des données privées fréquemment mises à jour (telles que les dossiers médicaux électroniques des patients), l'étude a sélectionné l'ensemble de données de dossiers médicaux électroniques publics MIMIC-IV, qui peut simuler les scénarios de données privées en évolution dynamique dans des applications réelles et fournir une base pour vérifier la praticité de la méthode.
* Données du référentiel
Cet ensemble de données est utilisé pour fournir des sources fiables et des définitions de vocabulaire faisant autorité pour les réponses du grand modèle. Le référentiel de niveau supérieur est MedC-K, qui contient 4,8 millions d'articles scientifiques biomédicaux, 30 000 manuels et toutes les publications de FakeHealth et PubHealth. Son contenu couvre un large éventail de domaines et fait autorité sur le plan académique ; le référentiel sous-jacent est le graphe UMLS, qui contient un vocabulaire médical et des relations sémantiques faisant autorité pour garantir l'exactitude de la terminologie médicale.
* Données de test
Cet ensemble de données est utilisé pour évaluer les performances de la méthode MedGraphRAG, y compris la partie test de 9 ensembles de données biomédicales à choix multiples dans MultiMedQA (y compris MedQA, MedMCQA, PubMedQA, sujets cliniques MMLU, etc.), qui est utilisé pour tester les performances de la méthode dans les réponses aux questions médicales de routine ; 2 ensembles de données de vérification des faits de santé publique FakeHealth et PubHealth sont utilisés pour évaluer la capacité de réponse fondée sur des preuves de la méthode ; en outre, l'étude a également collecté l'ensemble de tests DiverseHealth, qui contient 50 questions cliniques réelles couvrant un large éventail de sujets tels que les maladies rares et la santé des minorités, en mettant l'accent sur l'équité en santé, ce qui peut enrichir davantage les dimensions de l'évaluation.
MedGraphRAG : basé sur le partitionnement par fenêtre glissante, le clustering d'étiquettes et la recherche U
Comme le montre la figure ci-dessous,Le flux de travail global de MedGraphRAG comprend principalement trois liens principaux :Créez un graphique de connaissances basé sur des documents, organisez et résumez le graphique pour prendre en charge la récupération et répondez aux requêtes des utilisateurs en récupérant des données.
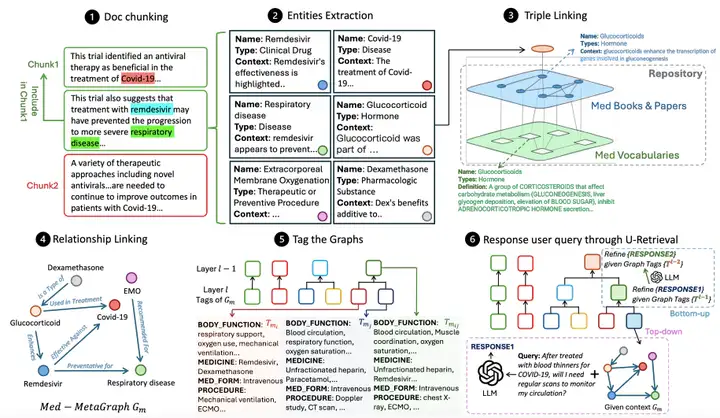
Medical Graph Construction effectue d'abord un découpage sémantique du document, en divisant le document en blocs de données conformes aux contraintes de contexte LLM.L'étude adopte une méthode hybride qui combine la séparation des caractères et la segmentation sémantique du sujet, c'est-à-dire en séparant d'abord les paragraphes par des sauts de ligne, puis en construisant LLM LG à travers le graphique pour juger de la pertinence du sujet entre le paragraphe et le bloc actuel afin de décider s'il faut diviser le bloc.Dans le même temps, une fenêtre coulissante à 5 segments est introduite pour réduire le bruit, et la restriction de balise LG est utilisée comme seuil strict pour la segmentation des blocs, en tenant compte à la fois de la logique sémantique et des contraintes de contexte du modèle.
Après la division des blocs, le processus d'extraction des entités entre en jeu. Grâce à LG et aux invites d'extraction, les entités pertinentes sont identifiées pour chaque bloc, et une sortie structurée contenant le nom, le type et la description du contexte est générée, ouvrant la voie à la liaison ultérieure des entités.La triple liaison est la clé pour garantir la précision.Un graphe référentiel (RepoGraph) est construit pour relier les documents RAG des utilisateurs à des sources fiables : la couche inférieure est le graphe UMLS (Med Vocabularies) contenant le vocabulaire et les relations médicales, et la couche supérieure est construite à partir de manuels médicaux et d'articles scientifiques (Med Books & Papers). Ensuite, les chercheurs définissent les entités extraites des documents RAG comme E1. Sur la base de la corrélation inter-entités, ces entités sont liées aux entités E2 extraites d'ouvrages ou d'articles médicaux. E2 est ensuite liée aux entités UMLS E3, formant une structure triple [entité RAG, source, définition], garantissant que chaque entité puisse être retracée jusqu'à une source claire et une définition standard. La liaison des relations est ensuite effectuée. Un LG, doté d'indices de reconnaissance de relations, identifie les relations entre les entités RAG en fonction de leur contenu et de leurs références, générant des phrases contenant les entités sources, les entités cibles et les descriptions des relations. Enfin, un graphe méta-médical orienté est généré pour chaque bloc de données.
Une fois le graphique créé, vous devez étiqueter les graphiques pour améliorer l'efficacité de la récupération.Contrairement à l'approche coûteuse de GraphRAG, basée sur la construction de communautés de graphes, cette méthode exploite la structure du texte médical et synthétise chaque métagraphe médical à l'aide d'étiquettes prédéfinies (symptômes, antécédents médicaux, fonction physique et médicaments) pour générer un résumé structuré. Cette méthode utilise ensuite un clustering hiérarchique agglomératif à seuil dynamique basé sur la similarité des étiquettes pour regrouper les graphes et générer un résumé plus abstrait et complet. Initialement, chaque graphe est traité comme un groupe indépendant. La similarité des étiquettes entre les paires de clusters est calculée de manière itérative, et les 201 paires de clusters TP3T présentant la plus grande similarité sont fusionnées pour générer une nouvelle couche de résumé des étiquettes. Ce processus est limité à 12 couches, garantissant un équilibre entre précision et efficacité.
L'étape finale de récupération U permet d'obtenir une réponse efficace à la requête en répondant à LLM LR.Tout d'abord, LR génère un résumé des étiquettes pour la requête utilisateur. Grâce à une récupération de précision descendante, en partant de l'étiquette de niveau supérieur, il associe les étiquettes les plus similaires couche par couche, localisant ainsi le métagraphe médical cible. En fonction de la similarité d'intégration entre la requête et le contenu de l'entité, il récupère les entités les mieux classées et leurs triplets voisins les plus proches, et utilise ces entités et relations pour générer une première réponse. Ensuite, la phase d'affinement de la réponse ascendante commence. LR ajuste la réponse en fonction du résumé des étiquettes de la couche précédente. Ce processus se répète jusqu'à atteindre le niveau cible (généralement 4 à 6 couches), générant ainsi une réponse qui équilibre la connaissance du contexte global et l'efficacité de la récupération.
MedGraphRAG : validé sur 6 modèles et 11 jeux de données pour obtenir le SOTA
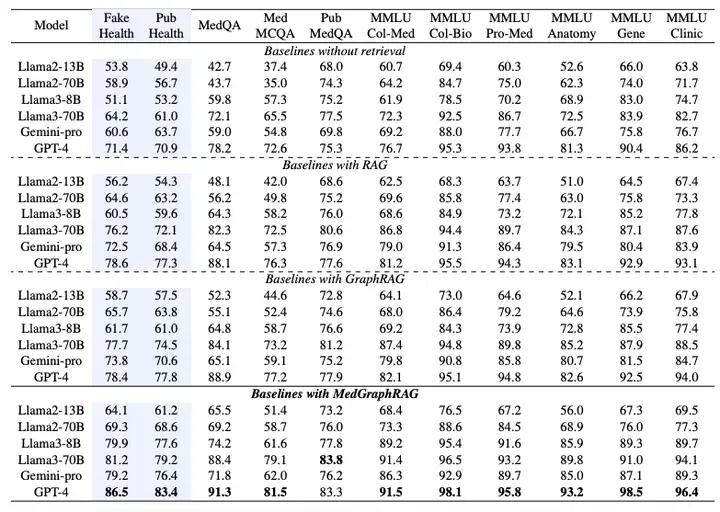
Pour vérifier les performances de MedGraphRAG, l’étude a sélectionné six grands modèles de langage et conçu plusieurs ensembles d’expériences, dont Llama2 (13B, 70B), Llama3 (8B, 70B), Gemini-pro et GPT-4.La principale comparaison est avec le RAG standard implémenté par LangChain et GraphRAG implémenté par Microsoft Azure.Toutes les méthodes sont exécutées sur les mêmes données RAG et données de test.
Comme le montre le tableau ci-dessous, la performance de l’évaluation à choix multiples est mesurée par la précision de la sélection de l’option correcte.Les résultats expérimentaux montrent que MedGraphRAG surpasse significativement la ligne de base sans fonction de récupération, le RAG standard et le GraphRAG :Par rapport à une base de référence sans recherche, il a obtenu une amélioration moyenne de près de 101 TP3T en vérification des faits et de 81 TP3T en réponse aux questions médicales. Comparé à GraphRAG, il a obtenu une amélioration d'environ 81 TP3T en vérification des faits et de 51 TP3T en réponse aux questions médicales. L'amélioration était encore plus significative pour les modèles plus petits (comme Llama2 13B), démontrant son intégration efficace des capacités de raisonnement du modèle et des connaissances externes. Appliqué à des modèles plus grands (tels que Llama70B et GPT-4), il a atteint des performances de pointe sur 11 ensembles de données, surpassant même les modèles optimisés sur des corpus médicaux tels que Med-PaLM 2 et Med-Gemini, établissant ainsi une nouvelle performance de pointe au classement des LLM médicaux.
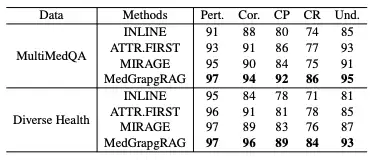
Dans l'évaluation de la génération au format long,Cette étude a comparé MedGraphRAG avec des modèles tels que Inline Search et ATTR-FIRST en termes de cinq dimensions : pertinence, exactitude, précision de citation, rappel de citation et compréhensibilité sur les benchmarks MultiMedQA et DiverseHealth.Les résultats sont présentés dans le tableau ci-dessous. MedGraphRAG a obtenu de meilleurs résultats dans tous les indicateurs, notamment en termes de précision des citations, de mémorisation et de compréhensibilité, grâce à ses réponses fondées sur des données probantes et à ses explications claires des termes médicaux.
Dans l'étude de cas, face à un cas complexe de bronchopneumopathie chronique obstructive (BPCO) et d'insuffisance cardiaque, les recommandations de GraphRAG ont ignoré l'impact des médicaments sur l'insuffisance cardiaque, tandis que MedGraphRAG a pu recommander des médicaments sûrs. Cela était dû au lien direct entre ses entités et ses références, évitant l'omission d'informations clés causée par l'imbrication des informations dans GraphRAG.
Intégration du Knowledge Graph et du modèle de langage étendu
À l’intersection de la médecine et de l’intelligence artificielle, l’intégration de graphes de connaissances et de grands modèles de langage devient une direction clé pour promouvoir les percées technologiques, fournissant de nouvelles idées pour résoudre des problèmes complexes dans le domaine médical.
Par exemple, le cadre KG4Diagnosis proposé par une équipe conjointe de l’Université de Cambridge et de l’Université d’Oxford,Il simule des systèmes médicaux réels grâce à une architecture multi-agents hiérarchique et combine des graphiques de connaissances pour améliorer les capacités de raisonnement diagnostique, couvrant le diagnostic automatisé et la planification du traitement pour 362 maladies courantes.Une équipe de recherche de l'Université Fudan a cartographié de manière exhaustive le protéome de la santé et de la maladie humaines. En analysant en profondeur les données du protéome plasmatique de 53 026 individus sur une période de suivi médiane de 14,8 ans, la carte couvre 2 920 protéines plasmatiques et 406 maladies préexistantes, 660 maladies nouvellement apparues pendant le suivi et 986 phénotypes liés à la santé.Découvrir de nombreuses associations protéine-maladie et protéine-phénotype,Fournir une base importante pour la médecine de précision et le développement de nouveaux médicaments.
Le système AMIE lancé par Google DeepMind,Intégrer les capacités de raisonnement à long contexte du grand modèle Gemini avec le graphe de connaissances,Grâce à une recherche dynamique dans les recommandations cliniques et les bases de connaissances sur les médicaments, un plan de prise en charge cohérent peut être généré pour plusieurs diagnostics. Par exemple, pour les patients atteints de bronchopneumopathie chronique obstructive (BPCO) et d'insuffisance cardiaque, des bêtabloquants cardiosélectifs peuvent être recommandés avec précision, évitant ainsi les risques d'interactions médicamenteuses des systèmes d'IA traditionnels.
Le graphique de connaissances biomédicales construit par AstraZeneca intègre 3 millions de documents et de données de recherche internes, et accélère le criblage de nouveaux candidats médicaments en analysant le réseau d'association médicament-cible-maladie.La carte comprend non seulement les indications des médicaments approuvés, mais couvre également les données d'« utilisation hors AMM » dans les essais cliniques.Aide à la décision pour la réutilisation de médicaments reconnus. De plus, la plateforme de graphes de connaissances d'IBM Watson Health intègre un milliard de données patients à des recommandations fondées sur des données probantes pour générer des plans de traitement personnalisés contre le cancer du poumon, incluant des tests génétiques, la prédiction de la sensibilité aux médicaments et des plans de suivi, réduisant ainsi l'erreur prédite de survie des patients à ± 2,3 mois.
Ces pratiques innovantes favorisent non seulement la mise à niveau itérative des technologies d'IA médicale, mais démontrent également un potentiel considérable pour améliorer la précision des diagnostics, accélérer le développement de médicaments et optimiser la prise de décision clinique. À mesure que la technologie progresse, l'intégration de graphes de connaissances et de grands modèles linguistiques permettra de lever les barrières informationnelles dans le domaine médical et d'insuffler une dynamique durable au développement des soins de santé à l'échelle mondiale.
Articles de référence :
1.https://mp.weixin.qq.com/s/WhVbnoso2Jf2PyZQwV93Rw
2.https://mp.weixin.qq.com/s/RWy4taiJCu3kMPfTzOWYSQ
3.https://mp.weixin.qq.com/s/lMLk








