Command Palette
Search for a command to run...
Sélectionné Pour l'AAAI 2025 ! Pour Résoudre Le Problème Des Limites Souples Et De La Cooccurrence Dans La Segmentation Des Images Médicales, l'Université Chinoise Des Géosciences Et d'autres Ont Proposé Le Modèle De Segmentation d'images ConDSeg
La segmentation des images médicales est une étape critique et complexe dans le domaine du traitement des images médicales. Il extrait principalement des parties ayant des significations particulières à partir d'images médicales pour fournir un support au diagnostic clinique, au traitement de réadaptation et au suivi des maladies. Ces dernières années, avec le soutien des ordinateurs et de l'intelligence artificielle, les méthodes de segmentation basées sur l'apprentissage en profondeur sont progressivement devenues la méthode dominante de segmentation des images médicales, et ses résultats associés ont également prospéré.
Parmi les résultats sélectionnés annoncés lors de la 39e conférence annuelle de l'AAAI sur l'intelligence artificielle (AAAI 2025), la principale conférence internationale sur l'intelligence artificielle, certains articles ont une fois de plus montré les progrès fructueux de la segmentation automatisée des images médicales.L'un des résultats, « ConDSeg : un cadre de segmentation d'images médicales générales via l'amélioration des fonctionnalités pilotées par le contraste », publié conjointement par une équipe de l'Université chinoise des géosciences et de Baidu, a attiré une large attention.
Pour répondre aux deux défis majeurs des « limites douces » et des phénomènes de cooccurrence dans le domaine de la segmentation d'images médicales, les chercheurs ont proposé un cadre général appelé ConDSeg pour la segmentation d'images médicales basée sur le contraste. Ce cadre introduit de manière innovante la stratégie de formation au renforcement de la cohérence (CR), le module de découplage des informations sémantiques (Semantic Information Decoupling, SID), le module d'agrégation de caractéristiques pilotée par le contraste (Contrast-Driven Feature Aggregation, CDFA) et le décodeur sensible à la taille (Size-Aware Decoder, SA-Decoder), etc., pour améliorer encore la précision du modèle de segmentation d'images médicales.
Adresse du document :
https://arxiv.org/abs/2412.08345
Le projet open source « awesome-ai4s » rassemble plus de 200 interprétations d'articles AI4S et fournit des ensembles de données et des outils massifs :
https://github.com/hyperai/awesome-ai4s
La précision de la segmentation des images médicales est confrontée à deux défis majeurs
Au cours de la dernière décennie, l’essor de l’intelligence artificielle a contribué au développement rapide de la segmentation automatisée des images médicales, libérant les médecins et les chercheurs de tâches fastidieuses. Cependant, compte tenu de la complexité et du professionnalisme des images médicales, il reste encore un long chemin à parcourir pour parvenir à une segmentation d’image entièrement automatisée, et la précision est un défi important qui ne peut être ignoré, car une fois la précision perdue, l’automatisation est hors de question.
Du point de vue actuel,Les « limites souples » et les phénomènes de cooccurrence dans les images médicales sont les principaux problèmes qui entravent l’amélioration de la précision de la segmentation des images médicales.
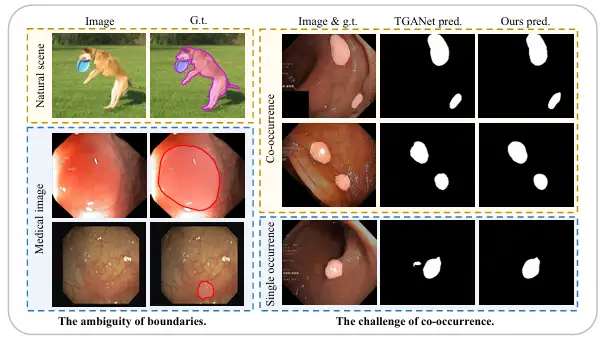
Premièrement, comparées aux images naturelles avec des limites claires entre le premier plan et l’arrière-plan, les images médicales ont souvent une « limite douce » floue entre le premier plan (comme les polypes, les glandes, les lésions, etc.) et l’arrière-plan. La principale raison en est qu’il existe une zone de transition entre le tissu pathologique et le tissu normal environnant, ce qui rend difficile la définition de la limite. De plus, dans la plupart des cas, les faibles effets d’éclairage et le faible contraste présentés par les images médicales brouillent encore davantage la frontière entre le tissu pathologique et le tissu normal, ce qui rend plus difficile la distinction des limites.
Deuxièmement, contrairement aux objets qui apparaissent de manière aléatoire dans les scènes naturelles, les organes et les tissus dans les images médicales sont très fixes et réguliers, il existe donc également un phénomène de cooccurrence répandu, c'est-à-dire que différentes caractéristiques d'image, tissus ou lésions apparaissent dans les images médicales en même temps. Par exemple, dans les images endoscopiques de polypes, les petits polypes apparaissent souvent avec des polypes de taille similaire, ce qui permet au modèle d'apprendre très facilement certaines caractéristiques de cooccurrence qui ne sont pas liées aux polypes. Cependant, lorsque le tissu pathologique apparaît seul, le modèle ne peut souvent pas faire de prédictions précises.
Afin de relever les défis susmentionnés, de plus en plus de méthodes de recherche se sont concentrées sur ce sujet ces dernières années. Par exemple, l'équipe du professeur associé Yue Guanghui de l'École de génie biomédical de l'École de médecine de l'Université de Shenzhen a publié un réseau de contraintes de limites BCNet qui peut être utilisé pour une segmentation précise des polypes. Il mentionne un module d'extraction de limites bilatérales qui peut capturer des limites en combinant des caractéristiques contextuelles peu profondes, des caractéristiques de position de haut niveau et une supervision supplémentaire des limites des polypes. Ce résultat a été publié dans l'IEEE Journal of Biomedical and Health Informatics sous le titre « Réseau de contraintes de limites avec intégration de fonctionnalités inter-couches pour la segmentation des polypes ».
Adresse du document :
https://ieeexplore.ieee.org/document/9772424
Par exemple, l'équipe du professeur Dinggang Shen, doyen fondateur de l'École d'ingénierie biomédicale de l'Université ShanghaiTech, et d'autres ont proposé un réseau d'agrégation de fonctionnalités à plusieurs niveaux, CFA-Net, qui peut être utilisé pour la segmentation des polypes. Il conçoit un réseau de prédiction de limites pour générer des fonctionnalités sensibles aux limites et utilise une stratégie hiérarchique pour fusionner ces fonctionnalités dans le réseau de segmentation. Ce résultat a été publié dans Pattern Recognition sous le titre « Cross-level Feature Aggregation Network for Polyp Segmentation ».
Adresse du document :
https://www.sciencedirect.com/science/article/abs/pii/S0031320323002558
Cependant, bien que toutes ces méthodes améliorent l’attention du modèle aux limites en introduisant explicitement une supervision liée aux limites, elles n’ont pas été en mesure d’améliorer fondamentalement la capacité du modèle à réduire spontanément l’incertitude dans les régions ambiguës. Par conséquent, dans des environnements difficiles, la robustesse de ces méthodes est encore faible et il existe encore des limites à l’amélioration des performances du modèle. Dans le même temps, l’incapacité à distinguer avec précision le premier plan et l’arrière-plan, ainsi qu’entre les différentes entités d’une image, reste un problème auquel sont confrontés la plupart des modèles.
Différent des méthodes précédentes,Dans une étude menée par une équipe de l'Université chinoise des géosciences et de Baidu, les chercheurs ont proposé un cadre général appelé ConDSeg pour la segmentation d'images médicales basée sur le contraste.Les innovations spécifiques sont les suivantes :
* En réponse au test de robustesse dans des environnements difficiles, les chercheurs ont proposé une stratégie de pré-formation de renforcement de cohérence (CR) pour améliorer la robustesse de l'encodeur et extraire des fonctionnalités de haute qualité. Dans le même temps, le module de découplage des informations sémantiques (SID) peut découpler les cartes de caractéristiques en régions de premier plan, d'arrière-plan et d'incertitude, et apprendre à réduire l'incertitude pendant la formation grâce à une fonction de perte spécialement conçue.
* Le module d'agrégation de caractéristiques piloté par le contraste (CDFA) proposé guide la fusion et l'amélioration des caractéristiques multicouches grâce aux caractéristiques de contraste extraites par SID. Le décodeur sensible à la taille (SA-Decoder) vise à mieux distinguer les différentes entités dans une image et à faire des prédictions distinctes pour les entités de différentes tailles afin de surmonter l'interférence des caractéristiques communes.
Les quatre innovations majeures de ConDSeg permettent d'améliorer la précision de la segmentation des images médicales
Dans l'ensemble,Le ConDSeg proposé dans cette étude est un cadre général de segmentation d’images médicales avec une architecture en deux étapes.Comme le montre la figure suivante :
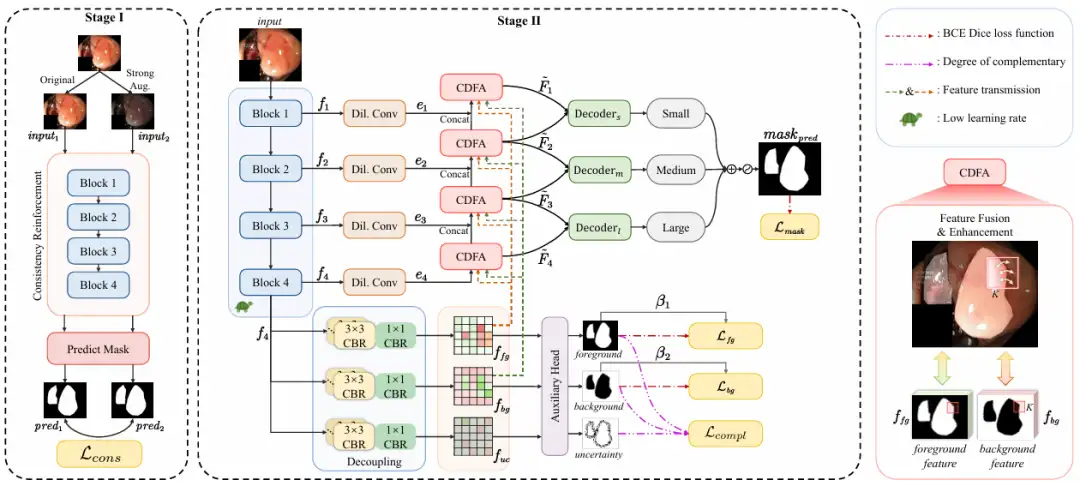
Dans la première étape,L’objectif de la recherche est de maximiser la capacité d’extraction de caractéristiques et la robustesse de l’encodeur dans les scènes à faible luminosité et à faible contraste.
Les chercheurs ont introduit la stratégie de pré-formation CR pour effectuer une formation préliminaire sur l'encodeur, ont séparé l'encodeur de l'ensemble du réseau et ont conçu une tête de prédiction simple (Predict Mask). En entrant l'image originale (Original) et l'image améliorée (Strong Aug.) dans l'encodeur, la cohérence entre les masques prédits est maximisée, la robustesse de l'encodeur sous différents défis d'éclairage et de contraste est améliorée et sa capacité à extraire des fonctionnalités de haute qualité dans des environnements difficiles est améliorée. Les méthodes d’amélioration incluent la modification aléatoire de la luminosité, du contraste, de la saturation, de la teinte, ainsi que la conversion aléatoire en images en niveaux de gris et l’ajout d’un flou gaussien.
Il convient également de mentionner que la perte de cohérence Lcons proposée par l’équipe de recherche est conçue sur la base d’une précision de classification au niveau des pixels. Il utilise des opérations de binarisation simples et un calcul de perte d'entropie croisée binaire (BCE) pour comparer directement les différences au niveau des pixels entre les masques prédits. Cette méthode est plus simple en termes de calcul et évite l’instabilité numérique, ce qui la rend plus adaptée aux données à grande échelle.
Dans la deuxième étape,L'ensemble du réseau est réglé avec précision et le taux d'apprentissage de l'encodeur est fixé à un niveau bas. Il est divisé en 4 étapes :
* Extraction de fonctionnalités, l'encodeur ResNet-50 extrait les cartes de fonctionnalités f₁ à f₄ avec différentes informations sémantiques à différents niveaux.
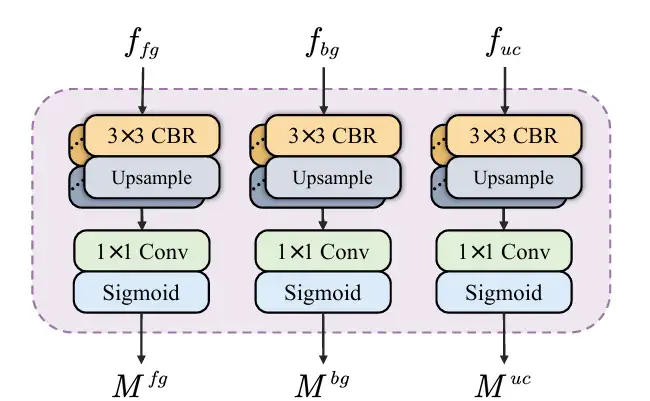
* Découplage des informations sémantiques : la carte de caractéristiques f₄ contenant des informations sémantiques profondes est entrée dans SID et découplée en une carte de caractéristiques contenant des informations de premier plan, d'arrière-plan et de zone incertaine. SID démarre avec trois branches parallèles, chacune composée de plusieurs modules CBR. Une fois la carte de caractéristiques f₄ entrée dans les trois branches, trois cartes de caractéristiques avec des informations sémantiques différentes sont obtenues, qui sont enrichies respectivement avec des caractéristiques de premier plan, d'arrière-plan et de zone incertaine. Ensuite, une tête auxiliaire prédit les trois cartes de caractéristiques et génère des masques pour le premier plan, l'arrière-plan et la zone incertaine. Grâce aux contraintes de la fonction de perte, l'apprentissage SID réduit l'incertitude et améliore la précision du masque entre le premier plan et l'arrière-plan. Comme le montre la figure suivante :
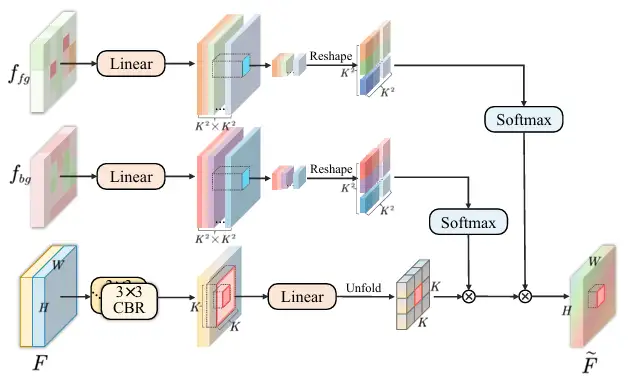
* Agrégation de fonctionnalités : les cartes de fonctionnalités f₁ à f₄ sont introduites dans le module CDFA, et les cartes de fonctionnalités à plusieurs niveaux sont progressivement fusionnées en fonction des cartes de fonctionnalités découplées pour améliorer la représentation des fonctionnalités de premier plan et d'arrière-plan. CDFA utilise non seulement les caractéristiques de contraste du premier plan et de l'arrière-plan découplées par SID pour guider la fusion de caractéristiques à plusieurs niveaux, mais aide également le modèle à mieux distinguer les entités à segmenter et l'environnement d'arrière-plan complexe. Comme le montre la figure suivante :
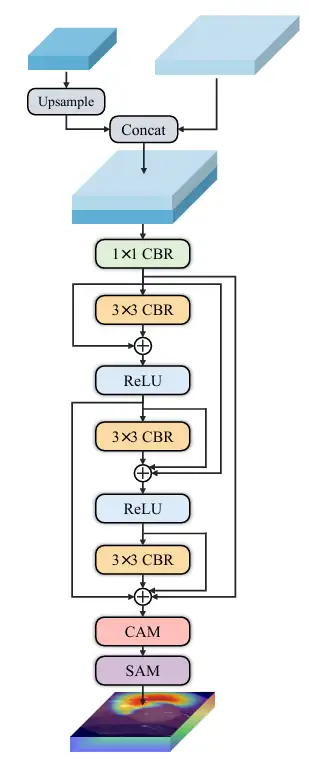
* Prédiction multi-échelle, les chercheurs ont établi trois décodeurs de petite, moyenne et grande taille - décodeur ₛ, décodeur ₘ et décodeur ₗ reçoivent respectivement la sortie de CDFA à un niveau spécifique, puis localisent plusieurs entités dans l'image en fonction de la taille. La sortie de chaque décodeur est fusionnée pour produire le masque final, de sorte que le modèle peut segmenter avec précision les grandes entités et localiser avec précision les petites entités, empêchant ainsi que les phénomènes de cooccurrence soient appris de manière incorrecte et résolvant le problème de singularité d'échelle du décodeur. Comme le montre la figure suivante :
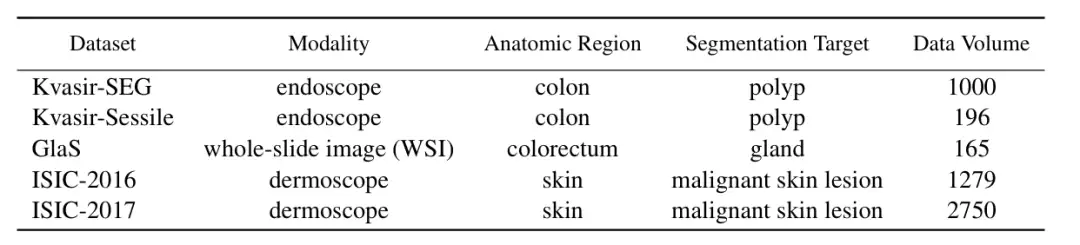
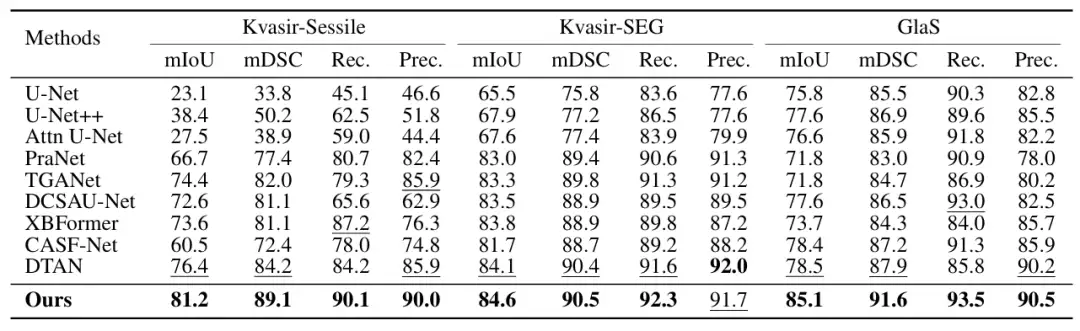
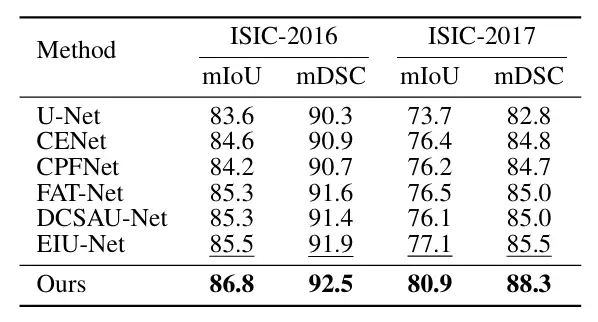
Afin de vérifier les performances de ConDSeg dans le domaine de la segmentation d'images médicales,Les chercheurs ont sélectionné cinq ensembles de données publiques (Kvasir-SEG, Kvasir-Sessile, GlaS, ISIC-2016, ISIC-2017, comme indiqué dans la figure ci-dessous) pour tester trois tâches d'imagerie médicale (endoscopie, images en coupe entière et dermatoscopie). Les chercheurs ont redimensionné les images à 256 × 256 pixels et ont défini la taille du lot à 4. L'optimiseur Adam a été utilisé pour l'optimisation.
Les principaux objets de comparaison incluent les méthodes les plus avancées telles que U-Net, U-Net++, Attn U-Net, CENet, CPFNet, PraNet, FATNet, TGANet, DCSAUNet, XBoundFormer, CASF-Net, EIU-Net et DTAN.Les résultats montrent que la méthode proposée atteint les meilleures performances de segmentation sur les cinq ensembles de données.Comme le montre la figure suivante :
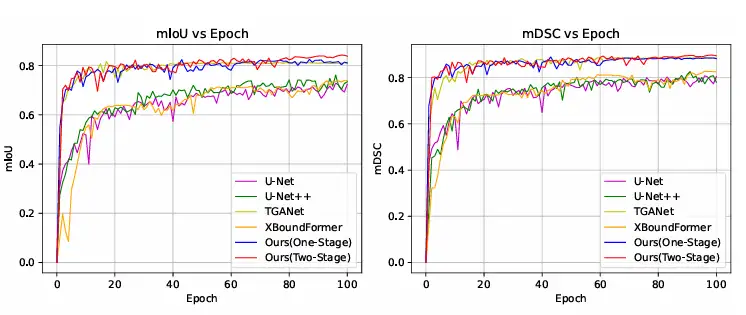
De plus, les chercheurs ont également comparé les courbes de convergence de formation avec d’autres méthodes sur l’ensemble de données Kvasir-SEG. Les résultats ont montré que ConDSeg peut atteindre des niveaux avancés même avec une seule étape de formation, et en utilisant le cadre ConDSeg complet, cette méthode a atteint la vitesse de convergence la plus rapide et les meilleures performances. Comme le montre la figure ci-dessous.
La segmentation des images médicales est devenue un sujet brûlant pour le capital et la technologie
La segmentation des images médicales joue un rôle important en médecine clinique et en recherche médicale. Les systèmes d’IA spécialement formés ont transformé les méthodes traditionnelles de segmentation d’images médicales grâce à leur grande efficacité et leur intelligence, ce qui en fait un outil auxiliaire indispensable pour le personnel médical et les chercheurs scientifiques. La raison pour laquelle la segmentation des images médicales a atteint un tel développement et de tels résultats est due au double moteur du capital et de la technologie.
En termes de capital, le domaine interdisciplinaire de l'IA et de la biomédecine est devenu un sujet brûlant dans la communauté des investisseurs ces dernières années, et cette année, l'imagerie médicale basée sur l'IA a pris les devants en réussissant un démarrage réussi. Le 28 janvier, la société espagnole d'imagerie médicale Quibim a annoncé avoir finalisé un financement de série A de 50 millions de dollars (environ 360 millions de RMB). Il convient de mentionner que la technologie principale de Quibim est l’analyse de l’intelligence artificielle basée sur des données d’imagerie médicale, et son QP-Liver est un outil de segmentation automatisé pour le diagnostic par IRM des maladies diffuses du foie.
En termes de technologie, la combinaison de l’IA et de la segmentation d’images médicales est depuis longtemps l’un des axes de recherche des grands laboratoires. Par exemple, une équipe du Laboratoire d'informatique et d'intelligence artificielle du Massachusetts Institute of Technology (MIT CSAIL), en collaboration avec des chercheurs du Massachusetts General Hospital et de la Harvard Medical School, a proposé un modèle général de segmentation interactive d'images biomédicales, ScribblePrompt, qui prend en charge les annotateurs utilisant différentes méthodes d'annotation telles que les graffitis, les clics et les cadres de délimitation pour effectuer de manière flexible des tâches de segmentation d'images biomédicales, même pour les étiquettes et les types d'images non formés.
Les résultats associés, intitulés « ScribblePrompt : segmentation interactive rapide et flexible pour toute image biomédicale », ont été acceptés par la principale conférence universitaire internationale ECCV 2024.
Adresse du document :
https://arxiv.org/pdf/2312.07381
De plus, sur la base de SAM 2 publié par Meta, l’équipe de l’Université d’Oxford a développé un modèle de segmentation d’images médicales appelé Medical SAM 2 (MedSAM-2), qui traite les images médicales comme des vidéos. Il fonctionne non seulement bien dans les tâches de segmentation d'images médicales 3D, mais débloque également une nouvelle capacité de segmentation à invite unique. L'utilisateur n'a qu'à fournir un indice pour un nouvel objet spécifique, et la segmentation des objets similaires dans les images suivantes peut être automatiquement complétée par le modèle sans autre saisie.
En bref, l’IA n’est plus une technologie haut de gamme. Le développement de la segmentation automatisée des images médicales a confirmé le potentiel de l’IA dans le domaine biomédical, et sa faisabilité commerciale a également été vérifiée à travers une histoire capitale après l’autre. À l’avenir, en tant que lien le plus important dans le domaine de l’imagerie médicale, la segmentation des images médicales bénéficiera sûrement de l’IA et suivra une voie de développement rapide. Des capitaux seront également introduits sur le marché biomédical plus large en raison du succès dans le domaine de la segmentation des images médicales, réalisant ainsi une boucle fermée parfaite de technologie, de capital et d'affaires.








