Command Palette
Search for a command to run...
VenusFactory Protein Engineering Design-Plattform
Datum
Größe
6.42 GB
Lizenz
Apache 2.0
GitHub
Paper-URL

1. Einführung in das Tutorial

VenusFactory wurde 2025 von einem gemeinsamen Team der Shanghai Jiao Tong University, des Shanghai Artificial Intelligence Laboratory und der East China University of Science and Technology entwickelt. Die zugehörigen Forschungsarbeiten sind folgende: VenusFactory: Eine einheitliche Plattform für den Datenabruf im Protein-Engineering und die Feinabstimmung von Sprachmodellen .
VenusFactory ist eine einheitliche Plattform, die speziell für die Protein-Engineering-Community entwickelt wurde und auf die Integration der biologischen Datenabfrage, des standardisierten Aufgaben-Benchmarkings und der modularen Feinabstimmung vortrainierter Proteinsprachenmodelle (PLMs) abzielt.
Die Plattform unterstützt die Ausführung über die Befehlszeile und eine codefreie Schnittstelle auf Gradio-Basis und integriert mehr als 40 proteinbezogene Datensätze und mehr als 40 gängige PLMs, sodass die Nutzung für Forscher in den Bereichen Informatik und Biologie einfach ist.
Das Tutorial bietet 7 Funktionsmodule:
- Training: Zero-Code-Modelltraining, unterstützt über 40 große Modelle und verwendet private Datensätze, um Ihre eigenen Modelle zu trainieren.
- Bewertung: Ein benutzerfreundliches Tool zur umfassenden Leistungsbewertung von Proteinmodellen.
- Vorhersage: Verwenden Sie das trainierte Modell, um die Funktion neuer Proteinsequenzen vorherzusagen.
- VenusAgent: Ein Protein-Engineering-Agent, der mit DeepSeek zusammenarbeitet, um die KI-Proteinberechnung zu ermöglichen.
- Quick Tools: Benutzerfreundliche Version, unterstützt die Vorhersage von Nullstichprobenmutationen (gerichtete Evolution) und die überwachte Vorhersage (Funktions- oder Eigenschaftsvorhersage).
- Erweiterte Tools: Erweiterte angepasste Version, die die Vorhersage von Nullprobenmutationen (gerichtete Evolution) und überwachte Vorhersage (Funktions- oder Eigenschaftsvorhersage) unterstützt.
- Download: Einfache Verknüpfung mit Proteindaten und Unterstützung von Multithread-Downloads aus wichtigen Datenbanken (RCSB, UniProt usw.).
Die in diesem Tutorial verwendeten Rechenressourcen sind eine einzelne RTX 4090-Karte. Das in diesem Tutorial verwendete Modell ist gespeichert in
/openbayes/input/input1Alle Daten werden im Verzeichnis gespeichert/openbayes/home/VenusFactoryVerzeichnis.
2. Bedienungsschritte
1. Starten Sie den Container
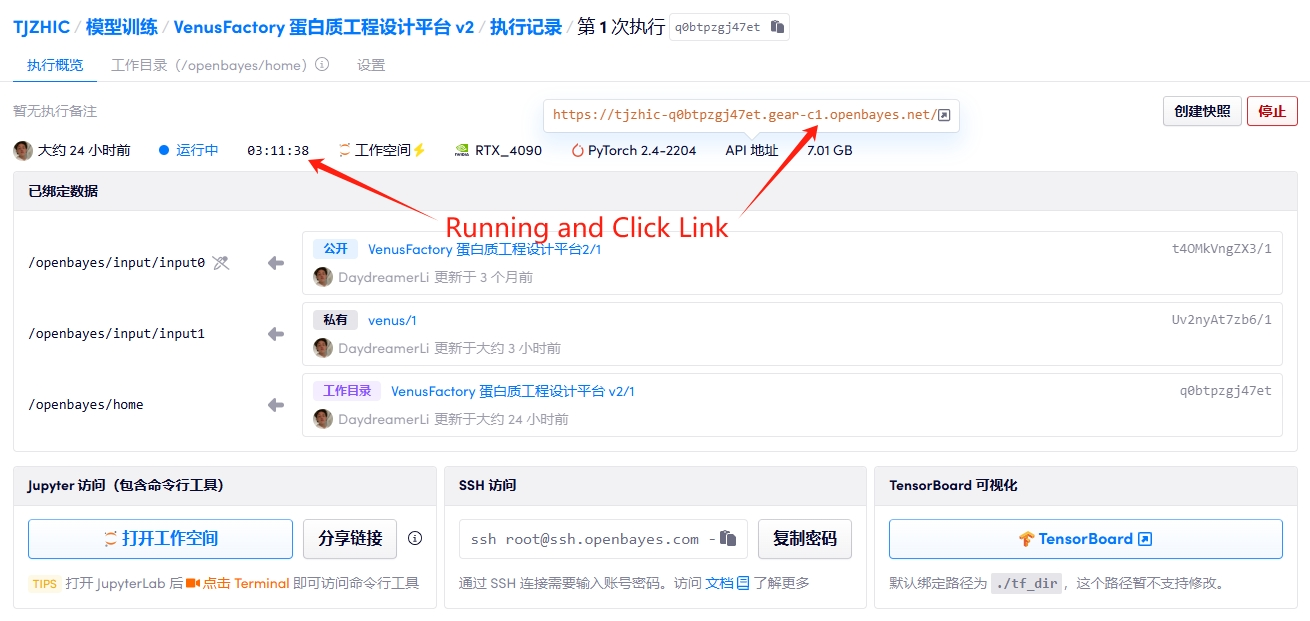
2. Handbuch
Wenn „Bad Gateway“ angezeigt wird, bedeutet dies, dass das Projekt initialisiert wird. Bitte warten Sie etwa 1–2 Minuten und aktualisieren Sie die Seite.
Das Benutzerhandbuch „Manuell“ von VenusFactory umfasst derzeit vier Module: Training, Auswertung, Vorhersage und Download.
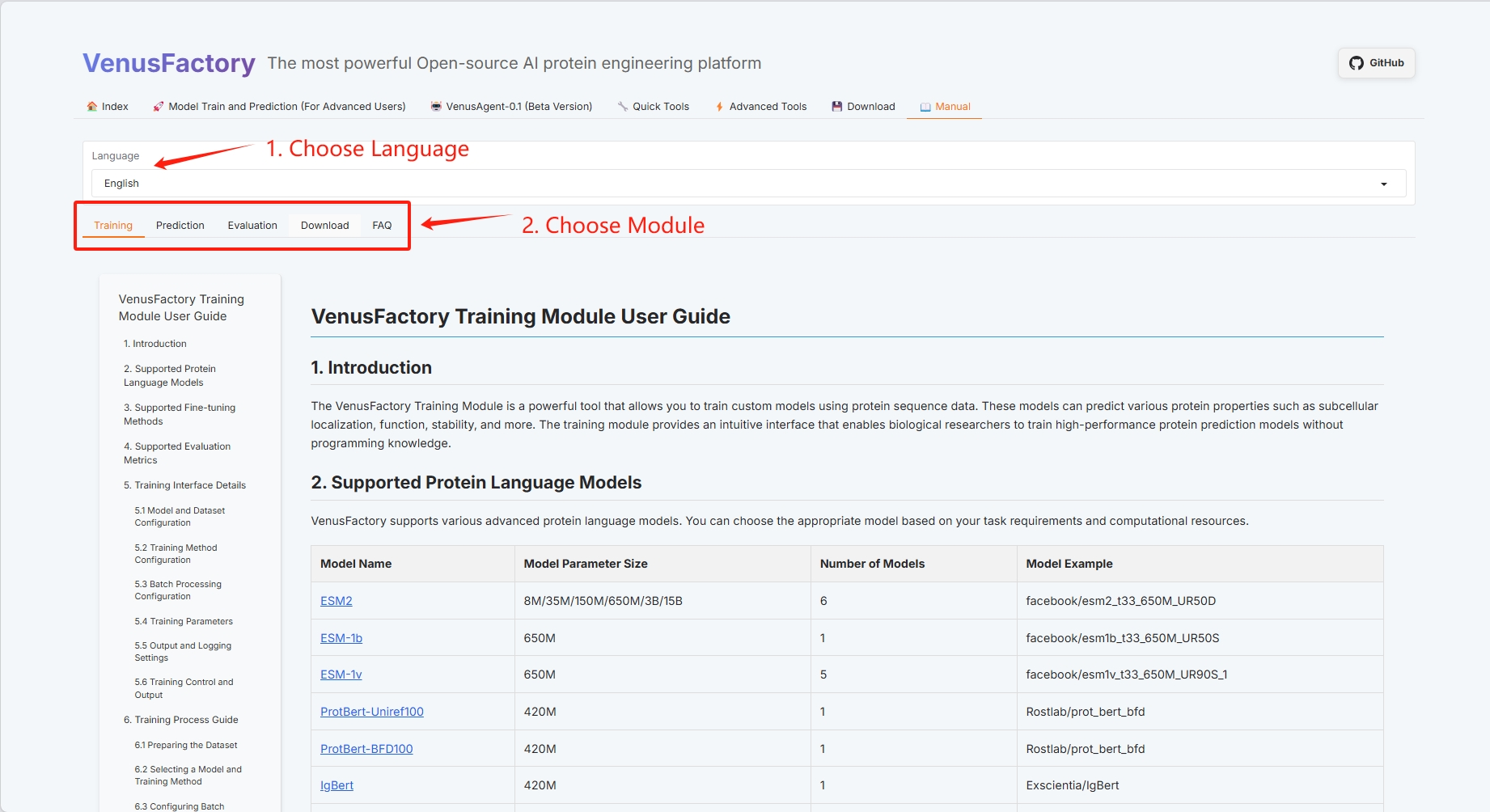
3. Spezifische Funktionsdemonstration
3.1 Schulung
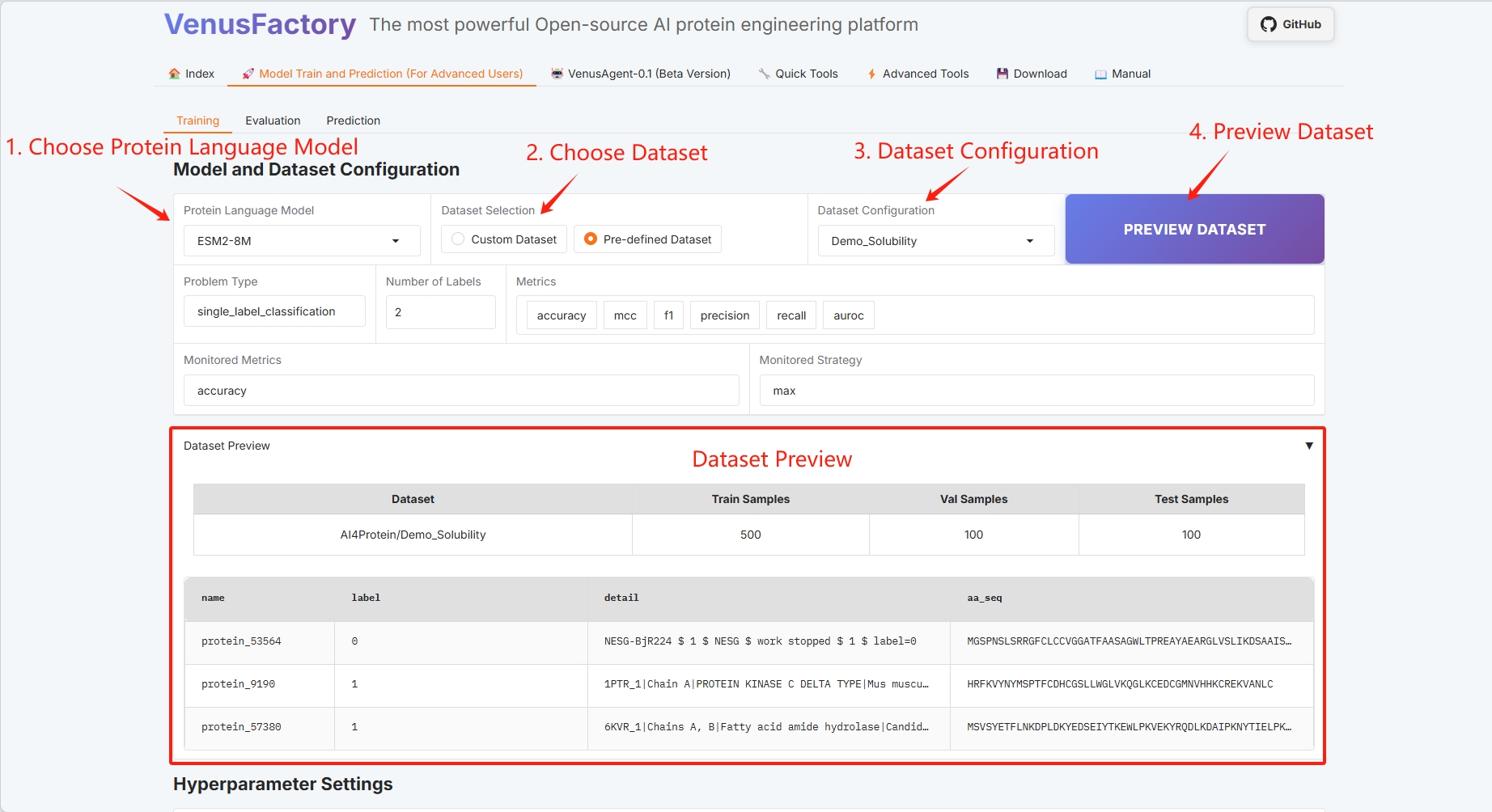
Klicken Sie im Modul „Modelltraining und Vorhersagetraining“ auf das Modul „Training“.
- Protein-Sprachmodell auswählen
- Datensatzauswahl
- Datensatzvorschau
- Konfiguration der Trainingsmethode (genaue Informationen finden Sie im Benutzerhandbuch)
- Batch-Konfiguration (weitere Einzelheiten finden Sie im Benutzerhandbuch)
Wenn die ausgewählten Modellparameter groß sind, ersetzen Sie bitte die Grafikkarte durch eine größere.
Legen Sie den Speicherpfad für das Trainingsmodell fest und klicken Sie auf „TRAINING STARTEN“, um mit dem Training zu beginnen.
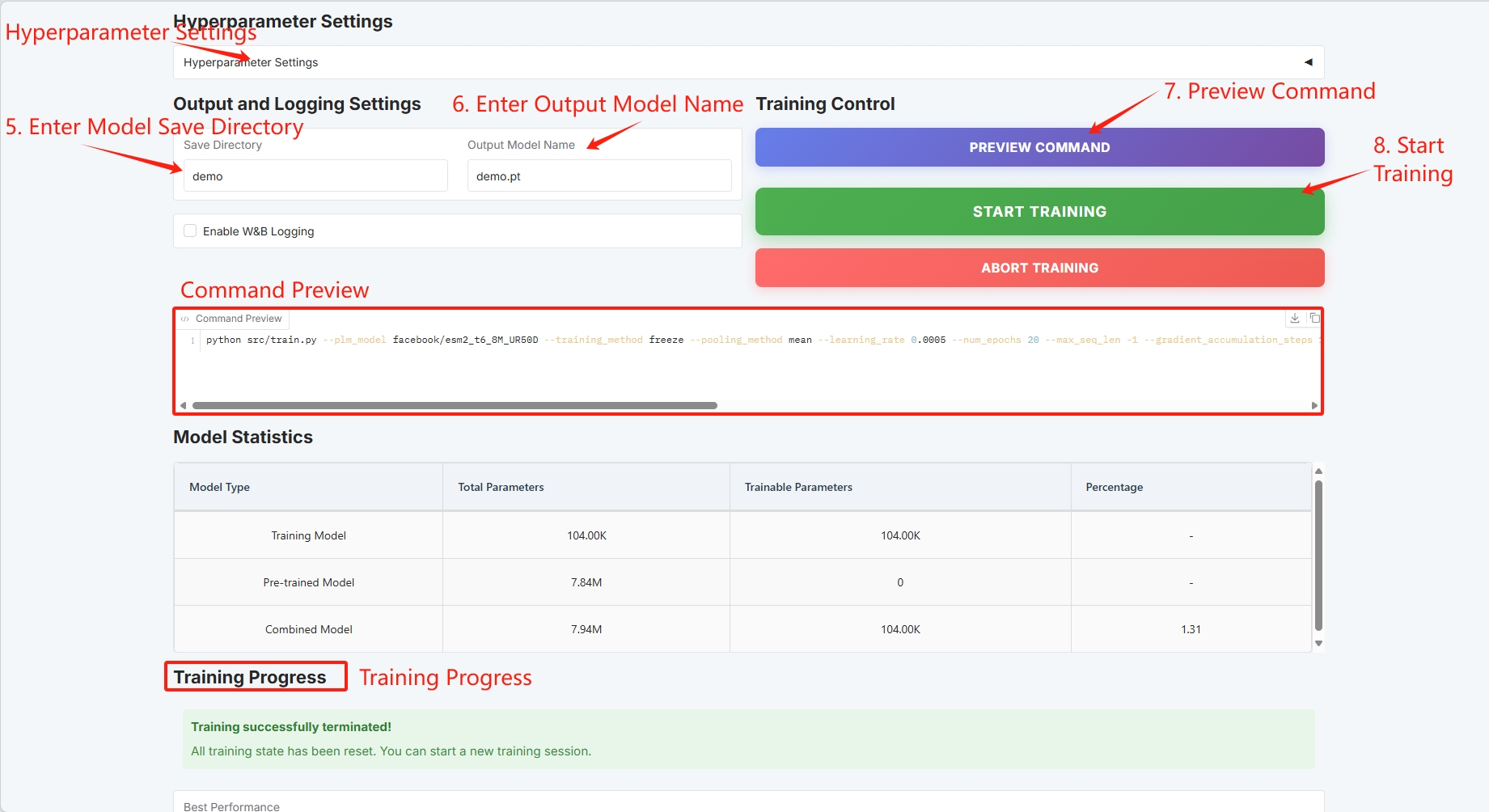
An dieser Stelle können Sie die Trainingsparameter und die Verlustkurve sehen
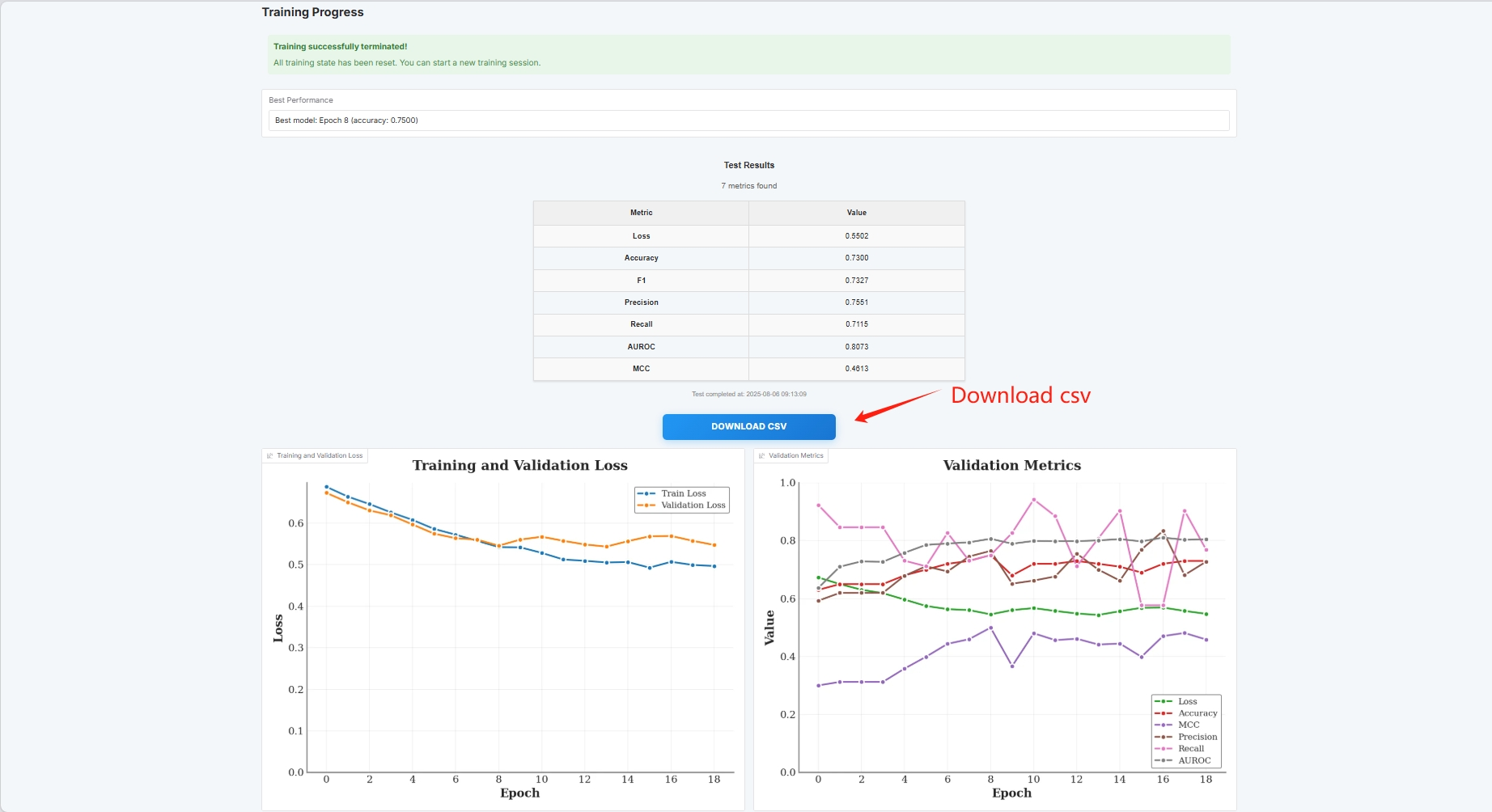
Wenn Sie Ihren eigenen Datensatz verwenden möchten, können Sie die benutzerdefinierte Datensatzkonfiguration verwenden. Geben Sie einfach den Pfad Ihres Datensatzes ein (weitere Informationen finden Sie in der Handbuchdokumentation).
3.2 Auswertung
Klicken Sie im Modul „Modelltraining und Vorhersagetraining“ auf das Modul „Auswertung“
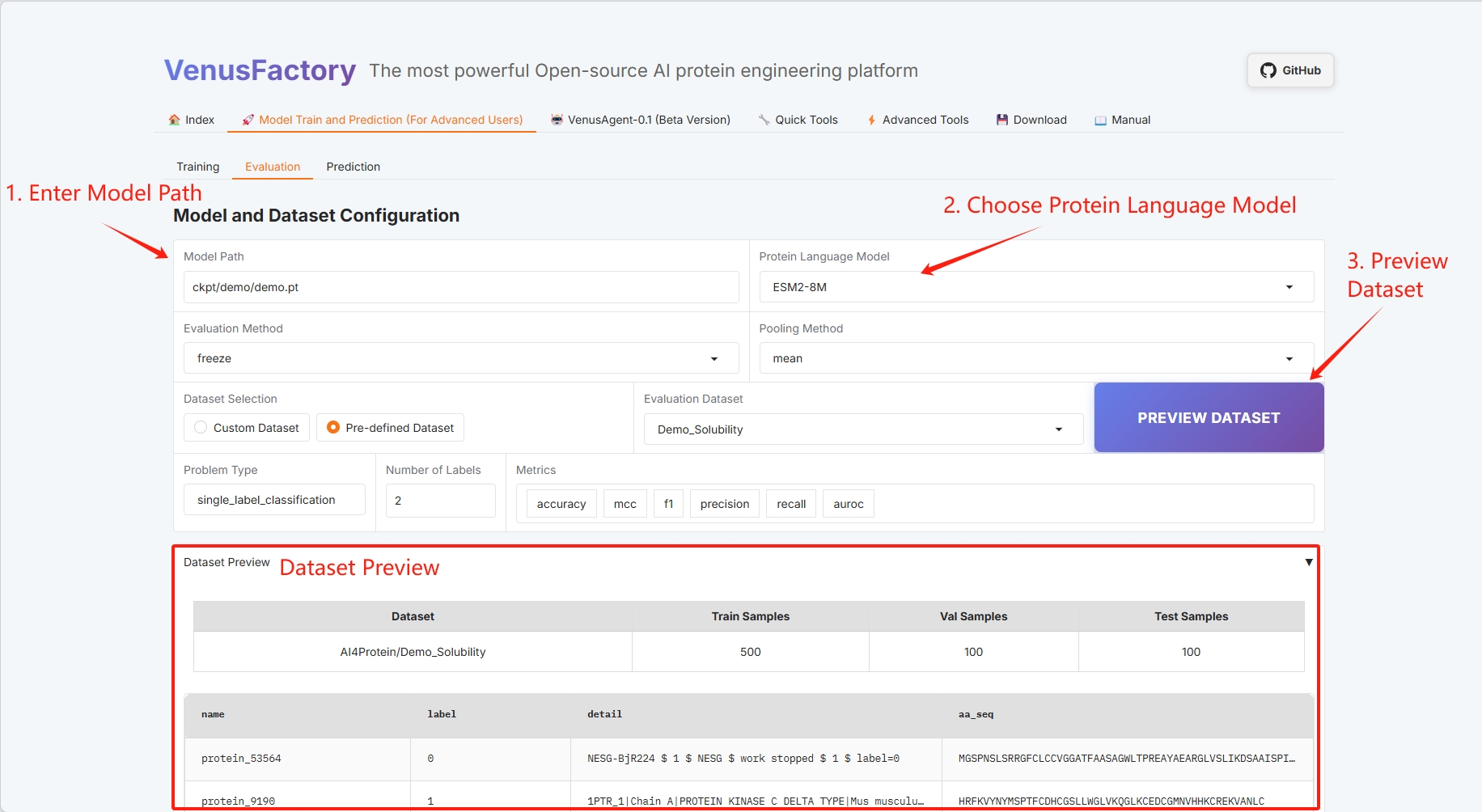
- Modellpfad und Auswahl des Proteinsprachenmodells
- Bewertungsmethode und Pooling-Methode (genaue Informationen finden Sie im Benutzerhandbuch)
- Datensatzauswahl
- Datensatzvorschau
- Fragetypen und Tags (weitere Informationen finden Sie im Benutzerhandbuch)
- Batch-Konfiguration (weitere Einzelheiten finden Sie im Benutzerhandbuch)
Legen Sie den Pfad zum Speichern des trainierten Modells fest und wählen Sie das Proteinsprachenmodell aus.
Batch-Konfiguration, klicken Sie auf „BEWERTUNG STARTEN“, um mit dem Training zu beginnen.
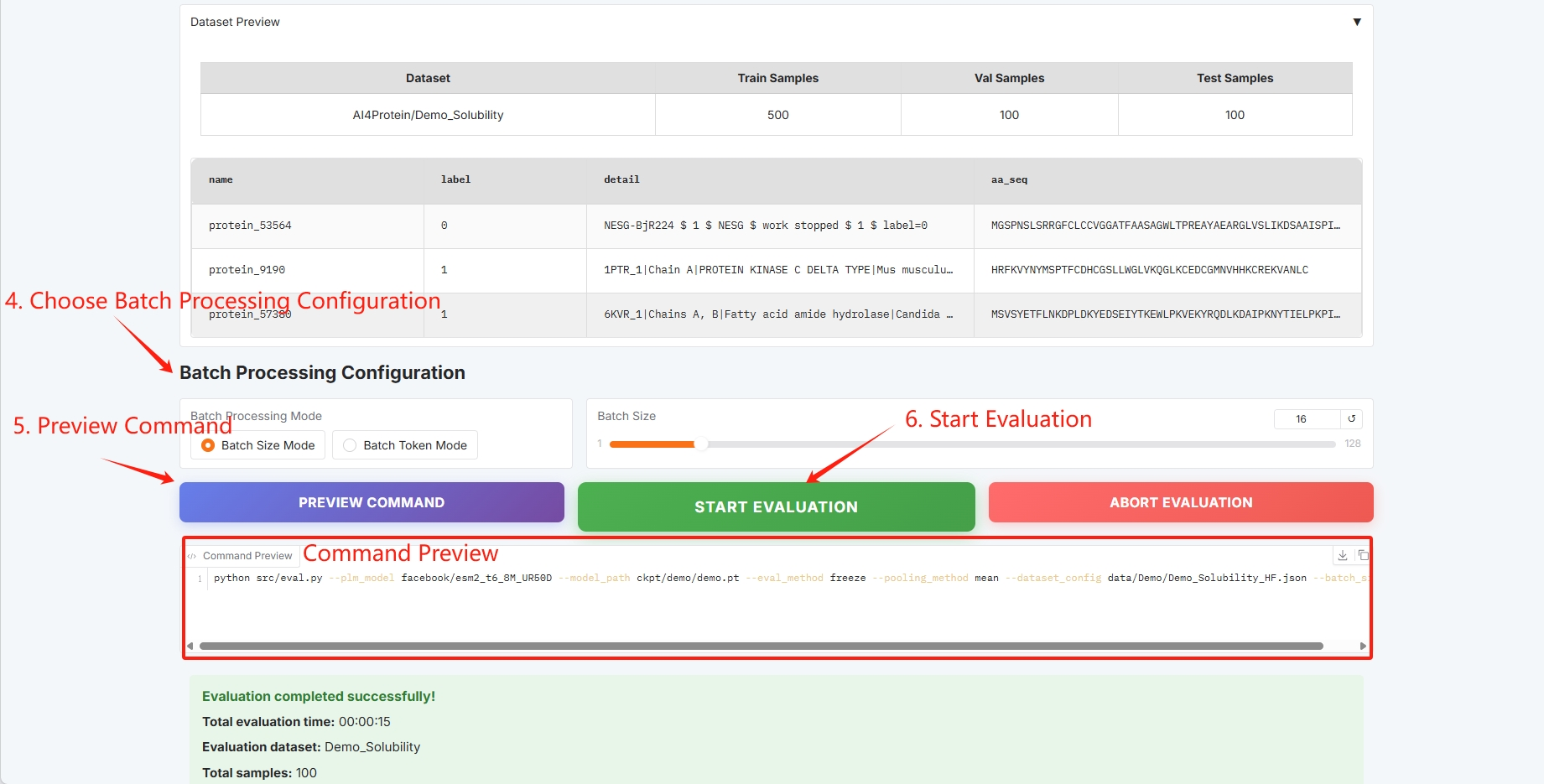
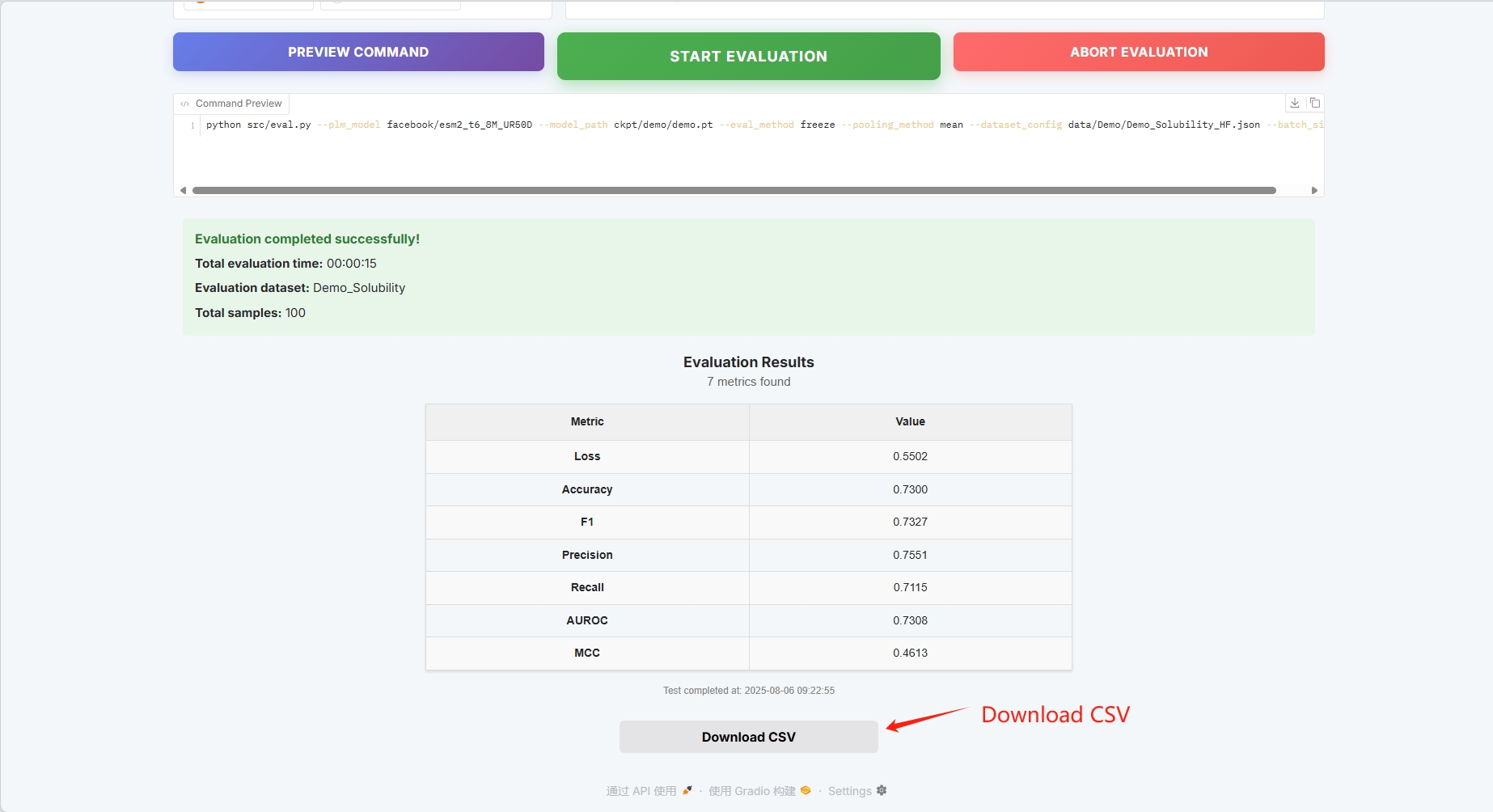
Die Auswertungsergebnisse sind wie folgt und können als CSV heruntergeladen werden
Wenn Sie Ihren eigenen Datensatz verwenden möchten, können Sie die benutzerdefinierte Datensatzkonfiguration verwenden. Geben Sie einfach den Pfad Ihres Datensatzes ein (weitere Informationen finden Sie in der Handbuchdokumentation).
3.3 Vorhersage
Klicken Sie im Modul „Modelltraining und Vorhersagetraining“ auf das Modul „Vorhersage“, um eine Einzelsequenzvorhersage und eine Stapelvorhersage durchzuführen.
- Modellkonfiguration
- Wählen Sie das Vorhersagemodul aus (weitere Informationen finden Sie im Benutzerhandbuch).
Legen Sie den Speicherpfad für das Trainingsmodell fest, wählen Sie das Proteinsprachenmodell aus und klicken Sie auf „VORHERSAGE STARTEN“, um mit dem Training zu beginnen.
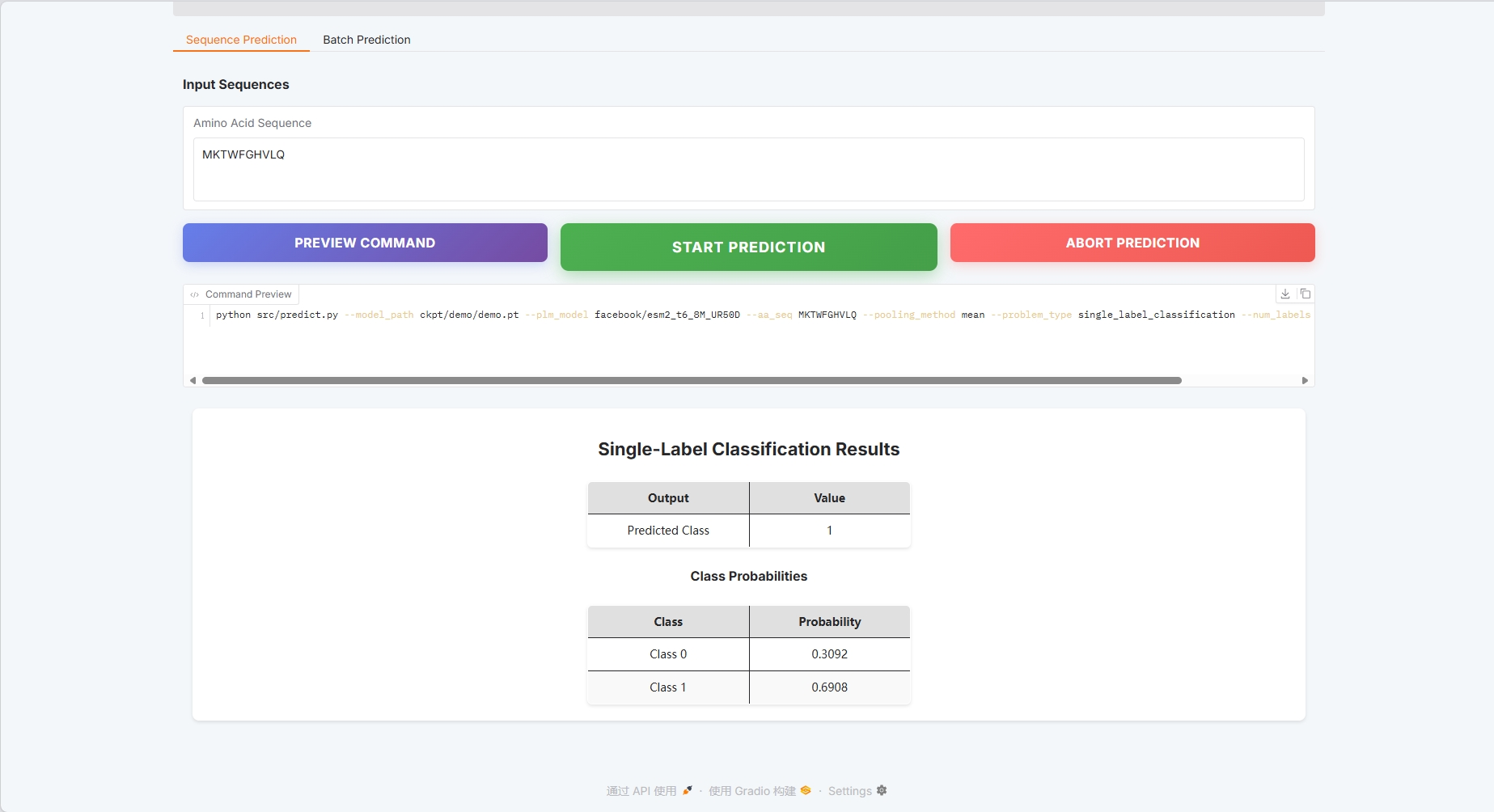
Einzelsequenzvorhersage
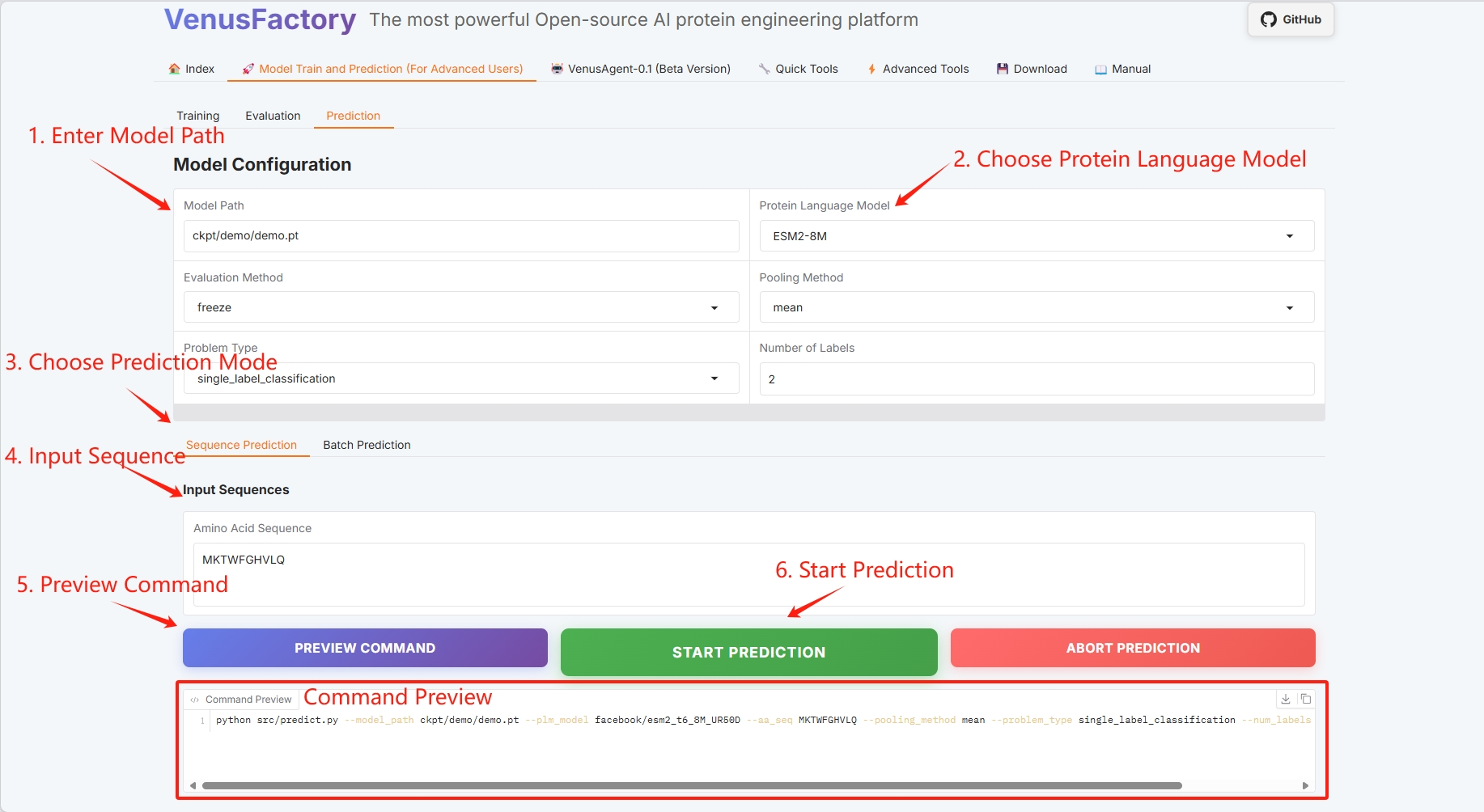
Beispiel einer Proteinsequenz: MKTWFGHVLQ
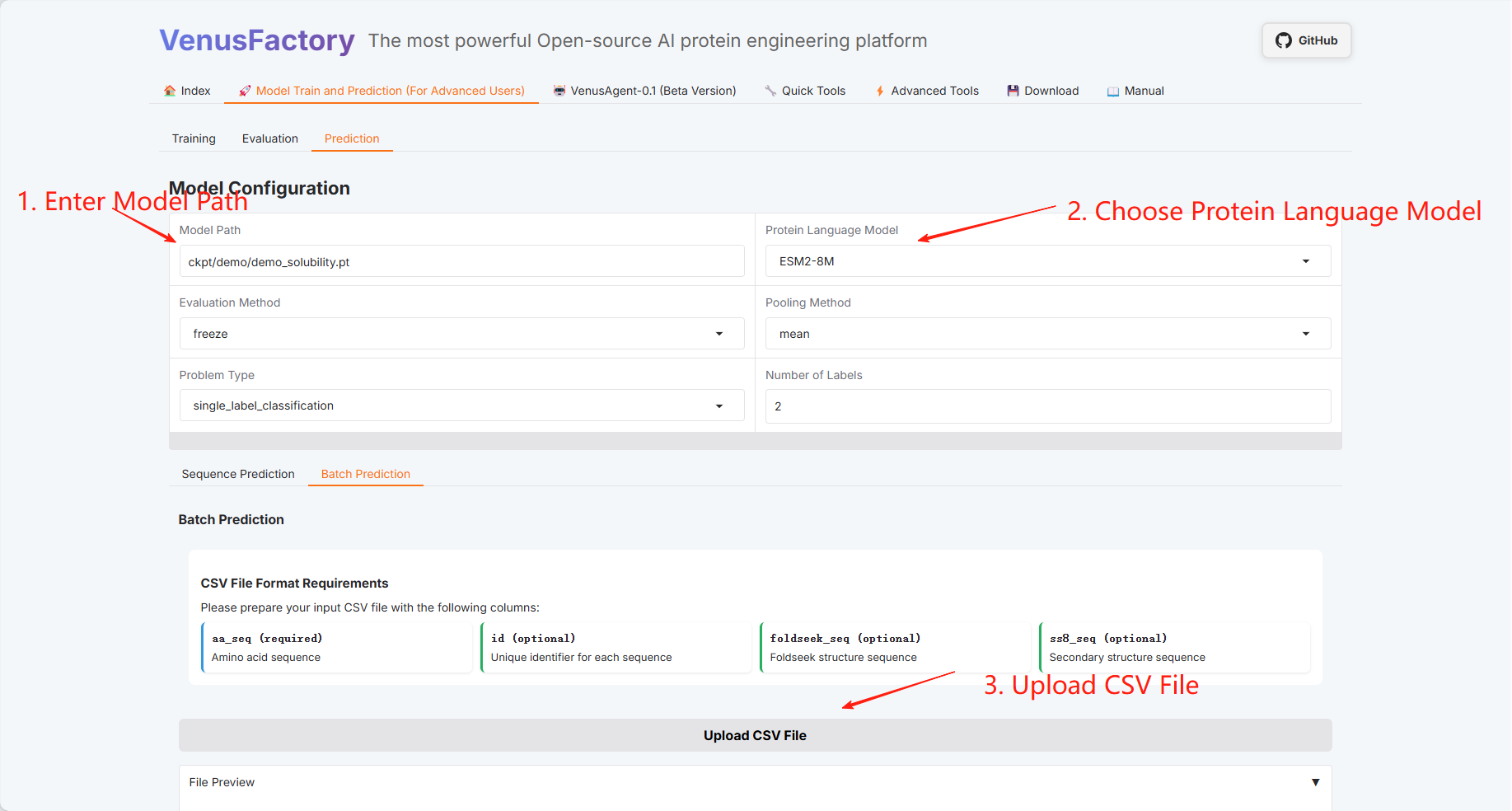
Batch-Vorhersage
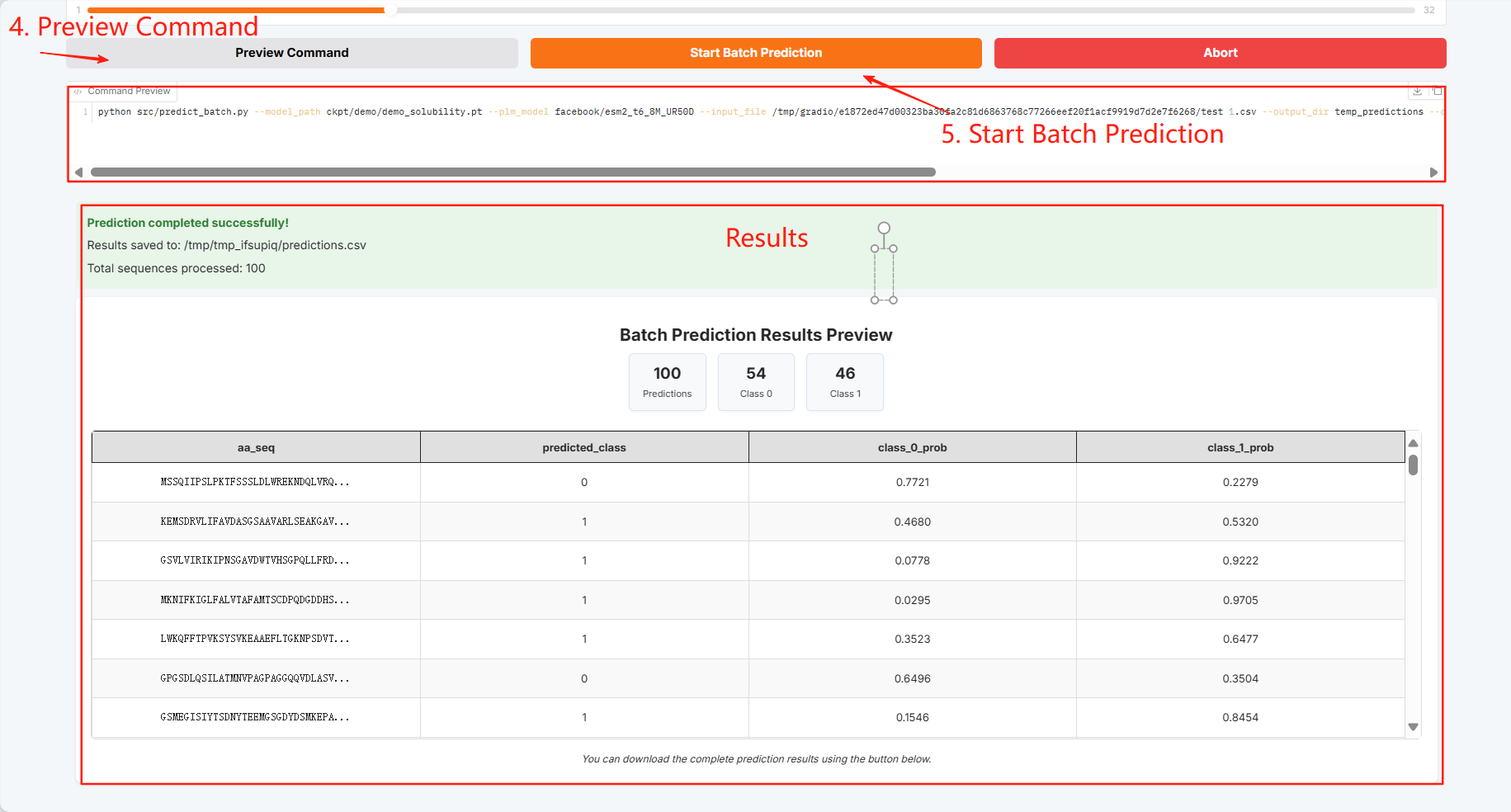
Batch-Vorhersageergebnisse können heruntergeladen und gespeichert werden
3.4 VenusAgent
Klicken Sie auf das Modul "VenusAgent"
Da VenusAgent das große DeepSeek-Modell aufrufen muss, bietet dieses Tutorial zwei Aufrufmethoden: Geben Sie den API-Schlüssel selbst ein oder verwenden Sie das auf der Plattform bereitgestellte Modell DeepSeek-R1-70B.
Sie können je nach den benötigten Funktionen unterschiedliche Grafikkarten auswählen. Die Anweisungen zur Kartenauswahl lauten wie folgt:
Bei Verwendung einer einzelnen RTX 4090-Grafikkarte unterstützt die VenusAgent-Funktion nicht die Verwendung lokal bereitgestellter großer Modelldienste (die Verwendung des DeepSeek-API-Schlüssels ist unbegrenzt).
Wenn Sie zwei RTX 4090-Grafikkarten verwenden, können Sie nach der Verwendung der VenusAgent-Funktion nicht sofort (nach 1–2 Minuten) andere Funktionen verwenden (bei Verwendung des DeepSeek-API-Schlüssels gibt es keine Einschränkung).
Wenn Sie zwei RTX A6000-Grafikkarten verwenden, sind die VenusAgent-Funktionen unbegrenzt.
Die in diesem Tutorial verwendeten Rechenressourcen sind eine einzelne RTX 4090-Karte. Das in diesem Tutorial verwendete Modell ist gespeichert in
/openbayes/input/input1Alle Daten werden im Verzeichnis gespeichert/openbayes/home/VenusFactoryVerzeichnis.
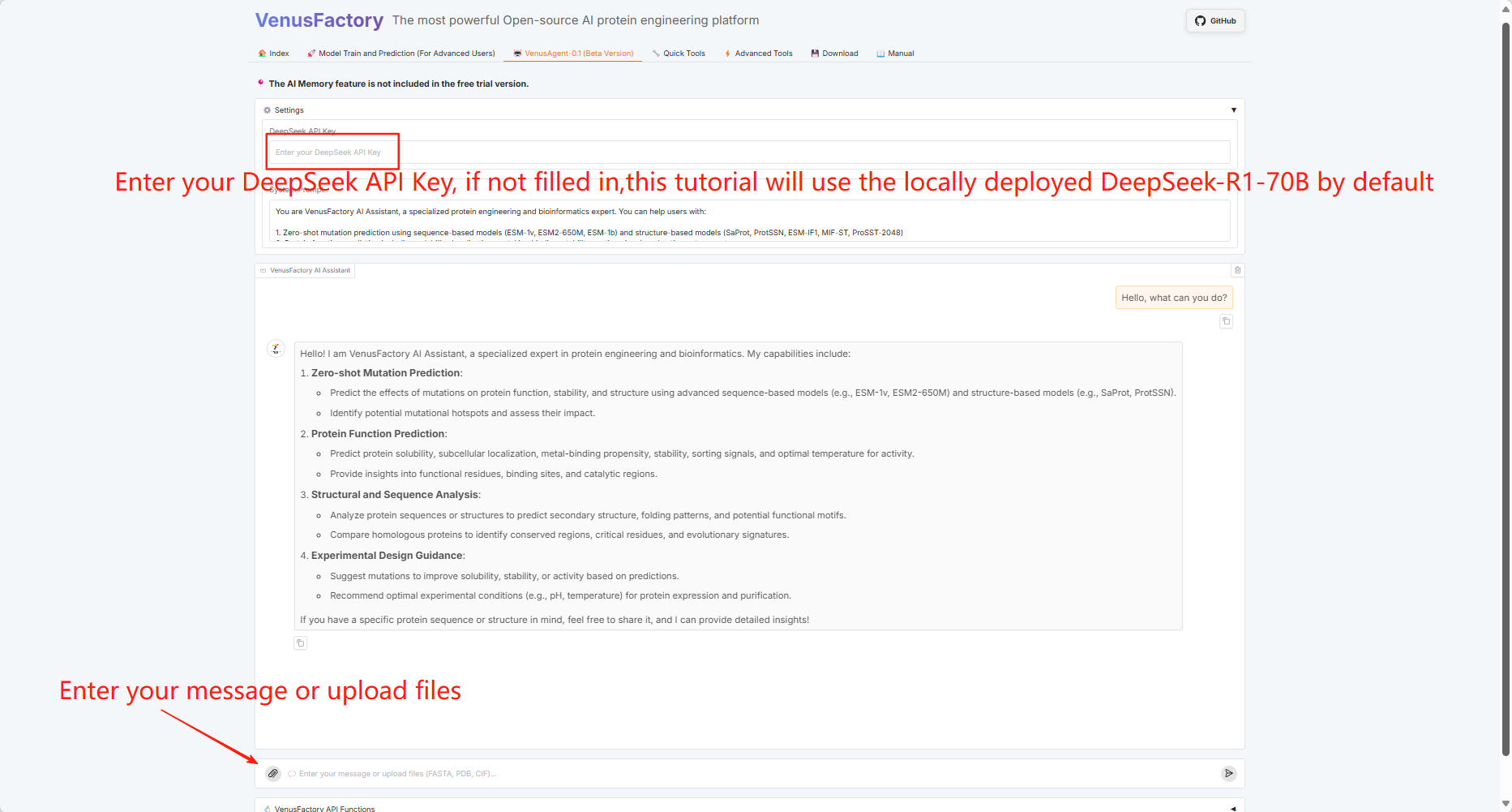
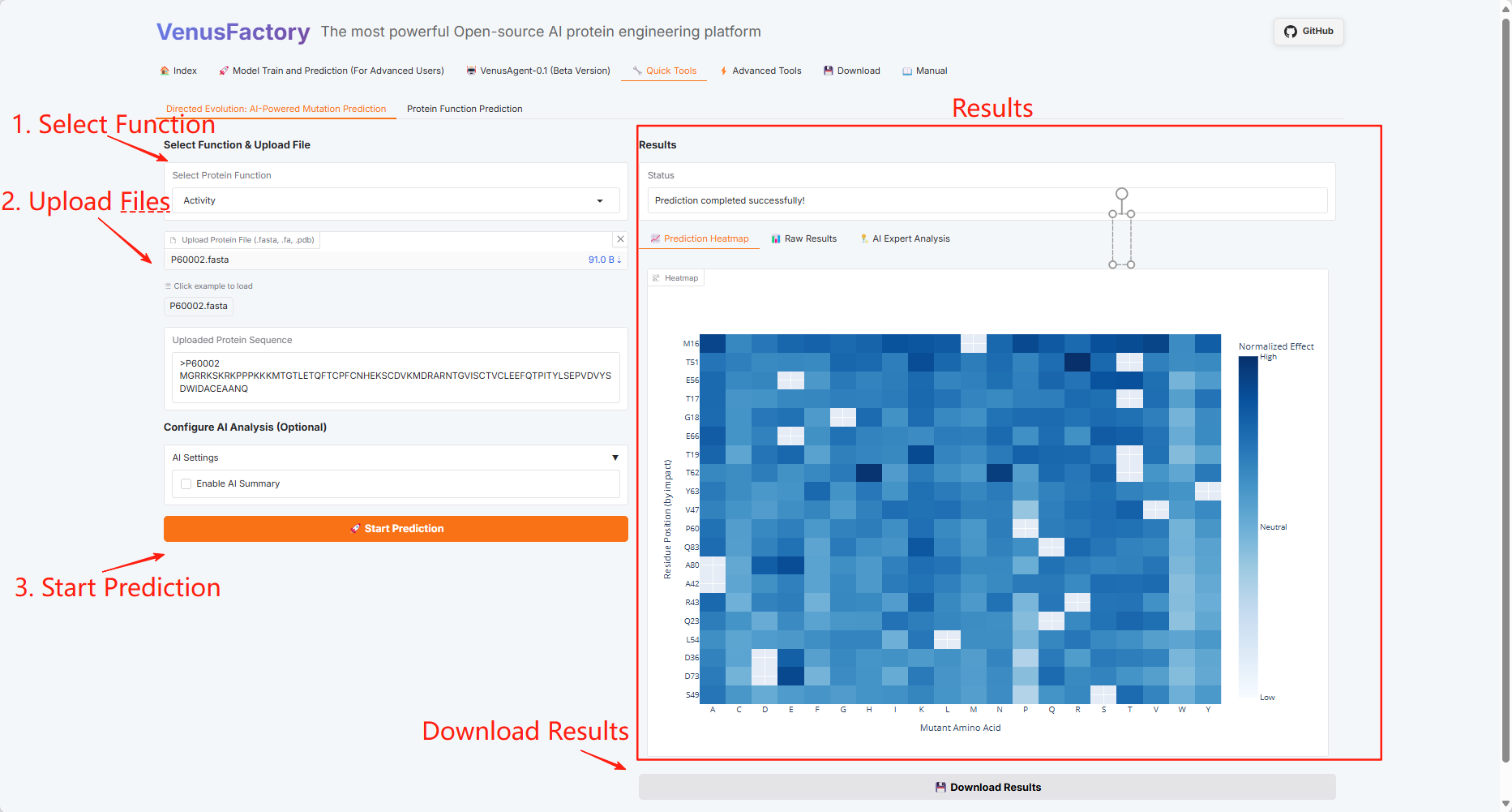
3.5 Schnellwerkzeuge
Klicken Sie auf das Modul „Quick Tools“, das zwei Funktionen enthält: Gerichtete Evolution: KI-gestützte Mutationsvorhersage und Proteinfunktionsvorhersage.
Gerichtete Evolution: KI-gestützte Mutationsvorhersage
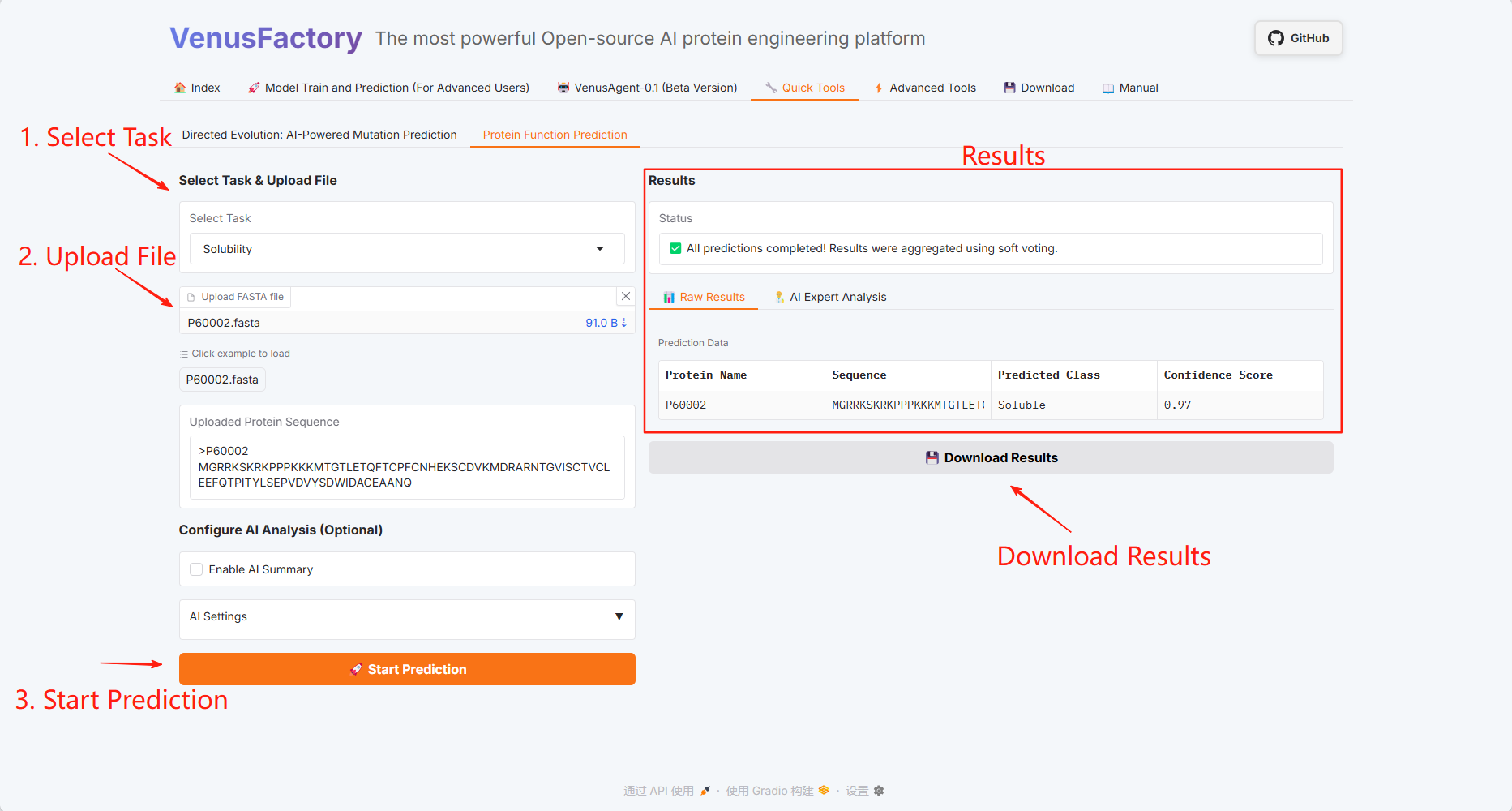
Vorhersage der Proteinfunktion
3.6 Erweiterte Tools
Klicken Sie auf das Modul „Erweiterte Tools“, das zwei Funktionen enthält: Gerichtete Evolution: KI-gestützte Mutationsvorhersage und Proteinfunktionsvorhersage.
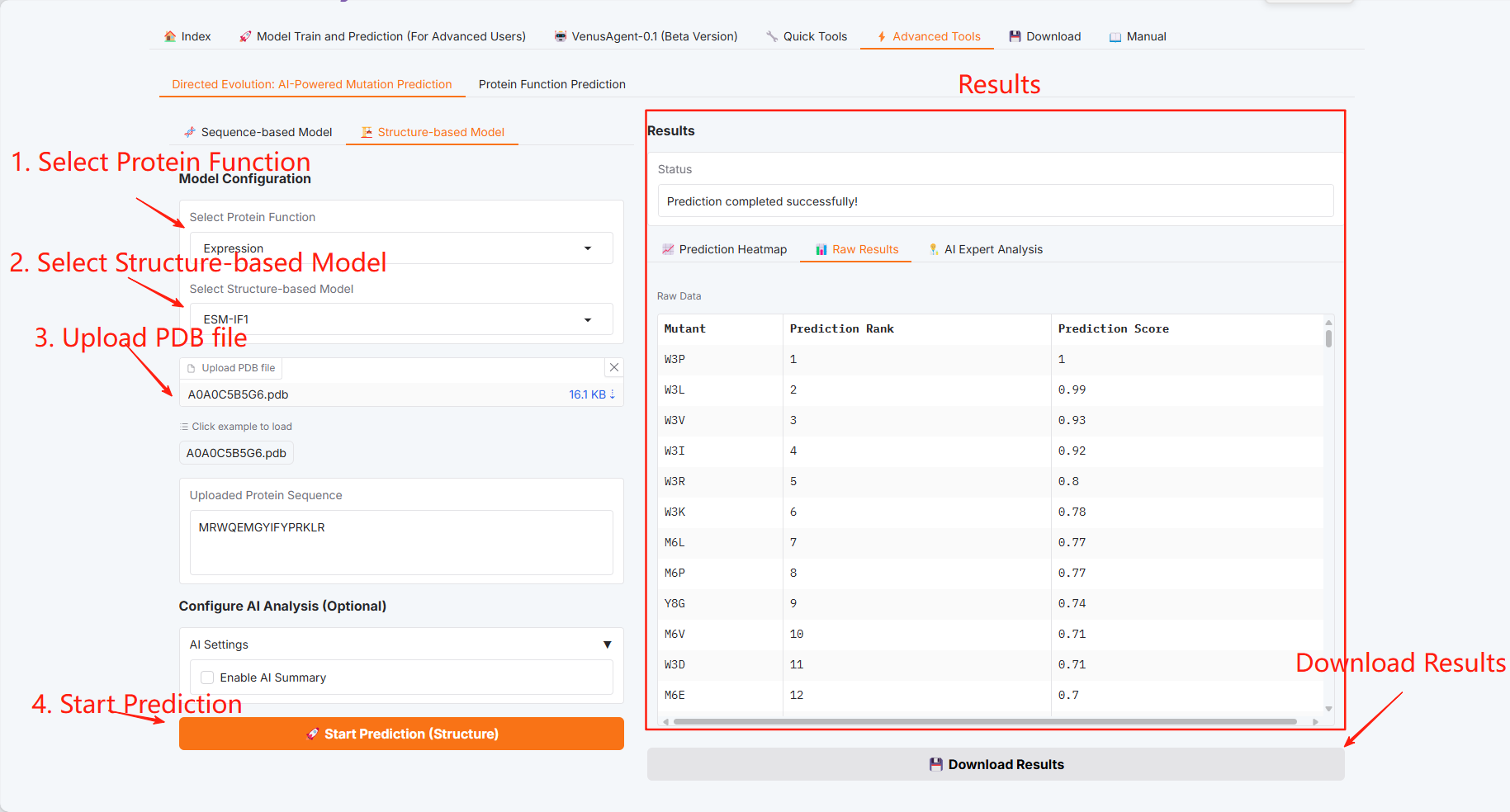
Gerichtete Evolution: KI-gestützte Mutationsvorhersage
Sequenzbasiertes Modell
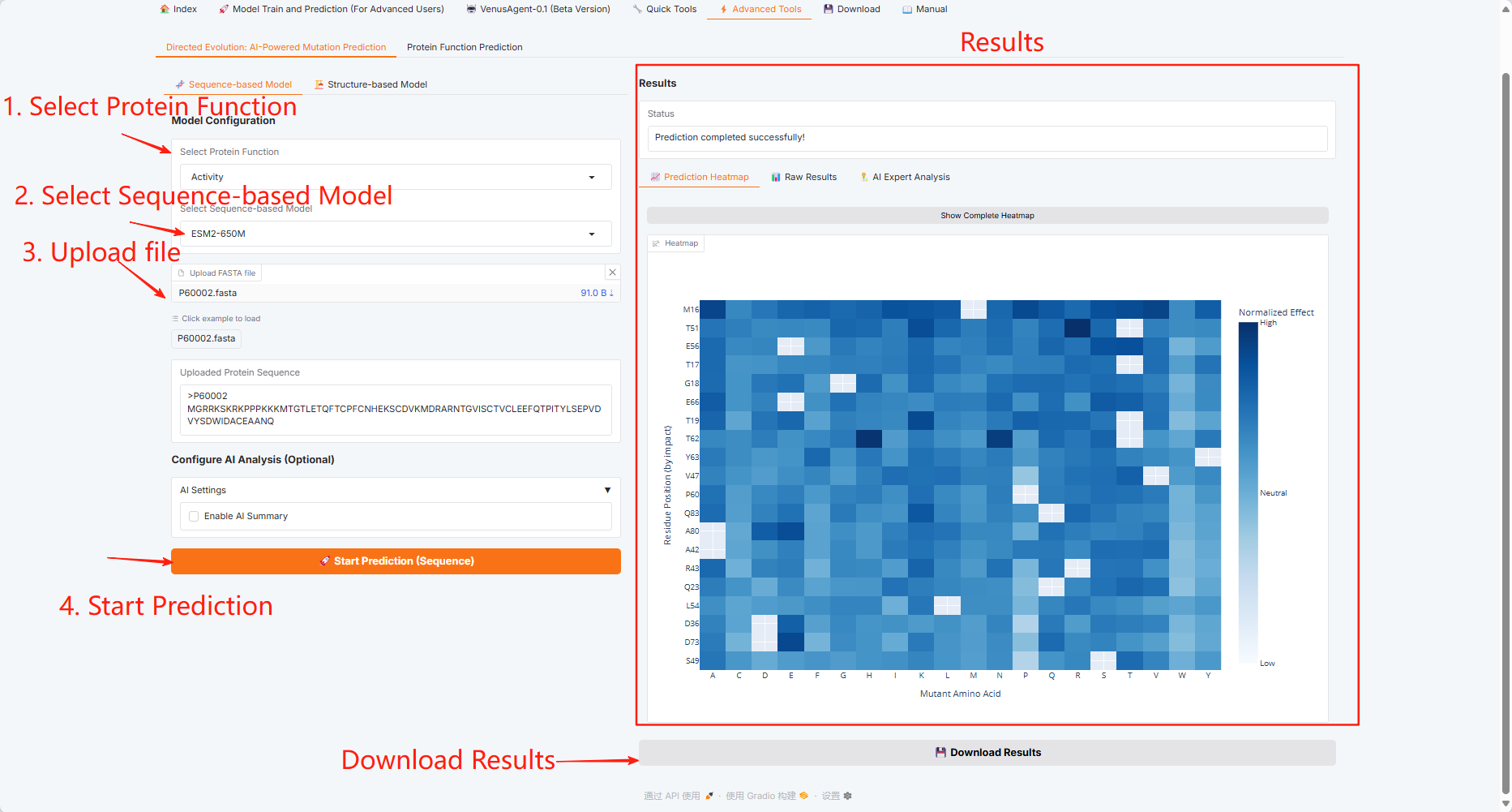
Strukturbasiertes Modell
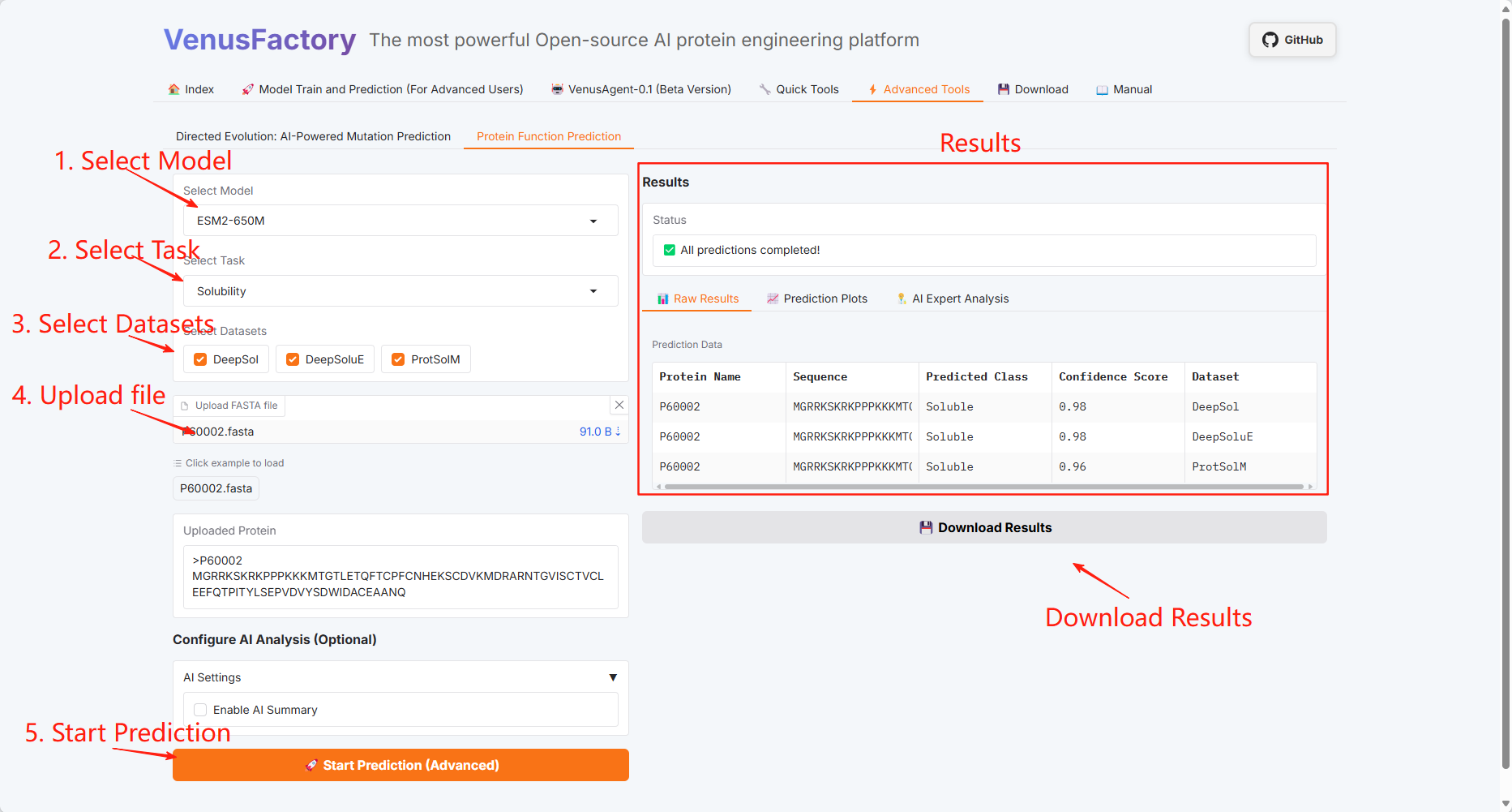
Vorhersage der Proteinfunktion
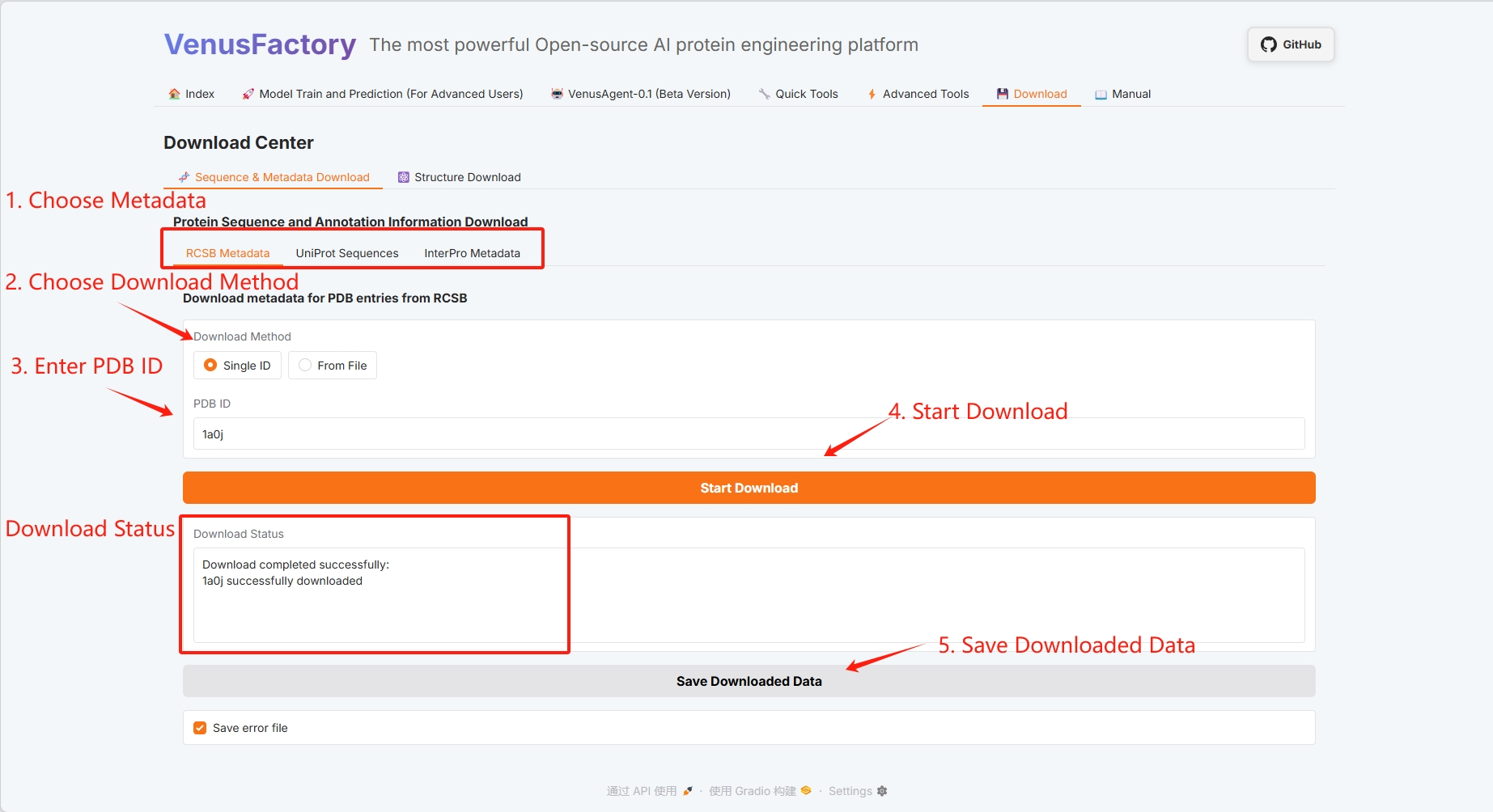
3.7 Herunterladen
Klicken Sie auf das Download-Modul, um Proteindaten in dieser Schnittstelle herunterzuladen.
3. Diskussion
🖌️ Wenn Sie ein hochwertiges Projekt sehen, hinterlassen Sie bitte im Hintergrund eine Nachricht, um es weiterzuempfehlen! Darüber hinaus haben wir auch eine AI4S-Austauschgruppe gegründet. Willkommen, Freunde, scannen Sie den QR-Code und kommentieren Sie [AI4S], um der Gruppe beizutreten, verschiedene technische Probleme zu besprechen und Anwendungsergebnisse auszutauschen ↓

Zitationsinformationen
Die Zitationsinformationen für dieses Projekt lauten wie folgt:
@inproceedings{tan-etal-2025-venusfactory,
title = "{V}enus{F}actory: An Integrated System for Protein Engineering with Data Retrieval and Language Model Fine-Tuning",
author = "Tan, Yang and Liu, Chen and Gao, Jingyuan and Wu, Banghao and Li, Mingchen and Wang, Ruilin and Zhang, Lingrong and Yu, Huiqun and Fan, Guisheng and Hong, Liang and Zhou, Bingxin",
editor = "Mishra, Pushkar and Muresan, Smaranda and Yu, Tao",
booktitle = "Proceedings of the 63rd Annual Meeting of the Association for Computational Linguistics (Volume 3: System Demonstrations)",
month = jul,
year = "2025",
address = "Vienna, Austria",
publisher = "Association for Computational Linguistics",
url = "https://aclanthology.org/2025.acl-demo.23/",
doi = "10.18653/v1/2025.acl-demo.23",
pages = "230--241",
ISBN = "979-8-89176-253-4",
}KI mit KI entwickeln
Von der Idee bis zum Launch – beschleunigen Sie Ihre KI-Entwicklung mit kostenlosem KI-Co-Coding, sofort einsatzbereiter Umgebung und bestem GPU-Preis.