Command Palette
Search for a command to run...
Online-Tutorial | Die Kosten Für Rechenleistung Sinken Drastisch! Apple Bringt Ml-simplefold Auf Den Markt, Ein Proteinfaltungsmodell Basierend Auf Stream Matching.

Im September 2025 brachte Apple das leichtgewichtige KI-Modell Ml-simplefold zur Vorhersage der Proteinfaltung auf den Markt. Als erstes Proteinfaltungsmodell auf Basis von Flow Matching wurde es in maßgeblichen Benchmarks wie CAMEO22 und CASP14 getestet.Nach der Reduzierung des Rechenaufwands zeigt SimpleFold immer noch eine Leistung, die mit Topmodellen wie AlphaFold2 und RoseTTAFold2 vergleichbar ist.Gleichzeitig weist auch die kleinere Version, SimpleFold-100M, eine konkurrenzfähige Leistung auf.
Traditionelle Proteinfaltungsmodelle bieten zwar eine beeindruckende Genauigkeit, basieren jedoch auf einer Vielzahl domänenspezifischer Architekturentwürfe und handgefertigter Funktionen. Die häufig verwendeten dreieckigen Update-Module, expliziten Paardarstellungsmechanismen und vielfältigen Trainingsziele dieser Modelle machen das Training rechenintensiv. Darüber hinaus sind Architektur und Hardware nicht skalierbar, was die Generierung vielfältiger Strukturen oder die Ensemble-Vorhersage erschwert.
Mit dem Ziel, die üblichen Mängel traditioneller Methoden zu beheben,SimpleFold schlägt ein allgemeines generatives Framework vor, das ausschließlich auf Transformer basiert und die Abhängigkeit von Proteinfaltungsmodellen von komplexen Architekturen aufhebt:
* Basierend auf der Flow-Matching-Technologie überspringt es komplexe Module wie die Mehrfachsequenz-Ausrichtung (MSA) und generiert die dreidimensionale Struktur von Proteinen direkt aus zufälligem Rauschen, wodurch der Rechenaufwand erheblich reduziert wird;
* Übernahme einer allgemeinen Architektur, die auf spezifische Module wie Dreiecksaktualisierung und Paardarstellung verzichtet, nur den Standard-Transformer verwendet und die Strukturwahrnehmung durch adaptive Schichten verbessert;
* Strukturelle Einschränkungen werden hinzugefügt, um dem Modell mehr Flexibilität und physikalische Konsistenz bei der Generierung dreidimensionaler Proteinstrukturen zu verleihen. Das mit 9M-Strukturdaten trainierte 3B-Parametermodell kann problemlos auf handelsüblicher Hardware ausgeführt werden, wodurch der Rechenaufwand deutlich reduziert wird.
„Ml-simplefold: Ein leichtes KI-Modell zur Vorhersage der Proteinfaltung“ ist jetzt auf der HyperAI Hyperneuron-Website (hyper.ai) im Bereich „Tutorials“ verfügbar. Starten Sie es mit einem Klick, um das neueste Tool zur Proteingenerierung kennenzulernen.
Link zum Tutorial:
Demolauf
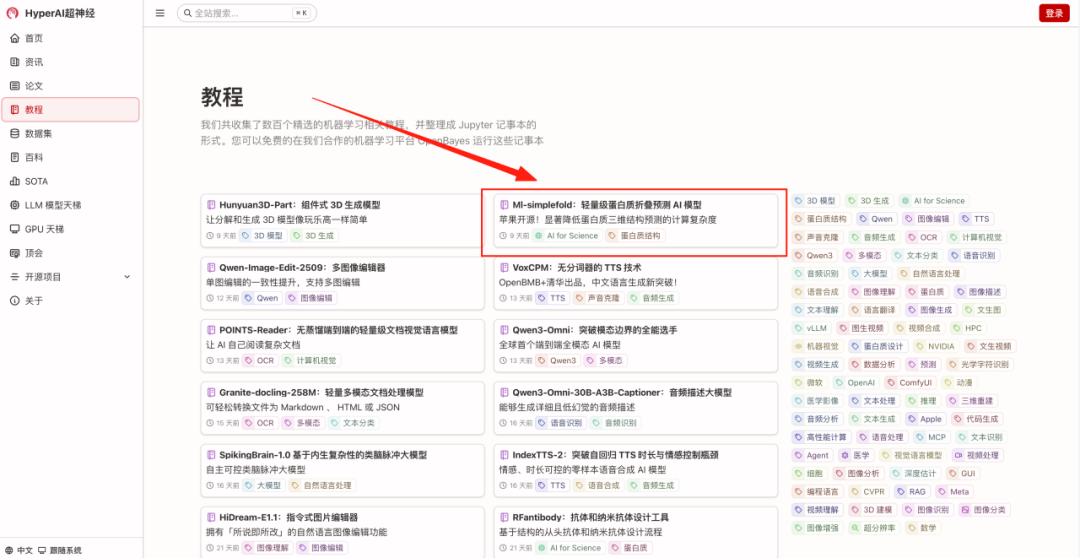
- Geben Sie die URL hyper.ai in Ihren Browser ein. Klicken Sie nach dem Aufrufen der Homepage auf die Seite „Tutorial“, wählen Sie „Protein Folding Model Based on Flow Matching“ und klicken Sie auf „Dieses Tutorial online ausführen“.
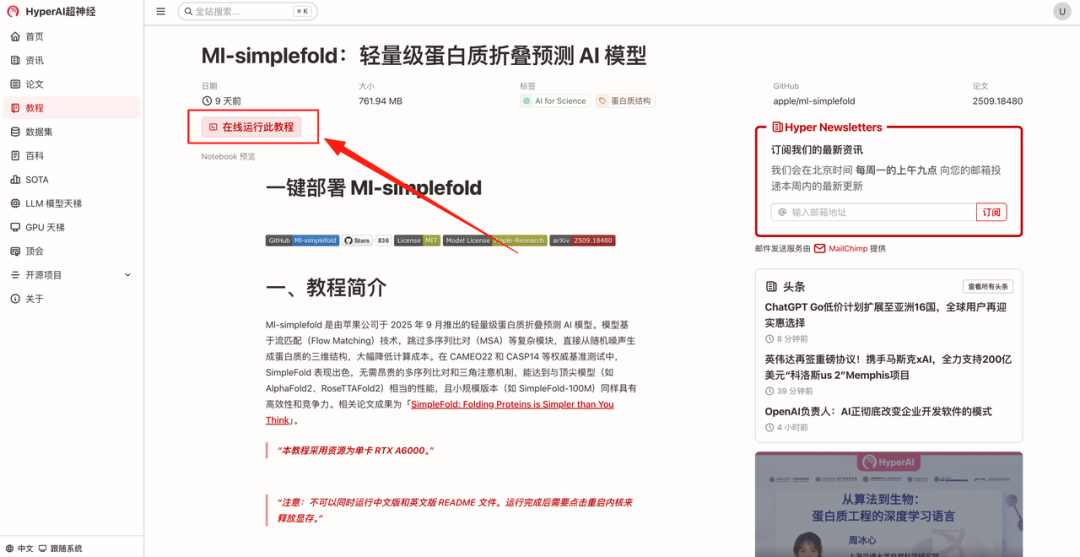
- Klicken Sie nach dem Seitensprung oben rechts auf „Klonen“, um das Tutorial in Ihren eigenen Container zu klonen.
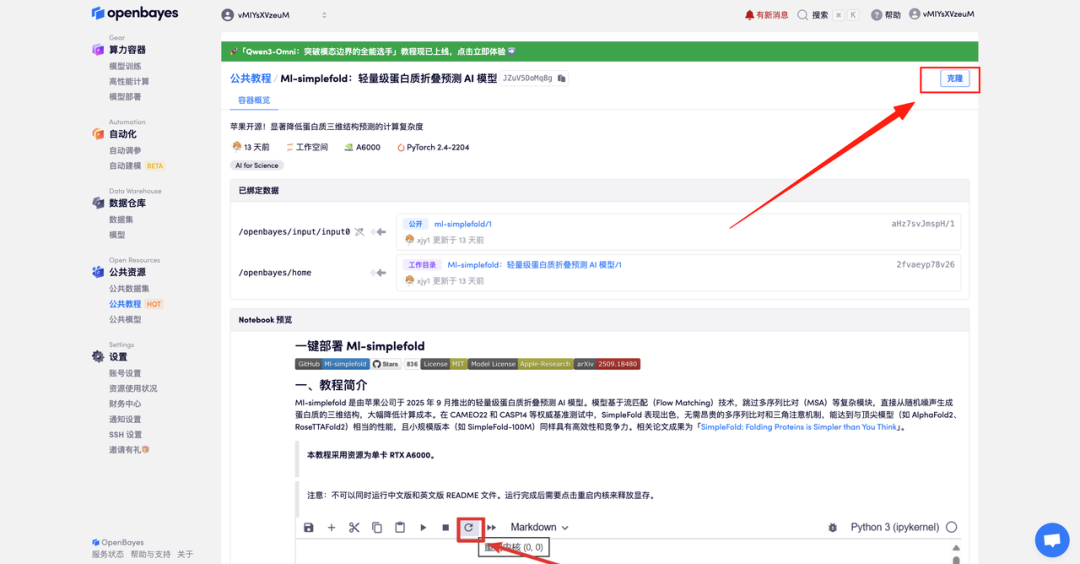
- Wählen Sie die Images „NVIDIA RTX A6000 48GB“ und „PyTorch“ aus und klicken Sie auf „Weiter“. Die OpenBayes-Plattform bietet vier Abrechnungsoptionen: Pay-as-you-go oder Tages-/Wochen-/Monatstarife. Neue Benutzer können sich über den unten stehenden Einladungslink registrieren und erhalten 4 Stunden kostenlose RTX 4090 und 5 Stunden kostenlose CPU-Zeit!
Exklusiver Einladungslink von HyperAI (kopieren und im Browser öffnen):
https://openbayes.com/console/signup?r=Ada0322_NR0n
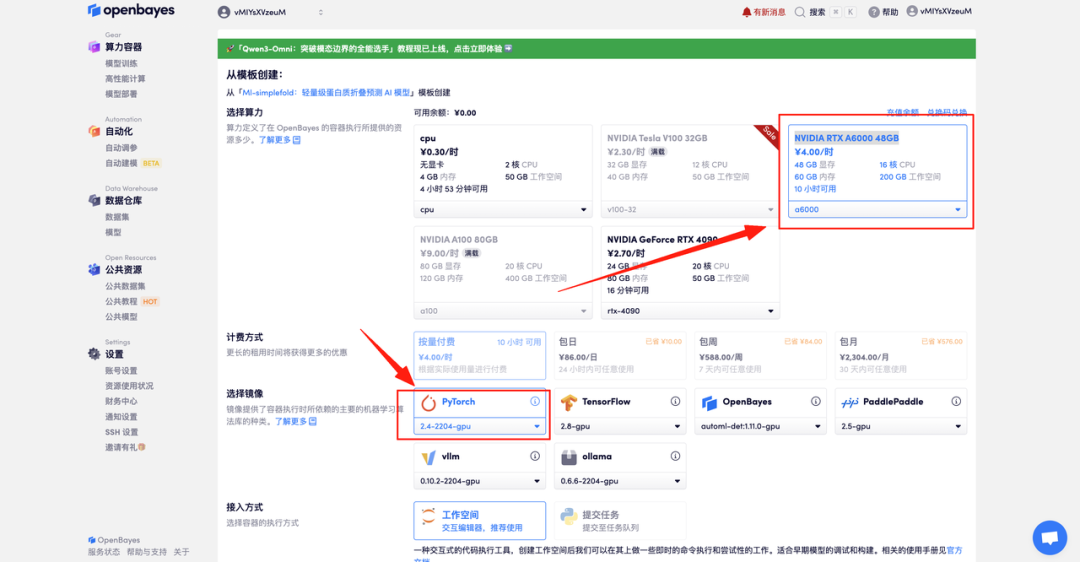
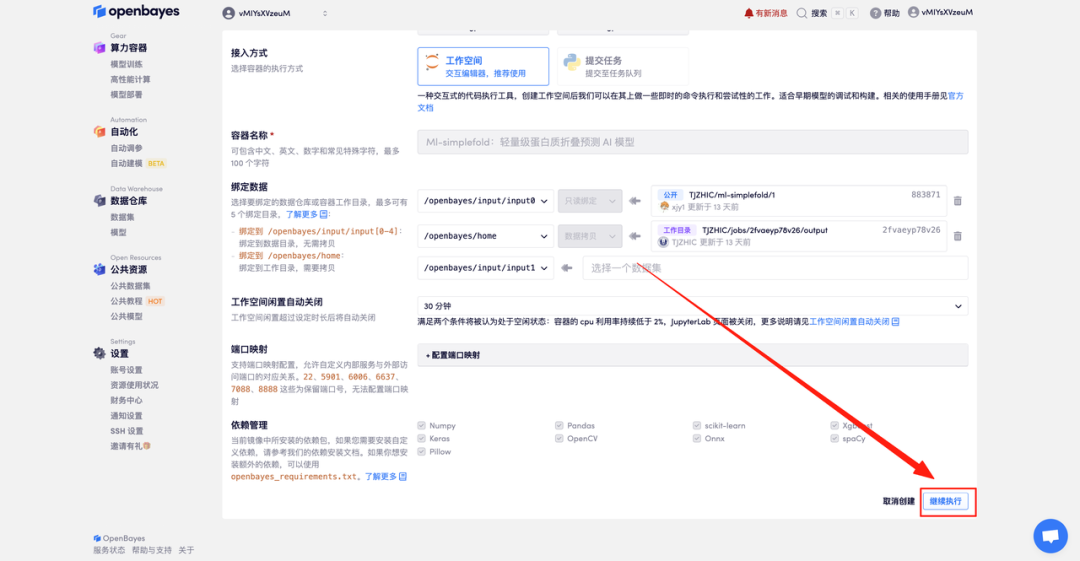
4. Warten Sie, bis die Ressourcen zugewiesen sind. Der erste Klonvorgang dauert etwa 2 Minuten. Wenn der Status auf „Läuft“ wechselt, klicken Sie auf „Arbeitsbereich öffnen“, um zur Demoseite zu gelangen.
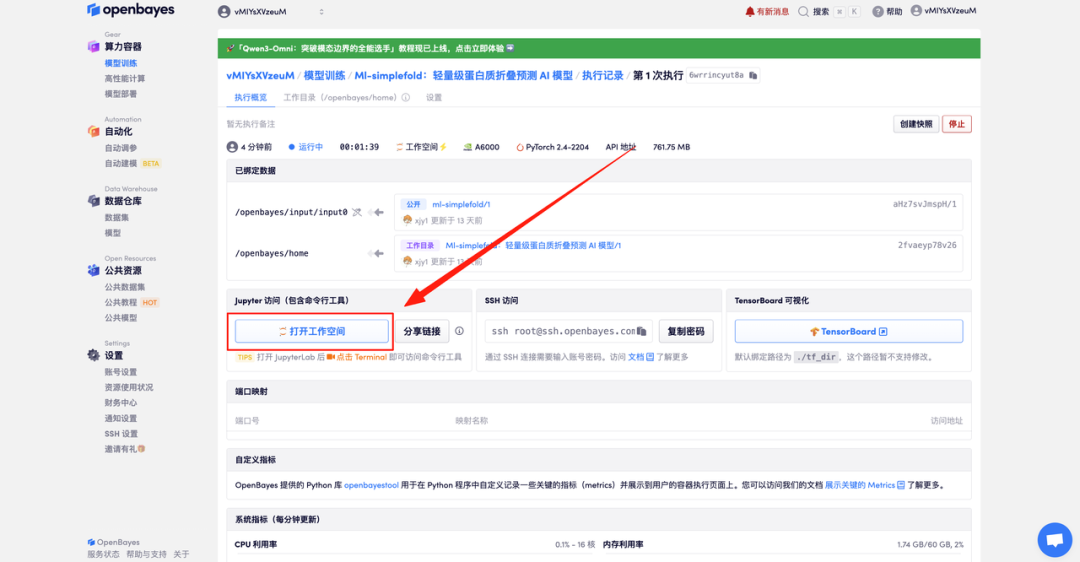
Effektdemonstration
Nachfolgend finden Sie die Ml-Simplefold-Nutzungsseite. Klicken Sie auf „README“, um zur Generierungsoberfläche zu springen.
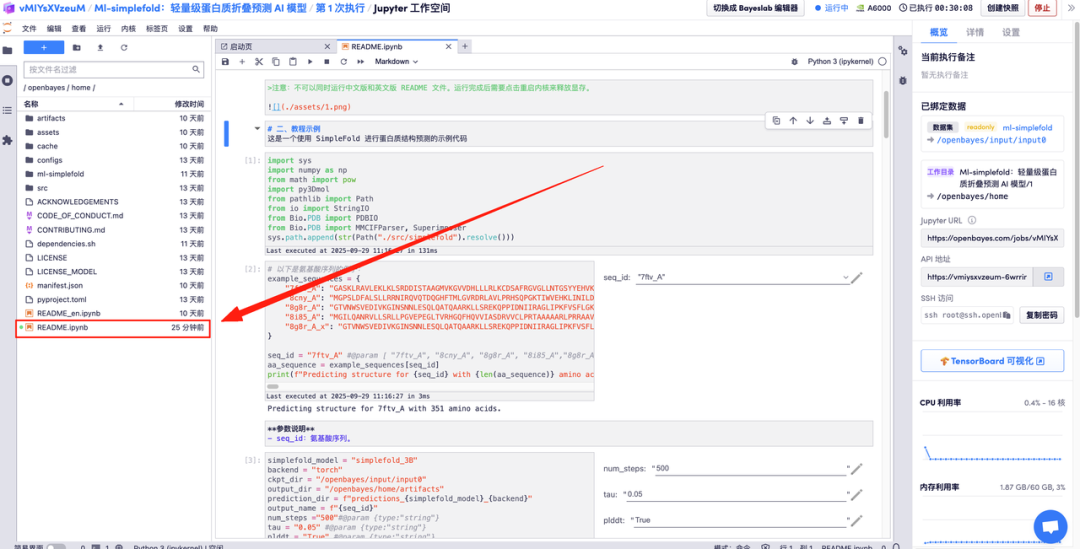
Geben Sie die Aminosäureparameterbeschreibungen in die entsprechenden Bereiche ein und klicken Sie auf „Ausführen“, um die vorhergesagte Proteindatei, die Gesamtatomstruktur und das 3D-Visualisierungsbild zusammen mit der GT-Struktur zu erhalten.
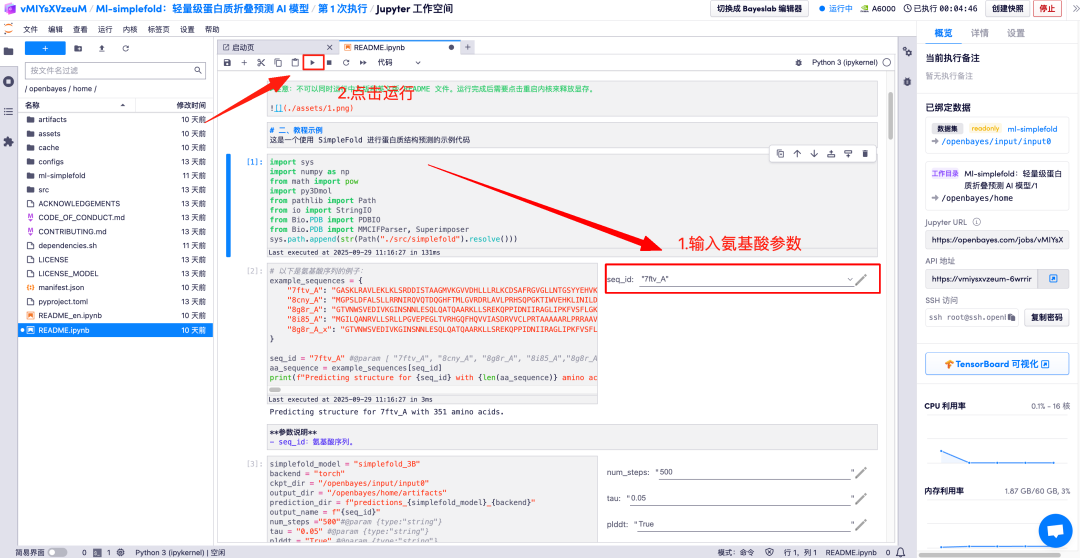
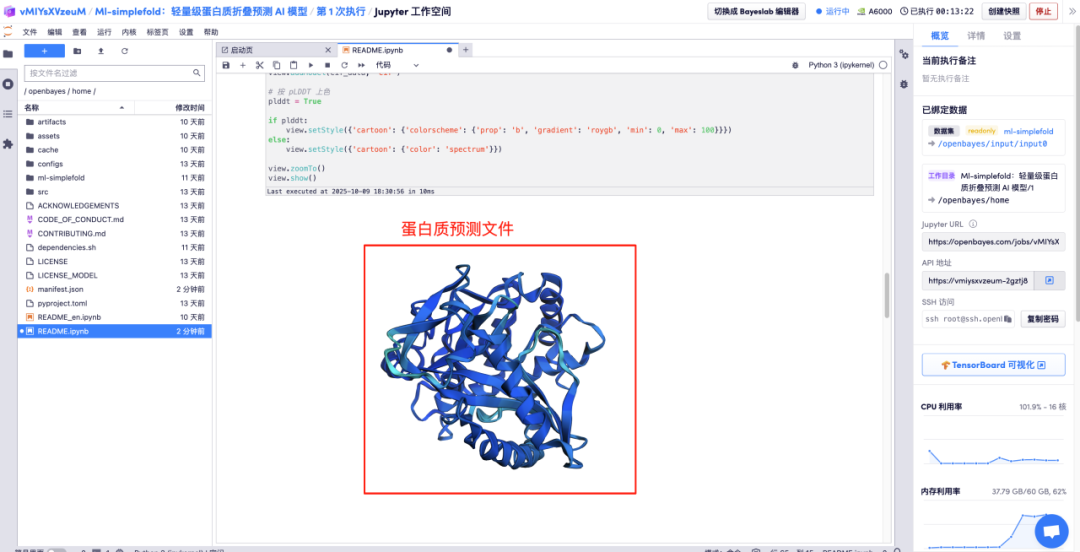
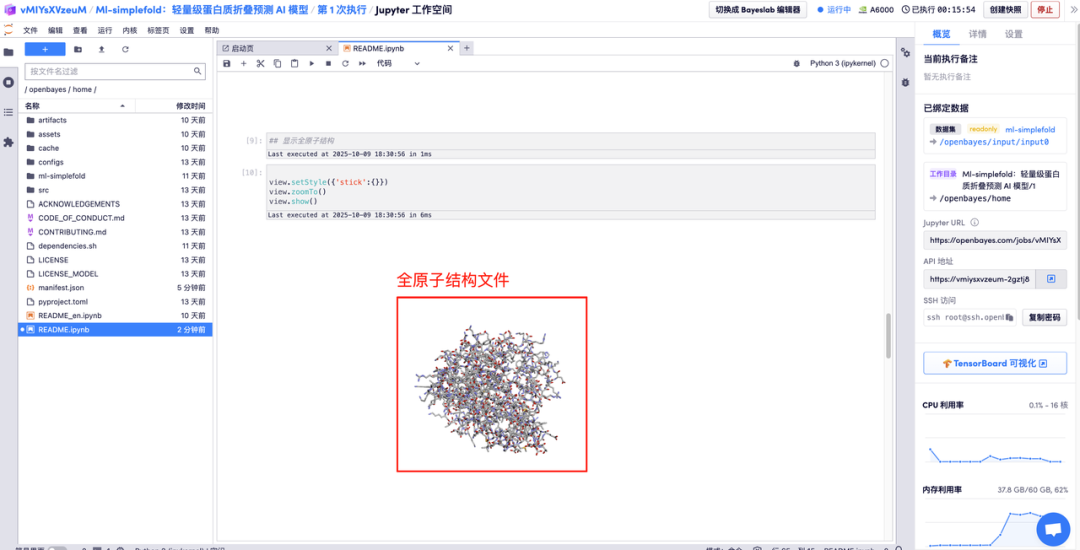
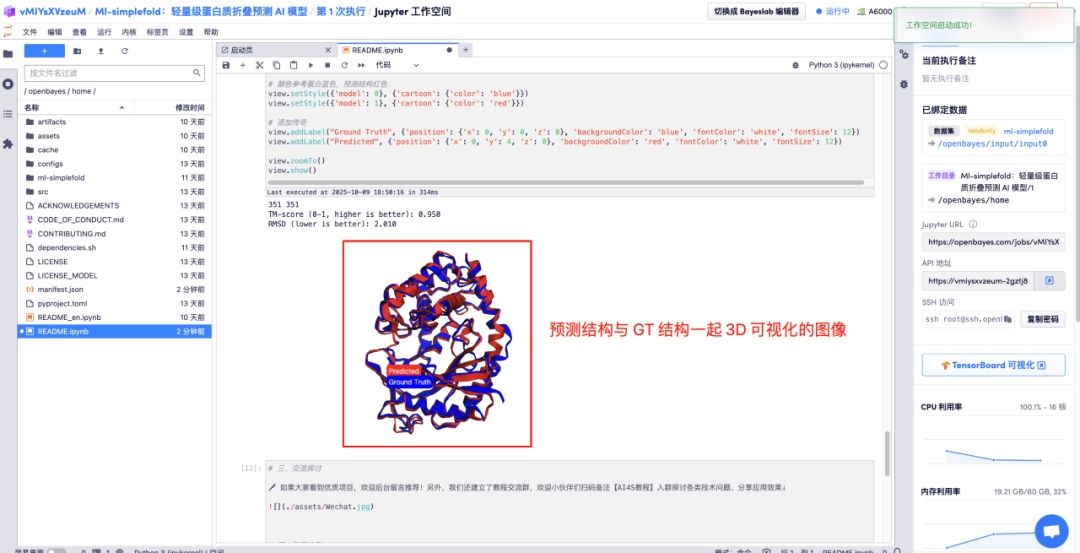
Das Obige ist das diesmal von HyperAI empfohlene Tutorial. Jeder ist herzlich eingeladen, vorbeizukommen und es auszuprobieren!
Link zum Tutorial: