Command Palette
Search for a command to run...
Online-Tutorial: RFdiffusion2 Ermöglicht Die Proteinerzeugung Auf Atomarer Ebene Basierend Auf Chemischen Reaktionen Und Erreicht in 41 Fällen Eine Erfolgsrate Von 100%

Bisher wurden mit dem generativen Proteindesign-Modell RFdiffusion vor allem Proteinstrukturen mit präzisen, festen Positionen durch die Beschreibung idealer aktiver Zentren generiert. Dieser Ansatz hatte jedoch zwei wesentliche Einschränkungen, die nur schwer zu überwinden waren:
* Die Geometrie des aktiven Zentrums kann nur auf der Ebene der Reste angegeben werden, und die Forscher müssen die möglichen Rückgratpositionen der Reste durch Aufzählung der Seitenkettenrotamere bestimmen.
* Die Positionen der katalytischen Rückstände müssen in der Sequenz vorbestimmt werden, was den Platz der zu beprobenden Lösungen stark einschränkt.
Als Reaktion auf damit verbundene Herausforderungen veröffentlichte das Institute for Protein Design der University of Washington im April 2025 ein neues generatives Modell, Rosetta Fold Diffusion 2 (RFdiffusion2).Dadurch wurde ein optimierter Weg zur Erzeugung von Proteingerüsten mit maßgeschneiderten aktiven Zentren auf der Grundlage einfacher chemischer Reaktionsbeschreibungen geschaffen, wodurch der langjährige technische Engpass bei der Katalysatorentwicklung überwunden wurde.Es bietet umfassende technische Unterstützung für eine Vielzahl von Anwendungen, einschließlich der Kunststoffzersetzung.
RFdiffusion2 verwendet einen neuen, tiefgreifenden generativen Ansatz, der Proteindesign basierend auf sequenzunabhängigen Beschreibungen der Positionen funktioneller Gruppen ermöglicht, ohne dass die Generierung von Rückrotationsisomeren erforderlich ist. Im Vergleich zu früheren Modellen konzentriert sich die Optimierung von RFdiffusion2 auf drei Schlüsselbereiche:
* Die Abhängigkeit von der Restzähl- und Sequenzindizierung wurde beseitigt und es wurden Flussabgleichs- und stochastische Zentrierungstechniken eingeführt, wodurch das Modell Proteinrückgrate direkt auf atomarer Ebene generieren kann.
* Unterstützt die indexfreie Generierung aktiver Zentren auf atomarer Ebene, kann Ligandeninformationen verarbeiten und während des Generierungsprozesses automatisch Ligandenkonformationen abtasten, wodurch die Designflexibilität verbessert wird;
* Verwendet einen neuen AME-Benchmark, einen rechnergestützten Proteindesign-Benchmark, der 41 verschiedene aktive Stellen abdeckt (die vorherige Generation deckte nur 16 ab).
In weiteren Experimenten entwickelte das Forscherteam Proteine um drei verschiedene katalytische Zentren herum. Die experimentellen Ergebnisse zeigten, dassRFdiffusion2 zeigt im Vergleich zu früheren Methoden erhebliche Leistungsverbesserungen und konnte in 41 Fällen erfolgreich Proteinrückgrate erzeugen, die die Einschränkungen erfüllen, mit einer Erfolgsrate von 100%;Unter denselben Bedingungen lag die Erfolgsrate von RFdiffusion1 bei nur etwa 39%. Man kann sagen, dass die Einführung von RFdiffusion2 einen entscheidenden Schritt vorwärts im KI-gesteuerten Proteindesign darstellt.
RFdiffusion2: Ein Proteindesign-Tool ist jetzt auf der HyperAI-Website (hyper.ai) im Bereich Tutorials verfügbar. Erleben Sie das neueste Tool zur Proteingenerierung mit nur einem Klick.
Link zum Tutorial:
https://hyper.ai/cn/tutorials/44096
Demolauf
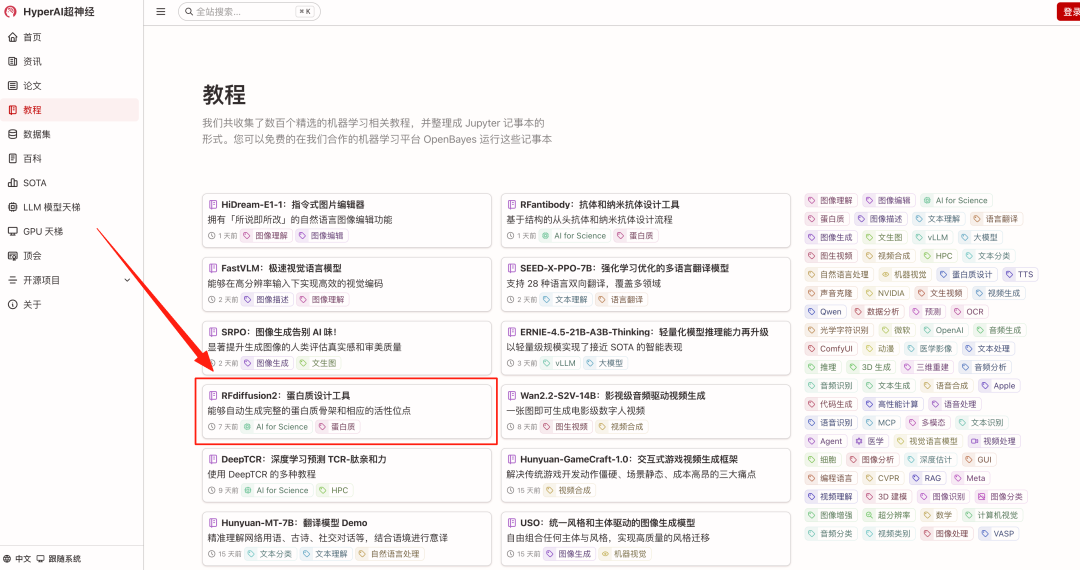
1. Geben Sie die URL hyper.ai in Ihren Browser ein. Klicken Sie auf der Startseite auf die Seite „Tutorials“, wählen Sie „RFdiffusion2: Protein Design Tool“ und klicken Sie auf „Dieses Tutorial online ausführen“.
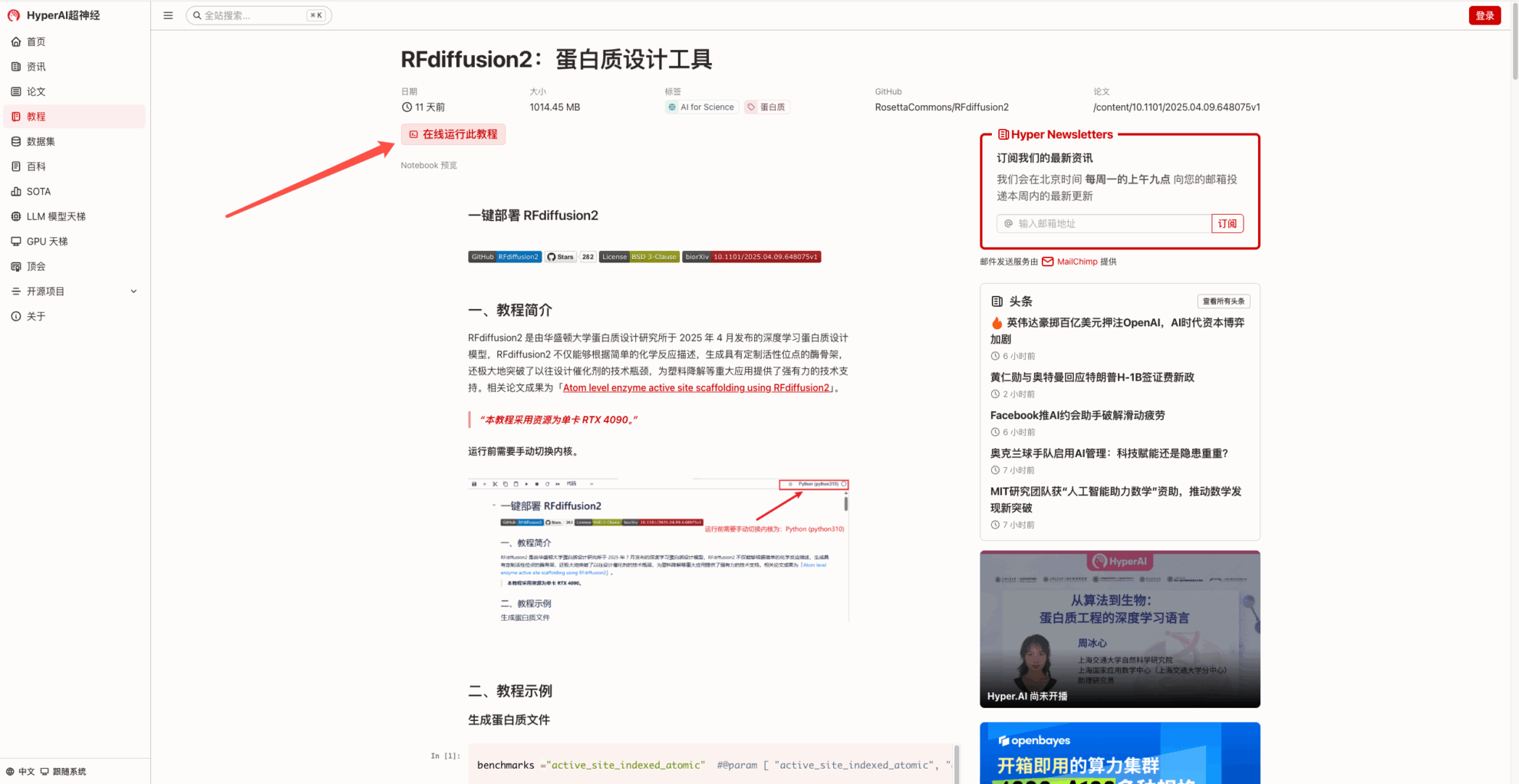
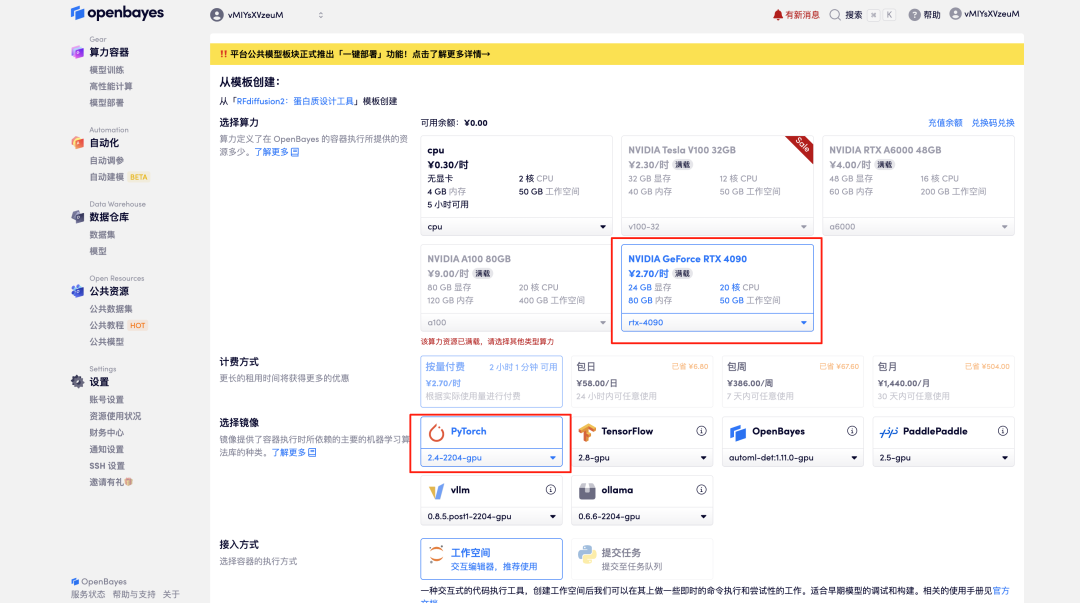
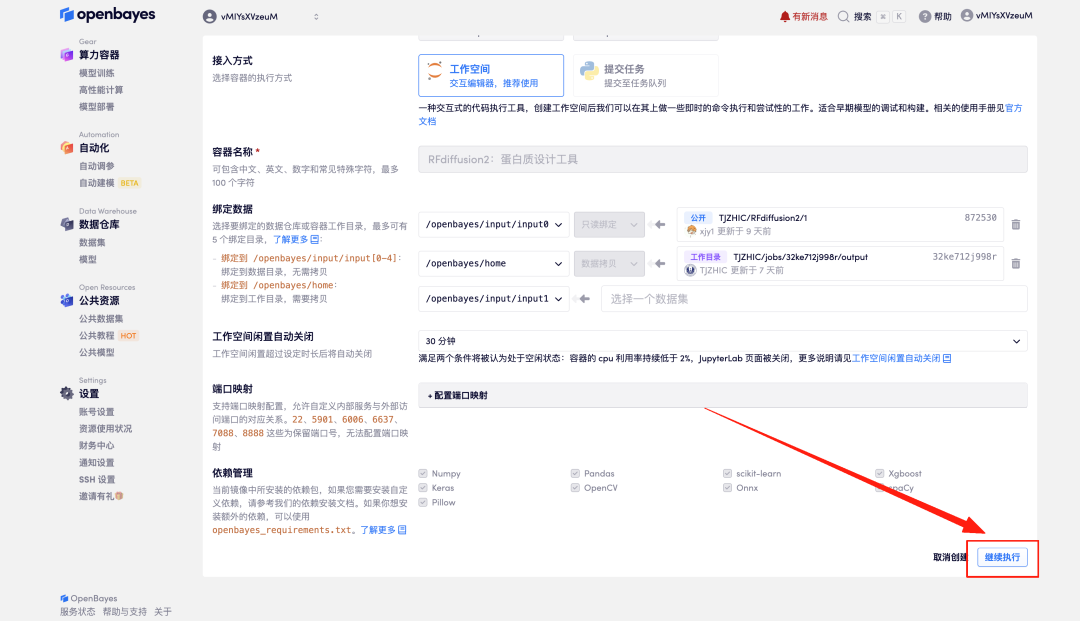
2. Klicken Sie nach dem Seitensprung oben rechts auf „Klonen“, um das Tutorial in Ihren eigenen Container zu klonen.
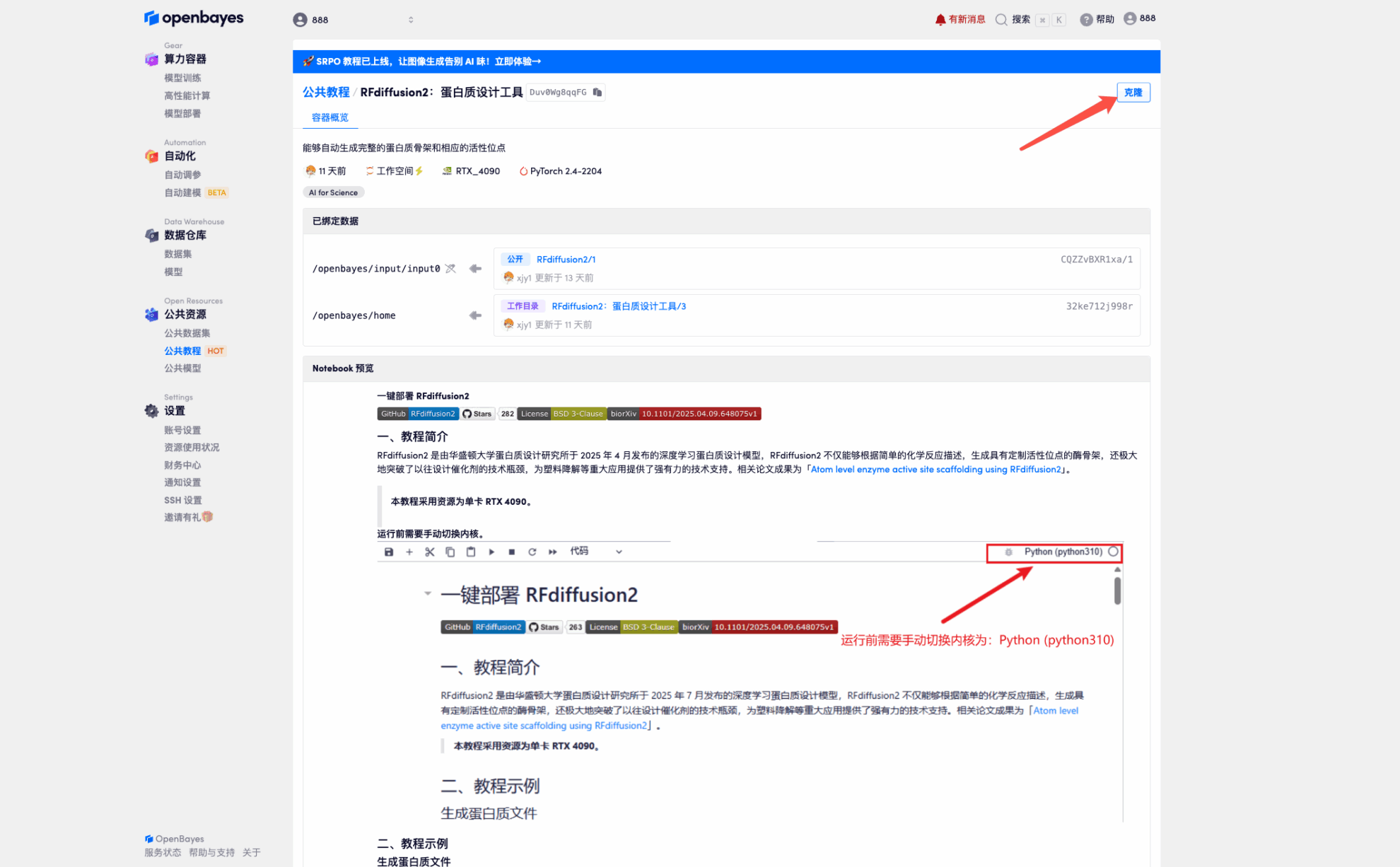
3. Wählen Sie NVIDIA GeForce RTX 4090 und PyTorch-Image aus und klicken Sie auf „Weiter“. Die OpenBayes-Plattform bietet vier Abrechnungsmethoden. Sie können je nach Bedarf zwischen „Pay as you go“ oder „täglich/wöchentlich/monatlich“ wählen. Neue Benutzer können sich über den unten stehenden Einladungslink registrieren, um 4 Stunden RTX 4090 + 5 Stunden CPU-freie Zeit zu erhalten!
Exklusiver Einladungslink von HyperAI (kopieren und im Browser öffnen):
https://openbayes.com/console/signup?r=Ada0322_NR0n
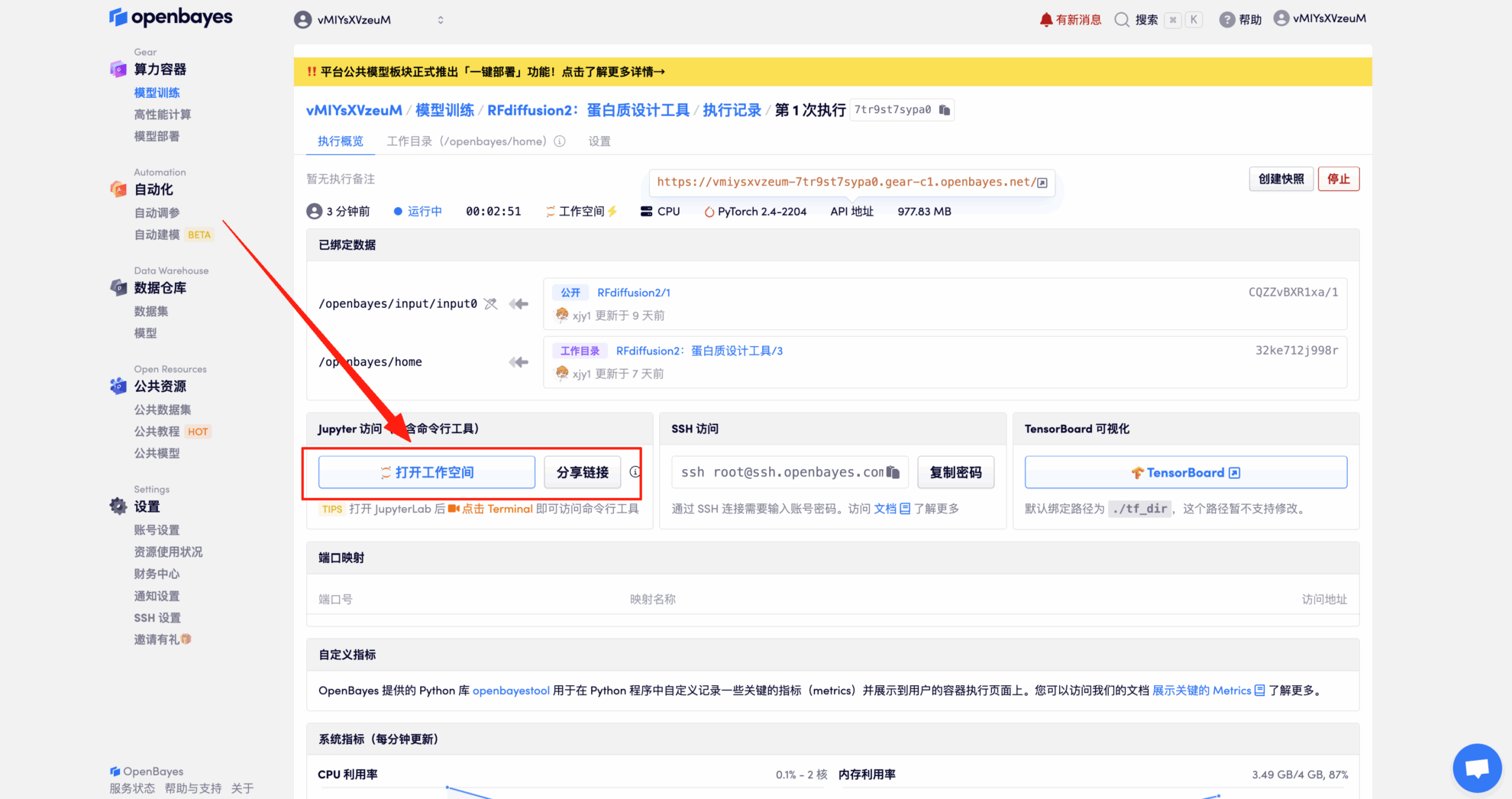
4. Warten Sie, bis die Ressourcen zugewiesen sind. Der erste Klon dauert etwa 2 Minuten. Wenn der Status auf „Läuft“ wechselt, klicken Sie auf den Sprungpfeil neben „Arbeitsbereich öffnen“, um zur Demoseite zu gelangen.
Effektdemonstration
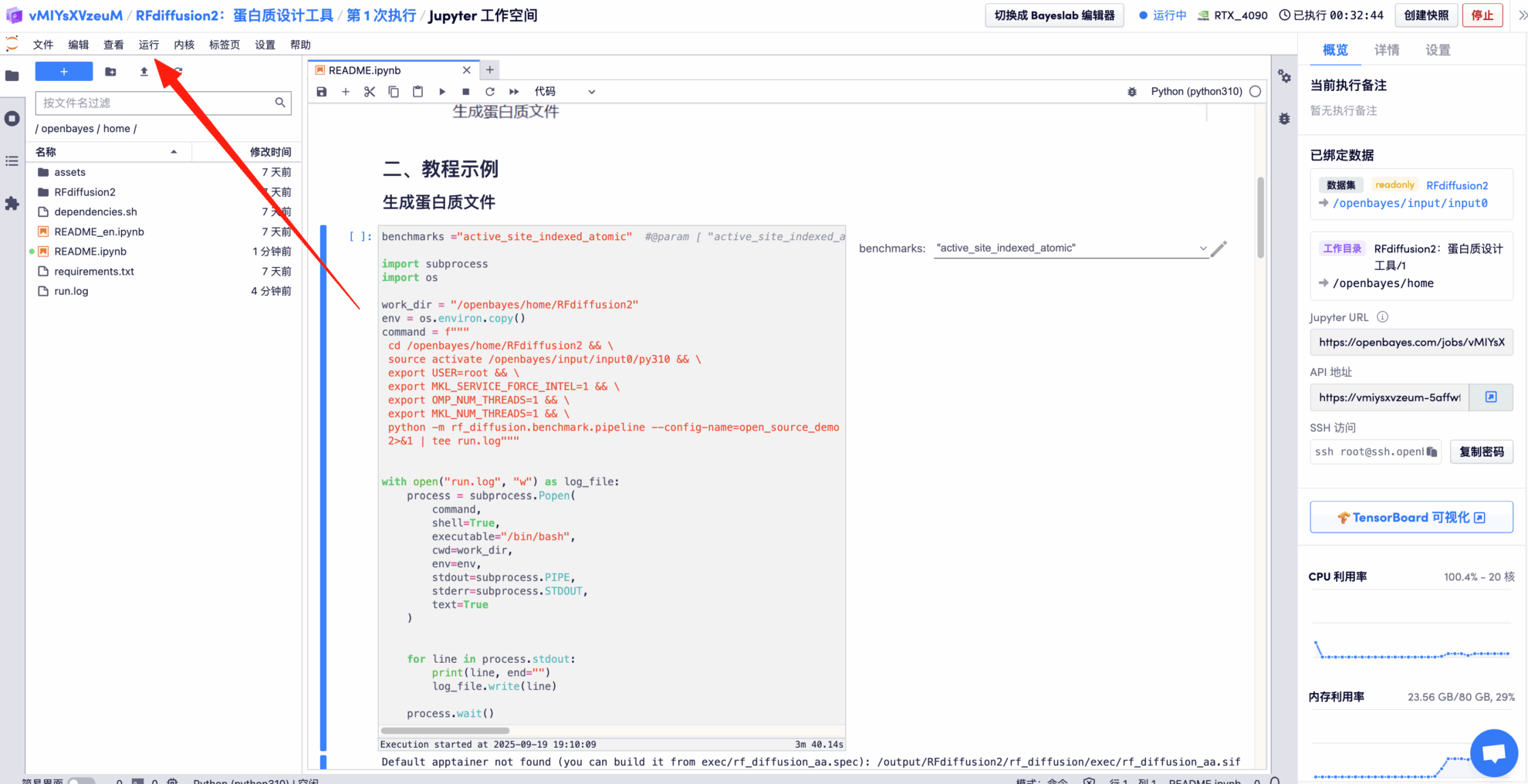
Nachdem Sie die Seite „Demo ausführen“ aufgerufen haben, geben Sie Benchmarks ein und klicken Sie auf „Ausführen“, um die Ausgabeergebnisse zu erhalten.
Hier nehmen wir "active_site_indexed_atomic" als Beispiel
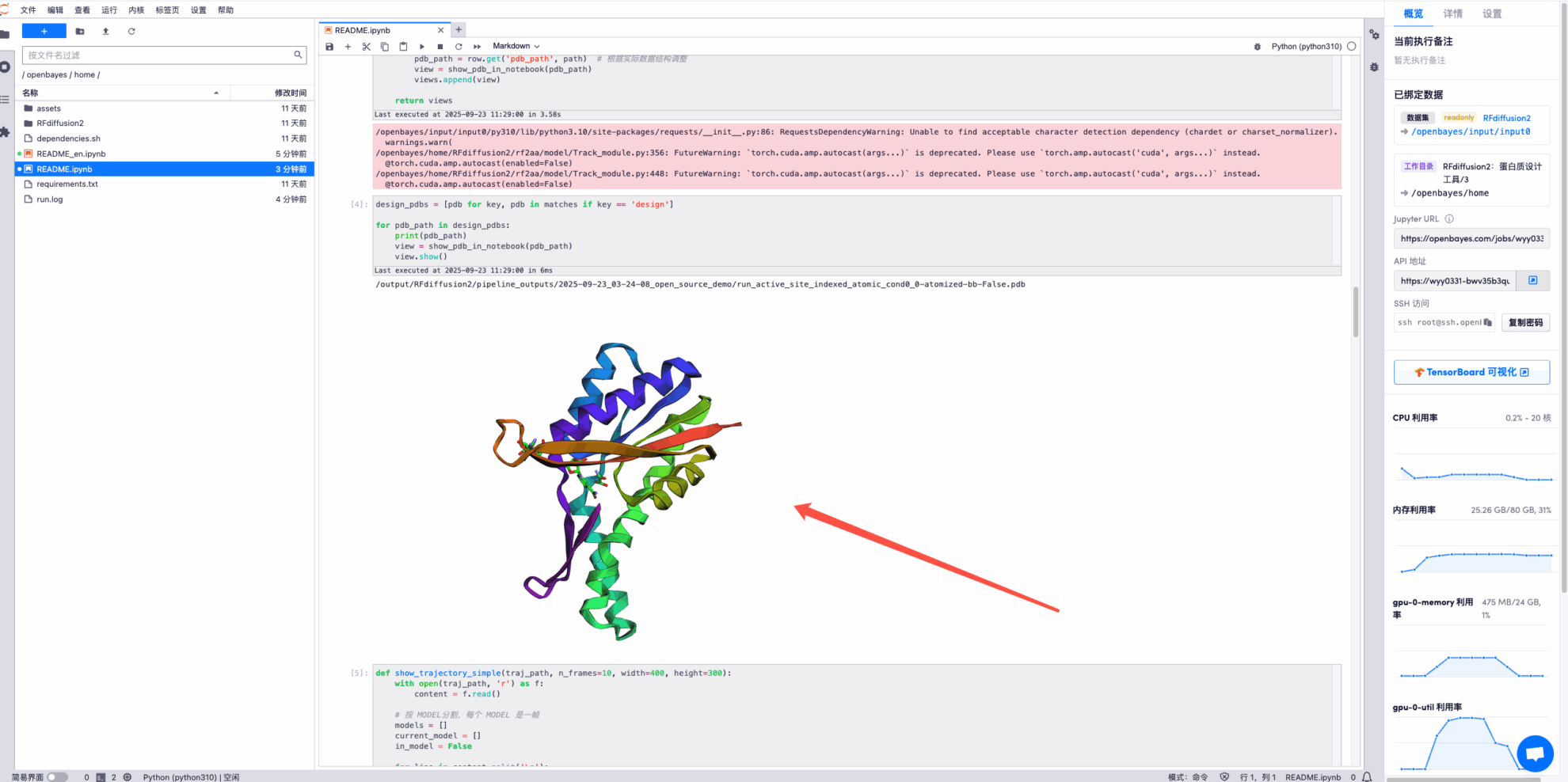
* Anzeige der endgültigen Proteindesigndatei
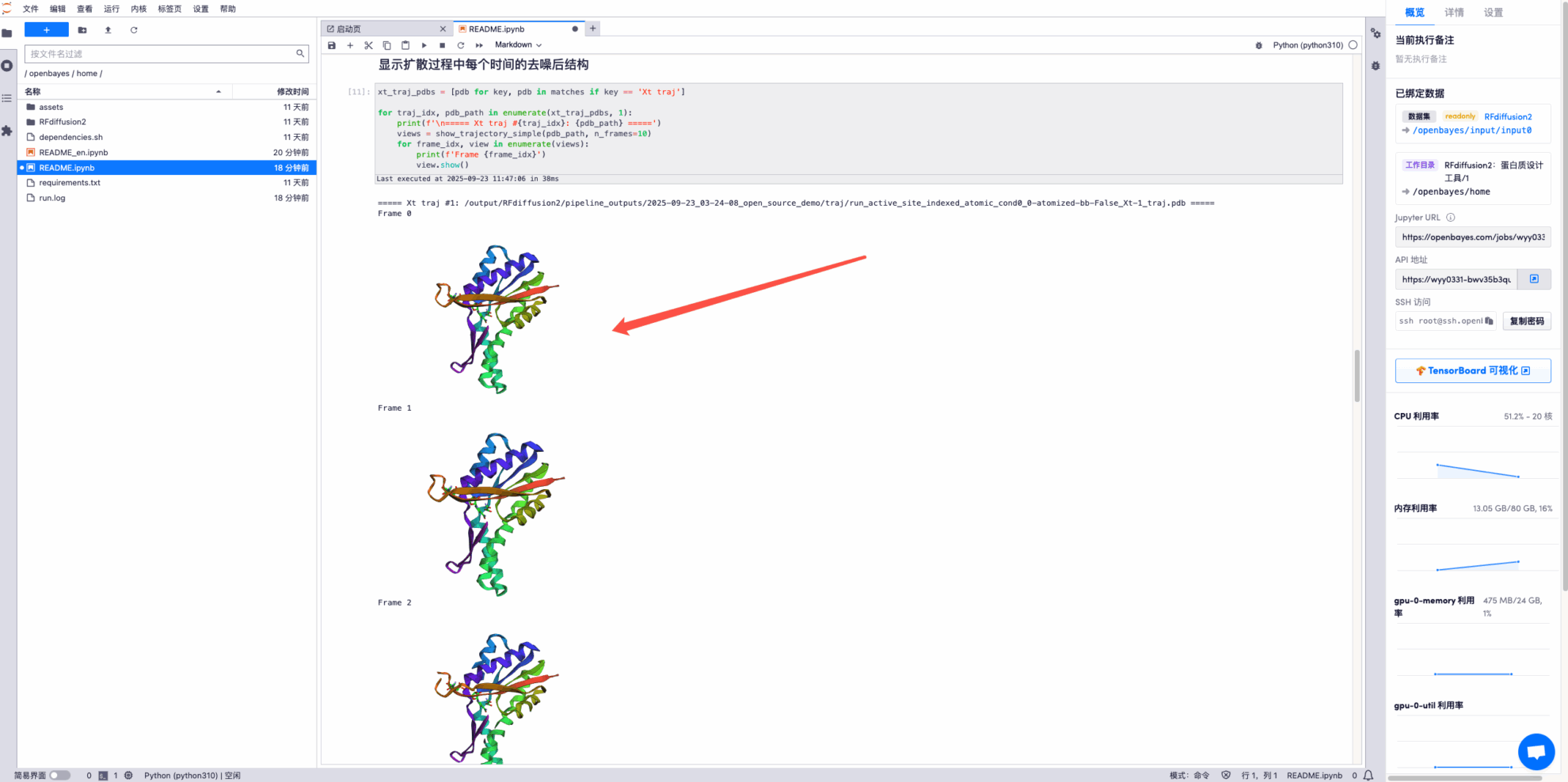
* Zeigen Sie die rauschfreie Struktur zu jedem Zeitpunkt während des Diffusionsprozesses an. Einige Beispiele:
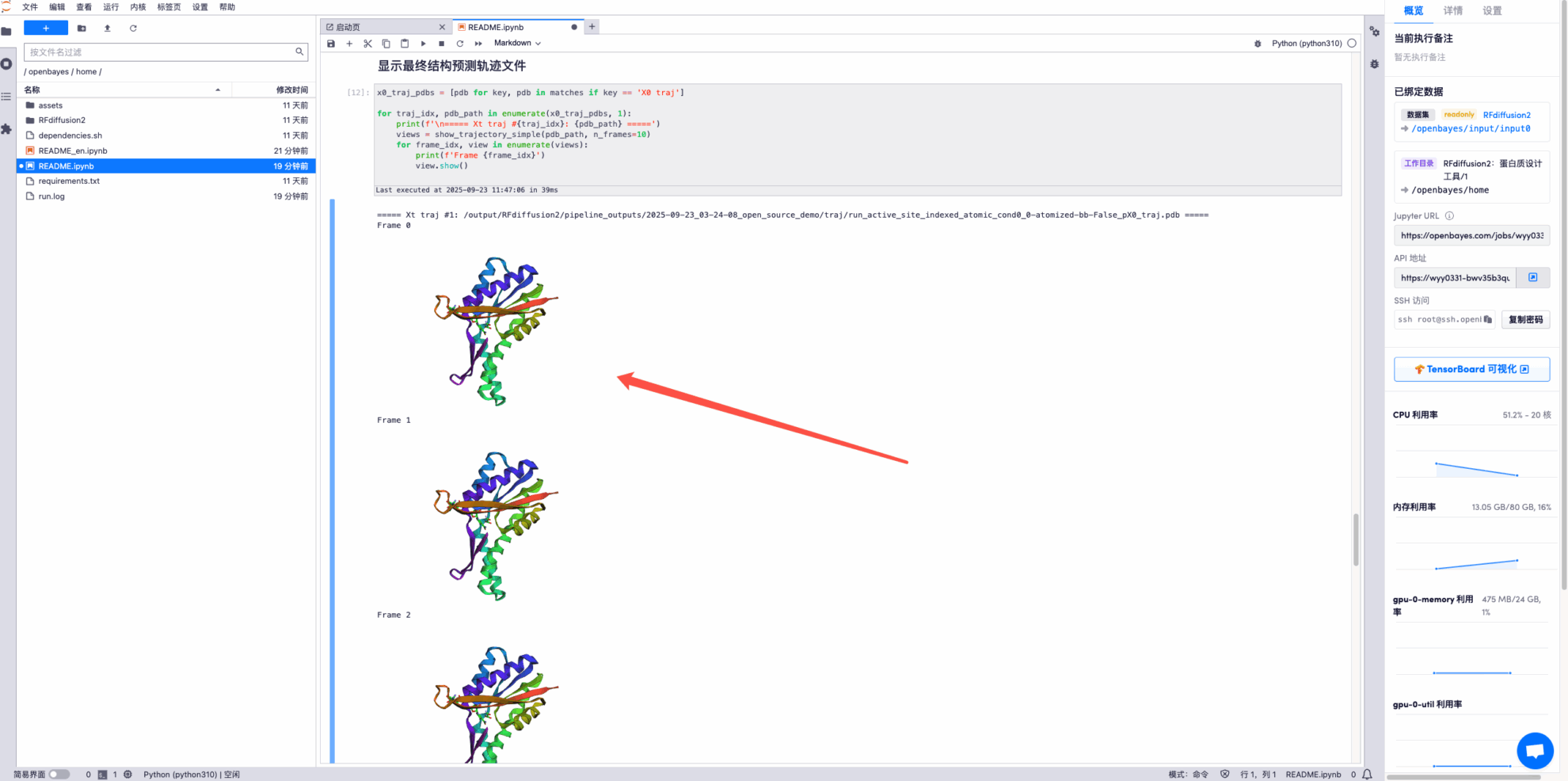
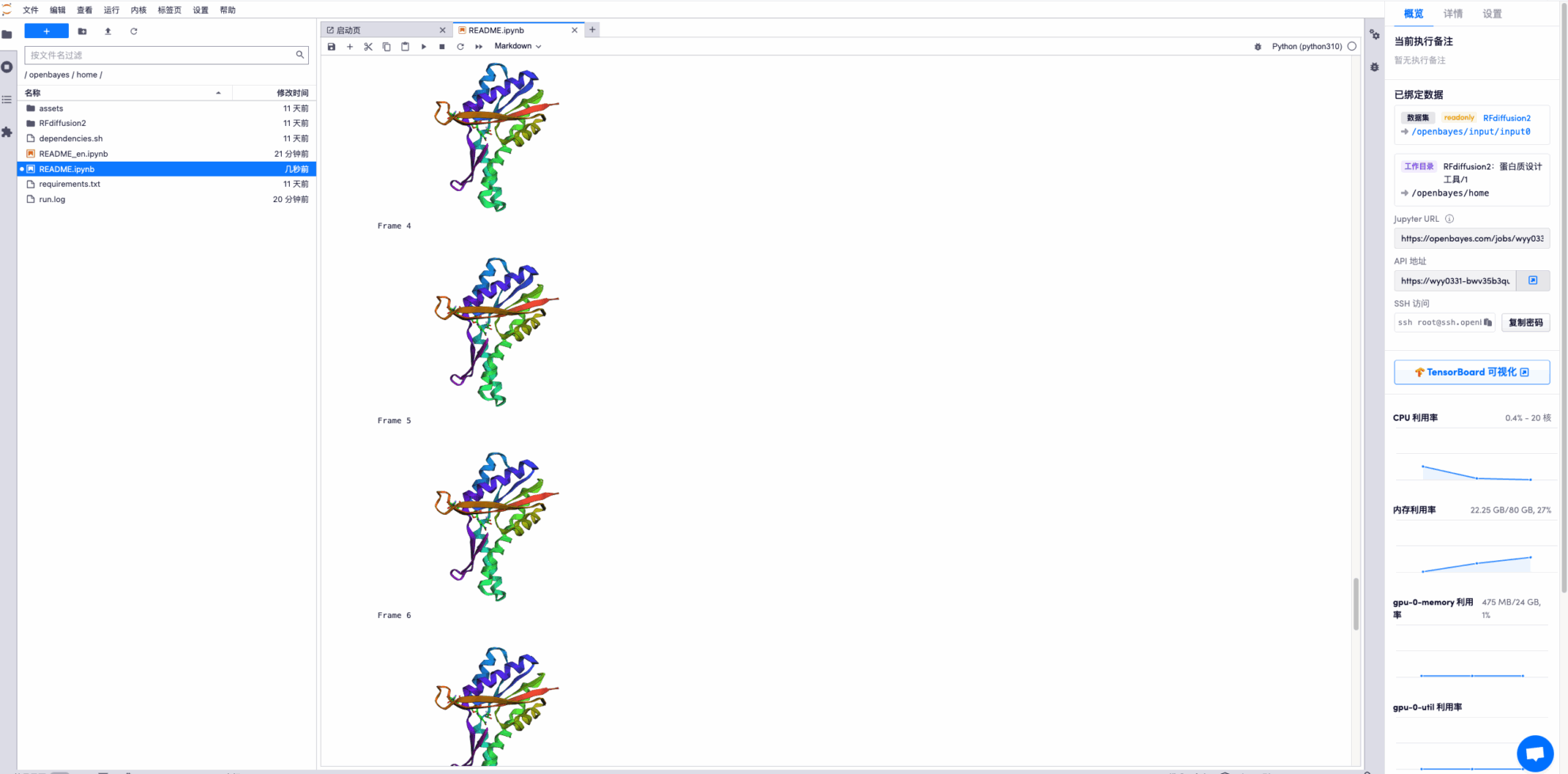
* Zeigen Sie die endgültige Strukturvorhersage-Trajektoriendatei an. Einige Beispiele sind wie folgt:
Das Obige ist das diesmal von HyperAI empfohlene Tutorial. Jeder ist herzlich eingeladen, vorbeizukommen und es auszuprobieren!
Link zum Tutorial: