Command Palette
Search for a command to run...
Von Der „Blindprüfung“ Bis Zur „präzisen Positionierung“ Verbessert Das AlphaPPIMI-Framework Die Generalisierungsfähigkeiten Erheblich Und Die Vorhersageleistung Der PPI-Schnittstellenregler Übertrifft Bestehende Methoden.

Innerhalb der komplexen regulatorischen Netzwerke des Lebens koordinieren Protein-Protein-Interaktionen (PPIs) die intrazelluläre Signalübertragung, den Energiestoffwechsel und die Genaktivität und sind somit grundlegend für die Aufrechterhaltung eines normalen Lebens. PPIs spielen eine zentrale Rolle bei der Aufrechterhaltung der physiologischen Homöostase im gesunden Zustand und bei abnormalen Veränderungen im Krankheitsverlauf. Studien haben gezeigt, dass eine Fehlfunktion von PPIs eng mit Krebs, neurodegenerativen Erkrankungen und verschiedenen Infektionskrankheiten verbunden ist. Daher ist die Entwicklung von Medikamenten, die auf PPIs abzielen, zu einem zentralen Bereich der Arzneimittelforschung und -entwicklung geworden.
Frühe Wissenschaftler untersuchten Protein-Protein-Interaktionen wie MDM2-p53 und bestätigten, dass das Eingreifen in solche Interaktionen das Potenzial zur Behandlung von Krankheiten hat und insbesondere neue Ideen für Krankheitsziele liefert, die zuvor schwer zu erreichen waren.Die Besonderheit von PPIs besteht jedoch darin, dass ihre Interaktionsschnittstellen normalerweise relativ flach sind und keine klaren Strukturmerkmale aufweisen, die für die Einbettung von niedermolekularen Arzneimitteln geeignet sind, was eine enorme Herausforderung für die Arzneimittelentwicklung darstellt.Insbesondere bei neu entdeckten PPIs oder solchen mit begrenzten Strukturinformationen ist es schwieriger, Moleküle zu finden, die ihre Funktionen regulieren können.
Die Forscher fanden heraus, dass die Schnittstelle von PPIs zwar breit und flach ist, es aber dennoch einige Schlüsselbereiche gibt – sogenannte „Hotspots“. Sie wirken wie „Schalter“ in der Interaktion und werden zu idealen Zielen für die Arzneimittelentwicklung.
Mit der rasanten Entwicklung von Technologien der künstlichen Intelligenz, insbesondere maschinellem Lernen und Deep Learning, hat sich der Prozess der Arzneimittelentwicklung für PPIs deutlich beschleunigt. Es sind zahlreiche innovative Algorithmen und Tools entstanden, darunter 2P2IHUNTER zur effizienten Identifizierung potenzieller PPI-Inhibitoren, PPIMpred für virtuelles Screening im großen Maßstab und SMMPPI, das nicht nur regulatorische Moleküle vorhersagt, sondern auch praktischen Nutzen in der COVID-19-Forschung zeigt. Trotz erheblicher Fortschritte bleiben Herausforderungen bestehen. Herkömmliche rechnergestützte Methoden, die stark auf Ähnlichkeitsscreening beruhen, haben Schwierigkeiten, die komplexen Interaktionsmerkmale von PPI-Schnittstellen vollständig zu erfassen. Darüber hinaus verfügen bestehende Modelle nur über begrenzte Generalisierungsmöglichkeiten über verschiedene Proteintypen hinweg, was die Effizienz der Arzneimittelentwicklung für neuartige Zielmoleküle beeinträchtigt.
In den letzten Jahren haben Transformer-basierte vortrainierte Sprachmodelle neue Ideen für die oben genannten Probleme geliefert. Diese Modelle können automatisch Schlüsselmerkmale aus einer großen Anzahl von Proteinsequenzen lernen und so Interaktionen intelligenter vorhersagen.
Basierend auf dieser Richtung hat das gemeinsame Forschungsteam der China University of Petroleum und der Yonsei University mehrere fortschrittliche Technologien integriert, um ein neues Framework namens AlphaPPIMI zu erstellen.Dieses Tool kombiniert vorab trainierte Modelle im großen Maßstab und adaptive Lernmechanismen, um die zentrale Herausforderung zu bewältigen, „Regulatoren zu finden, die speziell auf die PPI-Schnittstelle abzielen“.Durch die vollständige Ausnutzung der Vorteile vortrainierter Großmodelle und die effektive Modellierung komplexer Bindungsmuster durch ein spezielles Cross-Attention-Modul wurde die Generalisierungsfähigkeit des Modells über verschiedene PPI-Familien hinweg erheblich verbessert, was die zukünftige Entwicklung zielgerichteter PPI-Medikamente stark unterstützt.
Die entsprechenden Forschungsergebnisse wurden im Journal of Cheminformatics unter dem Titel „Alphappimi: a comprehensive deep learning framework for predicting PPI-modulator interactions“ veröffentlicht.

Papieradresse:
https://jcheminf.biomedcentral.com/articles/10.1186/s13321-025-01077-2
Folgen Sie dem offiziellen Konto und antworten Sie mit „AlphaPPIMI“, um das vollständige PDF zu erhalten
Weitere Artikel zu den Grenzen der KI:
https://hyper.ai/papers
Datensatz: Aufbau eines PPI-Datensatzsystems mit DLiP als Kern
Die Studie verwendete den DLiP-Datensatz als Trainingskern, der 120 PPIs und die entsprechenden 12.605 einzigartigen Regulatoren enthält.Es kann außerdem die Sequenz, die dreidimensionale Struktur und die experimentellen Aktivitätsdaten jedes Proteinkomplexpaars bereitstellen und bietet so umfassende Unterstützung für den Modellaufbau.
Zur unabhängigen Validierung erstellte das Forschungsteam zwei Benchmark-Testsets aus den Datenbanken DiPPI und iPPIDB. Beide Sets enthalten experimentell validierte Schnittstellenmodulatoren sowie deren Strukturen und Bindungsinformationen. Bei der Zusammenstellung der Daten führte das Team drei Qualitätskontrollen durch: Es wurden ausschließlich heterodimere PPIs beibehalten, Proben mit unklaren Bindungsstellen entfernt und der Umfang auf humane PPIs beschränkt. Darüber hinaus wurden Verbindungen, die auf mehrere Ziele wirken, separiert, um eine genaue Annotation zu gewährleisten.
Die endgültigen Details der beiden Benchmark-Sets lauten wie folgt:DiPPI enthält 201 Regulierungsbehörden, die 1.316 PPI-Zielen entsprechen.Jede Probe verfügt über eine Molekülstruktur, Proteinsequenz, Schnittstellenstruktur und ein aktives Tag; iPPIDB deckt 2.203 Regulatoren und 34 PPIs ab und alle Proteinsequenzen stammen aus der UniProt-Datenbank, wodurch die Datenkonsistenz gewährleistet wird.
Nach der Analyse der physikalischen und chemischen Eigenschaften der beiden Benchmark-Sets wurde festgestellt, dass sie sich hinsichtlich der Zielmerkmale der Schnittstelle und der chemischen Raumverteilung erheblich unterschieden, was die Verallgemeinerung des Modells erschweren würde.Durch die Berechnung des molekularen Fingerabdrucks von ECFP4 stellten wir außerdem fest, dass die durchschnittliche Tanimoto-Ähnlichkeit der Verbindungen in den beiden Benchmark-Sets sehr gering ist.Dies deutet darauf hin, dass die strukturelle Vielfalt dieser Verbindungen relativ hoch ist.
Für eine bestimmte PPI-Familie wählte die Studie auch Regulatoren, die für andere PPI-Familien selektiv sind, als potenziell inaktive Proben aus. Moleküle mit ähnlicher Struktur wie bekannte aktive Regulatoren wurden ausgeschlossen, um das Risiko falsch-negativer Ergebnisse zu verringern. Unter Berücksichtigung des Ungleichgewichts in der Anzahl positiver und negativer Proben reduzierte das Team die Anzahl der negativen Proben, um einen Datensatz mit einer ausgeglichenen Anzahl zu erstellen. Eine anschließende Sensitivitätsanalyse zeigte, dass die Modellleistung unabhängig von der Anpassung des Verhältnisses positiver und negativer Proben sehr stabil war und nicht stark vom Verhältnis abhing. Es ist zu beachten, dass verifizierte inaktive Verbindungen zwar vorhanden sind, diese jedoch nicht in den negativen Probensatz aufgenommen wurden, da sie ungleichmäßig verteilt sind und nach der Aufnahme zu Datenverzerrungen führen können.
Um den praktischen Anwendungswert dieser Methode zu überprüfen,Das Forschungsteam durchsuchte außerdem die „PPI-spezifische Bibliothek“ in der ChemDiv-Datenbank. Diese Bibliothek enthält 205.497 Verbindungen, die speziell für die Grenzflächeneigenschaften von PPIs entwickelt wurden.Dieses groß angelegte virtuelle Screening demonstrierte die Praktikabilität dieser Methode in Szenarien der Arzneimittelentwicklung.
AlphaPPIMI-Framework: Multi-Source-Feature-Extraktion, bidirektionale Cross-Attention und CDAN-Generalisierungsoptimierung
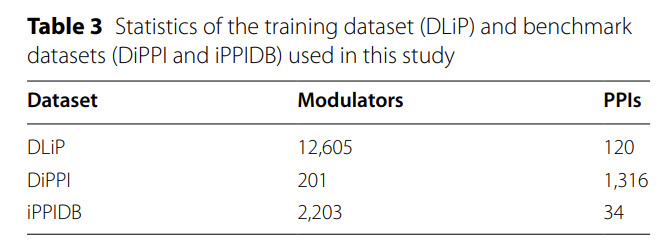
Wie in der Abbildung unten gezeigt,In dieser Studie wurde ein neues Berechnungsframework namens AlphaPPIMI entwickelt, das speziell der Vorhersage der Bindungsbeziehung zwischen PPIs und Regulatoren dient.Mit besonderem Fokus auf der gezielten Steuerung von Interaktionen an Schnittstellenbindungsstellen integriert dieses Framework mehrere erweiterte Module, darunter Uni-Mol2, ESM2, ProTrans, ECFP und PFeature, und strebt eine umfassende Extraktion PPI-bezogener Merkmale bei gleichzeitig effizientem Repräsentationslernen an.
In der Phase der molekularen Charakterisierung verwendete das Forschungsteam das Uni-Mol2-Modell mit 84 Millionen Parametern, um Atome, chemische Bindungen, geometrische Informationen und molekulare Fingerabdrücke zu integrieren.Für jeden Modulator wurde ein 768-dimensionaler globaler Merkmalsvektor generiert. Das Team integrierte zudem ECFP4-Fingerabdrücke, um einen 1.024-dimensionalen Binärvektor zur Erfassung wichtiger chemischer Informationen, wie beispielsweise der zyklischen Substruktur, zu generieren. Letztendlich wurden diese beiden Merkmalstypen kombiniert, um einen 1.792-dimensionalen Merkmalsvektor zu erzeugen, der molekulare Topologie, 3D-Geometrie und chemische Substruktur umfasst und zuverlässige Datenunterstützung für Vorhersagen der Grenzflächenbindung bietet.
Die Extraktion von Proteinmerkmalen erfolgt mithilfe von drei sich ergänzenden Ansätzen:Das auf der Transformer-Architektur basierende Modell ESM2-150M wurde anhand von 60 Millionen UniRef50-Sequenzen trainiert und generiert 640-dimensionale Feature-Vektoren, die speziell Aminosäurebeziehungen im Zusammenhang mit der Grenzflächenbildung erfassen. Das an über 45 Millionen Proteinsequenzen trainierte Modell ProtTrans liefert 1.024-dimensionale Embedding-Vektoren und erfasst evolutionäre Muster, die ESM2 ergänzen. Schließlich liefert die PFeature-Methode anhand von 19 Deskriptorkategorien Informationen zur Proteinstruktur und zu physikochemischen Eigenschaften. Die Kombination dieser drei Methoden erzeugt eine 3.366-dimensionale Proteindarstellung, die Proteinsequenzmuster und grenzflächenspezifische Eigenschaften umfassend abdeckt.
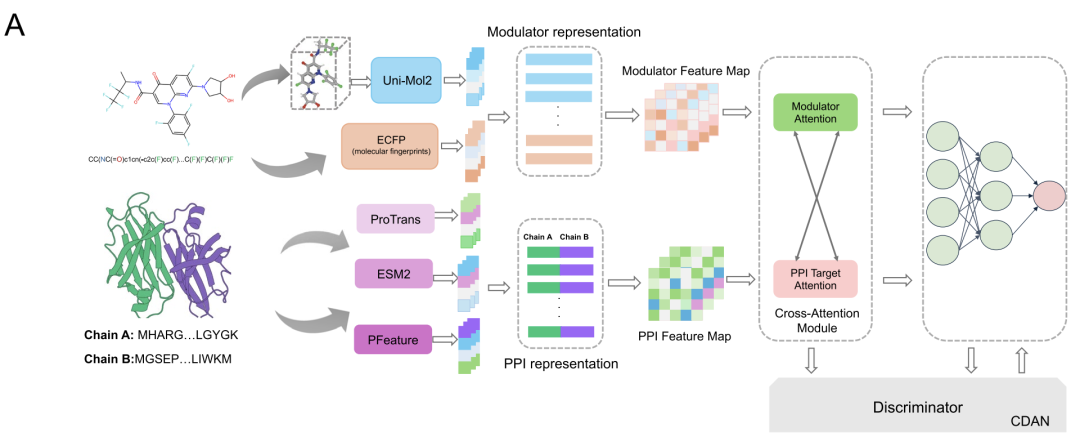
Um die komplexen Interaktionen zwischen Proteinen und Regulatoren zu modellieren, hat AlphaPPIMI ein bidirektionales Cross-Attention-Modul entwickelt, wie in der folgenden Abbildung dargestellt. Dieses Modul führt zunächst lineare Transformationen an der Regulator-Feature-Matrix FM und der Ziel-Feature-Matrix FP durch, die dann in das Attention-Submodul eingespeist werden. Dies ermöglicht einen bidirektionalen Informationsaustausch auf Schlüssel-Wert-Ebene. PPI-Features werden mithilfe der Aufmerksamkeitsgewichte der Regulatorquelle optimiert, während Regulator-Features mithilfe des PPI-gesteuerten Aufmerksamkeitsmechanismus angepasst werden.Dem Modul werden außerdem Restverbindungen und maximale Pooling-Operationen hinzugefügt.Es kann das Interaktionsmuster zwischen den beiden dynamisch erlernen, während die einzigartigen Informationen jeder Modalität erhalten bleiben, und letztendlich eine umfassendere Darstellung der Interaktion ausgeben.
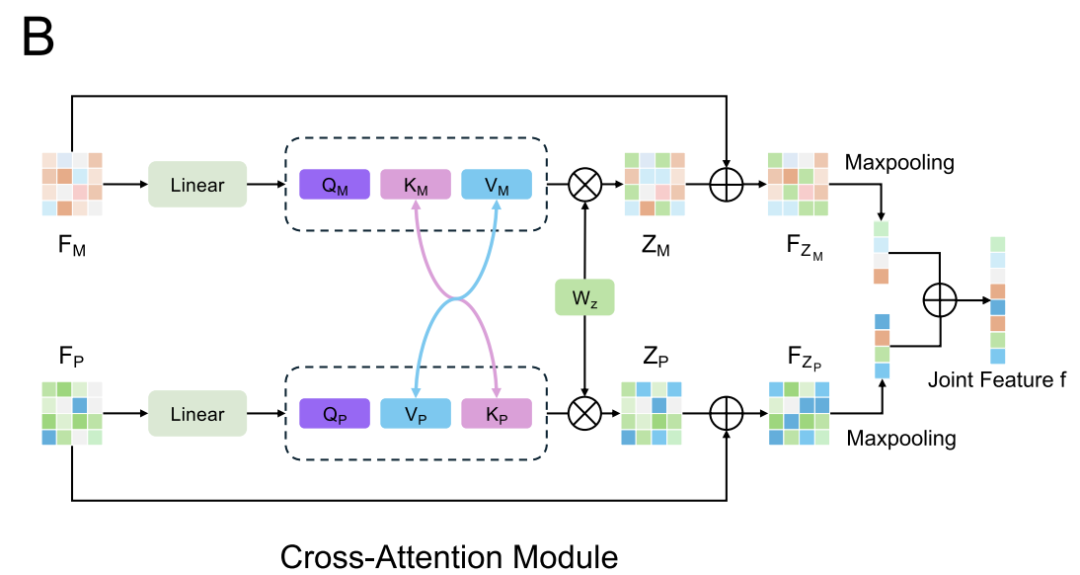
Angesichts der Unterschiede in der Merkmalsverteilung zwischen Datensätzen – DiPPI konzentriert sich beispielsweise auf schnittstellenspezifische Modulatoren, während allgemeine Datensätze wie DLiP solche Informationen nicht enthalten – führt AlphaPPIMI außerdem ein Conditional Domain Adversarial Network (CDAN) ein, wie in der folgenden Abbildung dargestellt. CDAN verwendet eine „gemeinsame Darstellung von Merkmalseinbettung und Klassifizierungsvorhersage“ als Bedingung für den Domänendiskriminator. Dadurch bleiben diskriminierende Merkmale erhalten, während eine Verteilungsausrichtung zwischen Quell- und Zieldomäne erreicht wird.Der Trainingsprozess verwendet ein Minimax-Spiel: Der Feature-Encoder und das Cross-Attention-Modul sind für die Generierung domäneninvarianter Darstellungen verantwortlich, während der Diskriminator zur Unterscheidung der Feature-Quellen verwendet wird.Dieser Mechanismus verbessert die Generalisierungsfähigkeit des Modells über verschiedene Proteinfamilien hinweg erheblich und bietet eine robustere Unterstützung für die Identifizierung neuer, auf Schnittstellen ausgerichteter Regulatoren.
Bewertung und Anwendungsüberprüfung der domänenübergreifenden Generalisierungsfähigkeit von AlphaPPIMI
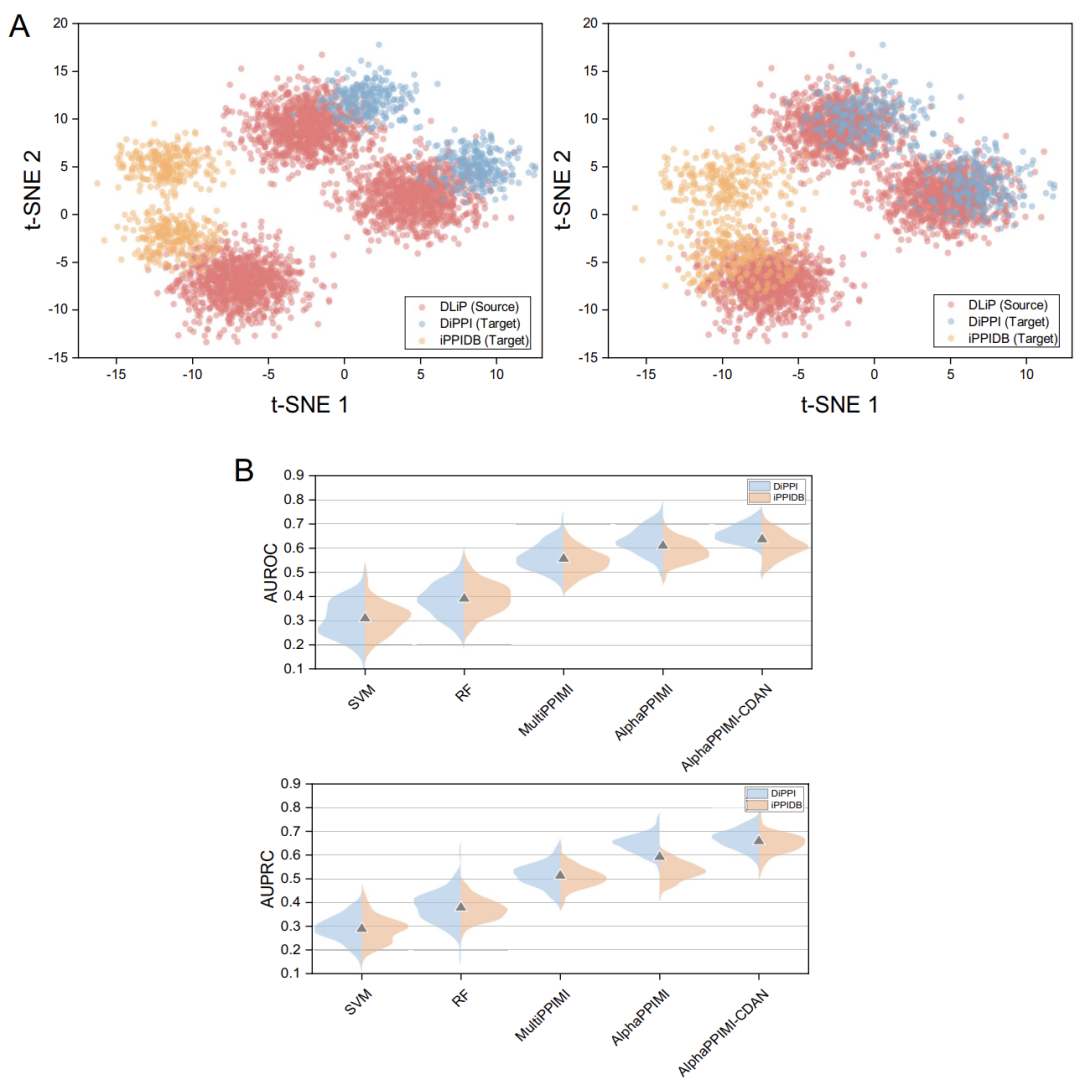
Um die domänenübergreifende Anpassungsfähigkeit von AlphaPPIMI bei der Vorhersage von PPI-Regulatoren zu testen, entwarf das Forschungsteam ein Migrationsexperiment.Der DLiP-Datensatz wird als „Quelldomäne“ (für das Modelltraining verwendete Daten) und die DiPPI- und iPPIDB-Datensätze als „Zieldomänen“ (für die Modellvalidierung verwendete Daten) betrachtet.
Experimentelle Ergebnisse zeigen, dass die Leistung aller Modelle bei Domänenverschiebung nachlässt, AlphaPPIMI jedoch robuster ist.Seine AUROC und AUPRC auf DiPPI sind deutlich höher als die von MultiPPIMI. Die Lücke zwischen der domänenübergreifenden und der domäneninternen Leistung unterstreicht die Notwendigkeit von Domänenanpassungsstrategien. Wie in der folgenden Abbildung gezeigt, wurde in der Studie außerdem die AlphaPPIMI-CDAN-Architektur vorgeschlagen, die eine domänenübergreifende Verteilungsanpassung durch bedingte Merkmalsausrichtung erreicht. Dieses Modell übertrifft die Basismodelle auf DiPPI und iPPIDB deutlich. Anders als herkömmliche Methoden der Kantenausrichtung leitet diese Methode die Merkmalsausrichtung basierend auf der kategoriebedingten Verteilung und erzeugt so diskriminierendere Darstellungen. Sie kann die durch subtile funktionale Unterschiede in PPI-Domänen verursachte Verteilungsverschiebung wirksam angehen und gleichzeitig den negativen Transfer abmildern, um die Robustheit und Generalisierung der domänenübergreifenden Vorhersage zu verbessern.
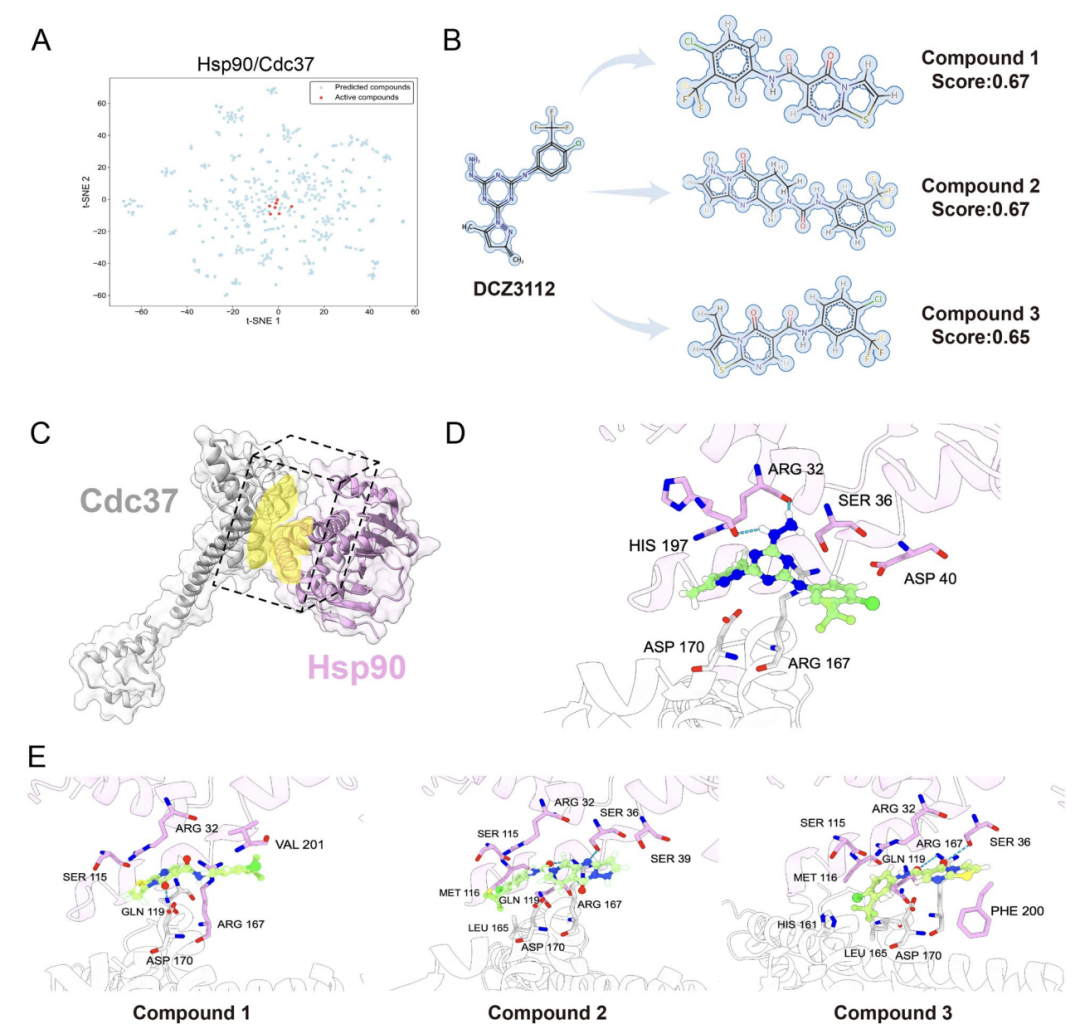
Bei der praktischen Anwendungsüberprüfung wurden in der Studie auch Hsp90-Cdc37-PPIs als Ziel genommen, die eine klare Schnittstellenstruktur aufweisen und ein wichtiges Ziel in der Krebsbekämpfung darstellen.Wie in Abbildung A unten gezeigt, hat AlphaPPIMI Verbindungen mit Vorhersagewerten > 0,8 in der ChemDiv-Bibliothek gescreent, deren chemischer Raum der Verteilung bekannter aktiver Inhibitoren nahe kam; wie in Abbildung B unten gezeigt, verwendeten die Forscher den verifizierten Inhibitor DCZ3112 als Referenz und filterten drei Kandidatenverbindungen durch strukturelle Ähnlichkeit und Pharmakophoranalyse heraus; wie in den Abbildungen D–E unten gezeigt, zeigte das molekulare Docking, dass diese Verbindungen ähnliche Wechselwirkungen wie Referenzmoleküle mit Schlüsselresten wie Arg32 und Ser36 eingehen können, wodurch ihr Hemmpotenzial verstärkt wird.
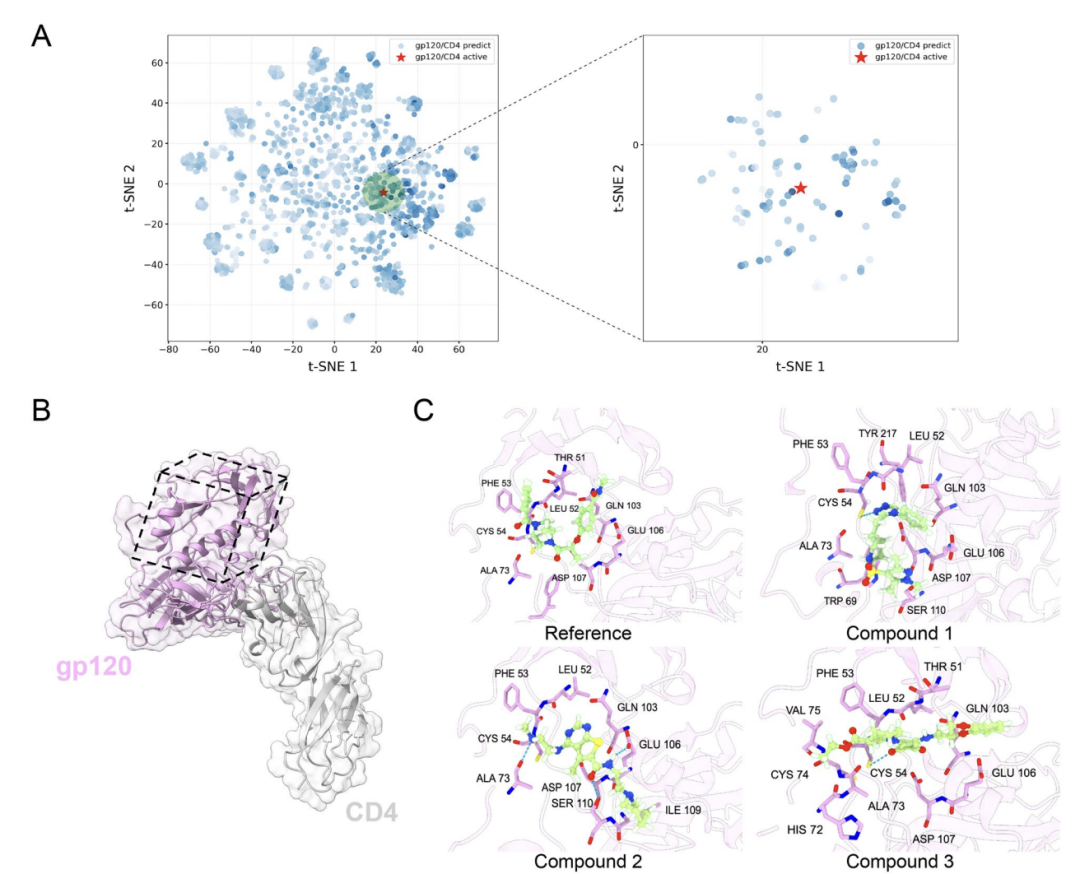
Was die Anwendung von AlphaPPIMI beim Screening allosterischer PPI-Modulatoren betrifft, so nahmen die Forscher, wie in Abbildung A unten dargestellt, die Interaktion zwischen HIV-1 gp120 und CD4 als Beispiel. AlphaPPIMI filterte Verbindungen mit einer vorhergesagten Wahrscheinlichkeit von über 0,8 heraus, deren chemischer Raum eine hohe Überlappung mit bekannten aktiven Inhibitoren aufwies. Wie in Abbildung BC unten dargestellt, zeigten die Ergebnisse des molekularen Dockings basierend auf der atypischen Schnittstellenstruktur (PDB: 6L1Y), dass die Kandidatenverbindungen mit Schlüsselresten wie THR51, LEU52 und PHE53 interagieren könnten. Dies weist darauf hin, dass AlphaPPIMI allosterische Inhibitoren entdecken kann, die auf die Schnittstelle schwer medikamentös behandelbarer PPIs abzielen, und bietet einen neuen Ansatz für die Entwicklung verwandter Medikamente.
Zusammenarbeit zwischen Industrie, Universitäten und Forschung fördert PPI-spezifische Medikamente von der Grundlagenforschung bis zur klinischen Anwendung
Bei der Erforschung und Entwicklung zielgerichteter PPI-Medikamente arbeiten Wissenschaft und Industrie eng zusammen, um die Grundlagenforschung auf diesem Gebiet schrittweise in Richtung klinischer Anwendung zu treiben.
An der akademischen Front erforschen viele Forschungsteams genauere und effizientere Methoden zur Vorhersage und Zielausrichtung von PPIs.Beispielsweise hat ein Team der Stanford University einen allgemeinen biomedizinischen KI-Agenten namens Biomni entwickelt.Dieser intelligente Agent ist in der Lage, komplexe Forschungsaufgaben in verschiedenen biomedizinischen Disziplinen, darunter Genetik, Genomik, Mikrobiologie, Pharmakologie und klinische Medizin, autonom zu erledigen. Die Entwicklung von Biomni markiert den Übergang der KI in der biomedizinischen Forschung vom Werkzeugnutzer zum autonomen Entscheidungsträger. Durch die Integration verteilter wissenschaftlicher Forschungsressourcen in umsetzbare, intelligente, agentenbasierte Verhaltenseinheiten überwindet Biomni nicht nur die fragmentierten Engpässe traditioneller Forschungsprozesse, sondern fördert potenziell auch die Entstehung einer disziplinübergreifenden, hochdurchsatzfähigen und autonomen wissenschaftlichen Entdeckungsmaschine.
Eine weitere repräsentative StudieDie Sun Yat-sen-Universität hat eine PPI-Vorhersagemethode vorgeschlagen, die auf der Fusionsmerkmalsextraktion und einem neuen unbeaufsichtigten Merkmalsauswahlmechanismus basiert.Umfangreiche Experimente zeigten, dass die vorgeschlagene Methode bei fünf Datensätzen, die sowohl Interaktionen innerhalb als auch zwischen Arten abdecken, gute Ergebnisse lieferte und 16 bestehende Methoden des maschinellen Lernens deutlich übertraf. Diese Forschung bietet nicht nur einen effizienten und zuverlässigen Rahmen für groß angelegte PPI-Vorhersageaufgaben, sondern zeigt auch eine breite funktionale Anpassungsfähigkeit und bietet eine neue Lösung für die Vorhersage von Wechselwirkungen zwischen Arzneimitteln und zwischen Arzneimitteln und Lebensmitteln.
Auch bei der industriellen Umsetzung treiben Unternehmen diese akademischen Durchbrüche aktiv in die Klinik voran. Beispielsweise ist AN2025 (generischer Name Buparlisib), das vom chinesischen Biopharmaunternehmen Adlai Nortye auf Basis einer weltweiten Lizenz von Novartis entwickelt wurde, ein Pan-Inhibitor, der speziell auf den PI3K-Signalweg abzielt. Der Wirkstoff befindet sich derzeit in einer weltweiten klinischen Phase-III-Studie, in erster Linie für die Behandlung von Patienten mit rezidiviertem oder metastasiertem Plattenepithelkarzinom im Kopf-Hals-Bereich, deren Krankheit trotz Anti-PD-1/PD-L1-Therapie fortgeschritten ist.
Ein weiteres Beispiel ist Iqirvo (Elafibranor), das vom renommierten französischen Pharmaunternehmen Ipsen auf den Markt gebracht wurde. Als erste neue PBC-Behandlung des letzten Jahrzehnts bestätigte es den klinischen Wert der PPI-Regulierung im nicht-tumorösen Bereich und schuf ein neues Behandlungsparadigma für komplexe Stoffwechselerkrankungen. Seine Zulassung förderte zudem eine eingehende Erforschung des Proteininteraktionsnetzwerks der PPAR-Familie.
Die enge Zusammenarbeit zwischen Wissenschaft und Industrie im Bereich der PPI-gerichteten Medikamente hat nicht nur die Umsetzung wissenschaftlicher Forschungsergebnisse in klinische Anwendungen beschleunigt, sondern auch die Effizienz und Erfolgsquote der Entwicklung neuer Medikamente deutlich verbessert. Von multimodalen KI-Vorhersagemodellen bis hin zu Medikamentenkandidaten mit klarem klinischen Nutzen – diese fachübergreifende Zusammenarbeit definiert den Weg biomedizinischer Innovationen neu. Durch die Integration weiterer Daten und Algorithmen sowie die Vertiefung der institutionen- und interdisziplinären Zusammenarbeit könnten PPI-gerichtete Medikamente in Zukunft noch mehr Durchbrüche bei der Behandlung komplexer Krankheiten bringen.
Referenzlinks:
1.https://mp.weixin.qq.com/s/ryYJ6T7qEjnjvkhBL4-dAA
2.https://mp.weixin.qq.com/s/7upIPYam1LR0TiGBYXmkOw
3.https://mp.weixin.qq.com/s/69GU1R5lXHdTLttlT8apyw








