Command Palette
Search for a command to run...
Die University of Pennsylvania Hat 386 Neue Antimikrobielle Peptide Aus Tiergiften Entdeckt Und Ein Deep-Learning-Modell Namens APEX Entwickelt, Um Potenzielle Antibiotikakandidaten Zu screenen.

Die Entdeckung des Penicillins im Jahr 1928 läutete eine neue Ära im Kampf gegen bakterielle Infektionen ein. Diese Antibiotika verbesserten die Fähigkeit der Menschheit, Infektionskrankheiten zu bekämpfen, erheblich. Der weit verbreitete und oft übermäßige Einsatz von Antibiotika in Bereichen wie Medizin und Landwirtschaft führte jedoch zu bakteriellen Resistenzen, die sich in besorgniserregendem Tempo verschlimmern.
Zu Beginn des 21. Jahrhunderts ist die Antibiotikaresistenz zu einer großen Herausforderung für die globale öffentliche Gesundheit geworden. Laut Angaben der Weltgesundheitsorganisation sterben jährlich weltweit etwa fünf Millionen Menschen direkt oder indirekt an medikamentenresistenten Infektionen. Der Hauptgrund für diese Krise ist das schnelle Wachstum und die Verbreitung antibiotikaresistenter Krankheitserreger. Gramnegative Bakterien, die von der WHO als „prioritäre Krankheitserreger“ eingestuft werden, sind dabei besonders problematisch. Aufgrund ihrer ausgeprägten Fähigkeit zur Entwicklung von Medikamentenresistenzen haben sie im „Krieg der Antibiotika gegen den Menschen“ weiterhin die Oberhand. Noch schwerwiegender ist, dass in den letzten JahrzehntenDie Forschung und Entwicklung neuer Antibiotika stagniert: Hohe Forschungs- und Entwicklungskosten und lange klinische Testzyklen halten viele Pharmaunternehmen davon ab, in dieses Feld einzusteigen. Die Zahl neuer Antibiotika ist stark zurückgegangen und hinkt der Entwicklung resistenter Bakterien weit hinterher.
Angesichts dieses Dilemmas haben Wissenschaftler begonnen, ihre Aufmerksamkeit auf eine potenzielle „Schatzkammer“ der Natur zu richten: Tiergift. Nach Millionen von Jahren natürlicher Evolution hat Tiergift eine extrem reiche molekulare Vielfalt hervorgebracht, darunter bioaktive Peptide und Proteine.Es kann nicht nur mit einer Vielzahl biologischer Ziele interagieren, sondern weist auch eine signifikante antibakterielle Aktivität auf.Tatsächlich ist die Anwendung von Gift in der biomedizinischen Wissenschaft bereits eine Erfolgsgeschichte. Ziconotid (Handelsname Prialt®), gewonnen aus dem Gift von Kegelschnecken, hat sich zu einem wichtigen Analgetikum zur Behandlung chronischer Schmerzen entwickelt; Captopril, gewonnen aus Schlangengift, ist ein häufig eingesetztes blutdrucksenkendes Medikament in der klinischen Praxis. Diese Beispiele liefern wichtige Erkenntnisse für die Erforschung von Gift im antimikrobiellen Bereich.
In diesem ZusammenhangEin Forschungsteam der University of Pennsylvania in den USA hat im Hochdurchsatzverfahren antimikrobielle Kandidatenpeptide aus Giftproteinen aus aller Welt gescreent und dabei 386 antimikrobielle Peptide mit neuartigen Strukturen aus Tiergiften aus aller Welt entdeckt.Unter ihnen zeigte das experimentell verifizierte Molekül 91.4% eine starke antibakterielle Aktivität und lieferte eine bahnbrechende Idee für die Entdeckung einer neuen Generation von Antibiotika.
Die entsprechenden Forschungsergebnisse wurden in Nature Communications unter dem Titel „Computational exploration of global venoms for antimicrobial discovery with Venomics artificial intelligence“ veröffentlicht.
Forschungshighlights:
* Im Rahmen dieser Studie wurde eine globale Giftdatenbank erstellt und eine Kandidatenbibliothek verschlüsselter Peptide generiert, die insgesamt 16.123 Giftproteine umfasste und mehr als 40 Millionen verschlüsselte Giftpeptide generierte;
* In dieser Studie wurde das Deep-Learning-Modell APEX verwendet, um die minimale Hemmkonzentration jedes Peptids gegen 34 Bakterienarten vorherzusagen. Dabei wurden letztendlich 386 Kandidatenpeptide mit antibakteriellem Potenzial und geringer Sequenzähnlichkeit zu bekannten AMPs herausgefiltert.
* Das Forschungsteam synthetisierte und testete 58 Peptide aus 386 VEPs, von denen 91.4% (53) signifikante hemmende Wirkungen auf mindestens einen Krankheitserreger hatten.

Papieradresse:
https://www.nature.com/articles/s41467-025-60051-6[*](https://arxiv.org/pdf/2507.09466*)
Folgen Sie dem offiziellen Konto und antworten Sie mit „APEX“, um das vollständige PDF zu erhalten
Datenbank: Sammlung von 16.000 Giftproteinen
Um neue antimikrobielle Kandidaten zu identifizieren, wurden im Rahmen der Studie Giftproteinsequenzen aus vier Datenbanken gesammelt: ConoServer, ArachnoServer, ISOB und VenomZone. Insgesamt wurden 16.123 Giftproteine erfasst. ConoServer konzentriert sich auf Conopeptide, ArachnoServer umfasst Spinnengifte, ISOB ist eine Datenbank mit Giften einheimischer Schlangenarten aus Bangladesch und VenomZone, zusammengestellt aus UniProtKB, umfasst Proteine aus sechs Taxa: Schlangen, Skorpione, Spinnen, Kegelschnecken, Seeanemonen und Insekten, die in dieser Studie von UniProt vertreten wurden.
Jeder dieser Datensätze weist einzigartige Eigenschaften und Verwendungszwecke auf. Wie die folgende Abbildung zeigt, zeigt eine Analyse der Artenüberschneidungen zwischen verschiedenen Datenbanken, dass UniProt mit 699 die größte Anzahl einzigartiger Arten enthält, was auf eine größere Proteinvielfalt hindeutet. ConoServer und ArachnoServer hingegen enthalten relativ kleine Untergruppen einzigartiger Arten, während ISOB überhaupt keine einzigartigen Arten aufweist.Die Existenz dieser Vielfalt ermöglicht es Forschern, antimikrobielle Peptide im Gift aus mehreren Blickwinkeln zu untersuchen, Proteininformationen aus verschiedenen Quellen zu integrieren und die Möglichkeit zu erhöhen, neue antimikrobielle Peptide zu entdecken.
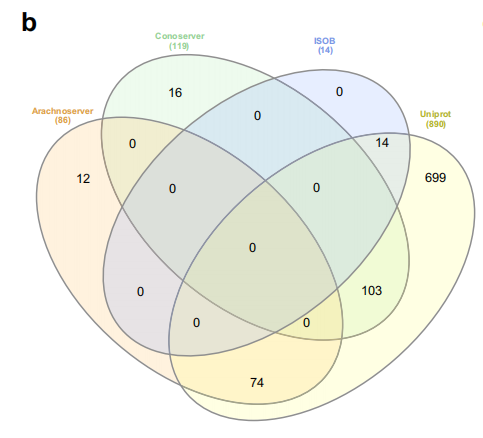
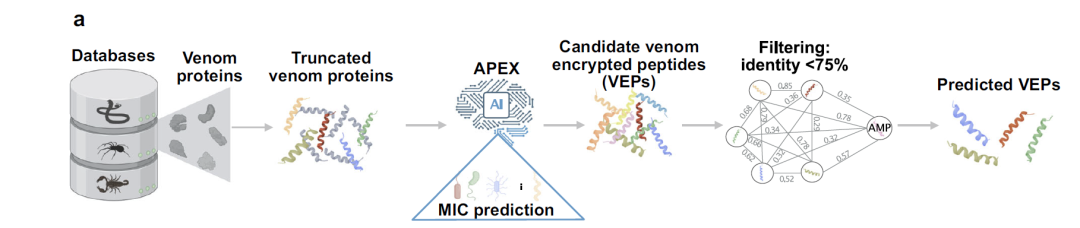
Wie in der Abbildung unten dargestellt, verarbeiteten die Forscher diese Giftproteine, um über 40 Millionen verschlüsselte Giftpeptide (VEPs) zu erzeugen. Das APEX-Deep-Learning-Modell wurde dann verwendet, um die antimikrobielle Aktivität dieser Peptide vorherzusagen, indem die minimale Hemmkonzentration (MHK) für jedes Peptid gegen Bakterienstämme geschätzt wurde, wobei die mittlere MHK als Maß für die antimikrobielle Aktivität diente. Durch weiteres Screening identifizierten die Forscher schließlich 7.379 VEPs mit einer mittleren MHK ≤ 32 μmol L⁻¹. Nach dem Filtern auf Sequenzähnlichkeit mit bekannten antimikrobiellen Peptiden wurden 386 Kandidaten identifiziert. Diese Kandidaten unterscheiden sich strukturell und funktionell von bekannten antimikrobiellen Peptiden und stellen wertvolle Ziele für die anschließende experimentelle Validierung dar.
APEX-Modellvorhersage: Ein Prozess zur Integration von Giftproteomdaten aus mehreren Quellen und zum Screening antimikrobieller Kandidatenpeptide
Das APEX-Modell ist ein Deep-Learning-Tool, das sich auf das potenzielle Mining und die Vorhersage antimikrobieller Peptide konzentriert.Sein Hauptvorteil liegt in der Fähigkeit, die antimikrobielle Aktivität bakterienstammspezifisch vorherzusagen. Nach systematischem Training kann das Modell die minimale Hemmkonzentration (MHK) von Peptiden für 34 verschiedene Bakterienstämme genau vorhersagen – ein Schlüsselparameter zur Messung der Wirksamkeit antimikrobieller Wirkstoffe.
Der Trainingsdatensatz des Modells besteht aus zwei Teilen:Intern gesammelte Peptidsequenzdaten und öffentliche Informationen zu antimikrobiellen Peptiden (AMPs), die aus der DBAASP-Datenbank bezogen wurden.Diese diversifizierte Datenunterstützung ermöglicht APEX eine hervorragende Leistung bei der Analyse der komplexen Zuordnungsbeziehung zwischen Peptidsequenzen und antibakterieller Aktivität und legt so eine solide algorithmische Grundlage für das nachfolgende Screening.
In Bezug auf das Architekturdesign verwendet APEX ein mehrzieliges Aufgabenmodell-Framework und eine „Drei-Schritte“-Strategie, um Kandidatenmoleküle für antimikrobielle Peptide von Giftproteinen abzugrenzen.Erste,Zur Konstruktion einer Peptidbibliothek wurde die Sliding-Window-Methode verwendet und Fragmente mit Längen im Bereich von 8 bis 50 Aminosäuren ausgewählt.Zweitens,Das APEX-Modell wurde verwendet, um die minimale Hemmkonzentration (MHK) dieser Peptide gegen verschiedene Bakterienstämme vorherzusagen;endlich,Eine weitere Selektion auf der Grundlage von Sequenzähnlichkeiten führte zu einer Gruppe von Gift-verschlüsselten Peptiden (VEPs) mit neuartigen Strukturen und hervorragender Aktivität.
Dieses Design ermöglicht die präzise Identifizierung von Kandidatenmolekülen mit potenzieller antimikrobieller Aktivität bei der Verarbeitung großer Mengen von Gift-verschlüsselten Peptiden (VEPs). Im Vergleich zu herkömmlichen experimentellen Screening-Methoden verbessert APEX die Effizienz der Entdeckung antimikrobieller Peptide deutlich und reduziert gleichzeitig die Abhängigkeit von ressourcenintensiven biochemischen Tests. Forschern steht damit ein schnellerer und kostengünstigerer Screening-Prozess zur Verfügung.
In der Praxis erfordert die Verwendung des APEX-Modells gewisse technische Vorbereitungen. Forscher müssen eine geeignete Laufzeitumgebung einrichten, einschließlich der Installation von Python 3.9, einer bestimmten Version von PyTorch und einer Reihe abhängiger Bibliotheken wie Numpy, Scipy und Matplotlib. Sobald die Umgebung eingerichtet ist, werden die Kandidatenpeptidsequenzen in einer Textdatei gespeichert. Durch Ausführen des angegebenen Befehls generiert das Modell eine CSV-Datei mit den MIC-Vorhersageergebnissen. Durch die Analyse dieser Vorhersagen können Forscher schnell Peptide mit hohem antimikrobiellen Potenzial für weitere experimentelle Überprüfungen identifizieren.
Überprüfung der antibakteriellen Aktivität von VEPs und Analyse ihres Wirkmechanismus
Um die antibakterielle Aktivität von Gift-verschlüsselten Peptiden (VEPs) zu überprüfen, führte das Forschungsteam eine Reihe von Experimenten durch.Im antimikrobiellen Aktivitätstest wurden 58 VEPs gegen eine Vielzahl von Krankheitserregern getestet.Die Ergebnisse sind in der folgenden Abbildung dargestellt. 53 davon (entsprechend 91,41 TP3T) zeigten Aktivität gegen mindestens einen Erreger.Alle VEPs aus der ArachnoServer-Datenbank zeigten antibakterielle Aktivität, was das starke antibakterielle Potenzial dieses Peptidtyps bestätigte.

Einige VEPs aus der UniProt-Datenbank zeigten jedoch eine relativ begrenzte Wirksamkeit. So zeigte UniprotKB-2 keine antibakterielle Aktivität, und UniprotKB-6 und UniprotKB-11 wirkten lediglich hemmend auf Enterococcus faecalis. Weitere Analysen ergaben, dass diese Peptide mit geringer Aktivität im Allgemeinen eine geringe Hydrophobie und Nettoladung aufwiesen – ein Befund, der darauf hindeutet, dassHydrophobie und Nettoladung spielen eine Schlüsselrolle bei der Förderung der Interaktion zwischen Peptiden und Bakterienmembranen, und ihre Konzentrationen können die antibakterielle Wirksamkeit von VEPs direkt beeinflussen.
In der Sekundärstrukturstudie beobachtete das Team mittels Zirkulardichroismus (CD), dass die Strukturveränderungen von VEPs stark von der Umgebung abhängen. In wässriger Lösung liegen VEPs überwiegend in ungeordneter Struktur vor; in einer simulierten Bakterienmembranumgebung (wie einem SDS-Mizellensystem) oder einem helikalen Induktionsmedium (wie einer Trifluorethanol-Wasser-Mischlösung) durchlaufen sie jedoch einen signifikanten Konformationsübergang und falten sich von einer ungeordneten Struktur zu einer α-Helix. Dieses Strukturmerkmal steht im Einklang mit dem Verhalten typischer antimikrobieller Peptide.Dies deutet darauf hin, dass VEPs möglicherweise speziell an Biofilm-bezogene Funktionen angepasst sind, was auch wichtige Hinweise auf ihren antibakteriellen Mechanismus liefert.
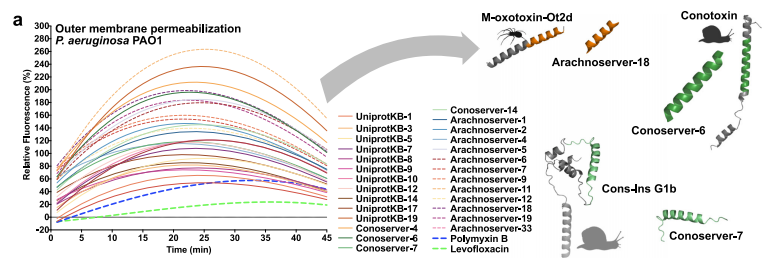
Im Rahmen der Wirkmechanismusforschung untersuchte das Team die Wirkung von VEPs auf bakterielle Außenmembranen und Zytoplasmamembranen mittels Fluoreszenztests. Die Ergebnisse sind in der folgenden Abbildung dargestellt. 23 VEPs können die bakterielle Außenmembran effektiv durchdringen, wobei Arachnoserver-18, ConoServer-6 und ConoServer-7 besonders gute Permeabilitätseigenschaften aufweisen. Im Experiment zur Depolarisation der Zytoplasmamembran war die Depolarisationswirkung der meisten getesteten VEPs besser als die der Kontrollgruppe, jedoch etwas schwächer als die einiger bekannter Peptidfamilien. InsgesamtDie antibakterielle Wirkung von VEPs beruht hauptsächlich auf der Depolarisation der zytoplasmatischen Membran. Dieser Mechanismus steht im Einklang mit der Wirkungsweise vieler bekannter antimikrobieller Peptide und Verschlüsselungspeptide und unterstreicht damit ihre Eignung als neues antibakterielles Mittel.
Wissenschaft und Industrie arbeiten zusammen, um neue antimikrobielle Medikamente mit geringer Resistenz zu erforschen und zu entwickeln
Um neue antimikrobielle Medikamente zu entwickeln, die mit geringerer Wahrscheinlichkeit Resistenzen hervorrufen, wird durch die Zusammenarbeit von Wissenschaft und Wirtschaft – von der Grundlagenforschung bis zur industriellen Umsetzung – ständig das Anwendungspotenzial antimikrobieller Peptide aus Giftstoffen erschlossen.
Im akademischen Bereich haben Forschungsteams aus verschiedenen Ländern eingehende Forschungen zur Entdeckung, Entwicklung und Mechanismusanalyse antimikrobieller Peptide in Giften durchgeführt. Das Team von Mu Yuguang an der Nanyang Technological University in Singapur befasste sich mit dem Problem, dass traditionelle Modelle bei der Vorhersage von Proteininteraktionen durch die Datenmenge und Merkmalsdimensionen eingeschränkt sind.Konstruierte den bislang größten strukturbasierten Datensatz zur Proteininteraktion.Sie entwickelten außerdem das Deep-Learning-Framework ProAffinity-GNN. Dieses Modell integriert auf innovative Weise Proteinsprachenmodelle mit Graph-Neural-Networks und verknüpft Sequenzinformationen eng mit räumlichen Strukturmerkmalen. Es übertrifft nicht nur bestehende Methoden in der Genauigkeit der Protein-Protein-Affinitätsvorhersage, sondern bietet mit seinen leistungsstarken Generalisierungsmöglichkeiten auch ein technisches Paradigma für die effiziente Vorhersage von Wechselwirkungen zwischen Giftpeptiden und bakteriellen Zielen. Darüber hinaus liefert die Analyse des Wirkmechanismus theoretische Unterstützung für die optimierte Entwicklung antimikrobieller Peptide.
Ein Team des Qilu Medical College der Shandong University verwendete das Diffusionsmodell im Bereich der künstlichen Intelligenz, um den „random perturbation-directed screening“-Prozess der Peptidevolution in der Natur zu simulieren.Es wurden erfolgreich 40 neue Peptidsequenzen erzeugt, von denen 25 eine klare antibakterielle oder antimykotische Aktivität zeigten.
In der Geschäftswelt konzentriert sich die israelische Bountica Company auf die Schwachstellen im Bereich der Lebensmittelkonservierung und nutzt mikrobielle Fermentationstechnologie zur Massenproduktion antimykotischer Peptide aus Gift. Seine Produktreihe VenomShield,Nur ein Teil pro Million ist erforderlich, um das Wachstum von Schimmel und Hefe in Brot und Saft zu hemmen, wodurch die Haltbarkeit im Vergleich zu herkömmlichen Konservierungsmitteln mehr als verdoppelt wird.
China Shanghai Hi-Tech Bioengineering Co., Ltd. kooperiert mit der Fudan-Universität,Gemeinsam haben wir weltweit drei relativ vollständige Datenbanken für antimikrobielle Proteine aufgebaut.Diese Datenbanken bieten nicht nur effiziente Werkzeuge für die Forschung und Entwicklung neuer biologischer antimikrobieller Enzyme und antimikrobieller Peptide, sondern beschleunigen durch systematisches Data Mining auch die Erforschung neuer Substanzen gegen medikamentenresistente Mikroorganismen.
Dies zeigt die enorme Vitalität der Forschung und Entwicklung antimikrobieller Peptide aus Giften, die sowohl die Grundlagenforschung als auch die industrielle Anwendung umfasst. Deep-Learning-Modelle analysieren Sequenz-Funktions-Beziehungen immer präziser und synthetische Biologietechniken optimieren kontinuierlich die Stabilität und Expressionseffizienz der Peptide. Diese von der „Überlebensweisheit“ der Natur inspirierten Moleküle könnten zu einer Schlüsselkraft im Kampf gegen Antibiotikaresistenzen werden und neue Schutzmechanismen für die globale öffentliche Gesundheit bieten.
Referenzartikel:
1. https://mp.weixin.qq.com/s/lwYWWIe9-Az22jlhiZ747A
2. https://mp.weixin.qq.com/s/sGHRf-ebaSRiqGiUxMMueA
3. https://mp.weixin.qq.com/s/0mJltGuaKTUwYDGK4wD9UA
4. https://mp.weixin.qq.com/s/R6Y_-38saZwqSSow0LNhzw








