Command Palette
Search for a command to run...
Basierend Auf 8 Millionen Realen Daten Verwendete Das Team Der Cornell University Graph-Neural-Networks, Um Das Überleben Von Lungenkrebspatienten Genau Vorherzusagen Und Entdeckte 3 Tödliche Subtypen
Vor zehn Jahren schockierten die Ergebnisse der CheckMate 017-Studie die Onkologie-Gemeinschaft. Im New England Journal of Medicine, im Journal of the American Medical Association und in anderen Fachzeitschriften wurde mehrfach berichtet, dass sich die Überlebensdaten von Patienten mit fortgeschrittenem Plattenepithelkarzinom der Lunge, die mit dem PD-1-Hemmer Nivolumab behandelt wurden, deutlich verbesserten: Die mediane Gesamtüberlebenszeit erhöhte sich von 6 Monaten bei Chemotherapie auf 9,2 Monate, und die 18-Monats-Überlebensrate war doppelt so hoch wie in der Chemotherapiegruppe. Diese Studie markiert den Beginn der Ära der Immun-Checkpoint-Inhibitoren (ICI), zeigt aber auch das Problem auf, dass Patienten mit fortgeschrittenem nicht-kleinzelligem Lungenkrebs (aNSCLC) unterschiedlich auf die Immuntherapie reagieren:Bei einigen Patienten blieben die Tumoren in der Studie über drei Jahre lang zurück, während bei anderen die Krankheit innerhalb weniger Monate fortschritt. Diese Heterogenität der Behandlungsreaktion ist im Zeitalter der Präzisionsmedizin zu einem Problem geworden.
Die Komplexität des Lungenkrebses ergibt sich aus seiner hohen Heterogenität. Nicht-kleinzelliger Lungenkrebs (NSCLC) macht 80%-85% des Lungenkrebses aus.Bei etwa 751 TP3T-Patienten wird die Krankheit im fortgeschrittenen Stadium diagnostiziert und die 5-Jahres-Überlebensrate beträgt nur 26,41 TP3T.Die unterschiedliche Expression von Biomarkern im Tumormikroumfeld, verschiedene Funktionszustände der Immunzellen und vielfältige Komorbiditäten der Patienten machen die pathologische Situation kompliziert. Patienten, die eine ICI-Behandlung erhalten, können aufgrund der hohen PD-L1-Expression davon profitieren, erfahren jedoch möglicherweise auch eine geringere Wirksamkeit aufgrund der geringen Tumormutationslast. Auch Komorbiditäten können die Behandlungsmöglichkeiten und die Prognose beeinflussen.
Um diesen Herausforderungen gerecht zu werden, vollzieht sich bei Diagnose- und Behandlungsplänen ein Wandel von der Einheitslösung hin zur präzisen Schichtung. In diesem Transformationsprozess ist nach und nach die prädiktive Medizin entstanden. Sein Hauptziel besteht darin, mehrdimensionale Daten, einschließlich elektronischer Gesundheitsakten und Omics-Informationen, zu integrieren, um für jeden Patienten den optimal geeigneten Behandlungsplan zu erstellen. Angesichts der kontinuierlichen Anhäufung umfangreicher biomedizinischer Daten und der rasanten Entwicklung der Technologie des maschinellen Lernens haben Forscher in den letzten Jahren damit begonnen, mithilfe von Methoden des unbeaufsichtigten maschinellen Lernens Patientengruppen mit ähnlichen Merkmalen zu clustern und so die Reaktion auf Behandlungen vorherzusagen. Leider sind traditionelle Methoden in der praktischen Anwendung jedoch häufig mit Einschränkungen verbunden.Es ist schwierig, die Konsistenz der Überlebensergebnisse zwischen den Patienten innerhalb der Gruppe sicherzustellen, was den Anwendungswert stratifizierter Ergebnisse in der klinischen Praxis einschränkt.
Um die oben genannten Probleme zu lösen, haben die Cornell University und Regeneron Pharmaceuticals das Graph Encoded Mixed Survival Model (GEMS) vorgeschlagen.Komplexe Beziehungen in den elektronischen Gesundheitsakten der Patienten wurden durch Graph-Neural-Netzwerke kodiert und mit Überlebensanalysemodellen kombiniert, um Subphänotypen mit konsistenten Merkmalen und Überlebensergebnissen zu identifizieren.Die Studie ergab, dass es herkömmlichen Methoden bei der Vorhersage des Gesamtüberlebens (OS) überlegen ist. Es wurden drei Subphänotypen mit unterschiedlichen klinischen Merkmalen und Überlebensmustern identifiziert, wodurch ein neuer Weg für die Präzisionsmedizin bei Lungenkrebs eröffnet wird.
Die entsprechenden Forschungsergebnisse wurden in Nature Communication unter dem Titel „Identification of predictive subphenotypes for clinical outcomes using real world data and machine learning“ veröffentlicht.

Papieradresse:
https://doi.org/10.1038/s41467-025-59092-8
Das Open-Source-Projekt „awesome-ai4s“ vereint mehr als 100 AI4S-Papierinterpretationen und stellt außerdem umfangreiche Datensätze und Tools bereit:
https://github.com/hyperai/awesome-ai4s
Aufbau einer Patientenkohorte mit fortgeschrittenem nicht-kleinzelligem Lungenkrebs basierend auf dem umfangreichen Realwelt-Datensatz von ConcertAI
In der Studie wurde der ConcertAI Patient360™ NSCLC-Datensatz aus der US Oncology Electronic Health Record (EHR)-Datenbank verwendet, um eine Kohorte von Patienten mit fortgeschrittenem nicht-kleinzelligem Lungenkrebs (aNSCLC) zu erstellen, die eine Erstlinienbehandlung (1 L) mit Immun-Checkpoint-Inhibitoren (ICI) erhalten.Bei diesem Datensatz handelt es sich um einen anonymisierten Datensatz auf Patientenebene mit Sitz in den USA, der aus dem ConcertAI-Netzwerk extrahiert wurde und mehr als 8 Millionen einzelne Patienten umfasst.Es wurden Daten aus über 900 onkologischen und hämatologischen Krebskliniken extrahiert, die Patienten repräsentieren, die in Gemeinschaftspraxen und akademischen Praxen in allen 50 US-Bundesstaaten behandelt wurden. Dazu gehörten Daten zu Datum und Art des Wiederauftretens der Krankheit, Histologie, PD-L1-Testinformationen, Tumorreaktion, ECOG-PS und Komorbiditäten.
Wie in der Abbildung unten dargestellt, wurden für diese Studie Patienten mit histologisch bestätigtem nicht-kleinzelligem Lungenkrebs (NSCLC) zwischen Januar 2015 und Januar 2023 (n = 17.265) ausgewählt, um eine retrospektive Beobachtungskohorte zusammenzustellen. Nach den Einschluss-/Ausschlusskriterien und dem Ausschluss von Patienten ohne gültige Aufzeichnungen zum Gesamtüberleben (OS),4.666 Patienten wurden in die Studie aufgenommen und die Patienten wurden durch einen 104-dimensionalen Vektor dargestellt, dessen Dimensionen demografische Informationen, Labortests und andere Variablen umfassten.
Basierend auf den vom U.S. Census Bureau definierten geografischen Regionen der klinischen Einrichtungen teilten die Forscher die Kohorte in eine Modellentwicklung (Regionen Nordosten, Süden und Westen, n=3.225) und eine Validierungsunterkohorte (Region Mittlerer Westen, n=1.441) mit ähnlicher Demografie auf, wobei die Validierungsunterkohorte einen höheren Anteil weißer Patienten und Patienten aus kommunalen medizinischen Einrichtungen hatte. Der Beobachtungszeitraum der Studie betrug 180 Tage vor dem Indexdatum. Das Gesamtüberleben (OS) wurde als die Zeit vom Indexdatum bis zum Tod aus beliebiger Ursache definiert, und das progressionsfreie Überleben (PFS) wurde als die Zeit vom Indexdatum bis zum ersten realen Progressionsereignis oder Tod aus beliebiger Ursache definiert. Ziel der Studie ist es, durch eine entsprechende Analyse dieses Datensatzes Probleme wie die Vorhersage des Überlebens von Patienten mit fortgeschrittenem nicht-kleinzelligem Lungenkrebs zu lösen.
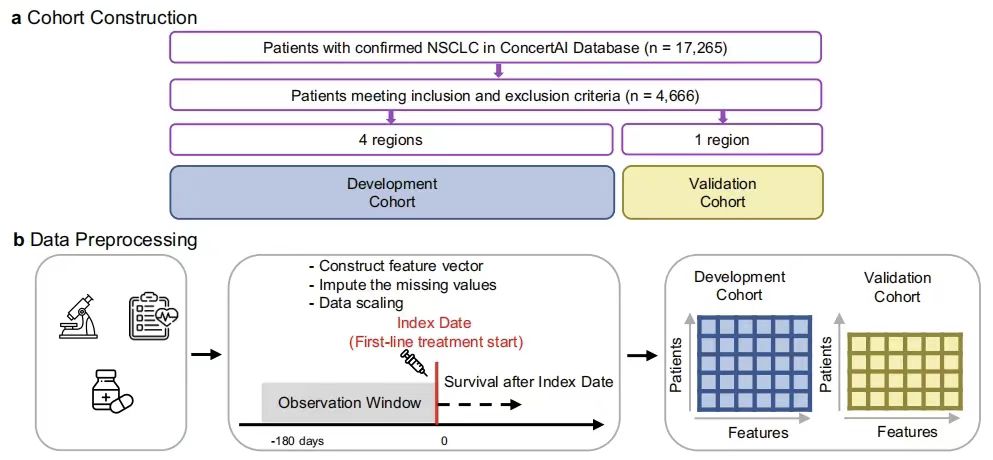
Konstruktion des GEMS-Modells: GNN-basierte Identifizierung von Überlebenssubphänotypen und Validierung der Vorhersageleistung für fortgeschrittenen nicht-kleinzelligen Lungenkrebs
In dieser Studie wurde das GEMS-Modell entwickelt, um prädiktive Subphänotypen zu identifizieren, die mit realen Gesamtüberlebensmerkmalen (OS) bei Patienten mit fortgeschrittenem nicht-kleinzelligem Lungenkrebs (aNSCLC) assoziiert sind.Seine Kernarchitektur umfasst GNN-Encoder, Cluster-Modul und Mixture Survival Predictor.
Unter diesen extrahiert der GNN-Encoder effektiv Patientendarstellungen höherer Ordnung, indem er die Graphstrukturbeziehung des 104-dimensionalen Merkmalsvektors des Patienten erfasst (der Variablen wie demografische Daten, Labortests und Metastasierungsstatus abdeckt). Die codierten Darstellungen werden in das Clustermodul eingegeben, um Subphänotypen mit Überlebensvorhersagewert als Grundkomponenten des Hybridmodells zu generieren.
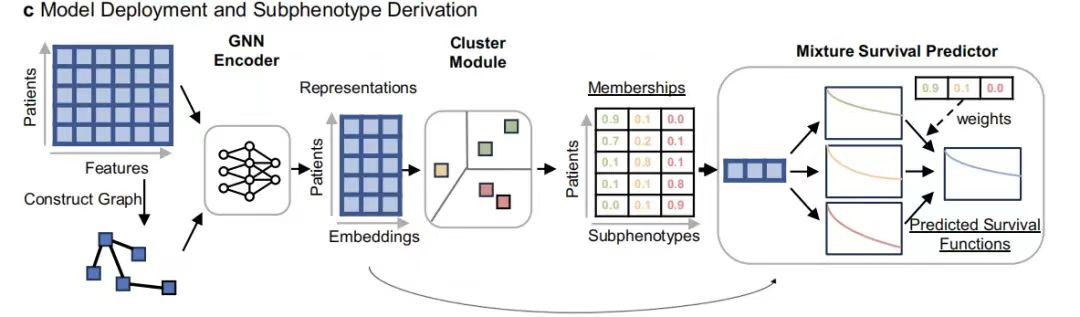
Das Modelltraining verwendete zunächst die Entwicklungskohorte (n = 3.225) als Datenunterstützung und verwendete den Konsistenzindex (C-Index) und den paarweisen Log-Rank-Score als Bewertungsindikatoren und verglich sie mit traditionellen Basismodellen wie Cox-proportionaler Risikoregression (CPH), Gradient Boosted Decision Tree (GBDT), Neural Survival Clustering (NSC) und unüberwachten Methoden wie K-Means und hierarchischem Clustering.
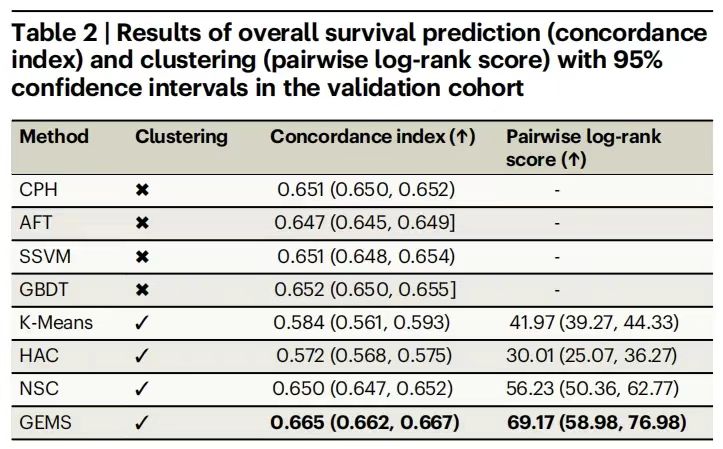
Die experimentellen Ergebnisse sind in der folgenden Tabelle dargestellt.GEMS zeigte gute Ergebnisse bei der Vorhersage des Gesamtüberlebens.Der durchschnittliche C-Index erreichte 0,665 (95% CI: 0,662–0,667) und lag damit deutlich über dem besten Basismodell GBDT von 0,652. der Log-Rank-Score lag bei 69,17 (95% CI: 58,98-76,98) und übertraf damit den NSC-Wert von 56,23 bei weitem. Dies bestätigt die effektive Nutzung der Datenfunktionen durch das Framework für überwachtes Lernen.
Dann,Diese Studie charakterisierte die Auswirkungen des GNN-Encoders auf GEMS weiter, indem sie die von Patienten und ihren GNN-Encodern abgeleiteten Darstellungen visualisierte.Es wird die Uniform Manifold Approximation and Projection (UMAP) verwendet. Wie in der folgenden Abbildung gezeigt, wurde durch die Visualisierung der Uniform Manifold Approximation Projection (UMAP) festgestellt, dass im vom GNN-Encoder ausgegebenen Patientendarstellungsraum Patientengruppen mit unterschiedlichen Gesamtüberlebenszeiten klar getrennt sind, während verschiedene Patiententypen im ursprünglichen Merkmalsraum gemischt verteilt sind, was intuitiv die Fähigkeit des Graph-Neuralnetzwerks widerspiegelt, komplexe Merkmalsbeziehungen zu modellieren.
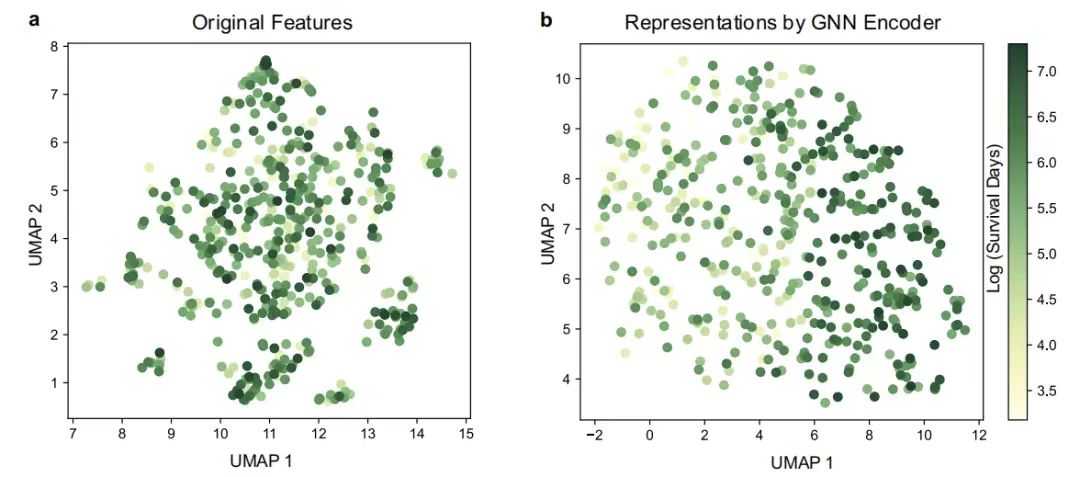
UMAP-Visualisierung von Patienten
Abbildung a: UMAP-Visualisierung der ursprünglichen Funktionen; Abbildung b: UMAP-Visualisierung der vom GNN-Encoder erhaltenen Merkmale
Wie in der Abbildung unten gezeigt,Die Forscher nutzten das Modell außerdem, um drei prädiktive Subphänotypen mit signifikanten Überlebensunterschieden zu identifizieren:Der Subphänotyp 1 (n = 1335) war durch einen hohen Frauenanteil (55.50%), leichte Komorbiditäten und eine geringe Metastasierungslast gekennzeichnet, mit einem durchschnittlichen Gesamtüberleben von 688 Tagen und der niedrigsten Verwendungsrate von Hustenstillern, β-Blockern und der Inzidenz von Knochen-/Hirn-/Nebennierenmetastasen. Die Überlebenskurve des Subphänotyps 2 (n = 420) zeigte einen mittelfristigen Risikoanstieg mit mittleren Komorbiditäten und metastasierender Belastung. Subphänotyp 3 (n = 1420) hatte einen weiblichen Anteil von 35,21% und ein durchschnittliches Gesamtüberleben von nur 321 Tagen, gekennzeichnet durch mehrere Medikamente, eine hohe Metastasierungsrate (Lebermetastasen 31,20%, Knochenmetastasen 51,48%) und schwere Komorbiditäten (Wasser- und Elektrolytstörungen 8,31%, Nierenanomalien 21,43%) und das komplexeste Koexistenzmuster von Metastasen-Komorbiditäten-Laboranomalien.
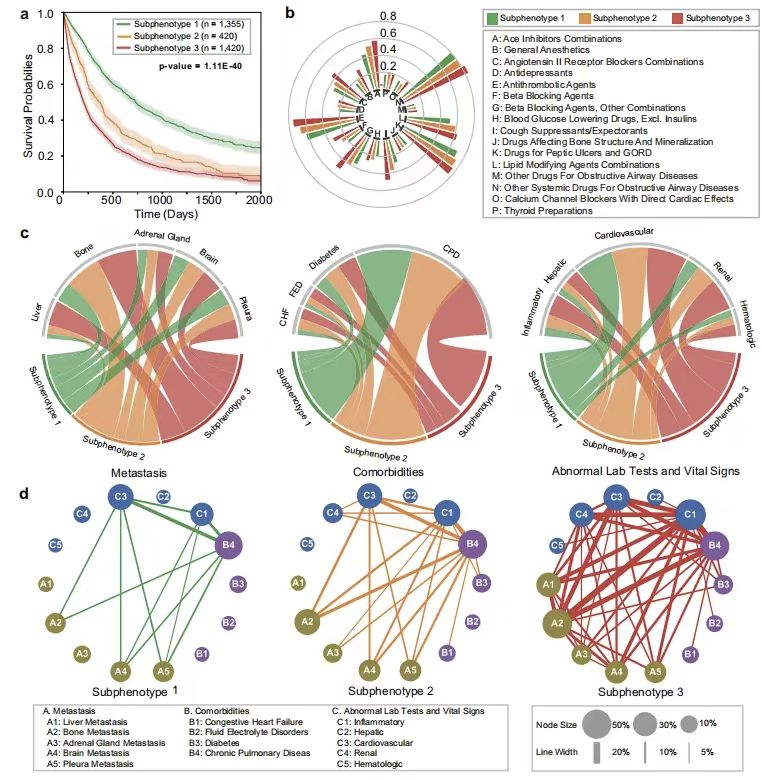
* Abbildung a: Kaplan-Meier-Kurven des Gesamtüberlebens für jeden Subphänotyp
* Abbildung b: Sunburst-Diagramm der Arzneimittelverabreichungsrate jedes Subtyps
* Abbildung c: Akkorddiagramm der Unterschiede in der Klassifizierung von Metastasen (links), Komorbiditäten (Mitte) und abnormalen klinischen Merkmalen
* Abbildung d: Die Häufigkeit verschiedener Subphänotypen
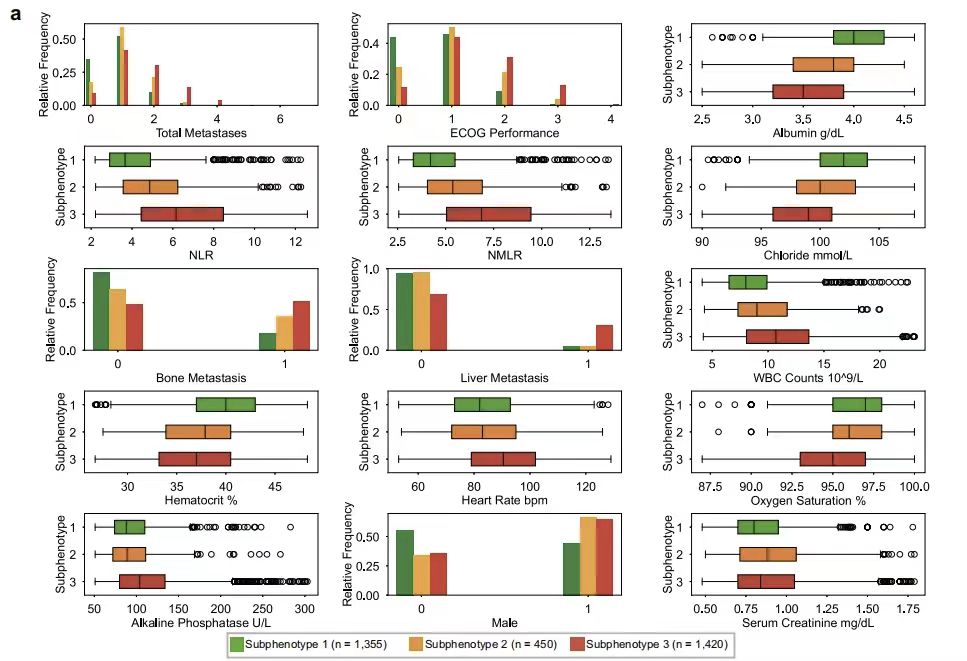
Um die unterschiedlichen Merkmale zwischen verschiedenen Subphänotypen besser zu verstehen, wurden in dieser Studie die Unterschiede in jeder Variable zwischen den Subphänotypen getestet. Wie in der folgenden Abbildung dargestellt, ergab die Analyse der wichtigsten prädiktiven Faktoren, dass der Leistungsstatus der Eastern Cooperative Oncology Group (ECOG-Leistung) und die Gesamtzahl der Metastasen (Gesamtmetastasen) die Hauptindikatoren zur Unterscheidung der Subphänotypen sind. Bei den Laborindikatoren sind das Neutrophilen-Lymphozyten-Verhältnis (NLR) und das Neutrophilen-Monozyten-Lymphozyten-Verhältnis (NMLR) charakteristische Parameter des Subphänotyps 2, während der Subphänotyp 1 mit normalen Albuminwerten (WBC Counts) und hohem Hämatokrit (Hematocrit) verbunden ist und der Subphänotyp 3 eng mit Indikatoren wie erhöhter Herzfrequenz (Heart Rate bpm), verringerter Sauerstoffsättigung (Oxygen Saturation) und erhöhter alkalischer Phosphatase (Alkalische Phosphatase) verbunden ist.
Die obigen Ergebnisse zeigen, dassDas GEMS-Modell ermöglicht nicht nur eine genaue Stratifizierung der Überlebensprognose von aNSCLC-Patienten,Darüber hinaus bietet es durch die Analyse subphänotypischer Merkmale eine auf realen Daten basierende klinische Entscheidungsgrundlage für die Formulierung individualisierter Behandlungsstrategien.
Die globale Revolution in der Präzisionsdiagnose und -behandlung von Lungenkrebs: Wie verändern KI und Multi-Omics-Technologien die Überlebenssituation?
Im Bereich der Diagnose und Behandlung von Lungenkrebs verändert ein durch künstliche Intelligenz (KI) und Präzisionsmedizin vorangetriebener Wandel die klinische Praxis. Ein Forschungsteam der Universität Toronto in Kanada hat eine KI-gestützte Bluttesttechnologie entwickelt, die EGFR-Mutationen in zirkulierender Tumor-DNA analysiert.Durch die Kombination von maschinellem Lernen mit klinischen Daten wird die Erkennungsrate von Personen, die von einer gezielten Behandlung profitieren, effektiv verbessert.Es ermöglicht Patienten mit EGFR-sensitiven Mutationen eine präzise Behandlung mit einem EGFR-Tyrosinkinase-Inhibitor (TKI), wodurch das mediane progressionsfreie Überleben deutlich verlängert wird.
Link zum Artikel:https://pubmed.ncbi.nlm.nih.gov/35624472/
Das „evA.I.-System“ des University College London verwendet 27-dimensionale klinische Daten.Sagen Sie Immun-Checkpoint-Inhibitor-Reaktionen (ICI) genau voraus und helfen Sie bei der Identifizierung von medikamentenresistenten Populationen.Dadurch wird die Wirksamkeit der Immuntherapie verbessert und das mediane Gesamtüberleben verlängert.
Link zum Artikel:https://pmc.ncbi.nlm.nih.gov/articles/PMC10957591/
In China werden an Universitäten und in Unternehmen weiterhin innovative Ergebnisse in der Forschung zur Präzisionsdiagnose und -behandlung von fortgeschrittenem nicht-kleinzelligem Lungenkrebs erzielt. Zum Beispiel,Das Team von Professor Zhang Peng von der Tongji-Universität und das Team der Chinesischen Akademie der Wissenschaften haben die erste internationale Studie zur Proteingenomikkarte für kleinzelligen Lungenkrebs abgeschlossen,Durch die Integration der multidimensionalen Omics-Daten von 112 Proben stellten wir fest, dass eine hohe Expression des HMGB3-Proteins mit einer schlechten Prognose verbunden war, und erstellten ein auf dem ZFHX3-Mutationsstatus basierendes Vorhersagemodell für den Nutzen einer Immuntherapie, wodurch ein neuer Weg für eine präzise Behandlung auf Grundlage der molekularen Typisierung eröffnet wurde.
Link zum Artikel:https://doi.org/10.1016/j.cell.2023.12.004
Die Shenzhen International Graduate School der Tsinghua-Universität und das Shenzhen People's Hospital haben gemeinsam das System „KI + Intelligente Pathologie“ entwickelt.Nach dem Deep Learning von mehr als 3.000 schwierigen Fällen kann es die histologischen Typen von schlecht differenziertem Lungenkrebs mit einer Genauigkeitsrate von 97% genau identifizieren.Verkürzen Sie den Entscheidungszyklus für eine zielgerichtete Therapie. Das von seinem Team entwickelte KI-Vorhersagemodell auf Basis von Blutglykoproteinmarkern kann drei Jahre im Voraus vor dem Lungenkrebsrisiko warnen, mit einer klinisch verifizierten Genauigkeitsrate von über 92% und bietet damit eine nicht-invasive Lösung für ein ultrafrühes Screening.
Link zum Artikel:https://www.nature.com/articles/s41598-025-98731-4
Referenzartikel:
1.https://mp.weixin.qq.com/s/LBcVbQUpTYRnKZ5I1KY_VA








