Command Palette
Search for a command to run...
In Nature Veröffentlicht: Russisches Forschungsteam Nutzt Maschinelles Lernen, Um Billionen Von Massenspektrometriedaten Zu Durchsuchen Und Unbekannte Chemische Reaktionen Zu Entdecken

Die Massenspektrometrie (MS) ist eine der Kerntechnologien der modernen chemischen Forschung. Durch die Messung des Masse-Ladungs-Verhältnisses (m/z) von Molekülionen kann die Massenspektrometrie wichtige Informationen über die Molekülformel, die Struktur und sogar den Reaktionsmechanismus einer Verbindung liefern. Durch die Entwicklung der hochauflösenden Massenspektrometrie (HRMS) konnte die analytische Genauigkeit auf den ppm-Bereich (parts per million) gesteigert werden, und die Methode ist zum „Goldstandard“ in der organischen Synthese, Metallkatalyse, Arzneimittelentwicklung und anderen Bereichen geworden. Mit der zunehmenden Automatisierung der Instrumente übersteigt jedoch die Menge der täglich in Laboren erzeugten Massenspektrometriedaten die Terabyte-Grenze (TB), was dazu führt, dass sich auf den Computern mehrere TB an Informationen anhäufen. Aber derzeitExperimentelle und MS-Daten basieren in hohem Maße auf manueller Analyse, und menschliche Faktoren können den Interpretationsumfang der Datenanalyse beeinträchtigen.Dies schränkt das Experiment erheblich ein.
Um diese Herausforderung zu bewältigen, haben Forscher der Russischen Akademie der Wissenschaften und anderer Institutionen eine innovative, auf maschinellem Lernen (ML) basierende Suchmaschine, MEDUSA Search, eingeführt.Die Ionenisotopenverteilung kann in hochauflösenden Mehrkomponenten-Massenspektrumdatenbanken bis auf TB-Ebene durchsucht werden.Der Ansatz nutzt einen isotopenzentrierten Suchalgorithmus, der durch zwei kollaborative Modelle des maschinellen Lernens erweitert wird, um die Entdeckung unbekannter chemischer Reaktionen zu unterstützen. Dieser Ansatz ermöglicht eine gründliche Überprüfung vorhandener Daten, um gültige Belege für chemische Hypothesen zu liefern und gleichzeitig den Bedarf an zusätzlichen Experimenten zu reduzieren. Darüber hinaus ist das Modell durch die Erweiterung des Basisansatzes in der Lage, automatisch Reaktionshypothesen zu generieren und neue chemische Transformationen aufzudecken. In,Der Heterocyclus-Vinyl-Kopplungsprozess in der Mizoroki-Heck-Reaktion stach in den Experimenten hervor und unterstrich die Fähigkeit des Motors, komplexe chemische Phänomene aufzulösen.
Die entsprechende Forschung mit dem Titel „Entdeckung organischer Reaktionen durch die auf maschinellem Lernen basierende Entschlüsselung von Massenspektrometriedaten im Terameterbereich“ wurde in Nature Communications veröffentlicht.
Forschungshighlights
* Unbekannte Reaktionen erforschen: Anstatt sich auf neue Experimente zu verlassen, nutzen Sie vorhandene Daten, um unbekannte chemische Reaktionen zu erforschen und so die Versuchskosten und den Ressourcenverbrauch zu senken.
* Effizienter Suchalgorithmus: Ein einzigartiger Suchalgorithmus für die Isotopenverteilung in Kombination mit einem Modell für maschinelles Lernen kann in umfangreichen Massenspektrometriedaten präzise nach Ionen suchen und Fehleinschätzungen reduzieren.
* Erweitern Sie Ihr chemisches Wissen: Entdecken Sie neue Reaktionswege und Produkte, wie beispielsweise den heterozyklischen Vinyl-Kopplungsprozess in der Mizoroki-Heck-Reaktion, und vertiefen Sie Ihr Verständnis chemischer Reaktionen.

Papieradresse:
Das Open-Source-Projekt „awesome-ai4s“ vereint mehr als 100 AI4S-Papierinterpretationen und stellt umfangreiche Datensätze und Tools bereit:
https://github.com/hyperai/awesome-ai4s
Datensatz: Über 20.000 Massenspektrometriebilder, die das Vorhandensein reaktiver Ionen bestätigen
Da den meisten Massenspektrometriesignalen eine professionelle Analyse fehlt, hat das Labor in den letzten Jahren eine riesige Menge an Daten gesammelt und gespeichert, und die in diesem Experiment verwendeten Daten stammen alle daraus. Diese Massenspektrometriedaten decken ein breites Spektrum an Studien zur chemischen Transformation ab.Das gesamte Datenvolumen übersteigt 8 TB, darunter mehr als 20.000 Massenspektren.Es werden hochauflösende Massenspektrometriedaten mehrerer Komponenten mit unterschiedlichen Auflösungen gespeichert, wodurch die Bestätigung der Anwesenheit von Zielionen in einem breiten Anwendungsbereich möglich ist.
MEDUSA-Suche Während des Reaktionsentdeckungsprozesses werden die generierten Ionenformeln mit der gesamten HRMS-Datenbank im Terascale-Format abgeglichen, um neue Reaktionswege und Produkte zu finden, und die Daten werden visualisiert.
Der Datensatz wurde mithilfe der t-SNE-Dimensionsreduktionstechnik visualisiert. Um die große Vielfalt des archivierten Datensatzes zu demonstrieren, erstellten die Forscher zwei t-SNE-Diagramme.Die gesammelten Moleküle wurden zufällig aus der PubChem-Datenbank und per Massenspektrometrie registrierten Verbindungen ausgewählt.Die in den analytischen Massenspektren registrierten Verbindungen decken den chemischen Raum gut ab. Jeder Punkt stellt ein Spektrum dar, ähnliche Massenspektren liegen im Diagramm nahe beieinander und verschiedene Mitarbeiter haben unterschiedliche Spektren aufgezeichnet, die miteinander verglichen wurden. Experimente haben gezeigt, dassDie Verbindungen in den Massenspektren sind im chemischen Raum weit verbreitet und die von verschiedenen Forschern aufgezeichneten Massenspektren variieren erheblich.Wie unten gezeigt.
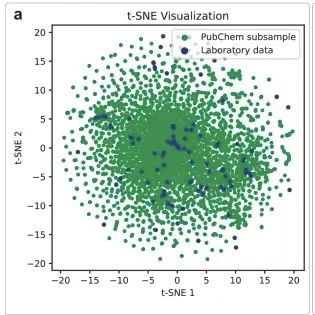
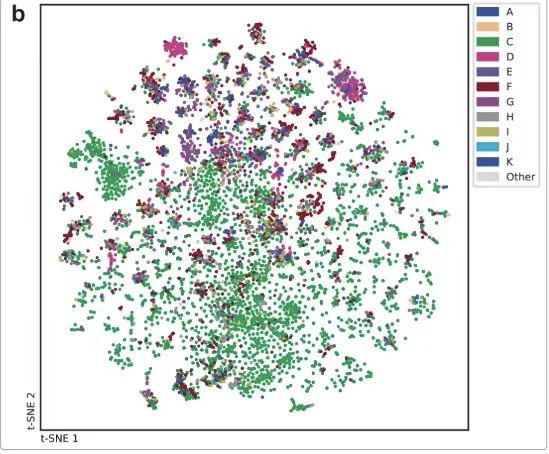
Die vielfältigen, durch die Studie generierten Daten wurden auf Figshare gespeichert.Dieses enthält ein 9 GB großes Massenspektrometrie-ZIP-Archiv,Alle genannten Discovery-Produkte werden abgedeckt und es sind zusätzliche Reaktions-Massenspektrometriedaten enthalten, die zum Testen der Suchmaschinenfunktionalität verwendet werden können. Einige Daten, die bei der Suche keine Ergebnisse lieferten, können aus Gründen der Vertraulichkeit oder des geistigen Eigentums nicht öffentlich zugänglich gemacht werden.
* figshare ist ein auf Cloud-Computing-Technologie basierendes Online-Daten-Repository, in dem Forscher ihre Forschungsergebnisse, einschließlich Daten, Datensätze, Bilder, Videos, Poster und Codes, speichern und teilen können.
HRMS-Datensatz zur hochauflösenden Massenspektrometrie:
Modellarchitektur: Entdeckung unbekannter chemischer Reaktionen durch Suche nach Isotopenverteilungen
MEDUSA Search ist eine auf maschinellem Lernen basierende Engine zur Analyse von Massenspektrometriedaten, mit der sich unbekannte chemische Reaktionen in umfangreichen Massenspektrometriedaten entdecken lassen.
Konkret besteht der in MEDUSA Search entwickelte Suchvorgang aus 5 Schritten.
Erste,MEDUSA Search verwendet als Eingabe die Molekülformel und Ladung des gesuchten Ions.Diese Formeln oder Ladungen können mithilfe eines Ansatzes zur Hypothesengenerierung aus dem Reaktionssystem abgeleitet oder manuell definiert werden (wie in Abbildung A unten dargestellt). Die Suchmaschine sucht dann nach allen Spektraldateien, die die beiden häufigsten Isotopologenspitzen des Eingabeions enthalten, wie in Abbildung B unten dargestellt. Isotopologe-Peaks werden durch ihr Masse-Ladungs-Verhältnis m/z dargestellt. Diese Spektraldateien werden als Kandidaten bezeichnet und die Forscher haben auch Berechnungen der Kosinus-Distanzschwelle für die Spektraldateien durchgeführt, wie in Abbildung C1 unten dargestellt. Anschließend werden alle Kandidaten-Massenspektren einem Algorithmus unterzogen, der gemäß einer Eingabeformel nach Isotopenverteilungen innerhalb eines einzelnen Spektrums sucht, wie in Abbildung C2 unten dargestellt.
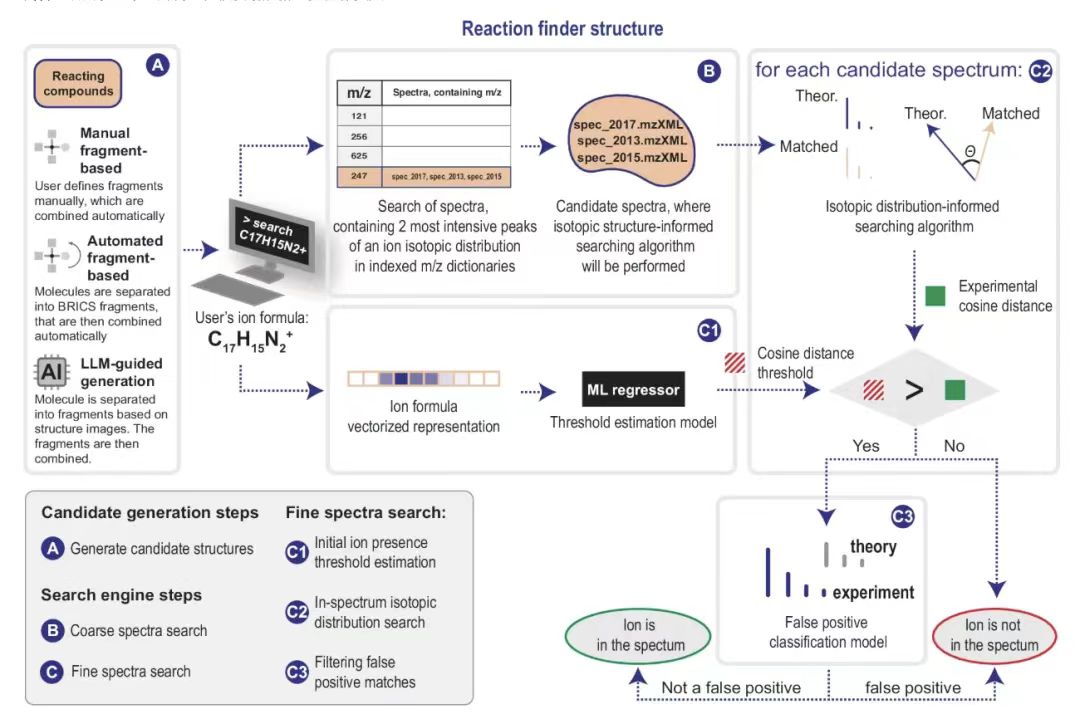
Vor der Suche sollten ForscherErstellen Sie eine Liste hypothetischer Reaktionswege basierend auf Vorkenntnissen über das Reaktionssystem(Wie in Abbildung A gezeigt). Dieses System ist auf brechbare Bindungen und die Rekombination der entsprechenden Fragmente ausgelegt. Durch Eingabe von Informationen zur chemischen Formel und Ladung kann das theoretische „Isotopenmuster“ des Ions berechnet werden. Suchen Sie im invertierten Index nach den beiden häufigsten Isotopologen-Peaks (Abbildung B). Massenspektren, die diese Peaks enthalten, werden als Kandidaten bezeichnet. Nach der groben Spektrumsuche wird jedes Kandidatenspektrum nach der Isotopenverteilung des Abfrageions durchsucht.Es sind drei Schritte erforderlich:
* Anfängliche Schätzung des Ionenpräsenzschwellenwerts:Die vom Suchalgorithmus für die Isotopenverteilung innerhalb eines Spektrums zurückgegebene Kosinusdistanz wird als Maß für die Ähnlichkeit zwischen der theoretischen und der übereinstimmenden Isotopenverteilung verwendet. Die automatische Bestimmung des Vorhandenseins oder Fehlens eines Ions in einem Spektrum hängt von der geschätzten maximalen Kosinusdistanz ab (d. h. dem Schwellenwert für das Vorhandensein von Ionen). Basierend auf einem Regressionsmodell für maschinelles Lernen (Abbildung C1) wird der Schwellenwert für die Ionenpräsenz mithilfe der eingegebenen Ionenformel bestimmt.
* Suche nach Isotopenverteilung innerhalb eines Spektrums:Der Suchalgorithmus für die intraspektrale Isotopenverteilung (Abbildung C2) gleicht die Spitzen im experimentellen Kandidatenmassenspektrum mit den Spitzen in der theoretischen Isotopenverteilung ab. Bei jedem Schritt wird die Kosinusdistanz berechnet, um den ähnlichsten Peak auszuwählen. Wenn kein Peak gefunden wird, wird er durch einen Peak ersetzt, dessen Intensität dem Median des Rauschens entspricht. Wenn der endgültige Kosinusabstand kleiner ist als der in Schritt geschätzte Schwellenwert für die Ionenpräsenz (wie in Abbildung C1 gezeigt), gilt das Ion als gefunden.
* Falsch positive Übereinstimmungen filtern:Ein zusätzlicher Klassifikator für maschinelles Lernen (Abbildung C3) wurde verwendet, um das Vorhandensein falsch positiver Ionen anhand von Informationen über benachbarte Peaks zu erkennen. Dieses Problem manifestiert sich normalerweise darin, dass nach einer Verteilung gesucht wird, die Teil einer anderen Verteilung ist. Eines der bekanntesten Beispiele beginnt mit M+1, obwohl auch M existiert.
Experimentelle Schlussfolgerung: Heterocyclische Vinyl-Kopplungsexperimente unterstreichen die Modellerkennungsfähigkeiten
Die 520 generierten Ionen wurden in der gesamten Terascale HRMS-Datenbank mit einer Gesamtrechenzeit von 3–4 Tagen (8–11 Minuten pro Ion) durchsucht. Experimentelle Ergebnisse zeigen, dass MEDUSA Search mehrere Isotopenverteilungsmuster erkennt.
Die Bildung katalytischer Umwandlungsprodukte hängt eng mit dem entsprechenden Reaktionsmechanismus zusammen.Mehrere Mizoroki-Heck- und Kreuzkupplungsreaktionen (wie Sonogashira, Suzuki, Buchwald-Hartwig usw.) wurden zuvor unter Verwendung von Pd/NHC-Komplexen mit verschiedenen NHC-Liganden und Halogensubstituenten als katalytische Komponenten durchgeführt. Bei der Untersuchung des Reaktionsmechanismus mittels ESI-MS-Spektroskopie des Reaktionsgemisches wurden die Kopplungsprodukte [NHC-H]⁺, [NHC-Ph]⁺, [NHC-O]⁺ und [NHC-N]⁺ gefunden. Basierend auf diesen Beobachtungen,Die Schlüsselrollen der R-NHC-Kopplung und der M-NHC-Bindungsspaltung bei der Entwicklung von M/NHC-Komplexen unter katalytischen Reaktionsbedingungen wurden aufgedeckt.Die Bildung katalytisch aktiver molekularer M/NHC-Katalysatoren und „NHC-freier“ Katalysatoren vom Cocktail-Typ wird aus der Perspektive der Anzahl der CC-Kopplungsreaktionen beschrieben, einschließlich der Bildung von H-NHC-Salzen und O-NHC-Kopplungen.
Bei der Sonogashira-Reaktion wurde ein bislang unbekanntes Ethinyl-NHC-Kupplungsprodukt isoliert und ein möglicher Reaktionsverlauf beschrieben. Die Ethinyl-NHC-Kupplungsprodukte sind hochreaktiv und können eine Vielzahl von Transformationen durchlaufen. Die hydrierten Derivate der Produkte wurden mit der beschriebenen Methode analysiert.Das ESI-MS-Spektrum des Sonogashira-Reaktionsgemisches zeigte das Vorhandensein des Produkts [NHC-(CH₂)₂-Ph]⁺.Wie unten gezeigt. Es wird vermutet, dass dieser Prozess über eine Transferhydrierungsreaktion erfolgt.
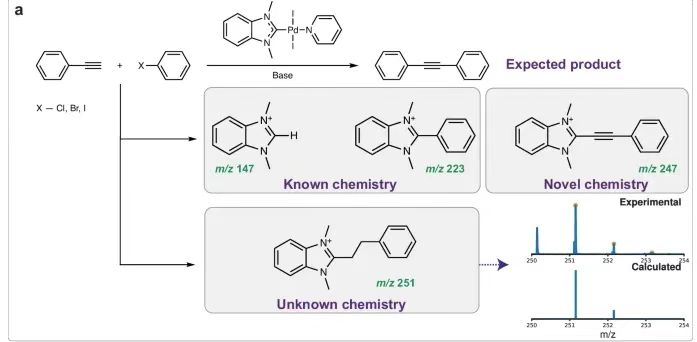
Unter der Katalyse des Pd/NHC-Komplexes [BIMePh]⁺ [BIMePdI₃]⁻,Die massenspektrometrische Analyse des Mizoroki-Heck-Reaktionsgemisches zwischen p-Methoxyiodbenzol und Butylacrylat ergab die Bildung von [BIMe (CH)₂COOBu]⁺.Die Molekülformel wurde durch Massenspektrometrie mit ultrahoher Auflösung bestätigt. Experimente mit der Bildung von [IPrCHC(Ph)COOBu]⁺ wurden verwendet, um zwischen homogener und heterogener Katalyse durch Quecksilber zu unterscheiden. Indem die Interferenz von Quecksilber mit den Reaktionsspezies ausgeschlossen wird und die anderen Bedingungen dieselben wie beim ursprünglichen Experiment bleiben. Die Molekülformel wurde außerdem durch Massenspektrometrie mit ultrahoher Auflösung bestätigt und die chemische Struktur durch MS/MS-Experimente verifiziert.
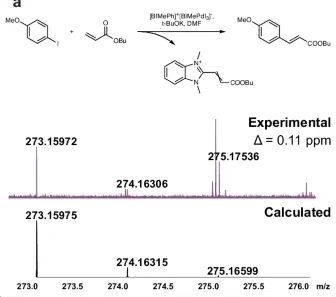
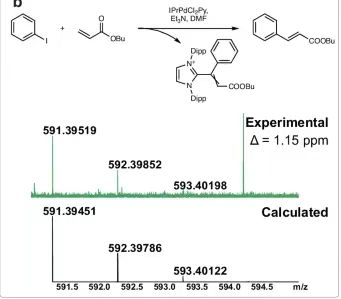
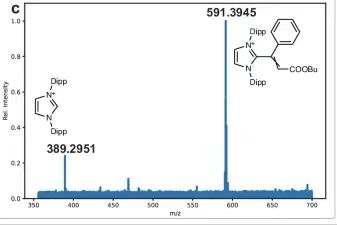
Es wurden Experimente mit 5 verschiedenen NHC-Liganden durchgeführt. Die Möglichkeit einer Vinyl-NHC-Kopplung während der Pd/NHC-Umwandlung im Rahmen der Mizoroki-Heck-Reaktion wurde getestet. In allen untersuchten Fällen wurden Vinyl-NHC-Produkte gefunden, unabhängig von den Liganden in den Komplexen, und alle Produkte wurden mit minimalen Fehlern definiert. Für die untersuchten Reaktionsgemische wie (BIMe)PdI₂Py, (SIMes)PdCl(allyl) und (PIPr)PdCl(allyl)Neben Vinyl-NHC wurde auch Ethyl-NHC nachgewiesen.Die m/z-Fehler der (IMes)PdCl(Allyl)- und (SIPr)PdCl(Allyl)-Komplexe sind sehr gering, weniger als 0,3 ppm, während die Fehler weniger als 1 ppm betragen. Bei allen MS-Experimenten wurde die Konfiguration so eingestellt, dass Übergänge während der Aufzeichnung der Massenspektren vermieden wurden. Für den betreffenden Vinyl-NHC-Kopplungsprozess wurde außerdem eine ESI-MS-Reaktionsüberwachung mittels Druckprobeninfusion durchgeführt, um zu bestätigen, dass Ionen in mehreren Modi der Reaktionsdatenerfassung beobachtet werden konnten.
Es wurde nachgewiesen, dass diese robuste, auf maschinellem Lernen basierende Rechenmaschine zur Reaktionsentdeckung Ionen unterschiedlicher Zusammensetzung verwenden kann.Ionensuchen können auf allen MS-Instrumenten mit einer Auflösung durchgeführt werden, die ausreicht, um Isotopenverteilungen zu beobachten.Durch die Kombination des entwickelten Systems mit anderen Computertechniken (z. B. Algorithmen zur Vorhersage von Ionenfragmenten anhand der Strukturformel oder Peptidsequenz, verschiedene Adduktrechner) könnte ein leistungsstarkes Analysetool für ein umfassendes Screening entstehen, das für die Beschleunigung von Entdeckungen in verschiedenen wissenschaftlichen Bereichen von entscheidender Bedeutung ist.
Auch,Mit dieser Methode wird auch das Forschungskonzept „Experimentieren in der Vergangenheit“ verwirklicht.Es schöpft den Wert vorhandener Daten voll aus, entdeckt neue Reaktionswege und Produkte, spart Forschungsressourcen, liefert neue Ideen und Methoden für die chemische Forschung und fördert die Entwicklung des Fachgebiets der organischen Chemie. Im Hinblick auf praktische Anwendungen kann es Pharmaunternehmen, Materialforschungs- und -entwicklungsunternehmen usw. dabei helfen, schneller neue Reaktionswege und Produkte zu finden, Forschungs- und Entwicklungskosten zu senken, die Forschungs- und Entwicklungseffizienz zu verbessern und ein leistungsstarkes Analysetool für die chemische Forschung bereitzustellen.
Automatisierte Analyse von Massenspektrometriedaten findet Eingang in die klinische Anwendung
Mit der kontinuierlichen Vertiefung der Massenspektrometrie-Technologie in der wissenschaftlichen Forschung und der industriellen Produktion hat die Automatisierungstechnologie begonnen, sich in Richtung klinischer Anwendung zu bewegen. Als wichtiger Bestandteil der Präzisionsdiagnosetechnologie kann die klinische Massenspektrometrie eine vollständige Automatisierung von der Probenentnahme über die Verarbeitung und Trennung bis hin zur Analyse erreichen. Laut der kürzlich in den USA veröffentlichten 17. Ausgabe des Global IVD Industry Report wird das Marktvolumen der weltweiten klinischen Massenspektrometriebranche im Jahr 2024 930 Millionen US-Dollar betragen.Es wird erwartet, dass dieser Wert im Jahr 2029 1,435 Milliarden US-Dollar erreichen wird. Von 2024 bis 2029 wird der Markt für klinische Massenspektrometrie voraussichtlich jährlich um durchschnittlich 9% wachsen und sich nach den Nukleinsäuretests zum am schnellsten wachsenden Marktsegment im IVD-Bereich entwickeln.
* IVD (In-vitro-Diagnostika) bezeichnet Medizinprodukte, In-vitro-Diagnostika-Reagenzien und Arzneimittel.
Betrachtet man den chinesischen Markt,Die klinische Massenspektrometriebranche befindet sich seit langem auf der Überholspur der Entwicklung und verzeichnet bedeutende Fortschritte in der Multi-Omics-Massenspektrometrie, bei Massenspektrometern für den Heimgebrauch und bei der automatisierten Massenspektrometrie.Laut dem „2024 Clinical Mass Spectrometry Industry Research Report“ wurden bis zum 31. Juli 2024 insgesamt 228 inländische klinische Massenspektrometrieprodukte (ausgenommen Qualitätskontrollprodukte und Kalibrierungsprodukte) von der NMPA zugelassen.
Was die zugelassenen Reagenztypen betrifft, ist die Zahl der in China zugelassenen, im Inland produzierten klinischen Massenspektrometrie-Geräte in den letzten fünf Jahren kontinuierlich gestiegen, und es gibt keine Anzeichen für eine Verlangsamung dieser Wachstumsrate. Bis zum 31. Juli 2024 waren 51 Reagenzien für Vitamintests, 46 Reagenzien für die Überwachung der Arzneimittelkonzentration und 45 Reagenzien für chronische Krankheiten und Hormone zugelassen. Von 2020 bis 2023 wird es jeweils 10, 12, 13 und 16 Modelle geben.
Unter den zugelassenen Geräten sind Flüssigkeitschromatographie-Massenspektrometrie-Geräte (LC-MS) in der Mehrzahl; insgesamt wurden 33 in China hergestellte LC-MS-Geräte zugelassen. Die zweitgrößte Gruppe sind Geräte für die inländische Matrix-unterstützte Laserdesorptions-Flugzeit-Massenspektrometrie (MALDI-TOF MS), von denen insgesamt 25 Modelle für den Einsatz in der mikrobiellen Erkennung, der Nukleinsäureerkennung und der Peptiderkennung zugelassen sind.
* Flüssigkeitschromatographie-Massenspektrometrie ist eine analytische chemische Technik, die die physikalischen Trennmöglichkeiten der Flüssigkeitschromatographie (LC) mit den Massenanalysemöglichkeiten der Massenspektrometrie (MS) kombiniert.
* Die matrixunterstützte Laserdesorptions-Flugzeit-Massenspektrometrie (MALDI-TOF MS) ist eine neue Art der Biomassenspektrometrie mit sanfter Ionisation, die in den letzten Jahren entwickelt wurde und häufig zur Identifizierung einer großen Anzahl von Bakterien und Pilzen eingesetzt wird.
Derzeit wird die klinische Anwendung von LC-MS in China erst seit relativ kurzer Zeit durchgeführt und steckt noch in den Kinderschuhen. Es gibt noch viele Defizite. Zahlreiche Faktoren wie IVD-Hersteller, medizinische Testlabore, Fach- und technisches Personal, Managementabteilungen und Richtlinien können die Anwendung der klinischen Massenspektrometrie-Erkennungstechnologie beeinflussen. Mit Blick auf die Zukunft wird die Kombination von Automatisierung und Intelligenz jedoch mit Sicherheit eine wichtige Entwicklungsrichtung sein. Die klinische Anwendung von LC-MS/MS wird sich weiterentwickeln und neben der Verbesserung der Erkennungseffizienz und -genauigkeit wird es den Ärzten weiterhin bei der Interpretation der Ergebnisse helfen und sie bei der klinischen Entscheidungsfindung unterstützen.
Quellen:
1.https://mp.weixin.qq.com/s/27drrM5lwawHRgRMWvHZRQ
2.https://mp.weixin.qq.com/s/pkd2I573on08syPkqdStOQ








