Command Palette
Search for a command to run...
Die Leistung Übertrifft Die Des Modells Der SAM-Serie Bei weitem. Die Universität Zürich Und Andere Haben Ein Allgemeines 3D-Basismodell Zur Blutgefäßsegmentierung Entwickelt, Das Für CVPR 2025 Ausgewählt wurde.
Vergleicht man den menschlichen Körper mit einer riesigen Stadt, dann sind die Blutgefäße zweifellos die „Straßen“ dieser Stadt. Arterien, Venen und Kapillaren entsprechen Autobahnen, Stadtstraßen und Landstraßen. Sie arbeiten zusammen, um Nährstoffe, Sauerstoff usw. über das Blut in verschiedene Teile des Körpers zu transportieren und so den effizienten und stabilen Betrieb dieser „Stadt“ aufrechtzuerhalten. Und wenn es Probleme mit diesen Straßen gibt, werden die Menschen natürlich krank.
Um zu prüfen, ob es Probleme mit diesen „Straßen“ gibt, ist die Blutgefäßsegmentierung ein wichtiges Mittel. Ebenso wie das Aufdecken von Problemen anhand von Verkehrsbildern im Städtebau ist dies eine zentrale Aufgabe in der medizinischen Bildverarbeitung.Durch die Segmentierung von Blutgefäßen kann die Struktur von Blutgefäßen aus medizinischen Bildern genau identifiziert und extrahiert werden.Es kann zur Analyse, Diagnose und Behandlung verschiedener Gefäßerkrankungen eingesetzt werden. Bei Herz-Kreislauf-Erkrankungen beispielsweise hilft die Segmentierung der Koronararterien den Ärzten dabei, den Grad der Stenose der Blutgefäße einzuschätzen und so geeignete Behandlungspläne für die Patienten zu erstellen.
In den letzten Jahren wurden bei der Segmentierung von Blutgefäßen mithilfe von Computer- und medizinischer Bildgebungstechnologie erhebliche Fortschritte erzielt. Allerdings bleibt die genaue und robuste Segmentierung vollständig verbundener Gefäße bei aufgabenspezifischer Bildgebung, insbesondere bei der 3D-Gefäßsegmentierung, eine Herausforderung. Einerseits liegt es an der Begrenzung der Blutgefäße selbst. Die komplexen und winzigen geometrischen Formen der Blutgefäße führen zu einer plötzlichen Schwierigkeit der Segmentierung.Auf der anderen Seite gibt es erhebliche Domänenlücken, die durch die Einschränkungen der Bildgebungsmethoden und -protokolle, spezifische Signal-Rausch-Verhältnisse, Gefäßmuster, Bildartefakte und Änderungen im Hintergrundgewebe verursacht werden.
Obwohl es viele Methoden zur Segmentierung medizinischer Bilder gibt, die auf Basismodellen basieren, wie z. B. SAM (Segment Anything Model) und SAM-Med3D und VISTA3D für medizinische 3D-Bilder,Allerdings weisen diese Modelle bei der Gefäßsegmentierung immer noch Einschränkungen auf.Daher bleibt die 3D-Gefäßsegmentierung für medizinisches Personal und Forscher eine arbeitsintensive Aufgabe, die eine umfangreiche manuelle Annotation auf Voxelebene erfordert, um eine noch genauere Gefäßbildanalyse zu erreichen.
Um dieses Problem zu lösen, hat ein Team der Universität Zürich, der ETH Zürich und der Technischen Universität München ein grundlegendes Modell vesselFM vorgeschlagen, das speziell für die 3D-Gefäßsegmentierung entwickelt wurde. Das Modell wird anhand eines umfangreichen Datensatzes (Dreal) und synthetischer Daten trainiert, die durch Domänenrandomisierung (Ddrand) und ein auf Flow-Matching basierendes generatives Modell (Dflow) generiert wurden.Es kann Segmentierungs- und Generalisierungsfunktionen erreichen, die bestehenden fortgeschrittenen Modellen in Zero-Shot-, Single-Shot- und Few-Shot-Szenarien überlegen sind.
Die zugehörige Forschung wurde unter dem Titel „vesselFM: A Foundation Model for Universal 3D Blood Vessel Segmentation“ veröffentlicht und für CVPR 2025 ausgewählt.
Forschungshighlights:
Die Studie schlägt ein universelles Basismodell für die 3D-Gefäßsegmentierung mit Zero-Shot- und Generalisierungsfunktionen vor, das von Forschern und medizinischem Personal sofort verwendet werden kann.
* Das Team hat den größten Datensatz zur 3D-Gefäßsegmentierung zusammengestellt, einschließlich sorgfältig verarbeiteter realer 3D-Gefäßbilder und passender Anmerkungen auf Voxelebene
* Die Studie schlägt eine feinkörnige Nachbarschaftsrandomisierungsstrategie für die 3D-Gefäßsegmentierung vor und führt Flow Matching in die 3D-Bilderzeugung im medizinischen Bereich ein.

Papieradresse:
https://go.hyper.ai/lVad9
Datensatz: 3 heterogene Datenquellen
Zum Trainieren verwendeten die Forscher drei heterogene Datenquellen.Der erste ist ein Datensatz Dreal (Diverse Real Data), der verschiedene reale Daten enthält; die zweite sind 2 synthetische Datenquellen,Dabei handelt es sich um den Domänen-Zufallsdatensatz Ddrand (Domain Randomization) und die gesammelten Daten aus dem auf Flow-Matching basierenden Generierungsmodell Dflow (Flow Matching-Based).
In,Dreal ist der bislang größte reale Datensatz, der für 3D-Blutgefäßsegmentierungsaufgaben verwendet wird.Eine breite Palette von Bildgebungsverfahren, die verschiedene anatomische Regionen unterschiedlicher Organismen abdecken,Enthält über 115.000 3D-Patches der Form 128³ aus 17 Anmerkungsquellen.Zu den umfangreichen klinischen Bildgebungsverfahren gehören insbesondere MRA, CTA, Röntgen, Zwei-Photonen-Mikroskopie und vEM. Die biologischen Proben stammen aus dem Gehirn, den Nieren und der Leber von Menschen und Versuchsmäusen, die Gefäßmuster mit unterschiedlichen strukturellen und funktionellen Eigenschaften für die Forschung liefern. Wie in der Abbildung unten gezeigt.
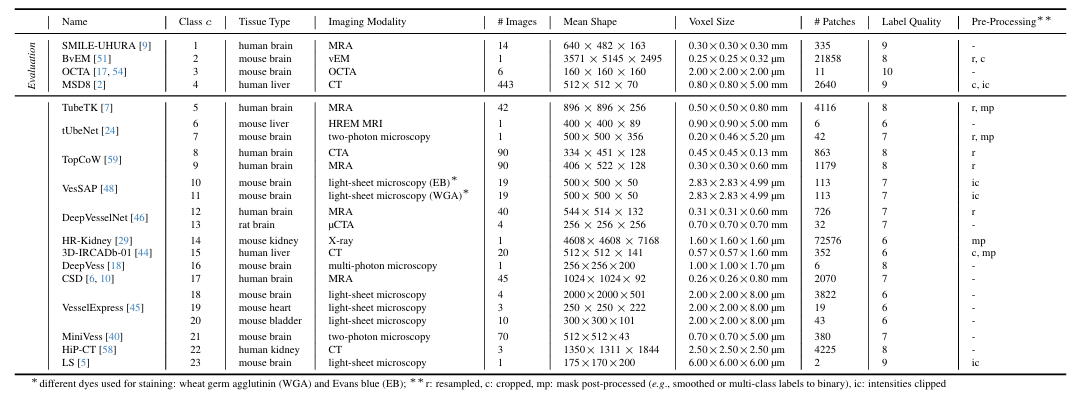
Anschließend untersuchten die Forscher die verschiedenen Gewebearten, Bildgebungsverfahren und Protokolle.Dreal ist weiter in 23 Datensätze unterteilt.Jeder Datensatz wird vorverarbeitet und schließlich werden Patches mit einer Zielform von 128³ aus den Bildern und ihren entsprechenden Beschriftungen extrahiert.
Die Forscher führten eine Domänen-Randomisierungsstrategie ein, um Ddrand zu erstellen, wie in der folgenden Abbildung dargestellt. Diese Methode erzeugt eine große Anzahl unterschiedlicher synthetischer Bild-Masken-Paare, indem sie eine Reihe räumlicher Transformationen und künstlicher Artefakte auf reale Gefäßdaten anwendet und so die Robustheit des Modells gegenüber realen Daten verbessert.Konkret lässt es sich in drei Schritte unterteilen, nämlich Vordergrundgenerierung, Hintergrundgenerierung und Hintergrundfusion.
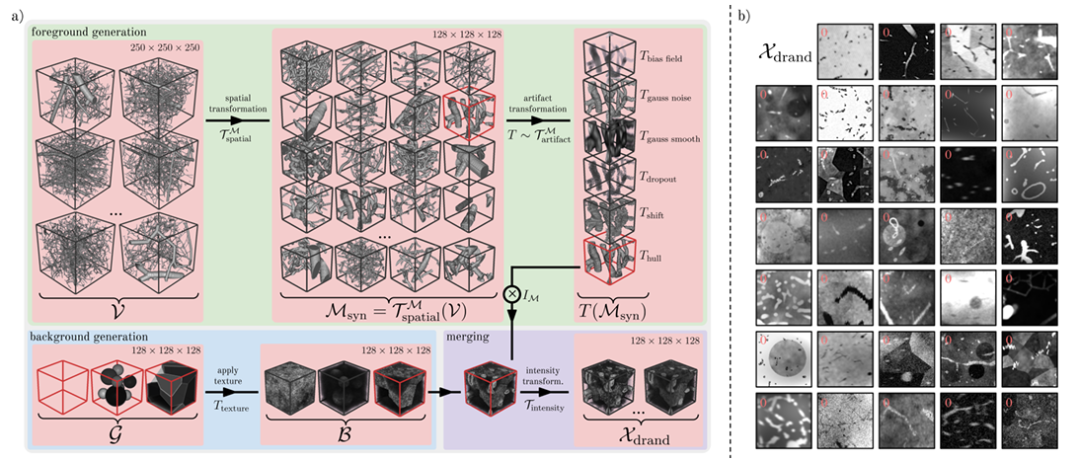
In der Phase der InteressentengenerierungDie Forscher verwendeten 1.137 Gefäßpflaster v mit einer Form von 250³, die von Wittmann et al. bereitgestellt wurden. als Grundlage für die synthetische Maske.Diese Gefäßplaques sind aus grafischen Darstellungen von Korrosionsabdrücken mit hoher Wiedergabetreue abgeleitet.
Der von den Forschern verfolgte Hauptansatz besteht darin, durch räumliche Transformationen wie zufälliges Zuschneiden, Umdrehen, Erweitern und Skalieren, zufällige elastische Deformation und binäre Glättung eine große Bandbreite realistischer Gefäßmuster zu erzeugen. Als nächstes simulierten die Forscher verschiedene Vordergrundartefakte, die in echten Gefäßbildern vorhanden sind, indem sie Artefakttransformationen wie Bias-Feld, Gaußsches Rauschen, Gaußsche Glättung, Dropout, Offset, Unschärfe usw. auswählten.
In der HintergrundgenerierungsphaseDie Forscher modellierten Hintergrundbilder, die eine Vielzahl von Hintergrundgeometrien mit unterschiedlichen Texturen enthielten.Es gibt drei Hauptvarianten: Kugel (sich nicht überlappende Kugeln), Polyeder (das Bild wird mithilfe von Voronoi in mehrere polyedrische Bereiche aufgeteilt) und geometrisches Bild ohne Hintergrund.
In der HintergrundfusionsphaseDie Forscher fügten den Vordergrund mit dem Hintergrund zusammen, indem sie Voxel-Addition und -Subtraktion durchführten oder indem sie Hintergrundintensitätswerte durch Maskenintensitätswerte ersetzten.Um den Bildbereich zu erweitern, verbesserten die Forscher das Bild kontinuierlich durch ein zufälliges Bias-Feld, fügten Gaußsches Rauschen hinzu, wendeten zufällige lokale Spitzen im k-Raum an, passten den Bildkontrast zufällig an, führten eine Gaußsche Glättung an einzelnen oder gemeinsamen σ-Werten aller räumlichen Dimensionen durch, fügten Rician-Rauschen und Gibbs-Rauschen hinzu, führten eine zufällige Gaußsche Schärfung durch und transformierten das Intensitätshistogramm zufällig, wodurch sie schließlich das synthetische Bild Xdrand erhielten (wie in Abbildung b oben gezeigt).
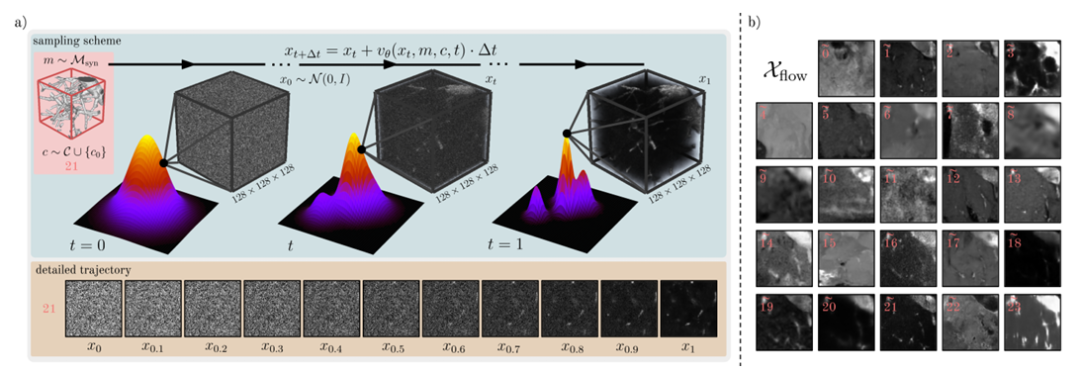
Um die Verteilung der realen Daten Dreal weiter anzureichern, trainierten und prüften die Forscher zusätzlich ein auf Flow Matching basierendes bedingtes Generierungsmodell F, um eine dritte Datenquelle Dflow zu generieren. Wie in der Abbildung unten gezeigt.

Modellarchitektur: Einführung tiefer generativer Modelle und Domänen-Randomisierungsstrategien
Gesamt,vesselFM ist ein allgemeines Basismodell, das speziell für die 3D-Gefäßsegmentierung entwickelt wurde und 3D-Gefäße in einer Vielzahl von Modalitäten und Gewebetypen präzise segmentieren kann.Während des Modellentwurfsprozesses trainierten die Forscher das Modell anhand von drei heterogenen Datenquellen, um seine leistungsstarken Segmentierungs- und Generalisierungsfunktionen zu erreichen.
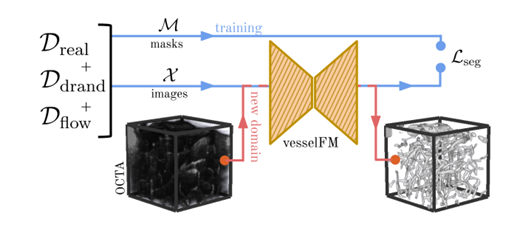
In diesem Prozess führten die Forscher zwei wichtige Schritte ein:Dabei handelt es sich um das tiefe generative Modell bzw. die Domänen-Randomisierungsstrategie.Der obige Abschnitt zur Einführung in den Datensatz geht näher auf die Strategie zur Domänenrandomisierung ein. Dieser Abschnitt konzentriert sich auf das tiefe generative Modell, das zur Generierung des Dflow-Datensatzes verwendet wurde.
Tiefe generative Modelle sind ein wichtiges Mittel zur Erzeugung synthetischer medizinischer Bilder. Sie basieren hauptsächlich auf dem Diffusionsmodell, das zur Erzeugung großer Mengen hochpräziser synthetischer Daten verwendet wird. Um Segmentierungsaufgaben für diese Daten durchführen zu können, ist eine genaue Übereinstimmung der Bild-Masken-Paare erforderlich. Methoden wie Med-DDPM und SegGuidedDiff sind darauf ausgelegt, diese Herausforderung zu meistern.Sie integrieren eine semantische Konditionierung durch kanalweises Verketten von Segmentierungsmasken in den Modell-Input, was zu Bild-Masken-Paaren führt, die konsistenten anatomischen Einschränkungen entsprechen.Ersteres ist auf die Synthese 3D-Gehirnbilder zugeschnitten, während Letzteres für die Generierung von 2D-Brust-MRTs und CTs des Abdomens konzipiert ist.
In dieser StudieDie Forscher wählten einen anderen Ansatz als das Diffusionsmodell und verwendeten einen generativen Modellierungsansatz namens Flow Matching.Es generiert neue Stichproben, indem es den kontinuierlichen Transformationsprozess von Daten von einer bekannten Verteilung in eine Zielverteilung lernt, was im Vergleich zum Diffusionsmodell eine bessere Leistung bei natürlichen Bildern zeigt.
Insbesondere verwendet das generative Modell F in der Studie ein θ-parametrisiertes Netzwerk, um ein gelerntes zeitabhängiges Geschwindigkeitsfeld v darzustellen, und ordnet dann die Stichprobe x₀~N(0,I) der Stichprobe x₁ der Datenverteilung durch eine gewöhnliche Differentialgleichung (ODE) zu. Gleichzeitig optimierten die Forscher zum Trainieren des Modells F auch das Ziel der Flussanpassung, um den Zeitskalenverlust zwischen der Vorhersagegeschwindigkeit und der Geschwindigkeit der Abtastung des wahren Werts zu minimieren.
Darüber hinaus übernimmt das Trainingsmodell F auch Masken- und Kategoriebedingungen.Die Maskenkonditionierung wird durch Verketten der Maskenkanäle mit dem Eingabebild xₜ erreicht.Die Forscher fügten den zeitlichen Einbettungen Klasseneinbettungen hinzu, um Klasseninformationen einzubinden, die dann durch Addition in die Zwischenmerkmalsebene eingefügt wurden. Um Dflow zu generieren, diskretisierten die Forscher schließlich x₁ durch Euler-Integration, um eine große Anzahl von Bildern Xflow abzutasten, wie in der folgenden Abbildung dargestellt.
Versuchsergebnisse: Die Leistung ist besser als beim derzeit fortschrittlichsten Modell
Um die Gültigkeit und Zuverlässigkeit von vesselFM zu überprüfen,Die Forscher führten eine vergleichende Auswertung durch.Und demonstrierte seine Segmentierungsfähigkeiten in Szenarien mit Nullstichproben, Einzelstichproben und wenigen Stichproben.
Speziell,In der Validierungsphase wurden vier klinische Datensätze verwendet: SMILE-UHURA, MSD8, OCTA und BvEM.Wir extrahieren drei Patches der Form 128³ aus diesen Auswertungsdatensätzen und verwenden sie, um Single-Shot- und Few-Shot-Segmentierungsaufgaben auf einem oder allen drei Patches zu definieren, um das Modell zu optimieren. Die restlichen Daten werden für Tests und Validierungen verwendet. Bei der Zero-Shot-Evaluierung wird das Modell ohne vorherige Feinabstimmung direkt auf die Testdaten angewendet.
Gleichzeitig dienten 4 speziell für die 3D-Blutgefäßsegmentierung entwickelte Basismodelle als Vergleichsobjekte.Dies sind: tUbeNet, VISTA3D, SAM-Med3D und MedSAM-2.
Mithilfe einer einzelnen NVIDIA RTX A6000 GPU haben die Forscher 10.000 Bildmasken von Modell F abgetastet und in 3 Tagen Dflow generiert. Um Ddrand zu verwalten, haben wir 500.000 Bild-Masken-Paare aus der Pipeline zur Domänenrandomisierungsgenerierung ausgewählt, jedes mit einer Form von 128³. Die Gewichte der drei verwendeten Datenquellen werden grob entsprechend ihrer Größe festgelegt, nämlich Ddrand (70%), Dreal (20%) und Dflow (10%).
Die konkreten Ergebnisse sind in der folgenden Abbildung dargestellt. vesselFM zeigt eine hervorragende Generalisierung und überlegene Leistung bei allen 4 Datensätzen und Aufgaben.Bei der Zero-Shot-Aufgabe ist der Dice-Score von vesselFM im MSD8-Datensatz um 5,86 Punkte höher als der von VISTA3D (VISTA3D wird mit 11.454 CT-Daten trainiert, die selbst Daten von MSD8 enthalten), was die starke induktive Voreingenommenheit von vesselFM weiter unterstreicht.
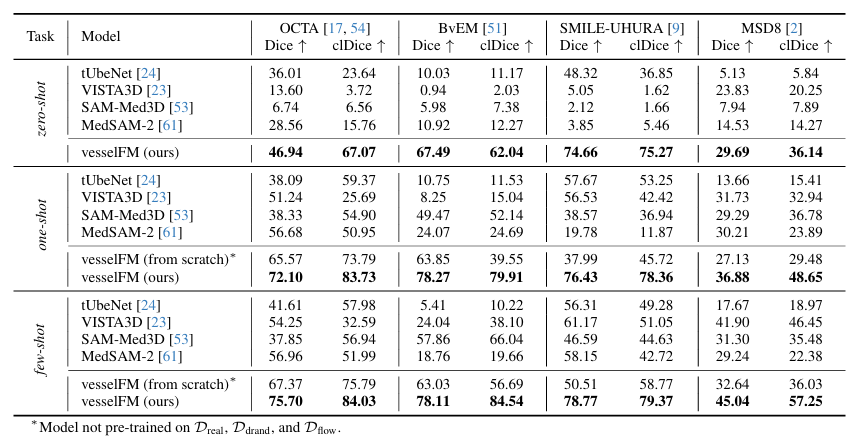
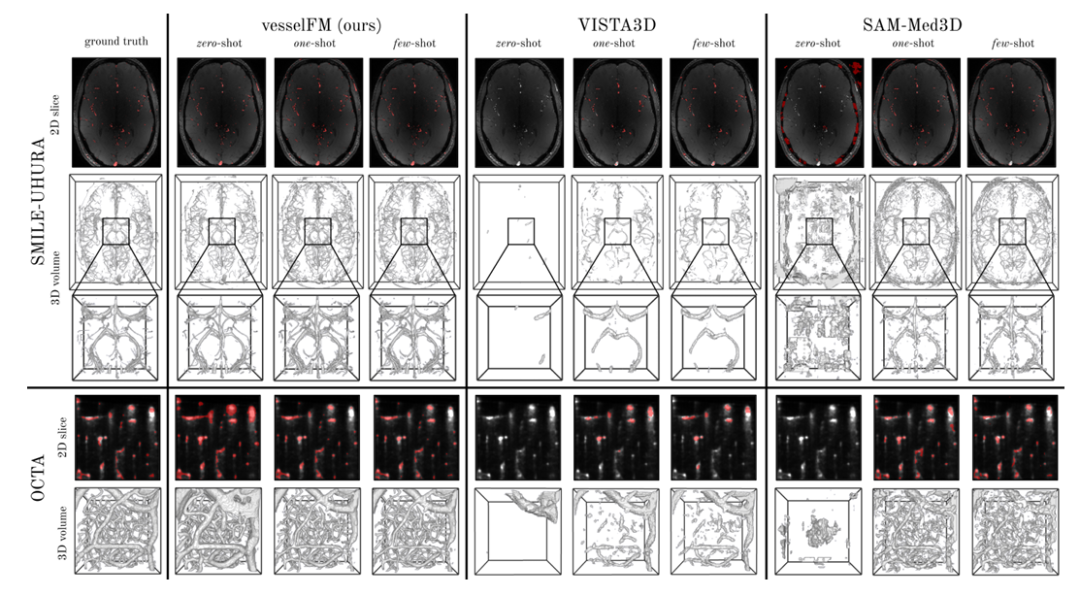
Im Gegensatz dazu weist das allgemeine 3D-Gefäßsegmentierungsmodell tUbeNet bei komplexeren Bildgebungsverfahren keine gute Leistung auf, und die beiden anderen allgemeinen Segmentierungsmodelle SAM-Med3D und MedSAM-2 sind nicht in der Lage, Gefäße in der Zero-Shot-Einstellung zu segmentieren. Es ist erwähnenswert, dass im SMILE-UHURA-Datensatz die Dice- und clDice-Werte von vesselFM in Zero-Shot-Szenarien in Few-Shot-Szenarien sogar die des Basismodells übertreffen.Qualitative Ergebnisse zeigen die überlegene Generalisierungsfähigkeit von vesselFM in Zero-Shot-Kontexten, ohne dass es unter annotatorspezifischen Verzerrungen leidet.Wie in der Abbildung unten gezeigt.
Deep Learning eröffnet neue Wege in der Erforschung der Blutgefäßsegmentierung
Zusammenfassend lässt sich sagen, dass die Forschung im Zusammenhang mit vesselFM zweifellos den Fortschritt der Forschung zur 3D-Gefäßsegmentierung gefördert und einen neuen Weg für die Behandlung und Erforschung von Gefäßerkrankungen aufgezeigt hat. Es wird erwartet, dass sie die Entstehung und Anwendung neuer fortschrittlicher Instrumente fördert und letztendlich das Ziel erreicht, den Patienten zu helfen.
Glücklicherweise,vesselFM ist mit diesem Unterfangen nicht allein.Mit der Entwicklung der Deep-Learning-Technologie und der zunehmenden Fülle medizinischer Daten ist die Verarbeitung medizinischer Bilder durch künstliche Intelligenz zu einer wichtigen Richtung der modernen medizinischen Reform geworden. Immer mehr Labore und Forschungseinrichtungen widmen sich diesem Gebiet und hoffen, durch ihre eigene Forschung die Herausforderungen der Gefäßerkrankungen zu lösen, vor denen die Menschheit steht.
So veröffentlichte beispielsweise ein Team der Chinesischen Akademie der Wissenschaften eine Studie mit dem Titel „VesselSAM: Nutzung von SAM für die Segmentierung von Aortic Vessels mit LoRA und Atrous Attention“. Die Studie schlug eine erweiterte Version von SAM mit dem Namen VesselSAM vor.Speziell für die Segmentierung von Aortengefäßen entwickelt.Das Modell integriert das Hole-Attention-Modul und die Low-Rank-Adaption (LoRA).Die wichtigsten Einschränkungen von SAM werden behoben und seine Fähigkeit, komplexe hierarchische Merkmale in medizinischen Bildern zu erfassen, wird verbessert.
Papieradresse:https://arxiv.org/abs/2502.18185
Ein Team der Shanghai Jiao Tong University in China hat zusammen mit Teams des Shanghai First People's Hospital, der Queen's University Belfast und der Louisiana State University eine Studie mit dem Titel „Self-Supervised Vessel Segmentation via Adversarial Learning“ veröffentlicht.In dieser Studie wird vorgeschlagen, zwei Generatoren durch kontroverses Lernen zu trainieren. Einer ist ein aufmerksamkeitsgesteuerter Generator und der andere ein Segmentierungsgenerator.Lassen Sie sie künstliche Blutgefäße synthetisieren und Blutgefäße aus Koronarangiographiebildern segmentieren und so die Merkmalsdarstellung von Blutgefäßen erlernen. Dieses Papier wurde auch für CVPR 2021 ausgewählt.
Ein Team der Universität Lissabon in Portugal und ein Team der Katholischen Universität Portugal veröffentlichten ebenfalls Forschungsergebnisse zur Gefäßsegmentierung.Sie schlugen 3DVascNet vor, eine auf Deep Learning basierende Software zur automatischen Segmentierung und Quantifizierung dreidimensionaler Netzhautgefäßnetzwerke.Die Software ermöglicht nicht nur eine genaue Segmentierung der Blutgefäße, sondern kann auch vaskuläre morphologische Messparameter wie Gefäßdichte, Verzweigungslänge, Gefäßradius und Verzweigungspunktdichte quantifizieren. Noch wichtiger ist, dass diese Software kostenlos ist.Gleichzeitig verbessert seine leistungsstarke Generalisierungsfähigkeit die Fähigkeit des medizinischen Personals, dreidimensionale Gefäßnetzwerke zu untersuchen.Die zugehörige Forschung wurde unter dem Titel „3DVascNet: Eine automatisierte Software zur Segmentierung und Quantifizierung von Gefäßnetzwerken von Mäusen in 3D“ veröffentlicht.
Papieradresse:https://www.ahajournals.org/doi/10.1161/ATVBAHA.124.320672
Zusammenfassend lässt sich sagen, dass die Blutgefäßsegmentierung als kritische und anspruchsvolle Aufgabe in der medizinischen Bildverarbeitung noch viele Probleme birgt, die gelöst werden müssen. Die wiederholten Versuche der Forscher haben uns jedoch zweifellos gezeigt, dass diese Probleme bald gelöst werden. Noch überzeugender ist die Annahme, dass Gefäßerkrankungen in naher Zukunft mit der zunehmenden Anwendung künstlicher Intelligenz möglicherweise schrittweise besiegt werden können.








