Command Palette
Search for a command to run...
Das Für CVPR 2025 Ausgewählte Team Des Harbin Institute of Technology Schlug Ein Hierarchisches Destillations-Multi-Instance-Lernframework HDML Vor, Um Gigapixel-Pathologie-Vollschichtbilder Schnell Zu Verarbeiten

Pathologische Bilder enthalten umfangreiche phänotypische Informationen, und die auf pathologischen Bildern basierende pathologische Diagnose gilt allgemein als „Goldstandard“ für die Krebsdiagnose. Darunter ist das Whole Slide Image (WSI) ein hochauflösendes digitales Pathologiebild, das mithilfe der digitalen Scantechnologie ganzer Objektträger pathologische Gewebeschnitte in digitale Bilder mit bis zu 1 Milliarde Pixel umwandelt. Es zeichnet sich durch eine hohe Auflösung, Panoramaanzeige und großes Datenvolumen aus. Es handelt sich um die gängige Methode der aktuellen medizinischen Diagnose und medizinischen Forschung.
Multi-Instance Learning (MIL) ist eine der wichtigsten Methoden zur Analyse von WSI und hat bei Aufgaben wie Tumorerkennung, Quantifizierung der Gewebemikroumgebung und Überlebensvorhersage gute Ergebnisse erzielt. Da WSI jedoch eine riesige Menge an Informationen enthält, ist die Schlussfolgerung mit MIL mit hohen Kosten verbunden. Das erste ist das Problem der Datenvorverarbeitung. Der WSI-Zuschneide- und Merkmalsextraktionsprozess ist sehr zeitaufwändig. Das zweite ist das Problem redundanter Patches. WSI enthält normalerweise redundante Patches, die am wenigsten zur Klassifizierung auf Taschenebene beitragen. Das Eliminieren irrelevanter Beispiele durch Aufmerksamkeitsbewertungen ist der einfachste Weg, das obige Problem zu lösen. Der vorhandene MIL-Algorithmus muss jedoch die Merkmale aller zugeschnittenen Blöcke extrahieren, bevor die Aufmerksamkeitswerte berechnet werden, was zweifellos zu einem Henne-Ei-Problem führt.
Basierend auf der obigen Analyse haben Professor Jiang Junjun, Associate Professor Jiang Kui vom Harbin Institute of Technology in China und Professor Zhang Yongbing vom Harbin Institute of Technology (Shenzhen) sowie andere eine innovative Lösung demonstriert, mit der die Denkzeit verkürzt werden kann. Das Team schlug ein hierarchisches Destillations-Multi-Instance-Lernframework (HDMIL) vor, das darauf abzielt, irrelevante Patches schnell zu identifizieren und dadurch eine schnelle und genaue Klassifizierung zu erreichen. Laut experimentellen Ergebnissen reduziert HDMIL die Inferenzzeit bei drei öffentlichen Datensätzen im Vergleich zu früheren fortschrittlichen Methoden um 28,6%.
Die entsprechenden Ergebnisse wurden unter dem Titel „Fast and Accurate Gigapixel Pathological Image Classification with Hierarchical Distillation Multi-Instance Learning“ veröffentlicht und für CVPR 2025 ausgewählt.
Forschungshighlights:
* Die vorgeschlagene Methode beschleunigt den Denkprozess und verbessert gleichzeitig die Klassifizierungsleistung. Sie erreicht ein Gleichgewicht zwischen Geschwindigkeit und Leistung, das mit herkömmlichen Methoden nicht erreicht werden kann, und bietet Inspiration für zukünftige Forschungen zur Multi-Instance-Klassifizierung.
*Diese Methode demonstrierte erstmals den auf Tschebyscheff-Polynomen basierenden Kolmogorov-Arnold-Klassifikator und wandte ihn auf die digitale Pathologie an, wodurch die Klassifizierungsleistung deutlich verbessert wurde
* Die vorgeschlagene Methode wurde durch eine große Anzahl von Experimenten verifiziert und hat zuverlässige und effektive Verifizierungsergebnisse auf 3 öffentlichen Datensätzen erzielt

Papieradresse:
https://arxiv.org/abs/2502.21130
Datensatz: Drei große öffentliche Datensätze bestätigen die Wirksamkeit
Um die Wirksamkeit des Experiments sicherzustellen, bewerteten die Forscher die Wirksamkeit der vorgeschlagenen Methode anhand von drei öffentlichen Datensätzen:
* Der Camelyon16-Datensatz wird zur Erkennung von Lymphknotenmetastasen bei Brustkrebs verwendet, wobei das Verhältnis von Trainingssatz und Validierungssatz gemäß dem offiziellen Trainingssatz 9:1 aufgeteilt wird und der offizielle Testsatz für Tests über alle Falten hinweg verwendet wird
*Der TCGA-NSCLC-Datensatz wurde zur Klassifizierung von Lungenkrebs verwendet. Der Datensatz wurde im Verhältnis 8:1:1 in Trainingssatz, Validierungssatz und Testsatz aufgeteilt.
*Bei Verwendung des TCGA-BRCA-Datensatzes zur Klassifizierung der Brustkrebs-Subtypen beträgt das Verhältnis von Trainingsdatensatz, Validierungsdatensatz und Testdatensatz ebenfalls 8:1:1
Es ist erwähnenswert, dass alle WSIs mit den von CLAM entwickelten Tools vorverarbeitet wurden und die Experimente einer 10-fachen Monte-Carlo-Kreuzvalidierung folgten.
Modellarchitektur: Die zweistufige Architektur umfasst Training und Argumentation und führt auf innovative Weise den Kolmogorov-Arnold-Klassifikator ein
Das vom Institut vorgeschlagene HDMIL-Framework umfasst zwei Teile: Training und Argumentation. In diesem Rahmen gibt es zwei Schlüsselkomponenten: Eine ist das dynamische Multi-Instance-Netzwerk (DMIN), das zur Klassifizierung hochauflösender WSI und zur Identifizierung von Instanzen dient, die nicht mit der Klassifizierung auf Beutelebene in Zusammenhang stehen; Das andere ist das Lightweight Instance Prescreening Network (LIPN), ein Netzwerk, das speziell auf WSI mit niedriger Auflösung zugeschnitten ist.
Vor dem Training verarbeiteten die Forscher die Eingabedaten zunächst gemäß dem Standardverfahren für pathologische WSI vor. Der Datensatz besteht aus S WSI-Pyramiden mit Folienbeschriftungen, wobei jedes Xᵢ ein Paar hochauflösender (20x) und niedrig auflösender (1,25x) WSIs enthält, die jeweils als Xᵢ,ₕᵣ und Xᵢ,ₗᵣ bezeichnet werden.
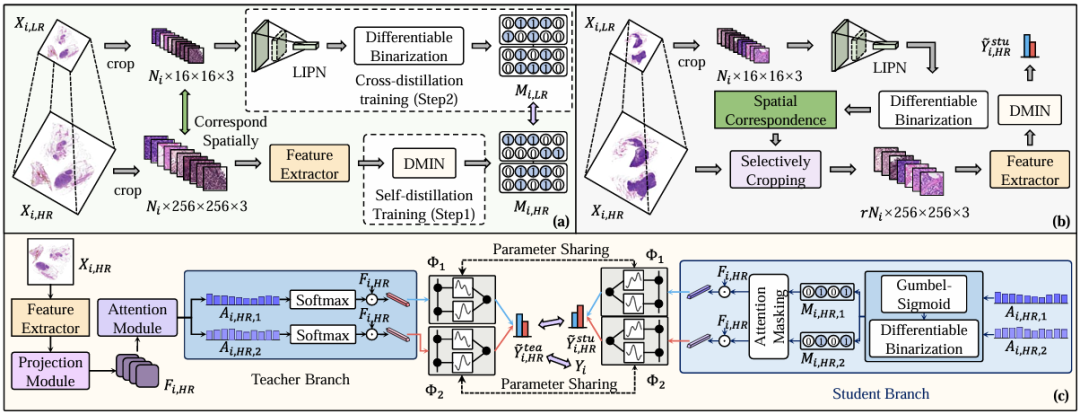
Insbesondere zeigt Abbildung a die Trainingsphase, wie in der folgenden Abbildung dargestellt. Die Forscher wendeten zunächst eine Selbstdestillations-Trainingsstrategie an, um DMIN mit hochauflösendem WSI (Xᵢ,ₕᵣ) zu trainieren, wodurch es eine Klassifizierung auf Beutelebene durchführen und irrelevante Regionen anzeigen konnte. Obwohl DMIN irrelevante Regionen in WSI erfolgreich identifizierte, verbesserte es die Inferenzgeschwindigkeit nicht. Denn DIMN muss die Features aller vom Feature-Extraktor generierten Patches verwenden, um zu bestimmen, welche Instanzen eliminiert werden sollen, und die patchweise Feature-Extraktion ist tatsächlich der Schlüssel zum Überwinden des Engpasses bei der WSI-Inferenzgeschwindigkeit.
Daher froren die Forscher dann das DMIN ein und verwendeten die resultierende Maske, um das LIPN zu extrahieren. Wie oben erwähnt, ist LIPN ein leichtes Instanz-Pre-Screening-Netzwerk, das auf WSI mit niedriger Auflösung zugeschnitten ist. Es wird durch Kreuzdestillation unter Verwendung von WSI mit niedriger Auflösung (Xᵢ,ₗᵣ) trainiert und kann schnell irrelevante Bereiche in WSI mit niedriger Auflösung identifizieren und so indirekt auf irrelevante Bereiche in WSI mit hoher Auflösung hinweisen.
Im Hinblick auf die konkrete Implementierung übernahmen die Forscher das weit verbreitete ResNet-50 als Merkmalsextraktor, ein auf ImageNet vortrainiertes Modell, und verwendeten eine leichtgewichtige Variante von MobileNetV4 für das Vorab-Screening-Netzwerk LIPN. Durch die oben genannten Schritte gelang es den Forschern, die binäre Wichtigkeit (wichtig oder nicht wichtig) jeder Region mit sehr geringem Rechenaufwand zu beurteilen.
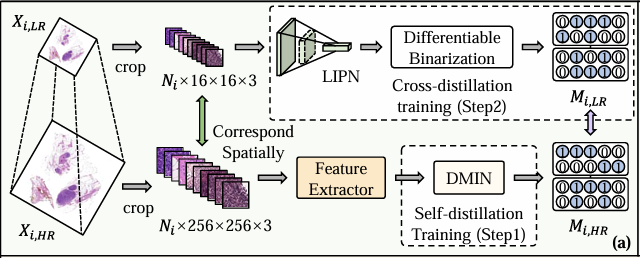
Abbildung c zeigt das Selbstdestillationstraining von DMIN auf hochauflösendem WSI (Xᵢ,ₕᵣ), wie unten dargestellt. Es ist ersichtlich, dass DMIN aus fünf Modulen besteht, darunter das Projektionsmodul, das Aufmerksamkeitsmodul, der Lehrerzweig, der Schülerzweig und die CKA-Klassifikatoren.
Insbesondere werden alle aus dem hochauflösenden WSI (Xᵢ,ₕᵣ) extrahierten Patches zuerst in den vortrainierten Feature-Extraktor eingegeben, um einen Satz von Beispiel-Features Iᵢ,ₕᵣ zu generieren, die dann zur Dimensionsreduzierung in das Projektionsmodul eingegeben werden, um einen neuen Feature-Satz Fᵢ,ₕᵣ zu erhalten, der dann in das Aufmerksamkeitsmodul eingegeben wird, um den nicht normalisierten Aufmerksamkeitswert zu berechnen.
Im Lehrerzweig wird das reduzierte Fᵢ,ₕᵣ mithilfe der Aufmerksamkeitsmatrix jeder Klasse linear gewichtet, um eine Darstellung auf Bag-Ebene für die endgültige Klassifizierung zu erstellen. Zur Berechnung der Bag-Level-Darstellung wird im Schülerzweig nur eine Teilmenge von Beispielen mit hohen Aufmerksamkeitswerten verwendet. Außerdem legen die Forscher Einschränkungen fest, um sicherzustellen, dass die Bag-Level-Darstellung so konsistent wie möglich mit der im Lehrerzweig unter Verwendung aller Instanzen erhaltenen Darstellung ist. Durch diese Methode wird das Aufmerksamkeitsmodul implementiert, um den Instanzen, die für die Klassifizierung auf Bag-Ebene wichtiger sind, mehr Aufmerksamkeit zu schenken und irrelevante Instanzen herauszufiltern. Gleichzeitig wendet der Optimierungsprozess auch den Gumbel-Trick an, um selektiv Instanzen mit höheren Aufmerksamkeitswerten für das End-to-End-Training zu verwenden und so das Auftreten nicht differenzierbarer Probleme zu vermeiden.
Um die Fähigkeiten des MIL-Klassifikators zu verbessern, schlugen die Forscher schließlich vor, ein Kolmogorov-Arnold-Netzwerk zu verwenden, um nichtlineare Aktivierungsfunktionen zu erlernen, anstatt feste Aktivierungsfunktionen im Klassifikator zu verwenden. Und durch die Entwicklung einer hybriden Verlustfunktion erreichten die Forscher drei Trainingsziele für DMIN. Der erste ist, dass der Lehrerzweig Xᵢ,ₕᵣ korrekt klassifizieren kann; Zweitens können die Klassifizierungsergebnisse bei Verwendung einiger Instanzen im Schülerzweig mit den Klassifizierungsergebnissen bei Verwendung aller Instanzen im Lehrerzweig übereinstimmen. und drittens sollte der Anteil der ausgewählten Instanzen kontrollierbar sein.
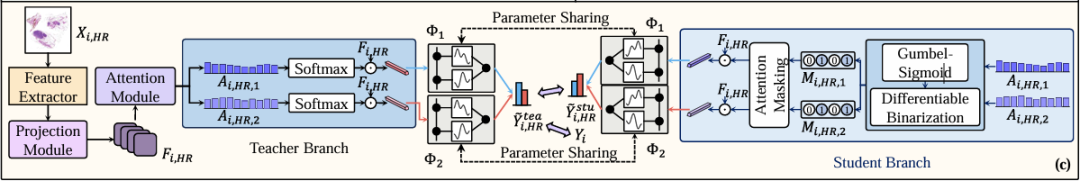
Abbildung b zeigt die Argumentationsphase, wie unten dargestellt. Der spezifische Prozess kann in drei Schritte unterteilt werden: Der erste Schritt besteht darin, alle Patches im WSI mit niedriger Auflösung (Xᵢ,ₗᵣ) mit insgesamt Nᵢ zuzuschneiden; Der zweite Schritt besteht darin, diese Patches in LIPN einzugeben, um klassifizierungsbezogene Bereiche zu identifizieren und Mᵢ,ₗᵣ zu generieren. Der dritte Schritt besteht darin, die entsprechenden Patches in Xᵢ,ₕᵣ basierend auf Mᵢ,ₗᵣ selektiv auszuschneiden, dann die verbleibenden Patches in den Feature-Extraktor und DMIN einzugeben und sie schließlich separat über die kategorieübergreifenden Studentenzweige zu berechnen, um die endgültigen Klassifizierungsergebnisse zu generieren.
Forschungsergebnisse: „Vereinfachtes“ HDMIL übertrifft bestehende fortgeschrittene Methoden immer noch
Basierend auf den drei Datensätzen von Camelyon16, TCGA-NSCLC und TCGA-BRCA verglichen die Forscher die Klassifizierungsleistung von HDMIL mit 11 MIL-Methoden, darunter Max-Pooling, Mean-Pooling, ABMIL, CLAMSB, CLAMMB, DSMIL, TransMIL, DTFDAFS, DTFDMAS, S4MIL und MambaMIL.
Es ist erwähnenswert, dass die Forscher verschiedene HDMIL-Konfigurationen getestet haben, nämlich HDMIL† und HDMIL. Ersteres bedeutet, dass nur DMIN zum Schlussfolgern verwendet wird, ohne dass eine Vorabprüfung durch LIPN erfolgt. Die spezifischen Ergebnisse sind in der folgenden Abbildung dargestellt:
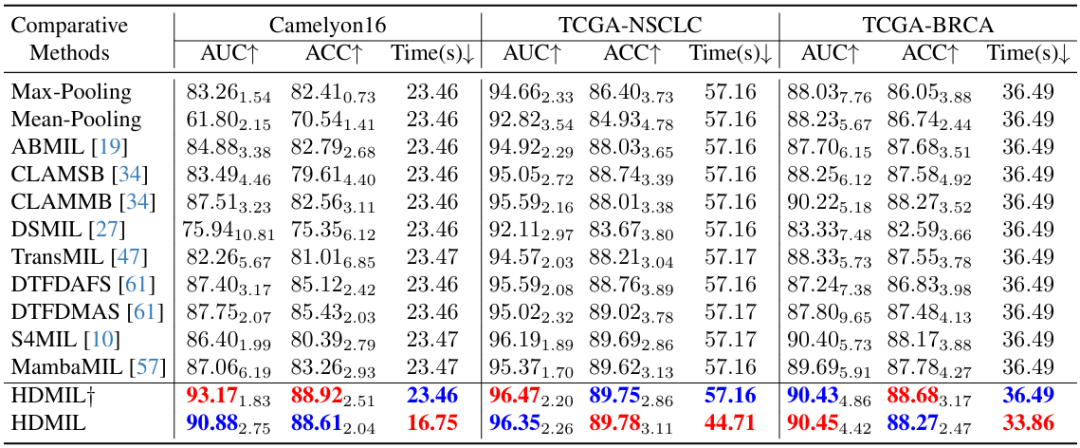
Es ist ersichtlich, dass sowohl HDMIL† als auch HDMIL bei den drei Datensätzen durchweg bessere Testergebnisse erzielen als bestehende Methoden. Beispielsweise erreichte HDMIL im Camelyon16-Datensatz eine AUC von 90,88% und eine Genauigkeit von 88,61%, was 3,13% bzw. 3,18% höher ist als bei der vorherigen besten Methode.
Gleichzeitig verbessert HDML die Geschwindigkeit, wenn der Datensatz groß genug ist, ohne die Klassifizierungsleistung zu beeinträchtigen. Beispielsweise enthalten TCGA-NSCLC und TCGA-BRCA beide etwa 1.000 WSIs, aber die Lücke in der Testleistung zwischen HDML† und HDML ist nicht groß, was beweist, dass HDML ein ausgezeichnetes Gleichgewicht zwischen Inferenzgeschwindigkeit und Klassifizierungsleistung erreicht hat.
Darüber hinaus ist HDMIL† hinsichtlich der Verarbeitungszeit mit anderen bestehenden Methoden vergleichbar, wobei HDMIL alle Methoden deutlich übertrifft, da HDMIL† dieselbe Anzahl hochauflösender Patches verarbeiten muss wie andere Methoden. HDMIL reduziert den Zeitaufwand für die Datenverarbeitung durch LIPN und reduziert so den Zeitaufwand für die Inferenz im Vergleich zu anderen Methoden bei den drei Datensätzen erheblich, wodurch Geschwindigkeitssteigerungen von 28,6%, 21,8% bzw. 7,2% erreicht werden.
Um die Auswirkungen der einzelnen Komponenten zu analysieren, führten die Forscher ein Ablationsexperiment durch, um die Auswirkungen jedes Moduls in HDML auf die Klassifizierungsergebnisse weiter zu veranschaulichen, wie in der folgenden Abbildung dargestellt. Die Forschungsergebnisse zeigen, dass das Ersetzen des herkömmlichen linearen schichtbasierten Klassifikators durch den vorgeschlagenen CKA-Klassifikator und die Einbeziehung der Selbstdestillation in das DMIN-Training die Klassifizierungsleistung erheblich verbessern.
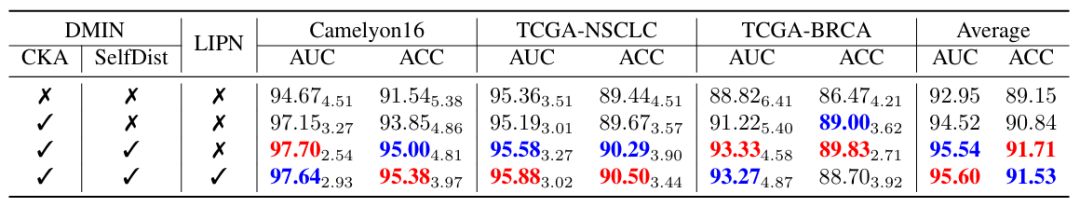
Generell handelt es sich bei dem HDMIL-Vorschlag zweifellos um eine neue Idee und einen neuen Versuch. Die Machbarkeit dieser Idee wurde durch zahlreiche Experimente bewiesen. Es bietet eine neue Methode zur Analyse pathologischer Bilder, insbesondere WSI, mithilfe der MIL-Methode und beschleunigt die dynamische Entwicklung der digitalen Pathologie.
Die digitale Pathologie floriert mit KI
In den letzten Jahren hat die rasante Entwicklung der digitalen Pathologie zu einer neuen Runde von Fortschritten in der Medizin und Biologie geführt und spielt insbesondere im Kampf gegen den Krebs, einen der größten Feinde der Menschheit, eine wichtige Rolle. Es ist erwähnenswert, dass der Vorschlag von HDMIL nicht der erste Versuch des Teams des Harbin Institute of Technology auf diesem Gebiet ist.
Im letzten Jahr enthielt CVPR 2024 eine Studie mit dem Titel „Virtuelle immunhistochemische Färbung für histologische Bilder mithilfe von schwach überwachtem Lernen“. Der Artikel erwähnte eine schwach überwachte Lernmethode namens Confusion-GAN für die virtuelle Immunhistochemie (IHC)-Färbung, die H&E-Bilder in IHC-Bilder umwandeln kann und so die umständlichen und hohen Kosten herkömmlicher Methoden der IHC-Färbung löst.

Adresse des Artikels: https://openaccess.thecvf.com/content/CVPR2024/papers/Li_Virtual_Immunohistochemistry_Staining_for_Histological_Images_Assisted_by_Weakly-supervised_Learning_CVPR_2024_paper.pdf
Neben denselben Autoren wie bei der oben genannten Forschung wurden auch Professor Jiang Junjun und Professor Zhang Yongbing als Co-Autoren dieses Artikels genannt, was die tiefgreifende Entwicklung und Ansammlung des Harbin Institute of Technology auf diesem Gebiet weiter bestätigt.
Natürlich verdienen auch Professor Jiang Junjun und Professor Zhang Yongbing als die beiden korrespondierenden Autoren des Artikels eine besondere Erwähnung. Professor Jiang Junjun ist derzeit ordentlicher Professor und Doktorvater an der School of Computer Science des Harbin Institute of Technology, Prodekan der School of Artificial Intelligence und stellvertretender Direktor des Forschungszentrums für intelligente Schnittstellen und Mensch-Computer-Interaktion. Er wurde für das National Youth Talent Program ausgewählt und ist außerdem akademischer Leiter des „Young Scientist Studio“ des Harbin Institute of Technology. Seine Forschungsrichtungen umfassen Bildverarbeitung, Computer Vision, Deep Learning (Forschungsschwerpunkte sind große Modelle und Bildverarbeitung, multimodale autonome unbemannte Systeme, generative künstliche Intelligenz usw.) und andere Bereiche.
Professor Zhang Yongbing ist derzeit Professor und Doktorvater an der School of Computer Science des Harbin Institute of Technology. Zu seinen wichtigsten Forschungsgebieten zählen Computer Vision, biomedizinische Bildverarbeitung und Computerbildgebung. Darüber hinaus hat Professor Zhang Yongbing mehrere Positionen inne. Er ist Mitglied vieler namhafter in- und ausländischer Verbände, darunter der China Computer Society, der China Artificial Intelligence Society, IEEE, SPIE, OSA usw. Er hat über 100 Beiträge auf führenden internationalen Konferenzen zum Thema künstliche Intelligenz veröffentlicht und ihm wurden über 50 Erfindungspatente erteilt. Derzeit konzentriert sich Professor Zhang Yongbings Forschung hauptsächlich auf die Anwendung künstlicher Intelligenz und Computervision in den Bereichen Biomedizin und medizinische Gesundheit.
Neben dem Harbin Institute of Technology widmen sich auch immer mehr Universitäten und Labore dem Bereich der digitalen Pathologie und leisten ihren Beitrag. So veröffentlichte beispielsweise ein Team der Technischen Universität Eindhoven in den Niederlanden eine Studie mit dem Titel „A Spatially-Aware Multiple Instance Learning Framework for Digital Pathology“, in der ein Modell namens Global ABMIL (GABMIL) vorgeschlagen wurde. Dieses Modell ist eine erweiterte Version des traditionellen ABMIL-Modells. Es kann räumliche Informationen über das Modul zum Mischen räumlicher Informationen in den Einbettungsvektor integrieren und dann das ABMIL-Netzwerk verwenden, um die Schnittbezeichnung vorherzusagen. Dadurch wird vermieden, dass bei der herkömmlichen MIL-Methode häufig ein Schlüsselfaktor bei der pathologischen Diagnose ignoriert wird – die räumlichen Interaktionsinformationen zwischen Bildblöcken.
Adresse des Artikels: https://arxiv.org/abs/2504.17379
Kurz gesagt: Die Integration von künstlicher Intelligenz und traditioneller Medizin ist unumkehrbar und jeder kann davon profitieren. Es lässt sich nicht leugnen, dass es diese „Forscher“, die sich für die Spitzenforschung einsetzen, sind, die uns die Möglichkeit geben, von den Anwendungsmöglichkeiten der wechselseitigen Integration von künstlicher Intelligenz und Medizin zu profitieren. Natürlich gibt es Grund zur Annahme, dass das Team des Harbin Institute of Technology durch eine langfristige, intensive Kultivierung hier weiterhin Fuß fassen und so die Entwicklung des gesamten Bereichs beschleunigen wird.








