Command Palette
Search for a command to run...
Die National University of Singapore/MIT Und Andere Erstellen Ein Auf SVM Basierendes Modell Zur Erkennung Mikrobieller Kontaminationen Und Geben Ergebnisse Innerhalb Von 30 Minuten aus.
Als wichtiger Bestandteil fortschrittlicher therapeutischer Arzneimittel (ATMPs) geben Zelltherapieprodukte (CTPs) Patienten mit seltenen und hartnäckigen Krankheiten Hoffnung. Allerdings ist der Produktionsprozess äußerst anfällig für mikrobielle Invasionen und eine mikrobielle Kontamination schwebt stets wie ein Schleier über diesem Hoffnungsschimmer. Traditionelle Sterilitätstestmethoden, wie etwa USP <71> (Sterilitätstestmethoden, die in Kapitel 71 der US-Pharmakopöe aufgeführt sind), die seit einem halben Jahrhundert verwendet werden, scheinen den neuen Anforderungen der Präzisionsmedizin nicht gerecht zu werden: Der zweiwöchige Kulturzyklus, die umständlichen Vorbehandlungsschritte und Trübungsbeobachtungen, die auf subjektiven Urteilen beruhen, bleiben nicht nur weit hinter der kurzen Haltbarkeit von Zellpräparaten zurück, sondern können Patienten aufgrund von Fehleinschätzungen auch einem Infektionsrisiko aussetzen.
Mit der rasanten Entwicklung von Zelltherapieprodukten ist die schnelle und genaue Erkennung mikrobieller Kontaminationen immer dringlicher geworden. Eine innovative Nachweismethode ist entstanden: die maschinenlerngestützte UV-Absorptionsspektroskopie. Es kombiniert optische Technologie mit leistungsstarken Methoden des maschinellen Lernens. Es sind keine großen Mengen an Trainingsdaten oder Wachstumsanreicherungsschritten erforderlich. Es wird nur eine kleine Probenmenge benötigt, um die Testergebnisse in nur 30 Minuten auszugeben, was einen starken Schutz für die Sicherheit von Zelltherapieprodukten bietet.
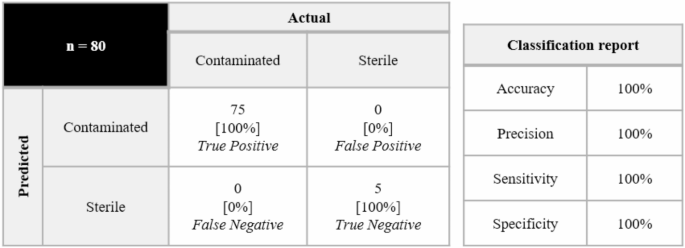
Kürzlich hat ein gemeinsames Forschungsteam der Singapore-MIT Research Alliance, des Singapore A*SRL Laboratory, der National University of Singapore und des Massachusetts Institute of Technology eine Nachweismethode vorgeschlagen, die Ultraviolett-Absorptionsspektroskopie mit maschinellem Lernen kombiniert und mit der sich mikrobielle Verunreinigungen im Zellkulturüberstand innerhalb von 30 Minuten nachweisen lassen. Diese Methode verwendet eine Ein-Klassen-Support-Vektor-Maschine (Ein-Klassen-SVM), um die charakteristischen Unterschiede zwischen Nicotinamid und Niacin im ultravioletten Spektrum zu analysieren, und weist beim Testen von sieben häufigen kontaminierenden Mikroorganismen eine durchschnittliche Richtig-Positiv-Rate von 92,7% auf. Selbst nach Ausschluss von Spenderproben mit abnormalem Niacinstoffwechsel erreicht die echte Negativrate immer noch 92% und übertrifft damit die Genauigkeit traditioneller, auf Erfahrung beruhender Beurteilungen bei weitem.
Die entsprechenden Forschungsergebnisse wurden im Nature-Journal Scientific Reports unter dem Titel „Machine learning aided UV absorbance spectroscopy for microbial contamination in cell therapy products“ veröffentlicht.

Papieradresse:https://hyper.ai/en/sota/papers/s41598-024-83114-y
Folgen Sie dem öffentlichen Konto „HyperAI Super Neural“ und antworten Sie mit „Microbial Contamination Monitoring“, um das vollständige PDF zu erhalten
Datensatz: Daten steriler MSC-Kulturproben, gesammelt mit einem kommerziellen Spektrometer
In dieser Studie drehten sich die Konstruktion und Analyse des Datensatzes eng um die Kultur mesenchymaler Stromazellen (MSC). Angesichts der breiten Anwendung der MSC-Therapie bei der Behandlung akuter Gewebeverletzungen, entzündlicher Erkrankungen und chronischer degenerativer Erkrankungen wählte das Forschungsteam sie als Demonstrationsobjekt aus und verwendete ein kommerzielles Spektrometer, um die Absorptionsspektren steriler MSC-Kulturproben als Basisdaten für das Training eines einklassigen Support Vector Machine-Modells (einklassiges SVM-Modell) zu erfassen. Die Studie verwendete eine Strategie zur Anomalieerkennung, um den Kontaminationsstatus von Zellkulturproben durch Erfassung der Unterschiede in spektralen Merkmalen vorherzusagen, und untersuchte den potenziellen Mechanismus des SVM-Modells bei der Identifizierung von Kontaminationen basierend auf der Hypothese spektraler Unterschiede zwischen Niacin- (NA) und Nicotinamid- (NAM) Metaboliten.
Während des Experiments inokulierten die Forscher 10 KBE E. coli in das MSC-Kultursystem von Spender A und konnten nach 21 Stunden erfolgreich das Kontaminationssignal erkennen. Nach dem Vergleich der Erkennungsleistung von Proben von sieben kommerziellen Spendern zeigte Spender A gute Ergebnisse bei der Identifizierung steriler Proben von anderen Spendern und wurde daher als Quelle der ersten Trainingsdaten ausgewählt. In nachfolgenden Studien gelang mit dieser Methode nicht nur die wirksame Erkennung von sieben Arten von Mikroorganismen mit Kontaminationswerten von nur 10 KBE, sondern auch die Überprüfung der Robustheit der Technologie durch spenderübergreifende Tests.
Um die Leistung des SVM-Modells gründlich zu analysieren, wurde in dieser Studie die Technik der Hauptkomponentenanalyse (PCA) eingeführt. Als Forschungsobjekte wurden mit Pseudomonas aeruginosa inokulierte Proben verwendet, da diese bei der Erkennung mittels Flüssigkeitschromatographie-Massenspektrometrie (LC-MS) die höchste NA-Konzentration aufwiesen. Die Verteilung der Trainingsdatensätze und kontaminierten Proben wurde mittels PCA visualisiert. Die Ergebnisse zeigten, dass kontaminierte und sterilisierte Proben räumlich deutlich voneinander getrennt waren, sodass effektiv zwischen sterilem und kontaminiertem Zustand unterschieden werden konnte. Die Ladevektoren der Hauptkomponente 1 (PC 1) und der Hauptkomponente 2 (PC 2) wurden weiter analysiert und die normalisierten UV-Absorptionsspektren von 100 μg/ml NA und NAM in PBS wurden kombiniert, um die Verbindung zwischen den spektralen Merkmalen und den Hauptkomponenten intuitiv darzustellen.
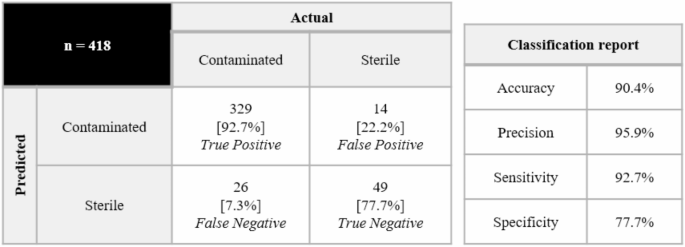
In der Phase der Überprüfung der Modellrobustheit wurden im Rahmen der Studie Abfallkulturmedium- und PBS-Sterilisationsproben von 6 gewerblichen Spendern (Spender-BG) gesammelt, um einen spenderübergreifenden Testdatensatz zu erstellen. Durch das Trainieren von SVM-Modellen aus verschiedenen Spenderquellen und die Kreuzvalidierung wurde festgestellt, dass das auf Grundlage der Spender A und B trainierte Modell eine höhere Vorhersagegenauigkeit aufwies. Die Analyse von 418 Testproben zeigte, dass die wahre Positivrate des Modells 92,71 TP3T erreichte und die Nachweisgrenze stabil bei 10 KBE gehalten wurde; Die Rate der echten Negativergebnisse von 77,71 TP3T deutete jedoch darauf hin, dass noch Verbesserungsbedarf besteht. Besonders ausgeprägt war dabei das Problem der falsch positiven Ergebnisse, das durch abnormale NA-Werte in der Probe des Spenders F verursacht wurde. Dies wies die Richtung für die anschließende Optimierung der Anpassungsfähigkeit des Modells an Stoffwechselunterschiede zwischen Spendern auf.
Maschinell lernende Ultraviolett-Absorptionsspektroskopie: Support Vector Machine als Kernalgorithmus
Damit maschinelles Lernen die Erkennung mikrobieller Kontamination besser unterstützen kann, wurde es in dieser Studie auf innovative Weise mit der Ultraviolett-Absorptionsspektroskopie kombiniert, um eine schnelle, empfindliche und kostengünstige Methode zur Erkennung mikrobieller Kontamination vorzuschlagen. Diese Methode verwendet Support Vector Machine (SVM) als Kernalgorithmus und identifiziert mikrobielle Kontamination präzise durch Analyse der Eigenschaften des Ultraviolettabsorptionsspektrums des Zellkulturmediums.
Für die Modellkonstruktion verwendeten die Forscher eine Support Vector Machine mit einer einzigen Klasse und wählten die Radialbasisfunktion (RBF) als Kernelfunktion aus. Dabei setzten sie den γ-Wert auf 0,002 und den ν-Wert auf 0,2. Der Trainingsdatensatz bestand aus Spender 5 von Tag 2 bis 7 der Passagen 2, 4 und 6 sowie mit PBS angereicherten sterilen Proben von Spender 8, die alle mit 1 gekennzeichnet waren, was den sterilen Status darstellt. Die Absorptionsdaten jeder Probe deckten den Wellenlängenbereich von 237 nm bis 300 nm ab, um sich auf die signifikanten spektralen Merkmale von Niacin (NA) und Nicotinamid (NAM) zu konzentrieren und Störungen durch Rauschen in anderen Bändern zu vermeiden. Alle Proben wurden vor dem Modelltraining mittelwertzentriert, um durch Instrumentendrift und andere Faktoren verursachte spektrale Abweichungen zu korrigieren und so die Genauigkeit des Modells zu verbessern.
Im Hinblick auf die Modellvalidierung und -anwendung wurde die Fähigkeit dieser Methode zur Erkennung niedrig konzentrierter Schadstoffe mithilfe der maschinenlerngestützten UV-Absorptionsspektroskopie demonstriert. Dazu wurden der MSC-Kultur von Spender A 10 KBE E. coli hinzugefügt und Überstandsproben dreimal im Abstand von 3 Stunden zwischen 9 und 24 Stunden entnommen. Die Ergebnisse sind in der folgenden Abbildung dargestellt. Das SVM-Modell konnte genau vorhersagen, dass die Probe nach 21 Stunden kontaminiert war, und die gesamte Erkennungszeit betrug ungefähr 21,5 Stunden.
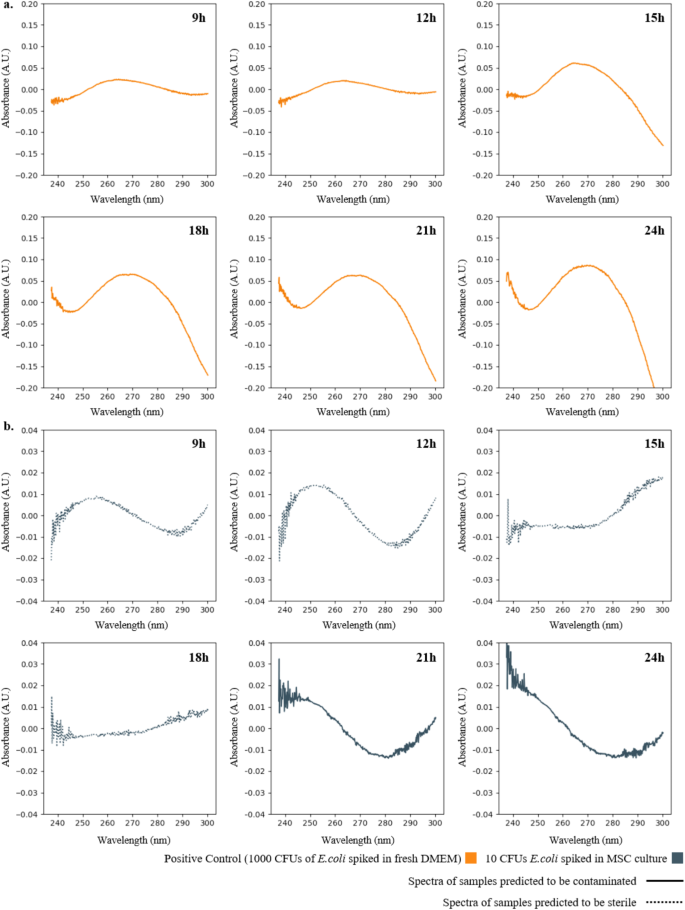
Darüber hinaus wurde in der Studie die maschinell lernende UV-Absorptionsspektroskopie mit bestehenden Methoden verglichen. Die Ergebnisse zeigten, dass die durch maschinelles Lernen unterstützte UV-Absorptionsspektroskopie-Methode eine Nachweiszeit (TTD) von 21 Stunden hatte, was vergleichbar ist mit dem USP <71>-Test (24 Stunden bis zur Beobachtung einer Trübung) und der Methode zur Berechnung des NA/NAM-Verhältnisses mittels LC-MS (18 Stunden). Allerdings erfordern sowohl BacT/Alert® 3D als auch USP <71> geschulte Bediener, um Proben aus Zellkulturen zu entnehmen und in verschiedene Wachstumsanreicherungsmedien zu impfen. Der Arbeitsablauf bei der UV-Absorptionsspektroskopie ist relativ einfach und erfordert weder eine zusätzliche Beimpfung in ein Wachstumsanreicherungsmedium noch zusätzliche Inkubationszeit und Probenvorbereitung. Somit entfallen die zusätzlichen Ressourcen und Kosten, die für den Beimpfungsschritt zur beobachteten Wachstumsanreicherung erforderlich sind.
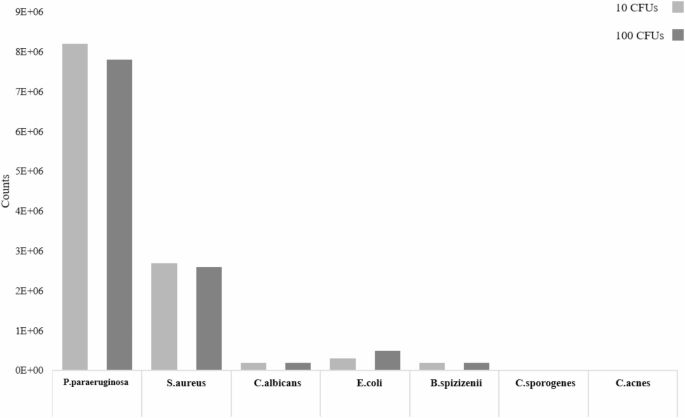
Um festzustellen, ob die vorgeschlagene, maschinenlerngestützte Methode der UV-Absorptionsspektroskopie auf andere Mikroorganismen anwendbar ist, verwendeten die Forscher PBS-sterilisierte Proben von Spender A und wendeten die Methode auf die Erkennung anderer langsam wachsender Organismen an, wie etwa Staphylococcus aureus (S. aureus), Pseudomonas aeruginosa (P. paraeruginosa), Bacillus subtilis (B. spizizenii), Clostridium perfringens (C. sporogenes), Hefen wie Candida albicans (C. albicans), Escherichia coli K-12 (E. coli) und Propionibacterium acnes (C. acnes), mit einer Nachweisgrenze (LoD) von nur 10 KBE. Darüber hinaus visualisierte die Studie auch die Leistung des SVM-Modells durch Hauptkomponentenanalyse (PCA) und stellte fest, dass sich die mit Pseudomonas aeruginosa beimpften Proben im PCA-Diagramm deutlich von den mit PBS sterilisierten Proben unterschieden, was darauf hindeutet, dass das Modell die Unterschiede in den spektralen Merkmalen effektiv erfassen kann.
Im Hinblick auf die Studie zur Robustheit des Modells sammelten die Forscher unter Berücksichtigung der Tatsache, dass Unterschiede zwischen Spendern die Leistung des Modells beeinträchtigen können, Abfallproben des Kulturmediums von sechs gewerblichen Spendern (Spender-BG) und bereiteten mit PBS sterilisierte Proben vor. Durch das Trainieren von SVM-Modellen auf der Grundlage verschiedener Spender und die Bewertung ihrer Vorhersagegenauigkeit anhand von Proben anderer Spender wurde festgestellt, dass die Modelle der Spender A und B höhere durchschnittliche Vorhersageraten hatten. Daher wurde in dieser Studie das anhand der Spender A und B trainierte SVM-Modell anschließend auf Proben der anderen 6 Spender angewendet. Die Ergebnisse zeigten, dass die maschinenlerngestützte UV-Absorptionsspektroskopie eine echte Positivrate von 92,71 TP3T erreichte und die Nachweisgrenze (LoD) für die sieben getesteten Mikroorganismen konstant bei 10 KBE blieb. Die Rate der echten Negativbefunde des Modells lag jedoch bei 77,7% und die falsch positiven Proben stammten hauptsächlich von Spender F. Die Analyse ergab, dass der NA-Wert in den Proben von Spender F höher war, was darauf schließen lässt, dass das Modell in Zukunft weiter optimiert werden muss, um seine Anpassungsfähigkeit an Unterschiede zwischen Spendern zu verbessern.
AI4S ermöglicht Zelltherapie: Wissenschaftliche Forschung, Industrie und Politik schreiten synergetisch voran
In den letzten Jahren haben Zelltherapieprodukte (CTP) von der wissenschaftlichen Forschung bis zur Geschäftswelt, von Laboren bis zur Industrialisierung große Erfolge erzielt.
Im Bereich der Grundlagenforschung gewann das Team TJ-AI4S der Tongji-Universität in Shanghai, China, die Global AI Drug Development Algorithm Competition. Die von ihnen vorgeschlagene Strategie zur Erweiterung molekularer Merkmale verbesserte die Generalisierungsfähigkeit des Modells und lieferte neue Ideen für den Aufbau der molekularen Fingerabdruckbibliothek der CTP-Schadstoffe. Das von der Beijing University of Posts and Telecommunications und der Peking University in China entwickelte UniBind-Framework analysiert Proteininteraktionen durch multiskalige Graph-Neuralnetzwerke und bietet eine rechnerische Grundlage für die Untersuchung der dynamischen Assoziationen zwischen Zytokinen und mikrobiellen Metaboliten in CTP.
Link zum Artikel: https://www.nature.com/articles/s41591-023-02483-5
Darüber hinaus hat ein Team der ShanghaiTech University in China die CAR-Toner-Plattform entwickelt, die KI-Algorithmen verwendet, um die Ladungsverteilung von CAR-Molekülen zu optimieren, wodurch die Effizienz der Zellexpansion erfolgreich verbessert und Chargenunterschiede reduziert werden. Diese Innovation verbessert nicht nur die Produktionseffizienz, sondern bietet auch neue technische Unterstützung für die standardisierte Produktion von CTP.
Link zum Artikel: https://www.nature.com/articles/s41422-024-00936-1
Auch aus der Branche gibt es gute Nachrichten. Das CAR-T-Medikament „Yikeda“ des chinesischen Biotechnologieunternehmens Fosun Kairui erreichte 2025 seine erste grenzüberschreitende Lieferung in China. Dahinter steht ein KI-gesteuertes Kühlketten-Logistikmanagementsystem, das die Aktivität der Zellen während des Transports bei extrem niedrigen Temperaturen sicherstellt. Darüber hinaus hat das US-Biotechnologieunternehmen A2 Bio durch KI-Screening universeller Spenderzellen eine groß angelegte Vorproduktion von CAR-T-Medikamenten erreicht, was die Produktionskosten erheblich gesenkt und die Wartezeit für die Behandlung verkürzt hat, was den Patienten mehr Hoffnung gibt.
Auch ausländische Forschungseinrichtungen haben im Bereich der KI-gestützten Zelltherapie fruchtbare Ergebnisse erzielt. Im Jahr 2025 entwickelte ein Team für Biomedizintechnik an der Duke University in den USA die PepPrCLIP-Technologie, die funktionale Kurzpeptide auf Grundlage des Proteinsprachenmodells ESM-2 entwarf und damit eine neue Strategie für eine präzise Krebsbehandlung ermöglichte. Das von IBM Watson Health in Zusammenarbeit mit der Stanford University in den USA entwickelte KI-Vorhersagemodell hat die Vorhersagegenauigkeit des Zytokinfreisetzungssyndroms (CRS) bei der CAR-T-Therapie auf 89% verbessert, indem es Genom-, Proteom- und mehrdimensionale klinische Daten des Patienten integriert und so klinische Interventionen bei Hochrisikopatienten im Voraus ermöglicht. Diese haben der Entwicklung im Bereich der Zelltherapie weltweit neue Dynamik verliehen.
Derzeit hat AI4S eine komplette Kette von der Grundlagenforschung bis zur klinischen Anwendung im CTP-Bereich aufgebaut, muss aber in Aspekten wie der Datenstandardisierung und den Mechanismen der einrichtungsübergreifenden Zusammenarbeit noch weiter verbessert werden. Mit Blick auf die Zukunft wird AI4S voraussichtlich mit verstärkter politischer Unterstützung und einer tiefgreifenden Integration von Industrie, Wissenschaft und Forschung größere Durchbrüche in Bereichen wie der personalisierten CTP-Vorbereitung, der Echtzeit-Rückverfolgbarkeit der Qualität und der Optimierung der grenzüberschreitenden Logistik erzielen.
Referenzartikel:
- https://mp.weixin.qq.com/s/VZI7pm-kO7CxNJyrn9-qQw
- https://mp.weixin.qq.com/s/0AoP5XSLOLzcTChQoIIfmw
- https://mp.weixin.qq.com/s/Avu5SbLetCFBVUwnDybfsw
- https://mp.weixin.qq.com/s/WoruStfCdYNDskAn_iFYtQ
- https://www.nsfc.gov.cn/publish/portal0/tab1128/info90687.htm








