Command Palette
Search for a command to run...
Das Team Der Shanghai Jiao Tong University Hat Die Zentrale Protein-Engineering-Designplattform VenusFactory Veröffentlicht, Die Über 40 Gängige Modelle Und Datensätze abdeckt.

Mit der rasanten Entwicklung künstlicher Intelligenz und datengesteuerter Methoden nähert sich das Protein-Engineering der Phase des KI-gestützten Designs. Forscher benötigen mehr denn je umfassende, qualitativ hochwertige Protein-Datensätze, leistungsfähigere und einflussreichere künstliche Intelligenzmodelle für Proteine sowie effizientere und standardisiertere Analyseplattformen, um aus riesigen biologischen Datenmengen wertvolle Informationen präzise zu gewinnen, die Entwicklung und Optimierung neuer Proteine zu beschleunigen und innovative Durchbrüche in der Biomedizin, der synthetischen Biologie und anderen Bereichen zu fördern.
In diesem Zusammenhang möchten immer mehr Praktiker der Biowissenschaften KI verstehen und KI-Technologie zur Unterstützung der Entwicklung von Protein-Engineering einsetzen. Allerdings weisen sowohl die neu gestaltete Open-Source-Lösung von David Baker als auch die großen Modelle der ESM-Reihe von Meta viele Schwierigkeiten bei der Verwendung auf, wie etwa die komplexe Logik des KI-Computing-Frameworks, die große Menge an Code und die Notwendigkeit einer soliden Grundlage in der Computerprogrammierung. Mit anderen Worten: Für Bioforscher und sogar Computerpraktiker ohne Erfahrung ist die Hemmschwelle zur Nutzung dieser Technologien immer noch ziemlich hoch. In dieser Hinsicht haben sich benutzerfreundliche Low-Code-Anwendungen allmählich zum Mainstream-Trend bei der Verwendung moderner Open-Source-Tools entwickelt. Sie können Forschern dabei helfen, die komplexe Modellkonfiguration und Codeimplementierung loszuwerden, sodass Informatiker und Biologen Deep-Learning-Modelle bequemer aufrufen oder trainieren und sich auf die wissenschaftliche Forschung selbst konzentrieren können.
Um die Anwendung und Entwicklung künstlicher Intelligenz im Bereich des Protein-Engineerings zu fördern, hat die Forschungsgruppe von Professor Hong Liang an der Shanghai Jiao Tong University in China VenusFactory entwickelt, eine offene One-Stop-Plattform, die speziell auf das Protein-Engineering zugeschnitten ist. Forscher können mühsame Datenabrufe, Modelltrainings, Aufgabenauswertungen, Modellbereitstellungen und andere Funktionen problemlos über die Schnittstelleninteraktion oder die Befehlszeile implementieren. Durch ein codefreies und prozessbasiertes Design vereinfacht die Plattform die komplexen KI-Engineering-Operationen der Vergangenheit und macht sie zu leichtgewichtigen Operationen, die auf Knopfdruck erledigt werden können. Forscher können Webdienste lokal starten und problemlos über 40 hochmoderne Protein-Deep-Learning-Modelle aufrufen, ohne komplexe Codes schreiben zu müssen. Dadurch wird die Vertraulichkeit privater Daten geschützt, die Schwelle für intelligente wissenschaftliche Forschung deutlich gesenkt und die eingehende Anwendung von KI im Bereich der Biowissenschaften beschleunigt.
Code und Daten sind Open Source unter: https://github.com/ai4protein/VenusFactory
Derzeit wurde die „VenusFactory Protein Engineering Design Platform“ im Tutorial-Bereich der HyperAI-Website gestartet. Das ausführliche Tutorial zur Verwendung ist am Ende dieses Artikels angehängt. Interessierte Leser können die Plattform über den folgenden Link erleben:
VenusFactory: Eine einheitliche Plattform, die Barrieren für Protein-KI-Anwendungen abbaut
Proteindaten sind stark gestreut. VenusFactory greift direkt auf die Quelle biologischer Daten zu Die KI-Proteinforschung ist in hohem Maße von biologischen Daten im großen Maßstab abhängig und die annotierten Daten sind in mehreren gängigen öffentlichen Datenbanken verteilt. Wissenschaftler müssen häufig zwischen mehreren Datenbanken wechseln, Daten manuell herunterladen und Skripte zum Konvertieren des Formats schreiben, was zu einer Verschwendung von Zeit und Energie für nicht praktische Forschungsarbeiten führt. VenusFactory stellt eine direkte Verbindung zu gängigen öffentlichen Datenbanken wie RCSB PDB, UniProt, InterPro usw. her. Der mehrfädige Hochgeschwindigkeitsdownload verbessert die Effizienz des Datenabrufs erheblich:
- Zentraler Zugriff auf Proteinsequenzen, dreidimensionale Strukturen und funktionelle Anmerkungen mit vollständiger Integration biologischer Informationen.
- Durch die Ausgabe im standardisierten Format werden Datenkompatibilitätsprobleme vermieden und das direkte KI-Training erleichtert.
- Der Multithread-Download-Mechanismus verbessert die Geschwindigkeit der Datenerfassung erheblich und ermöglicht es den Wissenschaftlern, sich auf die Forschung selbst zu konzentrieren.
Das Bewertungssystem für Protein-KI-Aufgaben ist nicht einheitlich. VenusFactory deckt fünf Kernaufgaben ab. Derzeit fehlen dem Bewertungssystem für Protein-KI-Modelle vorgefertigte, maßgebliche Benchmarkdaten und der Großteil der Forschung konzentriert sich immer noch auf die Optimierung einzelner Aufgaben. Wenn Forscher eine Lösung auswählen, müssen sie häufig viel zusätzliche Zeit für experimentelle Vergleiche aufwenden. VenusFactory integriert mehr als 40 hochmoderne Datensätze zur Protein-Engineering-Evaluierung und deckt fünf Kernaufgaben ab:
- Vorhersage der Proteinfunktion: Sagen Sie die funktionellen Markierungen von Proteinen voraus, um neue Enzyme und neue Ziele zu entdecken.
- Vorhersage der subzellulären Proteinlokalisierung:Sagen Sie die Lokalisierung von Proteinen in Zellen voraus, um die Diagnose von Krankheiten zu unterstützen.
- Beurteilung der Proteinlöslichkeit:Verbessern Sie die Effizienz von Nassexperimenten durch Vorabbeurteilung der Löslichkeit.
- Analyse der Auswirkungen von Proteinmutationen: Erforschen Sie die möglichen Auswirkungen von Genmutationen und entwickeln Sie die Präzisionsmedizin weiter.
- Andere Vorhersageaufgaben: Wie Metallionenbindung, Vorhersage von Proteinsortierungssignalen, Vorhersage der optimalen Temperatur usw.
Mithilfe dieser Benchmark-Datensätze und Evaluierungsergebnisse können Benutzer die Leistung verschiedener Modelle einfach vergleichen und Lösungen auswählen und optimieren. Gleichzeitig bietet VenusFactory auch die Funktion zum Herunterladen aller Datensätze, sodass Benutzer mit einem Klick die entsprechende Proteinsequenz, Struktur, Beschriftung und andere Informationen erhalten können.
Die Nutzung bestehender Protein-KI-Rechenwerkzeuge ist mit hohen Hürden verbunden und für Forscher ohne Informatik-Hintergrund schwierig. Die Verwendung aktueller Protein-KI-Modelle erfordert häufig fundierte Programmierkenntnisse und Kenntnisse im Bereich Deep Learning. Für die meisten Biologen ist das Trainieren, Feinabstimmen und Anwenden von KI-Modellen immer noch eine hochschwellige Aufgabe. VenusFactory integriert mehr als 40 der weltweit modernsten Proteinsprachenmodelle (PLMs) und deckt umfassende KI-Großmodelllösungen ab, wie etwa die Venus-Reihe (ProSST, Pro-Prime, PETA usw.), die ESM-Reihe (ESM2, ESM1b usw.), die Ankh-Reihe (Base, Large) und die ProtTrans-Reihe (ProtBert, ProtT5) usw.
- Vortrainiertes Modell-Ökosystem: Rufen Sie Open Source PLM direkt auf, ohne von Grund auf geschult werden zu müssen, und sparen Sie so Rechenressourcen.
- Leistungsstarke Feinabstimmung: Unterstützt modernste Methoden wie LoRA und SES-Adapter, um das Modell an spezifische biologische Aufgaben anzupassen.
- Multitasking-Unterstützung: Ob es um die Vorhersage der Proteinlöslichkeit oder der Mutanteneigenschaften geht, Sie können problemlos loslegen.
- Befehlszeilenmodus: Geeignet für Informatiker, ermöglicht flexibles Anpassen von Parametern und eine tiefgreifende Optimierung.
- Codefreie Weboberfläche: Geeignet für Biologen, Sie können KI-Aufgaben mit einfachen Klicks ausführen, keine Programmierkenntnisse erforderlich!
Um diese zentralen Herausforderungen zu bewältigen, hat VenusFactory eine KI-gestützte Protein-Engineering-Plattform aus einer Hand entwickelt. Sie bietet eine Komplettlösung von der Datenerfassung über die Aufgabenauswertung bis hin zur Feinabstimmung des Modells und ermöglicht es Biologen und Computerwissenschaftlern, ihre Forschung effizient voranzutreiben.
Open Source und Community-Aufbau zur Förderung wissenschaftlicher Innovationen
Die Zukunft der wissenschaftlichen Forschung liegt im offenen Teilen. VenusFactory verwendet die Apache 2.0-Lizenz. Alle Codes, Datensätze und Modellgewichte sind vollständig Open Source. Benutzer können die neuesten Ergebnisse kostenlos herunterladen, ändern und optimieren und mit Forschern auf der ganzen Welt teilen. Alle Daten, Modelle und Feinabstimmungscodes werden auf GitHub und Hugging Face gehostet. Dadurch wird sichergestellt, dass Wissenschaftler auf der ganzen Welt problemlos auf Experimente zugreifen und diese reproduzieren sowie ihre eigenen KI-Forschungsprojekte auf Basis von VenusFactory erstellen können.
Um den Lesern das Kennenlernen von VenusFactory zu erleichtern, hat HyperAI ein Ein-Klick-Bereitstellungstutorial für die „VenusFactory Protein Engineering Design Platform“ veröffentlicht. Im Folgenden finden Sie eine detaillierte Einführung in die Verwendung.
Link zum Tutorial: https://go.hyper.ai/ZqO3h
VenusFactory Protein Engineering Design-Plattform-Tutorial
Demolauf
1. Melden Sie sich bei hyper.ai an, wählen Sie auf der Tutorial-Seite die VenusFactory Protein Engineering Design Platform aus und klicken Sie auf „Dieses Tutorial online ausführen“.
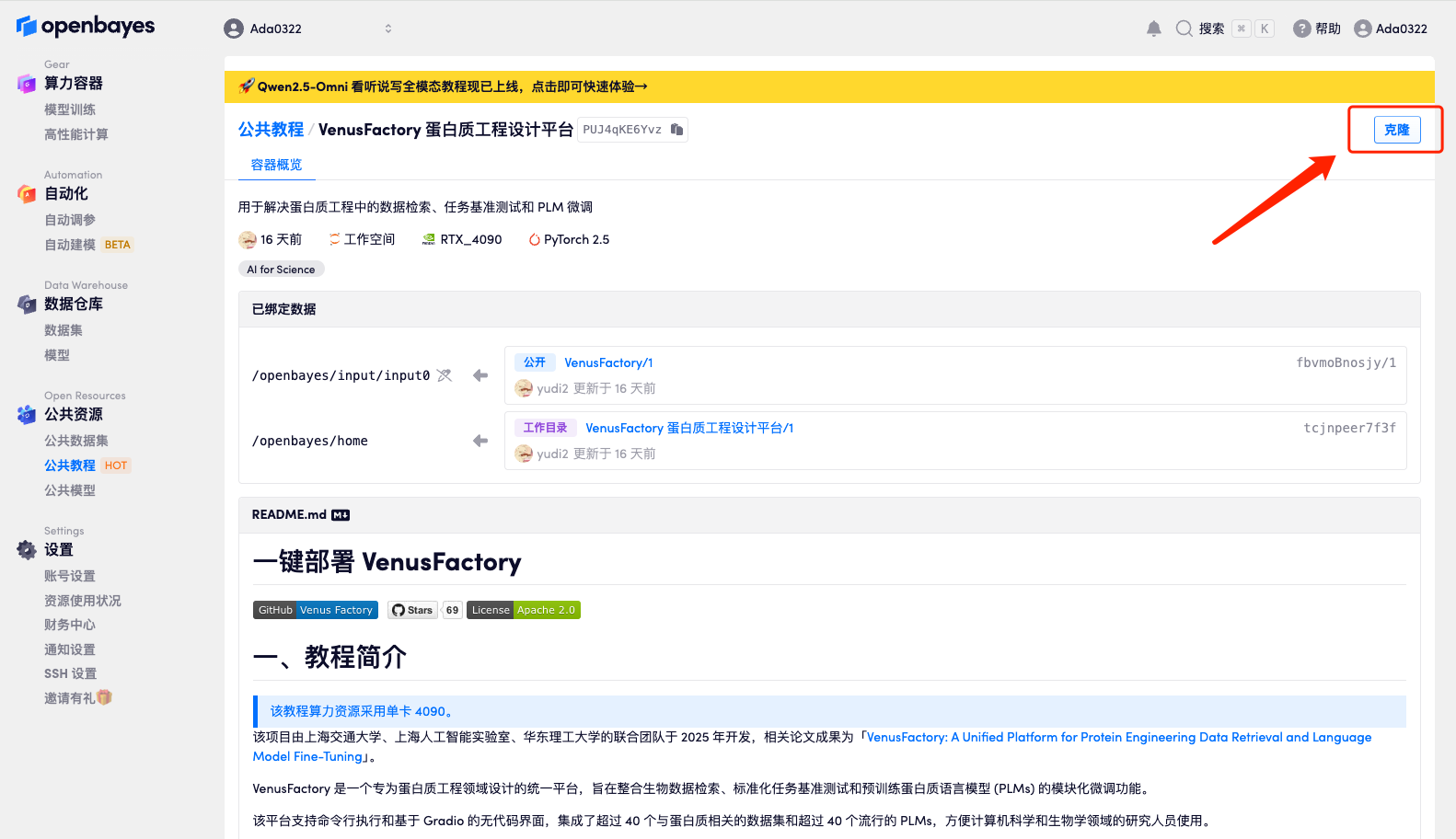
2. Klicken Sie nach dem Seitensprung oben rechts auf „Klonen“, um das Tutorial in Ihren eigenen Container zu klonen.
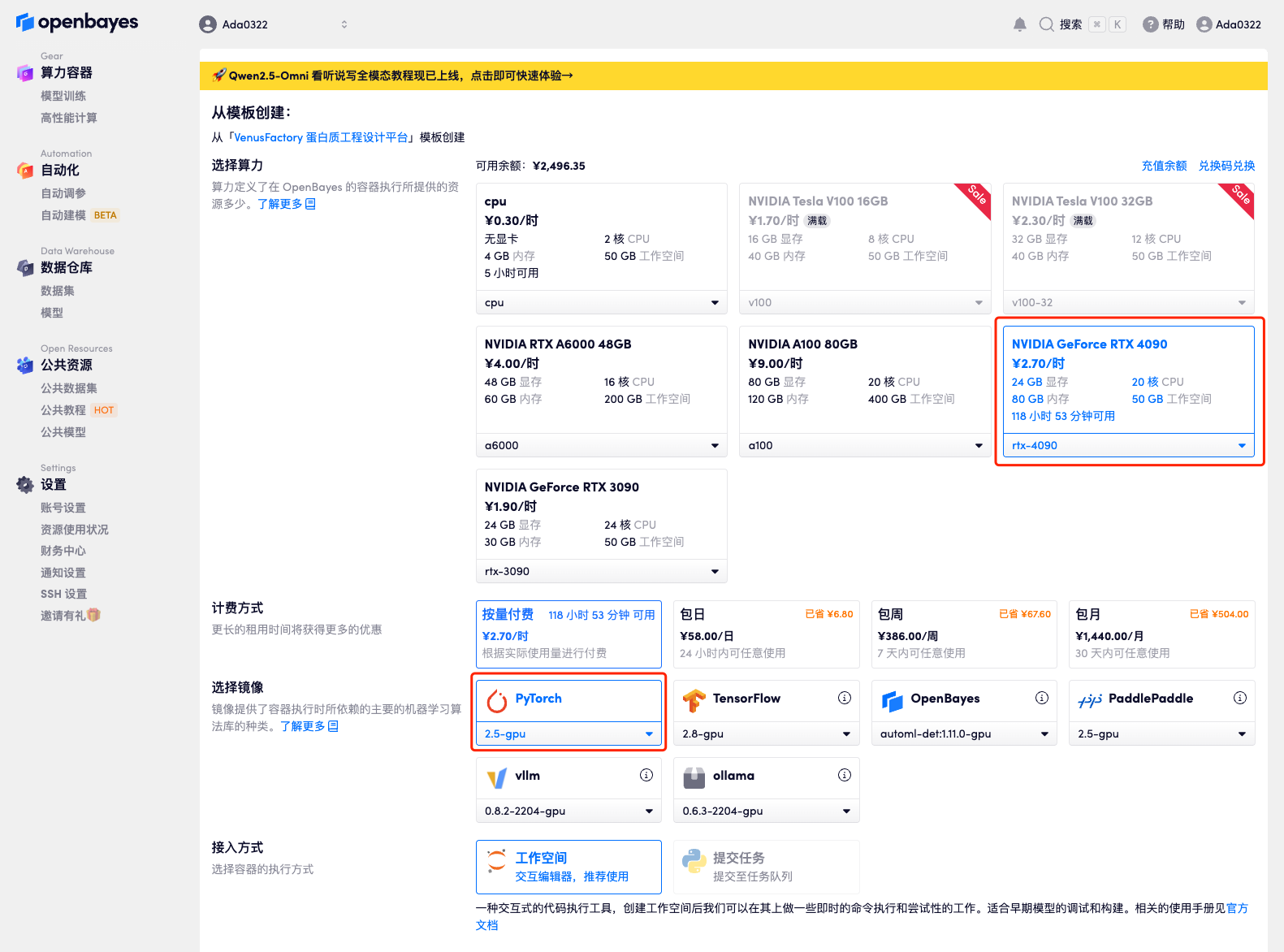
3. Wählen Sie NVIDIA GeForce RTX 4090 und PyTorch-Image aus und klicken Sie auf „Weiter“. Die OpenBayes-Plattform bietet vier Abrechnungsmethoden. Sie können je nach Bedarf zwischen „Pay as you go“ oder „täglich/wöchentlich/monatlich“ wählen. Neue Benutzer können sich über den unten stehenden Einladungslink registrieren, um 4 Stunden RTX 4090 + 5 Stunden CPU-freie Zeit zu erhalten!
Exklusiver Einladungslink von HyperAI (kopieren und im Browser öffnen):
https://openbayes.com/console/signup?r=Ada0322_NR0n
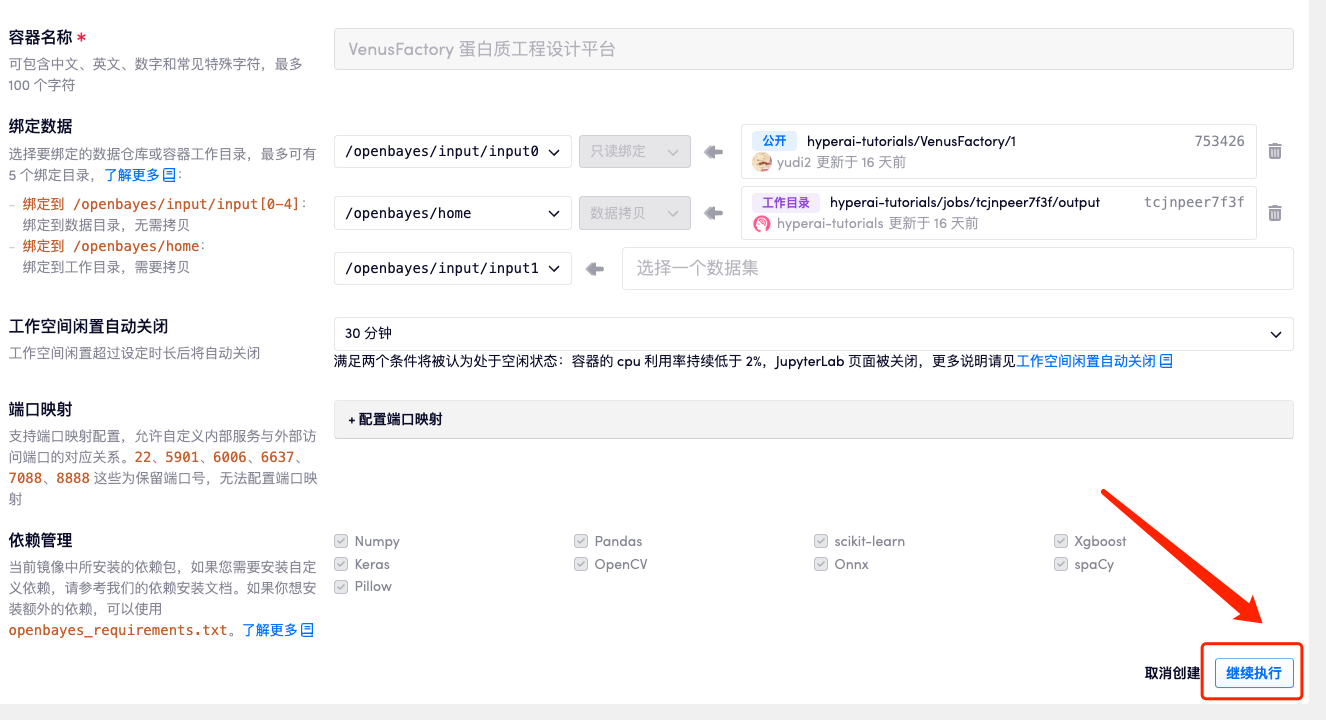
4. Warten Sie, bis die Ressourcen zugewiesen wurden. Der erste Klonvorgang dauert etwa 2 Minuten. Wenn sich der Status in „Läuft“ ändert, klicken Sie auf den Sprungpfeil neben „API-Adresse“, um zur Demoseite zu springen. Da das Modell groß ist, dauert es etwa 3 Minuten, bis die WebUI-Schnittstelle angezeigt wird, andernfalls wird „Bad Gateway“ angezeigt. Bitte beachten Sie, dass Benutzer vor der Verwendung der API-Adresszugriffsfunktion eine Echtnamenauthentifizierung durchführen müssen.
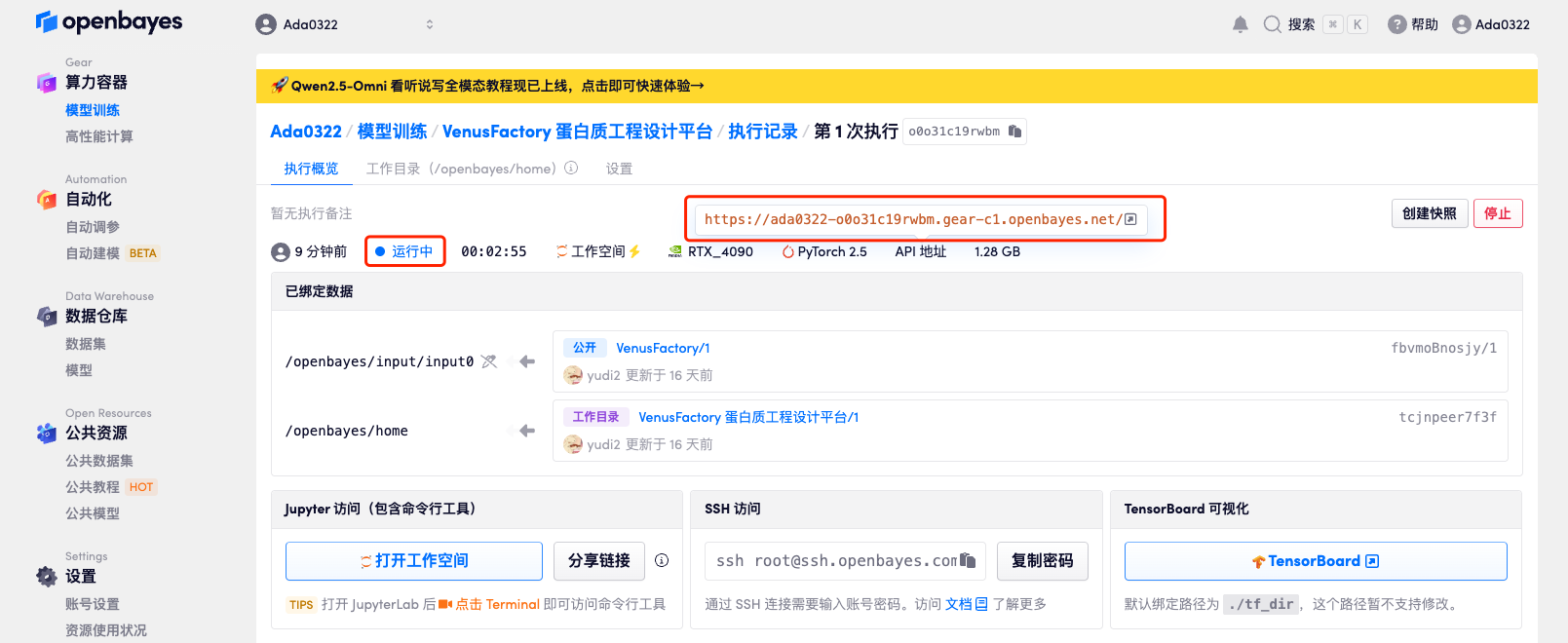
Effektanzeige
1. Dieses Tutorial umfasst vier Module: Training, Auswertung, Vorhersage und Download. Klicken Sie auf „Manuell“ und wählen Sie eine Sprache aus, um ausführliche Anweisungen für jedes Modul anzuzeigen.
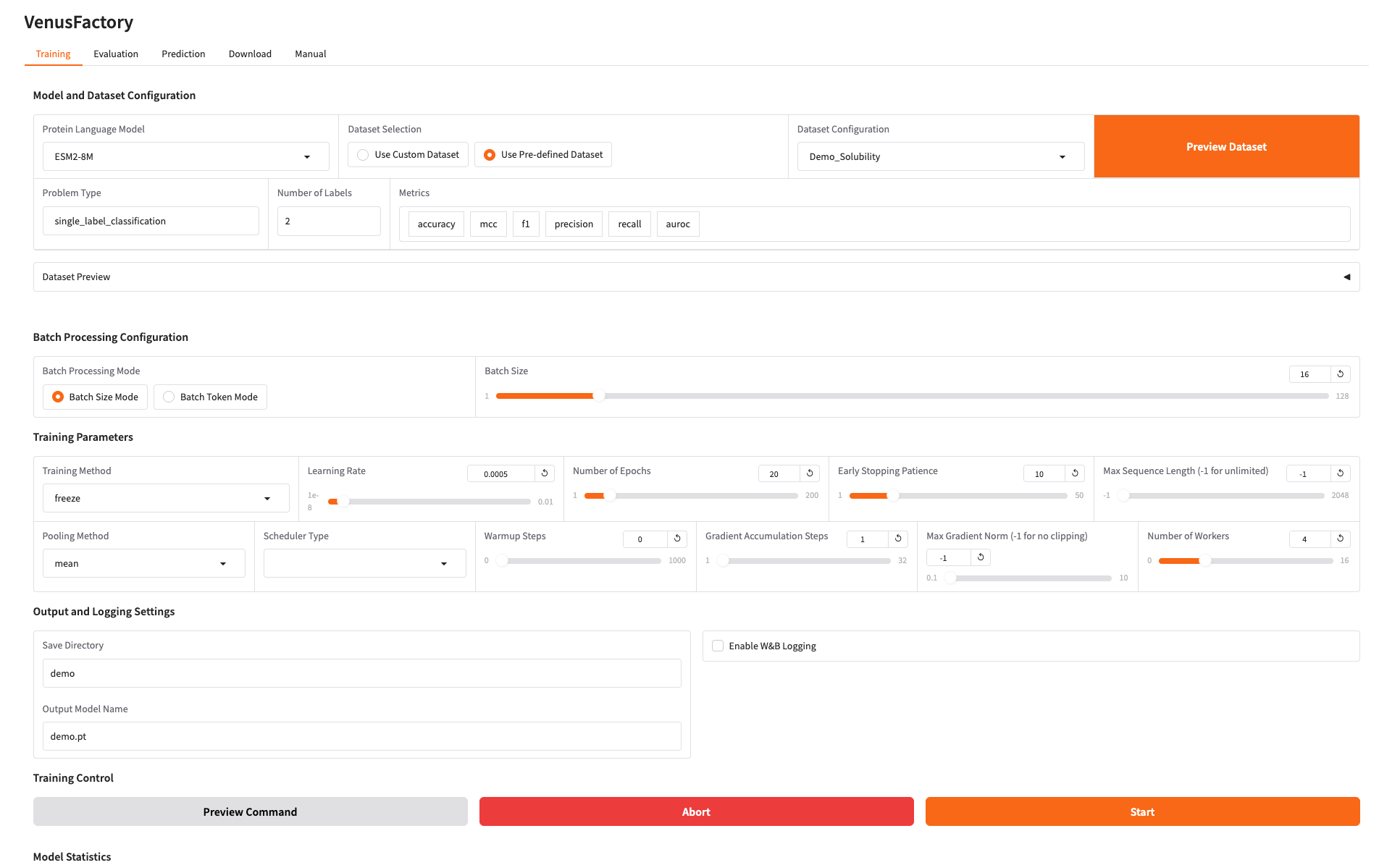
2. Trainingsmodul
Klicken Sie auf das Trainingsmodul, wählen Sie das Modell aus, das Sie im Protein Language Model trainieren möchten, und konfigurieren Sie die Trainingsdaten in der Dataset-Konfiguration
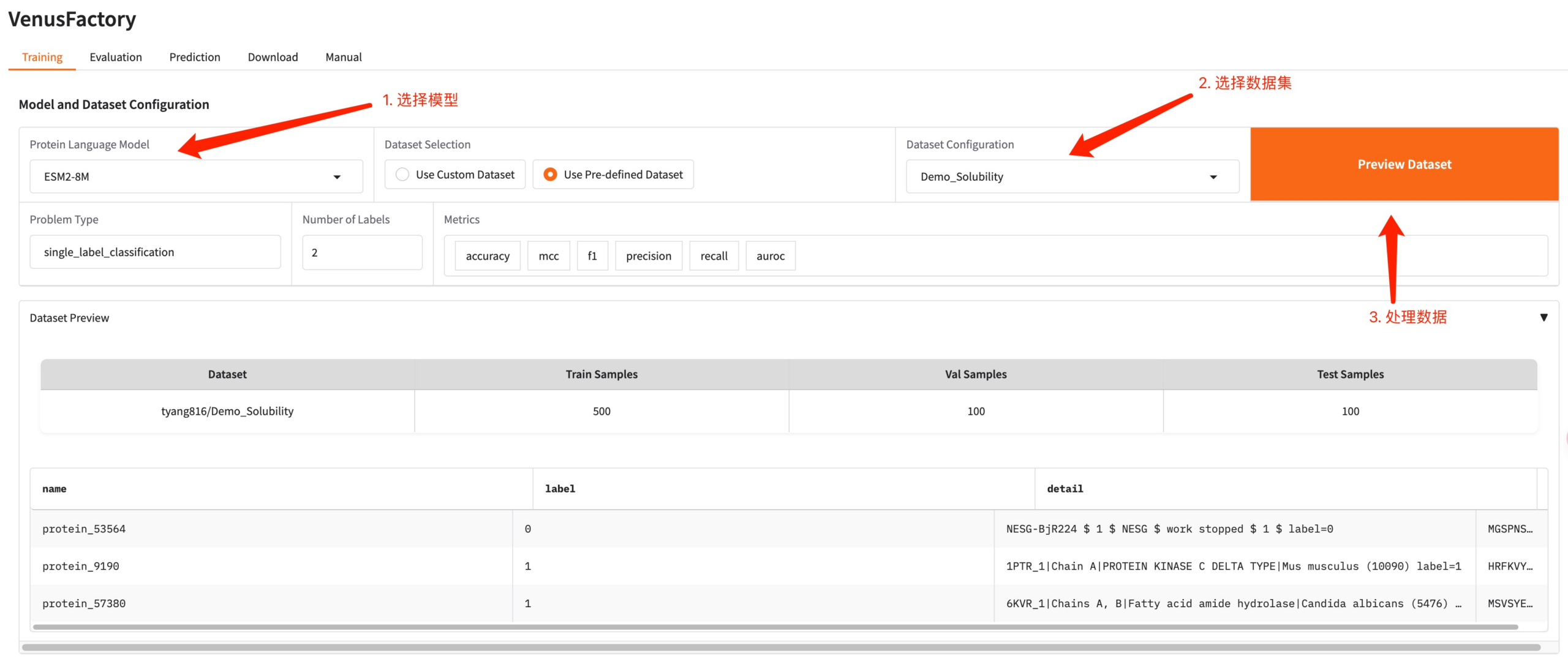
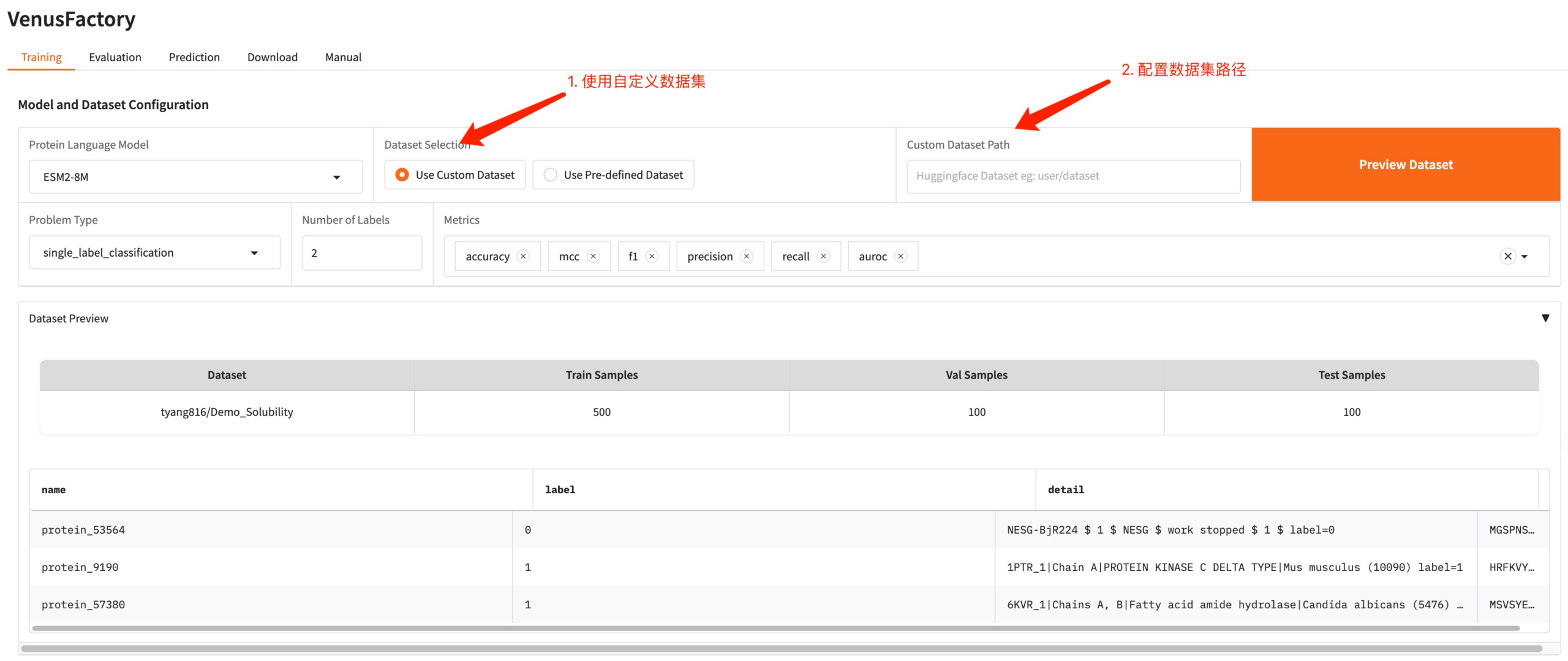
Wenn Sie Ihren eigenen Datensatz verwenden müssen, können Sie die Konfiguration „Benutzerdefinierten Datensatz verwenden“ verwenden und müssen nur den Datensatzpfad eingeben (weitere Informationen finden Sie in der Dokumentation zur manuellen Verwendung).
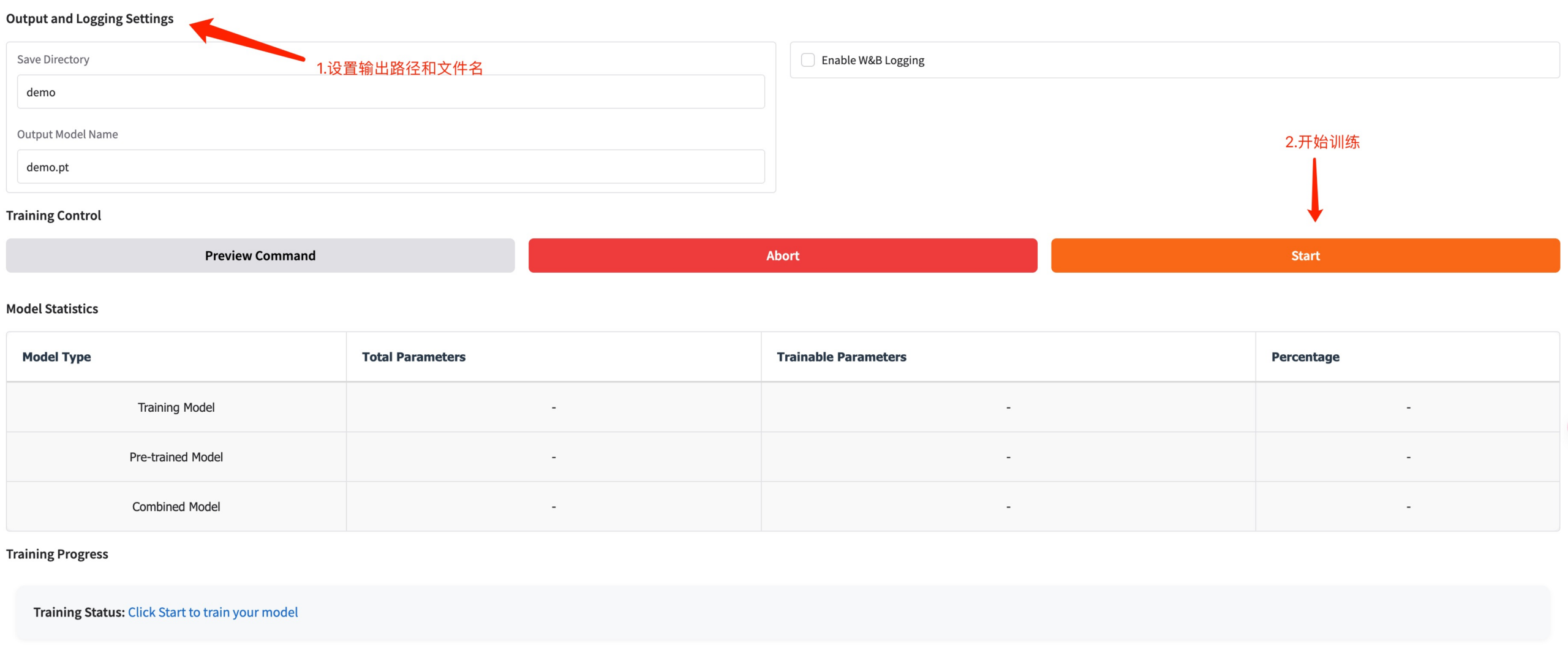
Legen Sie den Speicherpfad für das Trainingsmodell fest und klicken Sie auf „Start“, um mit dem Training zu beginnen.
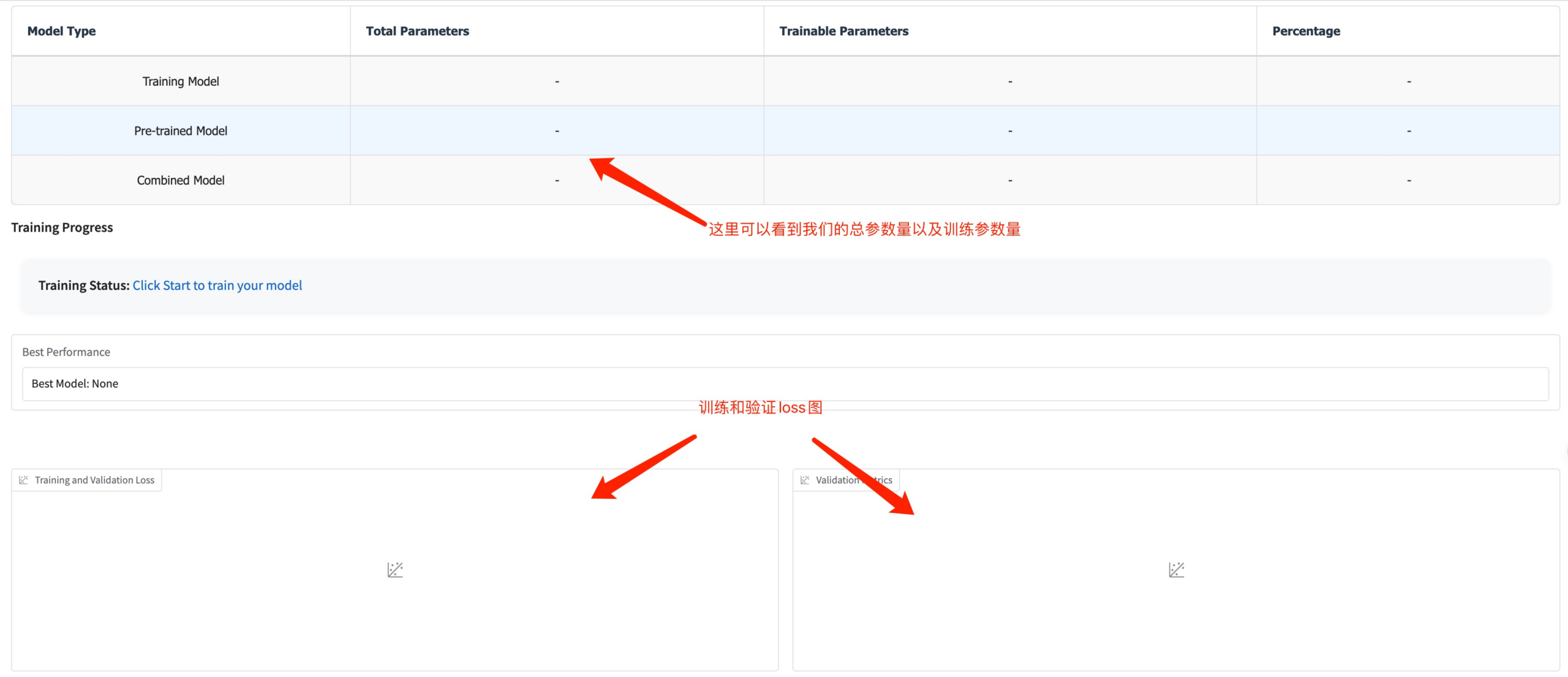
An dieser Stelle können Sie die Trainingsparameter und die Verlustkurve sehen
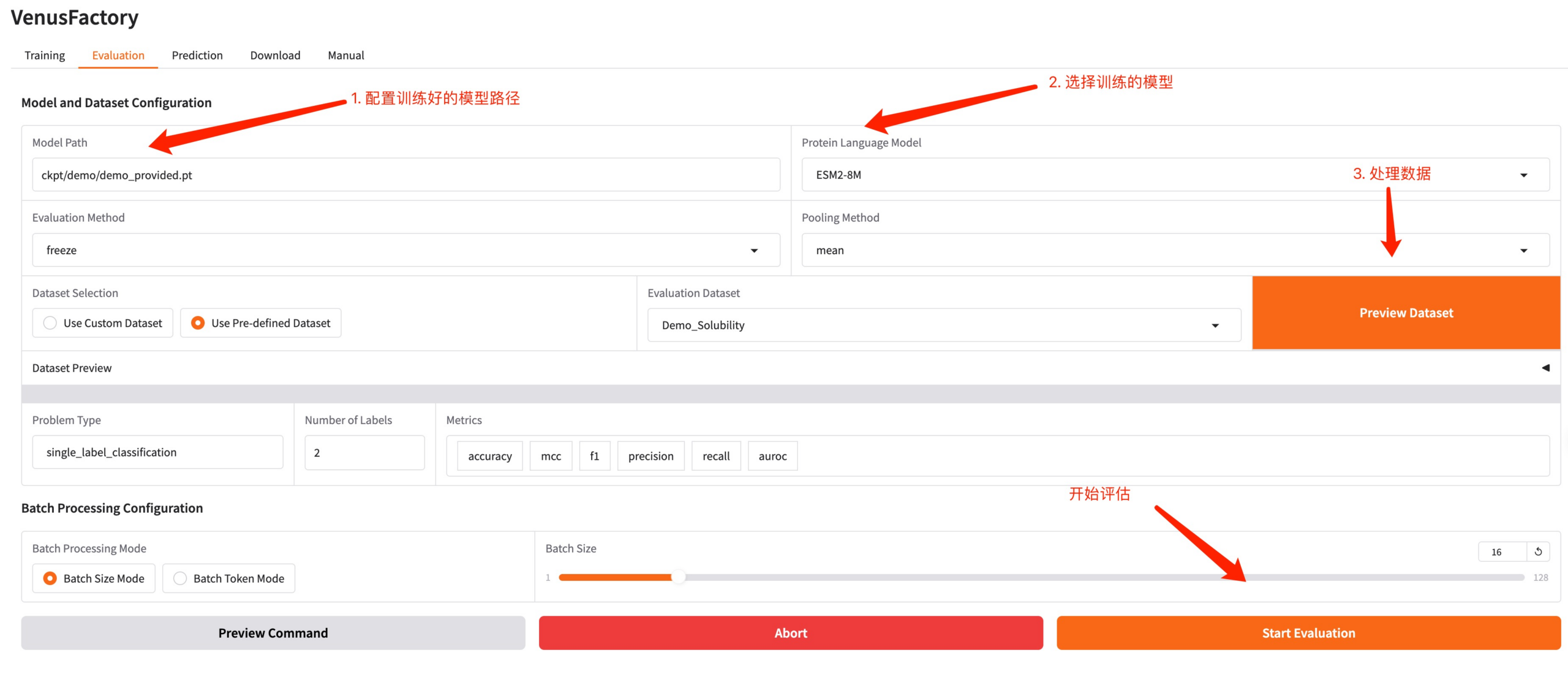
3. Evaluierungsmodul
Klicken Sie auf das Modul „Auswertung“, konfigurieren Sie den durch das Training generierten Modellpfad und das trainierte Modell, verarbeiten Sie die Daten, passen Sie die Hyperparameter an und starten Sie die Auswertung.
4. Vorhersagemodul
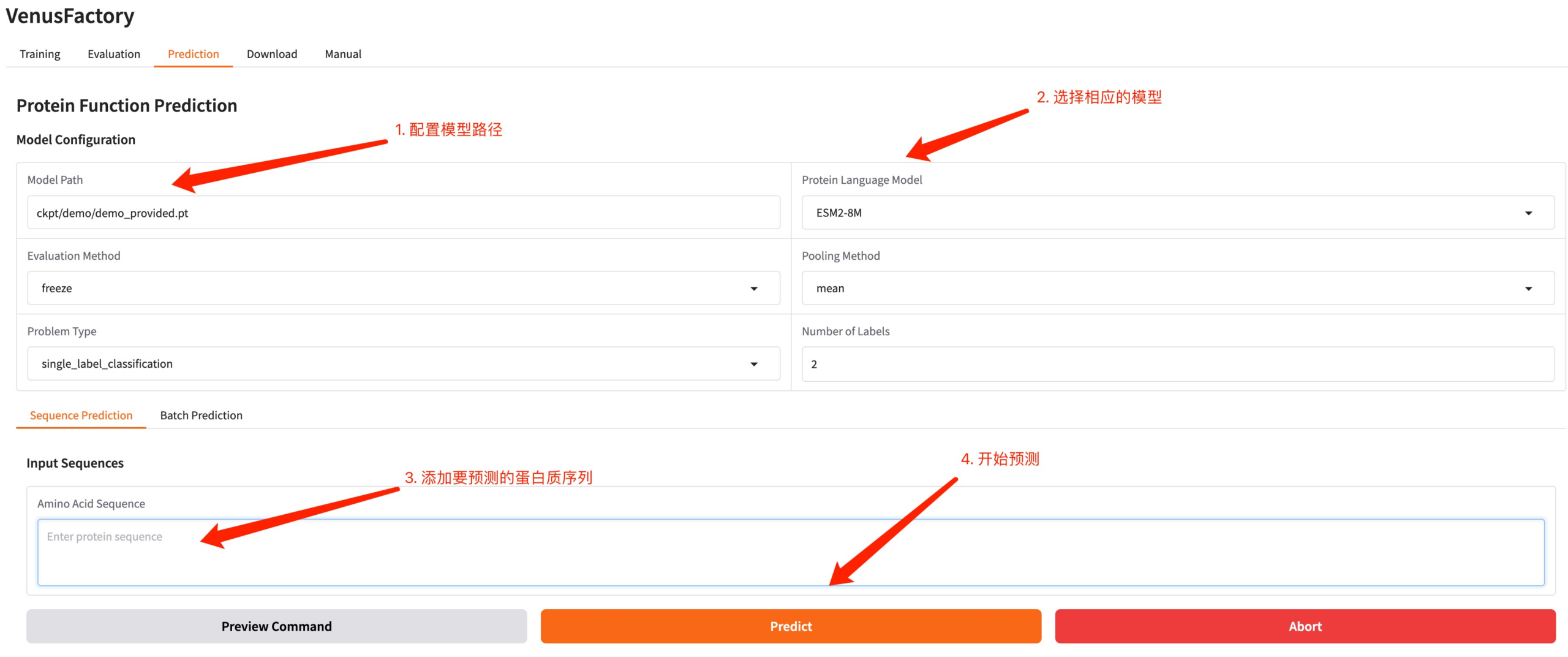
Klicken Sie auf das Modul „Vorhersagen“, konfigurieren Sie den durch das Training und das trainierte Modell generierten Modellpfad, geben Sie die Protein-Sequenz ein, die Sie vorhersagen möchten, und klicken Sie auf „Vorhersagen“, um eine Vorhersage zu treffen.
Beispiel einer Proteinsequenz: MKTWFGHVLQ
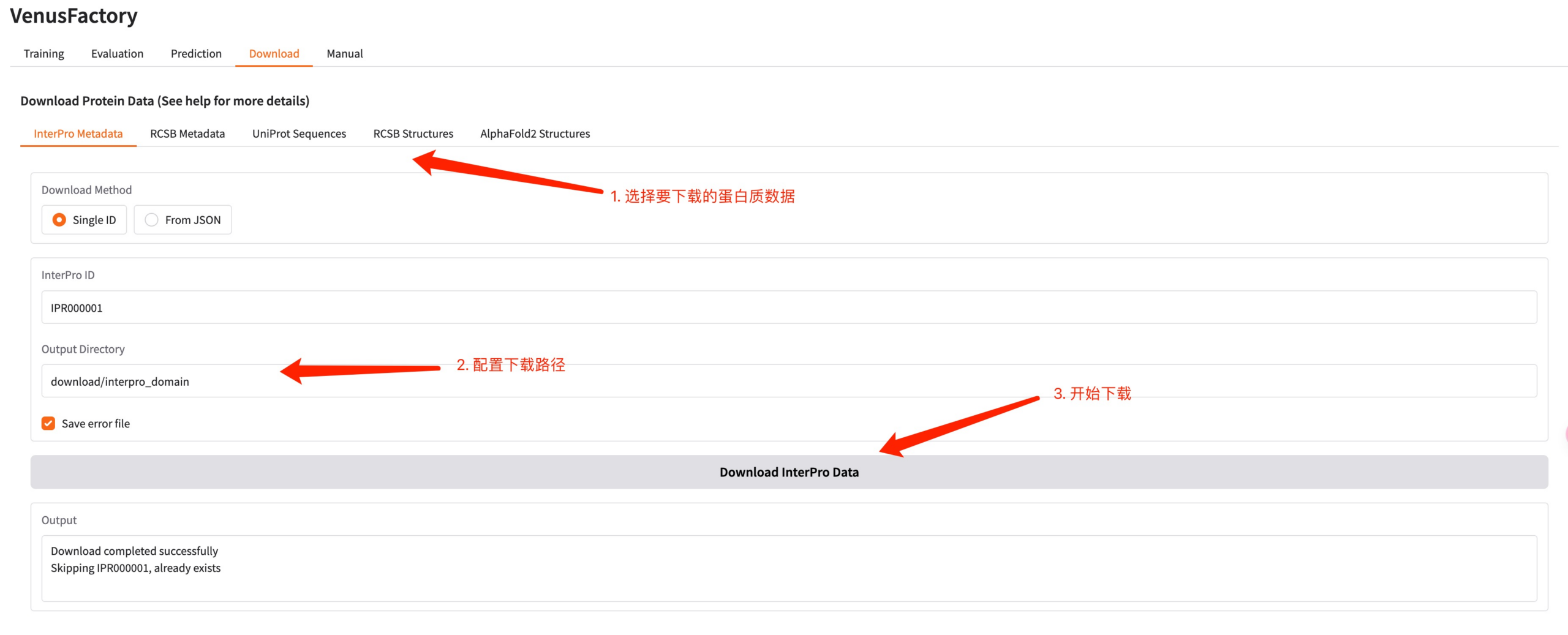
5. Modul herunterladen
Klicken Sie auf das Download-Modul, um Proteindaten in dieser Schnittstelle herunterzuladen.
Oben finden Sie ein ausführliches Tutorial zur Verwendung der „VenusFactory Protein Engineering Design Platform“. Jeder ist herzlich eingeladen, vorbeizukommen und es zu erleben!








