Command Palette
Search for a command to run...
Die Genauigkeit Wurde Um 5.2% verbessert. NVIDIA Und Andere Haben Ein Multimodales Medizinisches Bildsegmentierungsmodell Veröffentlicht, Um Eine Automatische Segmentierung Und Interaktion Von 3D-Bildern Zu erreichen.
Seit der Einführung des ersten klinischen CT-Scanners im Jahr 1971 hat die medizinische Bildgebung einen revolutionären Sprung von zweidimensionalen Schnitten zu dreidimensionalem Stereo vollzogen. Moderne Spiral-CTs mit 256 Reihen können Ganzkörper-Scandaten mit einer Schichtdicke von 0,16 mm in 0,28 Sekunden erfassen und mit der 7T-Ultrahochfeld-Magnetresonanztomographie lässt sich sogar die mikroskopische Richtung der Nervenfasern im Hippocampus erfassen. Wenn Ärzten jedoch diese dreidimensionalen Matrizen mit zig Millionen Voxeln vorgelegt werden, ist die genaue Segmentierung von Organen, Läsionen und Gefäßnetzen noch immer in hohem Maße von der manuellen, schichtweisen Darstellung abhängig. Studien haben gezeigt, dass die Lebersegmentierung für einen typischen Satz abdominaler CT-Bilder 45 bis 90 Minuten dauert, während die Annotation einer Strahlentherapieplanung mit Multiorganverknüpfung mehr als 8 Stunden dauern kann.Die durch die visuelle Ermüdung von Fachkräften verursachte Grenzfehlerrate kann 12% erreichen.
Dieses Dilemma hat zu einer äußerst aktiven Innovationsbewegung im Bereich der medizinischen Bildanalyse geführt. Vom frühen, auf Graustufenschwellenwerten basierenden Region-Growing-Algorithmus über die dreidimensionale U-Net-Variante V-Net, die Deep Learning integriert, bis hin zur TransUNet-Hybridarchitektur, die den visuellen Transformer einführt, haben Algorithmenentwickler ständig versucht, intelligente Navigationssysteme im Pixellabyrinth zu bauen. Die jüngsten Durchbrüche auf der MICCAI-Konferenz 2024 zeigten, dass einige Modelle eine gruppenübergreifende Konsistenz erreicht haben, die mit der von erfahrenen Radiologen bei Prostatasegmentierungsaufgaben vergleichbar ist, ihre Leistung in Fällen seltener anatomischer Variationen jedoch immer noch stark schwankt. Daraus ergibt sich ein tieferer technologisch-philosophischer Ansatz: Wie viel Vorwissen ist erforderlich, wenn KI versucht, den menschlichen Körper zu verstehen, und wie viele anatomische Erkenntnisse, die über die menschliche Wahrnehmung hinausgehen, kann sie generieren?
Kürzlich veröffentlichte ein interdisziplinäres Team bestehend aus NVIDIA, der University of Arkansas for Medical Sciences, den National Institutes of Health und der University of Oxford ein bahnbrechendes Forschungsergebnis: das multimodale medizinische Bildsegmentierungsmodell VISTA3D.Dieses Modell war der erste Schritt zur Merkmalsextraktion mit 3D-Supervoxel.Durch eine einheitliche Architektur wird die kollaborative Optimierung der automatischen 3D-Segmentierung (die 127 anatomische Strukturen abdeckt) und der interaktiven Segmentierung realisiert. In einem umfassenden Benchmarktest mit 14 Datensätzen erreicht es die fortschrittlichste automatische 3D-Segmentierung und interaktive Bearbeitung und verbessert die Zero-Sample-Leistung um 50%.
Die entsprechenden Forschungsergebnisse tragen den Titel „VISTA3D: A Unified Seamentation Foundation Model For 3D Medical Imaging“ und wurden als Preprint auf arXiv veröffentlicht.

Papieradresse:
https://doi.org/10.48550/arxiv.2406.05285
Paradigmenwechsel und Herausforderungen in der 3D-medizinischen Bildgebungstechnologie
In der digitalen Welle der medizinischen Bildanalyse erfährt die 3D-Automatiksegmentierungstechnologie einen Paradigmenwechsel vom „Spezialisten“ zum „Allgemeinmediziner“. Herkömmliche Methoden bauen dedizierte Netzwerkarchitekturen und maßgeschneiderte Trainingsstrategien auf, um unabhängige Expertenmodelle für jede anatomische Struktur oder jeden Pathologietyp zu erstellen. Obwohl dieses Modell bei bestimmten Aufgaben gute Ergebnisse liefert, ist es so, als würde man von Radiologen verlangen, wiederholt an einer Schulung zur Diagnose einzelner Organe teilzunehmen.Bei einem Ganzkörper-CT-Scan mit 127 anatomischen Strukturen muss das System Dutzende von Modellen parallel ausführen, und der Verbrauch der Rechenressourcen sowie die Komplexität der Ergebnisintegration steigen exponentiell.
Was den Ärzten in der klinischen Praxis noch mehr Sorgen bereitet, sind oft jene seltenen Fälle, die den üblichen anatomischen Atlas sprengen: Dabei kann es sich um neu entdeckte Verkalkungsherde im Nanomaßstab in der Leber von Versuchsmäusen handeln oder um ungewöhnliche Blutgefäßformen, die sich aufgrund anatomischer Abweichungen bei Transplantationspatienten gebildet haben. Diese Szenarien offenbaren grundlegende Mängel im bestehenden System:Durch übermäßiges Vertrauen auf voreingestellte Kategorien und geschlossenes Training wird es für das Modell schwierig, Nullstichproben zu lernen und sich an offene Domänen anzupassen.
Der Beginn eines Durchbruchs bei diesem Dilemma kommt aus dem Bereich der natürlichen Bildverarbeitung. Als große Sprachmodelle erstaunliche Generalisierungsfähigkeiten für verschiedene Aufgaben zeigten, begann man in der medizinischen Bildgebungsbranche, sich mit der Entwicklung intelligenter „Konversations“-Systeme zu befassen. Das von Meta vorgeschlagene SAM (Segment Anything Model) realisiert die revolutionäre Interaktion von „Click to Segment“ in zweidimensionalen Bildern und übertrifft mit seiner Zero-Sample-Leistung sogar einige professionelle Modelle. Bei der Übertragung dieses Paradigmas auf den Bereich der dreidimensionalen Medizin stößt die einfache Dimensionserweiterung jedoch auf grundlegende Herausforderungen: Die topologische Komplexität menschlicher Organe in kontinuierlichen Tomographie-Scans ist bei weitem nicht mit der eines fahrenden Fahrzeugs in einem Video vergleichbar.
Am Beispiel der Lebersegmentierung können zwischen benachbarten Schnitten gleichzeitig Pfortaderbifurkation, Tumorinfiltration und Metallartefakte von chirurgischen Klammern vorhanden sein. Dies erfordert, dass das Modell über echte dreidimensionale räumliche Denkfähigkeiten verfügt und nicht nur über eine einfache Zeitreihenverfolgung. Zuvor hatten Forscher versucht, die SAM-Architektur dreidimensional zu gestalten und die Systeme SAM2 und SAM3D entwickelt. Obwohl Fortschritte bei Aufgaben wie der Gefäßverfolgung erzielt wurden,Allerdings ist sein Dice-Koeffizient immer noch 9–15 Prozentpunkte niedriger als der des professionellen Modells.Insbesondere bei überlappenden Bereichen mehrerer Organe steigt die Fehlerquote dramatisch an.
Der tiefere Widerspruch liegt in der einzigartigen wissensabhängigen Natur medizinischer Daten. Wenn die natürliche Bildsegmentierung auf statistischen Merkmalen auf Pixelebene basieren kann,Die medizinische Bildanalyse muss anatomisches Vorwissen integrieren.Beispielsweise erfordert die Segmentierung der Bauchspeicheldrüse nicht nur die Identifizierung von Graustufenmerkmalen, sondern auch das Verständnis ihrer anatomischen Nähe zum Zwölffingerdarm. Dies hat zu einem neuen Paradigma des kontextbasierten Lernens geführt: Durch die Eingabe von Beispielbildern oder Textbeschreibungen wird das Modell angeleitet, sich an neue Kategorien anzupassen.
Allerdings sind die Probleme, die bei den Tests bestehender Systeme auftauchen, ziemlich ironisch: Die Forderung an Kliniker, qualitativ hochwertige Beispielanmerkungen bereitzustellen, widerspricht der ursprünglichen Absicht der automatisierten Segmentierung. und eine textgesteuerte semantische Ausrichtungsverzerrung kann dazu führen, dass ein Hilus-Cholangiokarzinom fälschlicherweise als normale Gefäßstruktur identifiziert wird. Das Paradoxon dieses technischen Weges spiegelt den grundlegenden Ansatz bei der Entwicklung medizinischer KI wider:Die Herstellung eines dynamischen Gleichgewichts zwischen der Anpassung an offene Domänen und der klinischen Sicherheit ist möglicherweise praktischer als die bloße Verfolgung der Algorithmusleistung.
VISTA3D: Ein einheitliches Segmentierungsbasismodell für die medizinische 3D-Bildgebung
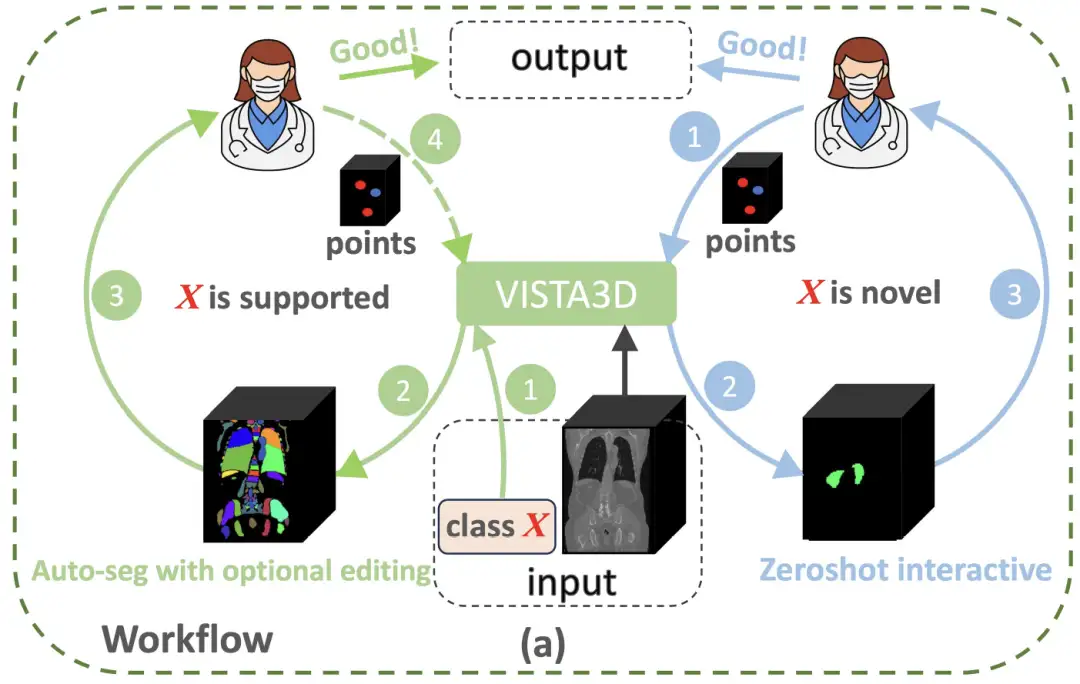
Um die Paradigmenbeschränkungen der medizinischen 3D-Bildanalyse zu durchbrechen,Das Forschungsteam von NVIDIA hat eine innovative Architektur entwickelt, die die Vorteile des zweidimensionalen Vortrainings mit dreidimensionalen anatomischen Merkmalen kombiniert – das VISTA3D-Modell.Wie in der folgenden Abbildung gezeigt, führt VISTA3D eine automatische Segmentierung (Auto-Seg) mit hoher Genauigkeit durch, wenn die Segmentierungsaufgabe X zu den 127 unterstützten Kategorien gehört (grüne Kreise links). Ärzte können die Ergebnisse bei Bedarf mit VISTA3D überprüfen und effizient bearbeiten. Wenn X eine neue Klasse ist (blauer Kreis rechts), führt VISTA3D eine interaktive 3D-Zero-Shot-Segmentierung durch.
Speziell,Die VISTA3D-Modellarchitektur verwendet ein modulares Designkonzept und erstellt einen 3D-Segmentierungskern auf Basis von SegResNet, das im Bereich der medizinischen Bildgebung umfassend erprobt ist.Diese U-förmige Netzwerkarchitektur hat bei internationalen, maßgeblichen Segmentierungsherausforderungen wie BraTS 2023 eine hervorragende Leistung bewiesen. Wie in der folgenden Abbildung gezeigt, aktiviert der Auto-Branch oben die automatische Aufteilungsfunktion sofort, wenn der Benutzer eine Klassenaufforderung bereitstellt, die zu den 127 unterstützten Kategorien gehört. Wenn der Benutzer 3D-Punktaufforderungen bereitstellt, aktiviert der interaktive Zweig unten die interaktive Segmentierungsfunktion. Wenn beide Zweige aktiviert sind, verwendet das algorithmusbasierte Zusammenführungsmodul die interaktiven Ergebnisse, um die automatischen Ergebnisse zu bearbeiten.
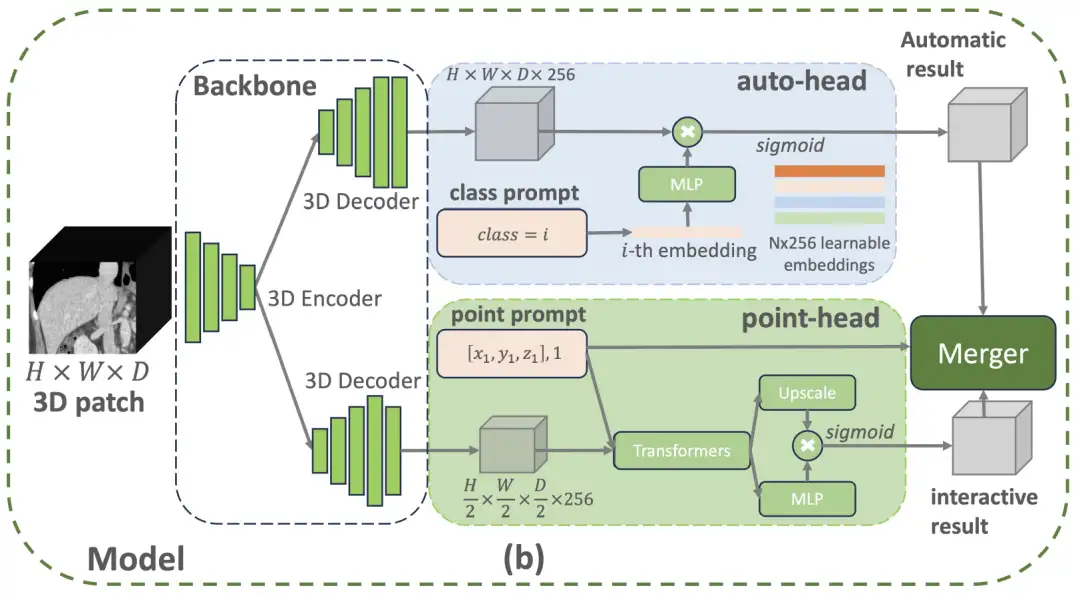
Unter ihnen verwendet der automatische Zweig intelligente Codierungstechnologie, um 127 menschliche Körperstrukturen zu verwalten. Wenn ein bestimmtes Teil lokalisiert werden muss, gleicht das System die Merkmalsinformationen im gescannten Bild genau ab und generiert durch intelligente Konvertierung das Segmentierungsergebnis.Dieses Design spart 60%-Speicherressourcen im Vergleich zu herkömmlichen Methoden und kann auch Lernverzerrungen durch unvollständige Anmerkungen vermeiden.Das manuelle Korrekturmodul verwendet eine dreidimensionale Klickpositionierungstechnologie: Stellen Sie zuerst die Bilddetails wieder her und optimieren Sie dann die Verarbeitungsgeschwindigkeit. Die vom Arzt angeklickte Stelle wird in Raumkoordinaten umgewandelt und intelligent mit den Scanfunktionen verknüpft. Bei leicht zu verwechselnden Strukturen wie der Bauchspeicheldrüse und Tumoren fügt das System automatisch Unterscheidungsmerkmale hinzu.
Durch intelligentes Zusammenspiel der beiden Module wird eine präzise Anpassung erreicht. Der Korrekturvorgang wirkt sich nur auf den lokalen Bereich aus, der mit der Klickposition verbunden ist, genau wie bei der Verwendung eines Präzisionsskalpells zum Ändern eines bestimmten Teils, ohne das Gesamtergebnis der Segmentierung zu zerstören.Diese dreidimensionale Optimierungslösung verbessert die Korrektureffizienz der Ärzte um 40%.Während der Modelltrainingsphase integrierte das Forschungsteam außerdem 11.454 CT-Scan-Datensätze, übernahm einen Mechanismus zur Pseudolabel-Generierung im Rahmen eines halbüberwachten Lernrahmens und kombinierte ihn mit einer vierstufigen progressiven Trainingsstrategie. Sie führten zunächst ein Vortraining mit einem gemischten Datensatz (einschließlich Pseudo-Labels und Super-Voxel-Annotationen) durch, optimierten dann die automatische Segmentierung bzw. die interaktiven Korrekturaufgaben und erreichten schließlich durch gemeinsames Training eine funktionale Integration. Letztlich erreicht das VISTA3D-Modell durch zentrale Innovationen mehrere Technologiesprünge.
Zunächst wurde das Modell systematisch anhand von 14 internationalen öffentlichen Datensätzen validiert, die 127 Arten anatomischer Strukturen und pathologischer Merkmale abdecken.Seine 3D-Automatiksegmentierungsgenauigkeit (Dice-Koeffizient 0,91±0,05) ist 8,3% höher als beim herkömmlichen Basismodell.Es unterstützt außerdem eine klickbasierte interaktive Korrektur, wodurch der Zeitaufwand für die manuelle Korrektur auf ein Drittel der herkömmlichen Methode reduziert wird. Zweitens erreicht die erste 3D-Supervoxel-Feature-Transfer-Technologie durch die Entkopplung der räumlichen Features des vortrainierten 2D-Backbone-Netzwerks eine 50% mIoU-Verbesserung bei Zero-Shot-Aufgaben wie der Pankreassegmentierung.Die Kennzeichnungseffizienz ist 2,7-mal höher als beim überwachten Lernen.Darüber hinaus erstellte das Forschungsteam einen einrichtungsübergreifenden multimodalen Datensatz.Während die Annotationsgenauigkeit von 97,21 TP3T beibehalten wird, werden die Datenannotationskosten auf 151 TP3T vollständig manueller Annotation komprimiert.
Forschungsfortschritte zur Integration von 3D-medizinischer Bildgebung und KI in China
In den letzten Jahren hat sich die Kombination aus dreidimensionaler medizinischer Bildgebungstechnologie und künstlicher Intelligenz mit der weitverbreiteten Anwendung der KI-Technologie im medizinischen Bereich allmählich zu einem Forschungsschwerpunkt entwickelt und in China erhebliche Fortschritte erzielt, was neue Möglichkeiten für die medizinische Diagnose und Behandlung mit sich bringt.
Im Jahr 2023 wird sich die Anwendung von KI in der medizinischen Bildgebung hauptsächlich auf die unterstützende Diagnose konzentrieren. KI kann schnell riesige Datensätze mit Bildern und Patienteninformationen durchsuchen, um die Diagnoseeffizienz zu verbessern. Beispielsweise können einige KI-integrierte Bildgebungssysteme winzige Anomalien erkennen, die mit bloßem Auge nur schwer zu erkennen sind, und so die Genauigkeit der Diagnose verbessern. Darüber hinaus kann KI frühere Bildgebungsscans aus der elektronischen Krankenakte eines Patienten abrufen und mit dem neuesten Scan vergleichen, wodurch Ärzte umfassendere Diagnoseinformationen erhalten. Zum Beispiel,Die Shanghai Jiao Tong University hat ein neues Arbeitsmodell PnPNet für die 3D-Segmentierung medizinischer Bilder vorgeschlagen.Das Problem der Verwechslung von Klassengrenzen wird durch die Modellierung der Interaktionsdynamik zwischen den sich überschneidenden Grenzbereichen und ihren angrenzenden Bereichen gelöst. Die Leistung ist SOTA und übertrifft Netzwerke wie MedNeXt, Swin UNETR und nnUNet.
* Papieradresse:
https://arxiv.org/abs/2312.08323
Im Jahr 2024 wird die Integration von 3D-medizinischer Bildgebungstechnologie und KI enger werden und die Forschungsrichtungen werden vielfältiger. Einerseits ist die Anwendung der KI-Technologie bei der dreidimensionalen Rekonstruktion medizinischer Bilder allmählich ausgereift und kann automatisch eine dreidimensionale Bildsegmentierung und -rekonstruktion durchführen, wodurch die Genauigkeit und Effizienz der Bildrekonstruktion verbessert wird. Andererseits wurden auch die Fähigkeiten der KI in der Bildanalyse weiter verbessert, was Ärzte bei der Diagnose von Krankheiten und der Ausarbeitung von Behandlungsplänen unterstützen kann. Darüber hinaus wird KI-Technologie auch bei der Bildnachbearbeitung, beispielsweise bei der Rauschunterdrückung, Bildverbesserung und beim Rendern, eingesetzt, um die Lesbarkeit und Ästhetik der Bilder zu verbessern. Zum Beispiel,Das West China Hospital der Sichuan University hat auf Grundlage der Lungenkrebs-Screening-Kohorte und der klinischen Lungenknoten-Kohorte der chinesischen Bevölkerung das datengesteuerte Chinese Lung Nodule Reporting and Data System (C-Lung-RADS) innovativ entwickelt.Es ist gelungen, das Malignitätsrisiko von Lungenknötchen präzise einzustufen und individuell zu behandeln.
* Papieradresse:
https://www.nature.com/articles/s41591-024-03211-3
Bis 2025 wird die Anwendung der KI-Technologie in der dreidimensionalen medizinischen Bildgebung umfangreicher und tiefer gehend sein. Zum Beispiel,Ein Forschungsteam der Peking-Universität hat kürzlich international ein „Renal Imaging Group Project“ gestartet.Es ist geplant, durch multimodale Bildgebungstechnologie und Algorithmen der künstlichen Intelligenz eine führende Rolle bei der Erstellung einer digitalen Karte der gesamten Niere zu übernehmen. Diese „digitale Niere“ kann den Mechanismus von Nierenerkrankungen deutlicher sichtbar machen und eine neue Richtung für die genaue Diagnose, die Entwicklung neuer Medikamente und die präzise Behandlung von Nierenerkrankungen aufzeigen.
gleichzeitig,Ein Team der China University of Geosciences und Baidu hat gemeinsam ein allgemeines Framework namens ConDSeg für die kontrastgesteuerte medizinische Bildsegmentierung vorgeschlagen.Dieses Framework führt auf innovative Weise eine Trainingsstrategie zur Konsistenzverstärkung, ein Modul zur semantischen Informationsentkopplung, ein kontrastgesteuertes Merkmalsaggregationsmodul und einen größenbewussten Decoder ein und verbessert so die Genauigkeit des medizinischen Bildsegmentierungsmodells weiter.
* Papieradresse:
https://arxiv.org/abs/2412.08345
Darüber hinaus haben die Kunming University of Science and Technology und die Ocean University of China eine bidirektionale, schrittweise Merkmalsausrichtung (BSFA)-Methode zur nicht ausgerichteten medizinischen Bildfusion vorgeschlagen. Im Vergleich zu herkömmlichen Methoden werden in dieser Studie nicht ausgerichtete multimodale medizinische Bilder gleichzeitig ausgerichtet und zusammengeführt. Dies geschieht durch einen einstufigen Ansatz innerhalb eines einheitlichen Verarbeitungsrahmens. Dadurch wird nicht nur die Koordination von Doppelaufgaben erreicht, sondern auch das Problem der Modellkomplexität, das durch die Einführung mehrerer unabhängiger Merkmalscodierer entsteht, effektiv reduziert.
* Papieradresse:
https://doi.org/10.48550/arXiv.2412.08050
Allerdings ist die Forschung zur Kombination medizinischer 3D-Bildgebungstechnologie mit KI auch mit einigen Herausforderungen verbunden. Themen wie Datenschutz, Transparenz der Algorithmen, Fähigkeit zur Modellgeneralisierung und behördliche Aufsicht bleiben zentrale Themen, die angegangen werden müssen. Mit der kontinuierlichen Weiterentwicklung der Technologie und der Verbesserung der Vorschriften könnten diese Probleme in Zukunft schrittweise gelöst werden, wodurch die breitere Anwendung der KI-Technologie im Bereich der medizinischen Bildgebung gefördert wird.








