Command Palette
Search for a command to run...
Neue Erfolge Vom David Baker-Team! RFdiffusion Entwickelt Sich Weiter, Um Ein De-novo-Antikörperdesign Mit Atomarer Präzision Zu Erreichen
Im späten 19. Jahrhundert führte der deutsche Bakteriologe Emil Adolf von Behring umfangreiche Forschungen zum Diphtherietoxin durch. Zu dieser Zeit war die Diphtherie wie die Sense des Todesgottes und forderte gnadenlos das Leben vieler Kinder. Im Rahmen des Experiments injizierte Behring Kaninchen Spuren von Diphtherietoxin, in der Hoffnung, ihre Reaktionen zu beobachten und einen Weg zu finden, das Toxin zu bekämpfen. Einige Tage später stellte er zu seiner Überraschung fest, dass die Kaninchen nicht nur nicht starben, sondern auch eine Resistenz gegen nachfolgende Injektionen von Diphtherietoxin entwickelten. Weitere Untersuchungen ergaben, dass im Kaninchenserum eine Substanz vorhanden war, die das Diphtherietoxin neutralisieren kann.Dies war das erste Mal, dass Menschen die Existenz von Antikörpern entdeckten.Diese zufällige Entdeckung öffnete den Vorhang für die Anwendung der Antikörperforschung und zeigte das große Potenzial auf, das in der Nutzung der körpereigenen Kraft zur Bekämpfung von Krankheiten liegt.
Nach mehr als einem Jahrhundert eingehender Forschung haben die Wissenschaftler ihre Anstrengungen auf dem Gebiet der Antikörperforschung fortgesetzt, konnten jedoch noch keinen effizienten Weg entwickeln, der sich vollständig auf Computerdesign stützt, um neue Antikörper gegen spezifische Epitope zu erzeugen. Heute,Die Entdeckung therapeutischer Antikörper beruht noch immer hauptsächlich auf zwei traditionellen Methoden: der Immunisierung von Tieren oder dem zufälligen Screening von Bibliotheken.Angesichts der zunehmend komplexen Herausforderungen bei Krankheiten treten die Grenzen dieser traditionellen Methoden immer deutlicher zutage. Es bedarf dringend neuer Durchbrüche, um eine effizientere und präzisere Entwicklung von Antikörpern gegen spezifische Ziele zu erreichen und so neue Hoffnung in die Antikörperforschung zu bringen.
Um das Problem der Antikörperentdeckung mit herkömmlichen Methoden zu lösen, kombinierte das Team von David Baker, Professor für Biochemie an der University of Washington, und seine Mitarbeiter das fein abgestimmte computergestützte Proteindesign des RF-Diffusionsnetzwerks mit einem Hefe-Display-Screening.Wir haben erfolgreich Antikörper mit variablen schweren Ketten (VHHs) und Einzelketten-ScFvs mit variablen Fragmenten erzeugt, die mit atomarer Präzision an spezifische Epitope binden können.Die Machbarkeit des De-novo-Designs von Antikörperdomänen wurde nachgewiesen. Dieser Ansatz bietet einen rationalen Rahmen für die computergestützte Entwicklung, das Screening, die Isolierung und die Charakterisierung neu entwickelter Antikörper und erreicht so eine Präzision auf atomarer Ebene bei der Struktur- und Epitop-Zielausrichtung.
Die entsprechenden Forschungsergebnisse wurden als Preprint auf bioRxiv unter dem Titel „Atomically accurate de novo design of antibodies with RFdiffusion“ veröffentlicht.

Papieradresse:
https://doi.org/10.1101/2024.03.14.585103
Das Open-Source-Projekt „awesome-ai4s“ vereint mehr als 200 AI4S-Papierinterpretationen und stellt umfangreiche Datensätze und Tools bereit:
https://github.com/hyperai/awesome-ai4s
Technologische Innovationen in der Antikörperforschung: Durchbrüche und Herausforderungen der KI
Als dominierende Kategorie der Proteintherapeutika sind Antikörper aufgrund ihrer hohen Spezifität und geringen Nebenwirkungen zur zentralen Behandlungsmethode in den Bereichen Tumore, Autoimmunerkrankungen usw. geworden. Bis 2025 wurden weltweit mehr als 160 Antikörpermedikamente von den Aufsichtsbehörden zugelassen, und ihr Marktvolumen ist in den letzten zehn Jahren jährlich um durchschnittlich 15% gewachsen.Man geht davon aus, dass dieser Wert in den nächsten fünf Jahren 445 Milliarden US-Dollar erreichen wird.
Jedoch,Die traditionelle Technologie zur Antikörperentwicklung beruhte lange Zeit auf der Immunisierung von Tieren und der zufälligen Durchmusterung von Bibliotheken.Es bestehen erhebliche Engpässe. Zur Immunisierung von Tieren sind mehrere Antigeninjektionen erforderlich, um das Immunsystem des Tieres zur Produktion von Antikörpern anzuregen.Der Prozess dauert normalerweise 6–12 Monate.Darüber hinaus ist es aufgrund der unterschiedlichen Immunreaktionen einzelner Tiere schwierig, hochwirksame Antikörper gegen komplexe Antigene wie Membranproteine zu erhalten. Obwohl zufällige Bibliotheksscreeningmethoden (wie etwa die Phagendisplay-Technologie) den Screening-Bereich erweitern können, ist es mit ihnen schwierig, Immunreaktionen auf Antigene wie etwa entfaltete Proteine oder glykosylierte Epitope zu stimulieren, und die Affinität der gescreenten Antikörper ist im Allgemeinen gering.
Um die Beschränkungen der traditionellen Technologie zu überwinden, haben sich computergestütztes Design und künstliche Intelligenz nach und nach zu neuen Richtungen in der Antikörperforschung und -entwicklung entwickelt. In frühen Studien konnte die Leistung von Antikörpern erfolgreich verbessert werden, indem Rückstände in vorhandene Antikörpergerüste eingebettet, die Konformation der komplementärbestimmenden Region (CDR-Schleife) optimiert und die Interaktionsschnittstelle mithilfe des Rosetta-Algorithmus transformiert wurde. Zum Beispiel,Eine Studie aus dem Jahr 2018 erhöhte die Affinität von Anti-PD-1-Antikörpern durch computergestütztes Design um das 20-fache.
In den letzten Jahren hat die Deep-Learning-Technologie die Generierung von Antikörpersequenzen weiter vorangetrieben. Im Jahr 2023 entwickelte ein Team der Stanford University mithilfe neuronaler Netzwerke breitbandige neutralisierende Antikörper gegen die neuen Coronavirus-Varianten. Ihre In-vitro-Aktivität war dreimal höher als bei herkömmlichen Methoden. Im Jahr 2024Das von David Bakers Team entwickelte RFdiffusionsmodell ermöglicht die De-novo-Entwicklung von Bindungsproteinen, ohne dass eine vordefinierte Rückgratstruktur erforderlich ist.Diese Technologie simuliert den physikalischen Prozess der Proteinfaltung, um eine Bindungsschnittstelle zu erzeugen, die die Form des Zielepitops in hohem Maße ergänzt, und hat erfolgreich zur Entwicklung neuer Inhibitoren gegen Hämagglutinin des Influenzavirus geführt. Allerdings ist diese Studie nur auf Epitope mit regelmäßigen Sekundärstrukturen wie α-Helices und β-Faltungen anwendbar, während Antikörper normalerweise auf komplexen Ringstrukturen (wie CDR-H3) beruhen, um eine Bindung zu erreichen, was ihre direkte Anwendung im Antikörperdesign immer noch schwierig macht.
Die Entwicklung strukturell präziser Antikörper von Grund auf, also ohne Homologie zu bekannten Antikörpern, bleibt ein ungelöstes Problem. Zu den zentralen Herausforderungen zählen unzureichende dynamische Konformationssimulationen, ein Mangel an qualitativ hochwertigen Daten und lange experimentelle Verifizierungszyklen.
Der Bindungsprozess von Antikörpern beinhaltet Konformationsänderungen in flexiblen CDR-Schleifen, und mit vorhandenen Algorithmen ist es schwierig, solche dynamischen Interaktionen genau zu simulieren. Gleichzeitig schränkt der Mangel an Strukturdaten zu Antikörper-Antigen-Komplexen die Generalisierungsfähigkeit von Deep-Learning-Modellen ein. Obwohl computergestütztes Design die anfängliche F&E-Zeit erheblich verkürzen kann, dauern Expressionsreinigung und Aktivitätstests immer noch mehrere Wochen und stellen einen Engpass im geschlossenen Technologiekreislauf dar.Zukünftige Durchbrüche könnten auf der Entwicklung hybrider Algorithmen beruhen, um physikalische Modelle mit generativen KI-Tools zu integrieren.Daten aus der Einzelzellsequenzierung und Kryo-Elektronenmikroskopie werden auf Grundlage der Erstellung einer maßstabsübergreifenden Datenbank integriert. Eine Echtzeit-Iteration wird durch Roboterautomatisierung auf Grundlage des F&E-Modells „Dry and Wet Closed Loop“ erreicht.
Von der Immunisierung von Tieren bis zum computergestützten Design stellt die Innovation der Antikörpertechnologie nicht nur einen Paradigmenwechsel im Bereich der Biomedizin dar, sondern spiegelt auch das Potenzial der interdisziplinären Grenzüberschreitung wider. Mit der Entwicklung der künstlichen Intelligenz und der synthetischen Biologie könnte die Vision, Antikörper von Grund auf neu zu entwickeln, allmählich Wirklichkeit werden und ein neues Kapitel der Präzisionsmedizin aufschlagen.
Es ist erwähnenswert, dass auf dem Gebiet des computergestützten Antikörperdesigns in jüngster Zeit entscheidende Durchbrüche erzielt wurden. Im März 2025 entwickelte das Team von David Baker ein neues KI-Proteingenerierungstool namens RFdiffusion (genannt: neue RFdiffusion).Eine neue Version des Modells, die speziell für variable Antikörperregionen wie CDR-Loops optimiert ist, wurde veröffentlicht.Dem Team war es in einer Studie im März 2024 gelungen, kurzkettige funktionelle Antikörperfragmente wie Nanobodies zu erzeugen. Aufgrund der Komplexität der Antikörperstruktur wies die damalige Version (genannt: gewöhnliche RF-Diffusion) jedoch noch Einschränkungen bei der Entwicklung komplexerer Antikörperstrukturen auf.
Nach mehr als einem Jahr der Algorithmus-Iteration ist die neue RFdiffusion nun in der Lage, durch die Einführung eines Trainings für eine Datenbank mit Antikörper-Antigen-Komplexstrukturen ein vollständigeres Einzelketten-Variablenfragment-scFv zu erzeugen, das den natürlichen menschlichen Antikörpern näher kommt. Dieser Fortschritt markiert denMithilfe künstlicher Intelligenz ist es gelungen, schwere und leichte Antikörperketten mit vollständigen Antigen-Bindungsdomänen zu koordinieren, ohne dass dafür Vorlagen erforderlich wären.Gibt neue Hoffnung für die Antikörperentwicklung.
Neue RFdiffusion: De novo Antikörperdesign mit atomarer Präzision
Um die RF-Diffusion an das Antikörperdesign anzupassen, nahm das Forschungsteam einige darauf basierende Feinabstimmungen vor. Wie in der folgenden Abbildung gezeigt, verwendet es während des Trainingsprozesses das AlphaFold2/RF2-Framework zur Darstellung des Protein-Rückgrats und fügt dem Protein-Framework durch eine Reihe von „Zeitschritten (T)“ Rauschen hinzu, bis das Framework nicht mehr erkennbar ist. Bei jedem Zeitschritt sagt RFdiffusion die rauschfreie Struktur voraus, optimiert durch Minimierung des mittleren quadratischen Fehlers mse zwischen der wahren Struktur X₀ und der vorhergesagten Struktur pX₀. Nach dieser einzigartigen Trainingsmethode RFdiffusion ist in der Lage, ausgehend von zufälligen Restverteilungen zum Zeitpunkt der Inferenz schrittweise neue Proteinstrukturen zu generieren.
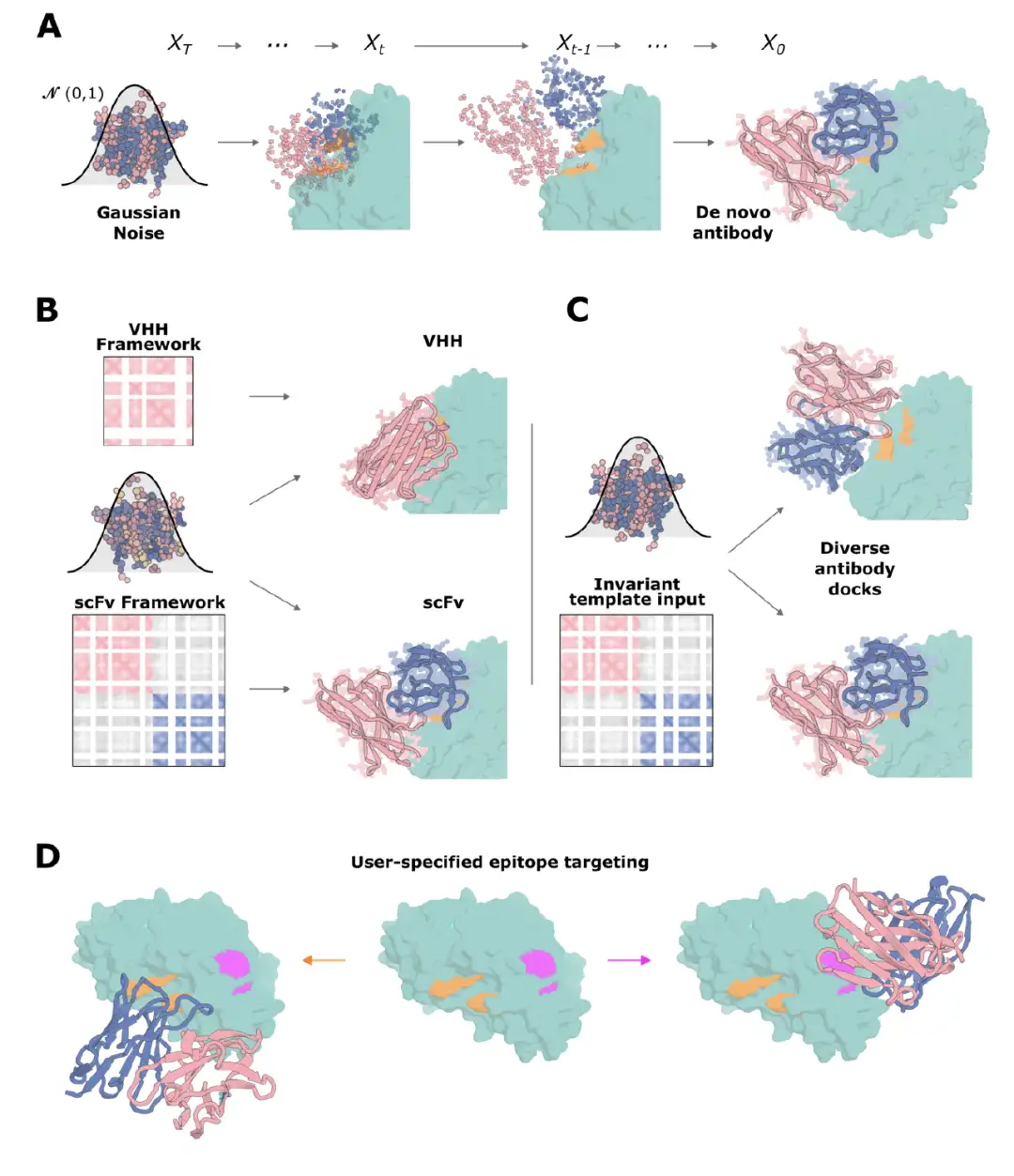
Anschließend wurde in der Studie die neuartige RF-Diffusion angewendet, um Einzeldomänen-Antikörper-VHHs zu entwickeln. VHHs werden auf der Grundlage der variablen Domänen von Schwerketten-Antikörpern von Kameliden entwickelt. Aufgrund ihrer geringeren Größe ist die Zusammenstellung der Gene, die die Designs kodieren, einfacher und kostengünstiger als bei einkettigen variablen Fragmenten (scFv) oder antigenbindenden Fragmenten (Fab).
Obwohl VHHs nur drei CDR-Schleifen haben, also weniger als die sechs herkömmlicher Antikörper, ist die durchschnittliche Interaktionsoberfläche von VHHs der herkömmlicher Antikörper sehr ähnlich.Dies deutet darauf hin, dass der Ansatz zum Entwurf von VHHs auch auf den Antikörperentwurf anwendbar ist.
Für den Entwurf von VHHs wählten die Forscher ein weit verbreitetes chimäres VHH-Framework als Grundlage, das auf eine Reihe krankheitsbezogener Ziele abzielte, darunter Clostridium difficile Toxin B (TcdB), Influenza H1 Hämagglutinin HA usw., und führten ein CDR-Loop-Sequenzdesign im Zielkontext durch ProteinMPNN durch und führten dann ein Screening mithilfe des fein abgestimmten RoseTTAFold2-Netzwerks durch. Um bei der Entwicklung von Influenza-HA die Bedingungen des Versuchsdesigns mit den Berechnungsparametern in Einklang zu bringen, verwendeten die Forscher für Affinitätsmessungen kommerziell hergestellte monomere HA-Produkte, die in Insektenzellen exprimiert werden.
Die Ergebnisse zeigen, dassRFdiffusion ermöglicht die Entwicklung von VHHs, die spezifisch mit Ziel-Epitopen interagieren.Die Moleküle mit der höchsten Affinität zur RSV-Stelle III, Influenza-HA, RBD und TcdB sind in der folgenden Abbildung dargestellt. Die CDR-Schleifen unterscheiden sich erheblich von denen natürlicher VHHs, was darauf hindeutet, dass das Design über den Rahmen des Trainingsdatensatzes hinausging. Für TcdB ist das Zielepitop die Frizzled-7-Schnittstelle und es gibt keine Antikörper oder VHHs, die auf diese Stelle im PDB abzielen. Darüber hinaus neutralisierte TcdB VHH die Toxizität von TcdB in CSPG4-Knockout-Zellen.
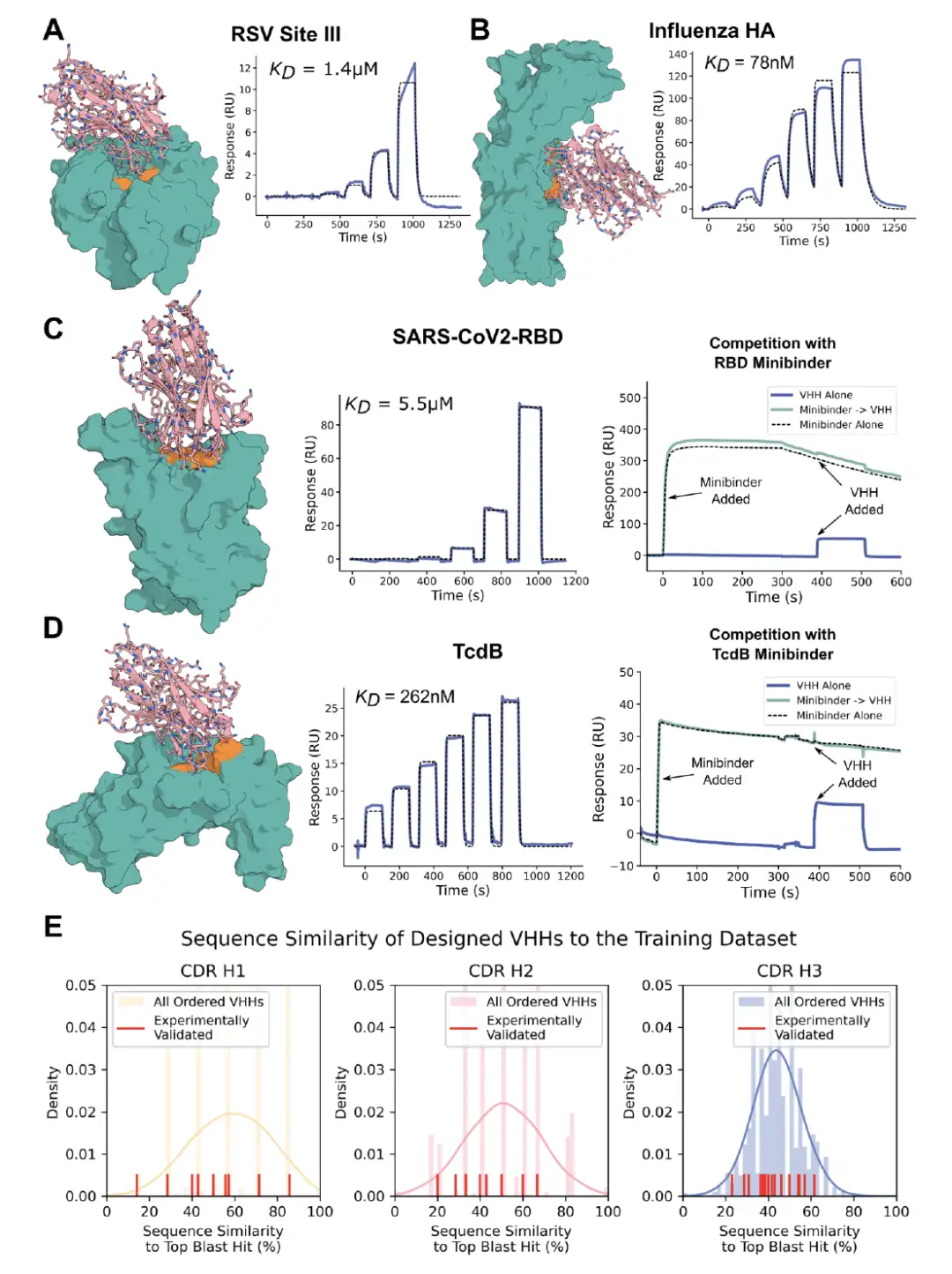
* Abbildung AC: Testergebnisse des VHH-Designs, das auf RSV III-Locus, Influenza HA, RBD und TcdB abzielt
* Abbildung E: Unterschied zwischen VHH-Design und Trainingsdatensatz
Die Forscher untersuchten außerdem die Fähigkeit der RFdiffusion, schwere und leichte Ketten in einem Einzelketten-variablen Fragment-scFv-Format zu konstruieren. Anders als bei VHHs ist das Design von scFv komplizierter und erfordert die Konstruktion aller sechs CDRs und ihrer Andockmuster sowohl auf der schweren als auch auf der leichten Kette. Die Gensynthese von scFv ist jedoch mit Herausforderungen verbunden. Einerseits ist die scFv-Sequenz lang und mit herkömmlichen Oligonukleotidpaaren schwer zusammenzusetzen. Andererseits weisen scFvs eine hohe Sequenzhomologie auf, was eine spezifische Paarung erschwert.
Zu diesem Zweck entwickelten die Forscher ein schrittweises Montageprotokoll, um den Aufbau schwerer und leichter Ketten zu erreichen.Sie können gezielt nach Designmodell gepaart oder in Design-Teilmengen mit ähnlichen gezielten Bindungsmodi kombiniert und gemischt werden.Experimentelle Ergebnisse zeigten, dass neue scFvs, die durch die Kombination von Schwer- und Leichtkettenpaaren unterschiedlichen Designs erzeugt wurden, mit einer ähnlichen Häufigkeit an das Zielepitop binden konnten wie das ursprüngliche Design. Darüber hinaus interagieren die CDRs der schweren und leichten Ketten in entworfenen Ensembles mit der gleichen Bindungsorientierung mit unterschiedlichen Regionen und können ohne Verlust der strukturellen Präzision kombiniert werden, während eine zufällige Paarung selten zu vorhergesagten Bindern führt.
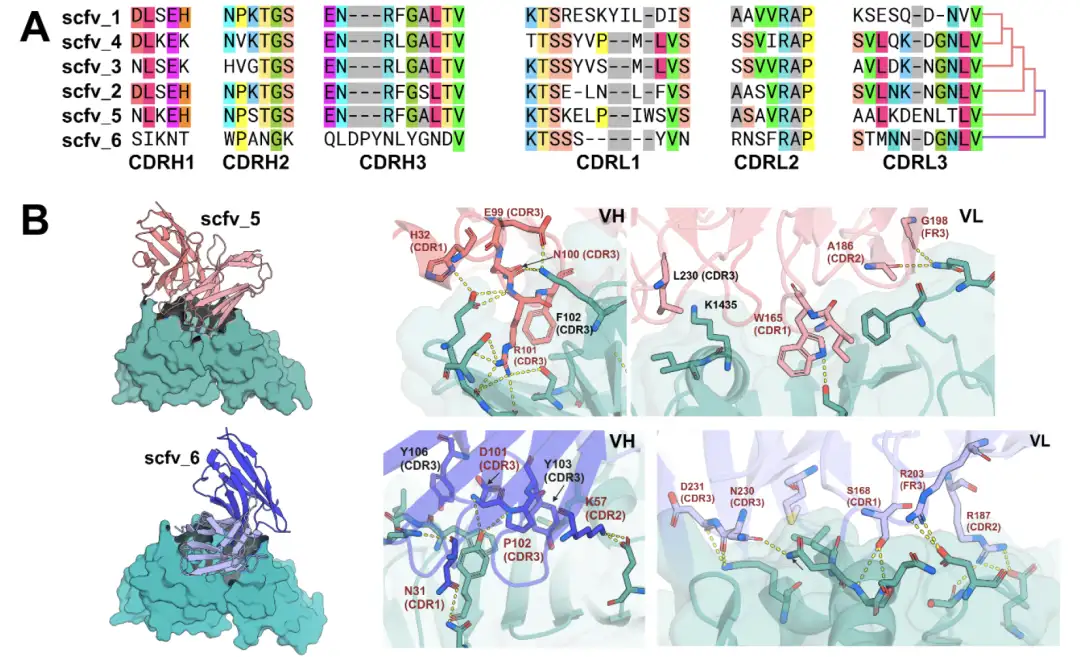
* Abbildung A: Mehrfachsequenz-Alignment von 6 scFvs, die an TcdB binden
* Abbildung B: Von AlphaFold3 vorhergesagte Struktur von scFv5 und scFv6 im Komplex mit der TcdB-Rezeptorbindungsdomäne
David Baker: Die Evolution von Antikörpern und RF-Diffusion
Tatsächlich ist diese Studie nur die Spitze des Eisbergs der Forschungsergebnisse von David Baker. Tatsächlich hat das Team von David Baker an der Spitze der Computerbiologie eine Reihe von Durchbrüchen erzielt, von der gezielten Bekämpfung von Viren bis hin zur Behandlung von Krankheiten durch Proteindesign auf Basis künstlicher Intelligenz.

Unter ihnen ist das RF-Diffusionsmodell für sie zu einer wichtigen Grundlage geworden, um das Paradigma der Entwicklung von Antikörpermedikamenten zu ändern. Im Jahr 2021 entwickelte das Team von David Baker RosettaFold basierend auf dem AlphaFold2-Framework.Seine Kernfähigkeiten beschränken sich auf die Vorhersage der dreidimensionalen Strukturen bekannter Proteine.
* RoseTTAFold Open-Source-Adresse:
https://github.com/RosettaCommons/RoseTTAFold
Das Forschungsteam erkannte bald, dass die wahre Revolution eher in der „Generierung“ als in der „Reproduktion“ liegt. Sie kombinierten also das Diffusionsmodell und den Proteinfaltungsalgorithmus und brachten 2023 die erste Generation der RFdiffusion auf den Markt. Dieses Modell ist wie der Schlüssel zum Reverse Engineering: Bei der traditionellen Methode muss die Struktur aus der Aminosäuresequenz abgeleitet werden.Durch RF-Diffusion kann umgekehrt ein neues Proteinskelett basierend auf den funktionellen Zielanforderungen erzeugt werden.In frühen Tests gelang es dem Unternehmen, einen Nanokörper zu entwickeln, der an Influenza-Hämagglutinin bindet. Allerdings wies die CDR-Loop-Region des von ihm erzeugten Antikörpers noch immer Konformationsabweichungen auf und die Kryo-Elektronenmikroskopie zeigte eine mittlere quadratische Abweichung von 1,2 Å in der Bindungsschnittstelle.
* Papieradresse:
https://www.science.org/doi/10.1126/science.abj8754
Diese Einschränkung war der Grund für die Einführung einer wichtigen Verbesserung im Jahr 2024 – eines dynamischen Rückhaltesystems.Das Forschungsteam fügte dem Modell physikalische und chemische Parameterbeschränkungen der Antigen-Antikörper-Bindungsstelle hinzu, sodass der Generierungsprozess nicht nur die strukturelle Stabilität berücksichtigt, sondern auch die dynamischen Wechselwirkungen zwischen Molekülen simuliert.
Die verbesserte RFdiffusion zeigte bei der Entwicklung des neuen Coronavirus-Spike-Protein-Antikörpers hervorragende Ergebnisse: Die damit konstruierte flexible Ringstruktur fixierte die konservierten Epitope präzise auf der Virusoberfläche, was durch Experimente bestätigt wurde.Seine Bindungsaffinität erreicht 0,8 nM und ist damit 15-mal stärker als bei natürlichen Antikörpern.Noch bemerkenswerter ist, dass dieses Modell beginnt, „nicht medikamentös behandelbare“ Ziele herauszufordern: Der für den IL-23-Rezeptor entwickelte Miniatur-Antikörper enthält nur 58 Aminosäuren, kann aber bei einer hohen Temperatur von 80 °C und in einer Pepsin-Umgebung aktiv bleiben, wodurch erstmals die orale Verabreichung von Antikörpern möglich wird.
* Papieradresse:
https://www.biorxiv.org/content/10.1101/2024.03.14.585103v2
Im Jahr 2025 erreichte RFdiffusion das Stadium der multimodalen Fusion. Das Team integrierte Einzelzellsequenzierungsdaten und Strukturbibliotheken aus der Kryo-Elektronenmikroskopie, wodurch das Modell in der Lage war, personalisierte Antikörper direkt auf der Grundlage der Eigenschaften des Immunrepertoires des Patienten anzupassen. Im jüngsten Fall wurden einem Patienten mit medikamentenresistentem Lungenkrebs Tumor-Neoantigene in das System injiziert.Durch RF-Diffusion wurden innerhalb von 36 Stunden 12 Kandidaten-Antikörper erzeugt, von denen drei in Organoid-Modellen eine signifikante tumortötende Wirkung zeigten.Das Modell ist nicht mehr auf die Entwicklung von Antikörpern beschränkt: Es erforscht die Synthese artenübergreifender Proteinelemente, etwa die Fusion mechanosensitiver Ionenkanäle von Tiefsee-Barytoskopiebakterien mit menschlichen Antikörpern, um intelligente Arzneimittel zu entwickeln, die Veränderungen des pH-Werts im Tumormikroumfeld erkennen können.
* Papieradresse:
https://www.nature.com/articles/s41586-024-08393-x
Man kann erkennen, dass sich RFdiffusion von einem „Protein-3D-Drucker“ zu einem „Lebensfunktionsarchitekten“ wandelt und die Grenzen der synthetischen Biologie neu definiert. Noch interessanter ist, dass diese Entwicklung noch lange nicht zu Ende ist und Innovationen in der Antikörpertechnologie dem biomedizinischen Bereich zu neuen Höhen verhelfen.








