Command Palette
Search for a command to run...
Oxford/Amazon/Westlake University/Tencent Und Andere Schlugen Ein Multimodales, Mehrdomänen- Und Mehrsprachiges Medizinisches Modell M³FM Vor, Das Für Die Klinische Nullstichprobendiagnose Verwendet Werden Kann

Ich glaube, dass viele Freunde, die Marvel-Filme lieben, von dieser Szene begeistert waren. Im Film „Iron Man 2“ sammelte der Butler Jarvis mit künstlicher Intelligenz Starks Blutproben und verwendete Deep-Learning-Algorithmen, um die Probendaten schnell zu modularisieren. Es analysierte den Palladiumgehalt in Starks Körper genau und schnell und machte bei der Veröffentlichung eines Berichts sogar bereichsübergreifende Vorschläge, wie etwa: „Vorhandene Elemente können Palladiummetall nicht ersetzen, und es müssen neue Elemente synthetisiert werden.“Obwohl es sich hierbei nur um ein Filmmaterial von einigen zehn Sekunden handelt, demonstriert es perfekt die Automatisierung, Intelligenz und prozessbasierten Funktionen der intelligenten Gesundheitsversorgung.
Um im wirklichen Leben jedoch dasselbe Ergebnis zu erzielen, muss das medizinische Personal komplizierte Prozesse wie Blutentnahme und -untersuchung, Bildanalyse, Datenvergleich, Berichterstellung und Krankheitsklassifizierung durchlaufen. Und dies gilt nur aus einer Makroperspektive. Wenn wir es zerstören, wird die Situation noch schlimmer. Medizinische Bilder sind die häufigste Art der klinischen Diagnose. Sie können beispielsweise klinische Befunde beschreiben und eine Grundlage für die weitere Diagnose der Krankheit bieten. Wenn es jedoch darum geht, einen Bericht über medizinische Bilder in natürlicher Sprache präzise, präzise, vollständig und verständlich zu beschreiben, empfinden viele medizinische Mitarbeiter dies als mühsam und langweilig.Statistiken zeigen, dass selbst erfahrene Ärzte im Durchschnitt 5 Minuten oder länger brauchen, um einen Bericht fertigzustellen.
Auch wenn die Science-Fiction die Realität noch nicht vollständig erhellt hat, ist glücklicherweise bereits ein Lichtschimmer durch die Risse in der Dunkelheit erschienen. An der Schnittstelle zwischen künstlicher Intelligenz und medizinischer Gesundheit haben immer mehr wissenschaftliche Forscher umfangreiche Forschungen durchgeführt und Methoden zur automatischen Berichterstellung entwickelt. Mit diesen Methoden werden automatisch Berichtsentwürfe erstellt, die das medizinische Personal prüfen, ändern und als Referenz verwenden kann. Einerseits können sie die zeit- und arbeitsintensiven Arbeitsaufgaben des medizinischen Personals effektiv lösen und andererseits durch Automatisierung die Wahrscheinlichkeit menschlicher Fehler reduzieren.
Kürzlich veröffentlichte npj Digital Medicine, eine Zeitschrift der international renommierten Fachzeitschrift Nature Portfolio, eine Studie mit dem Titel „Ein multimodales, mehrdomänenbasiertes, mehrsprachiges medizinisches Grundlagenmodell für die klinische Zero-Shot-Diagnose“.Es erwähnt ein multimodales (Bild und Text), multidomänen (CT und CXR) und mehrsprachiges (Chinesisch und Englisch) medizinisches Grundlagenmodell M³FM (Multimodal Multidomain Multilingual Foundation Model), das für die klinische Nullstichprobendiagnose verwendet werden kann und die Krankheitsberichterstattung und Krankheitsklassifizierung unterstützt.Die Forscher demonstrierten die Wirksamkeit dieser Methode anhand von neun Benchmark-Datensätzen für zwei Infektionskrankheiten und 14 nicht übertragbare Krankheiten und übertrafen damit frühere Methoden.
Die Studie verfügt über eine luxuriöse Autorenriege. Neben Teams der Oxford University, der University of Rochester, von Amazon und anderen Institutionen sind auch Dr. Zheng Yefeng vom Medical Artificial Intelligence Laboratory der Westlake University und Dr. Wu Xian, Leiter des Tianyan Research Center des Tencent Youtu Lab, dabei.

Papieradresse:
https://www.nature.com/articles/s41746-024-01339-7
Das Open-Source-Projekt „awesome-ai4s“ vereint mehr als 200 AI4S-Papierinterpretationen und stellt umfangreiche Datensätze und Tools bereit:
https://github.com/hyperai/awesome-ai4s
Datenverlust ist bei bestehenden Methoden immer noch ein Problem
Die medizinische Bildgebung bildet die Grundlage für medizinische Bildberichte und die Klassifizierung von Krankheiten und spielt eine wichtige Rolle bei der anschließenden klinischen Diagnose. Daher ist die Erforschung entsprechender Automatisierungsmethoden ganz selbstverständlich zu einem der Forschungsschwerpunkte im wissenschaftlichen Forschungsbereich geworden. Trotz der fruchtbaren Forschungsergebnisse bestehen aus praktischer Sicht jedoch noch viele Defizite.Eine der größten Herausforderungen besteht darin, dass es nur wenige oder gar keine Daten gibt.
einerseits,Die Erstellung von Krankheitsberichten ähnelt der bildbasierten Sprachgenerierungsaufgabe, bei der das Ziel darin besteht, beschreibenden Text zur Beschreibung des Eingabebilds zu generieren. Herkömmliche Basismethoden basieren häufig in hohem Maße auf großen Mengen hochwertiger medizinischer Trainingsdaten, die von Klinikern kommentiert werden. Deren Erfassung ist kostspielig und zeitaufwändig, insbesondere bei seltenen Krankheiten und nicht-englischen Sprachen.
Insbesondere bei neuen oder seltenen Erkrankungen fehlen in der Regel ausreichende Daten zur Wirksamkeit, um im Frühstadium dieser Erkrankungen trainieren zu können. So konnten beispielsweise im Frühstadium der neuen Coronavirus-Pneumonie, die Ende 2019 weltweit wütete, nur begrenzte Daten gesammelt werden. Dies führte dazu, dass die Trainingszeit des Systems die Dauer der ersten Wellen der Epidemie bei weitem übertraf. Laut dem „2024 China Rare Disease Industry Trend Observation Report“ gibt es weltweit mehr als 7.000 bekannte seltene Krankheiten. Konservativen, auf Beweisen basierenden Daten zufolge beträgt die Prävalenz seltener Krankheiten in der Bevölkerung etwa 3,51 TP3T bis 5,91 TP3T und die Zahl der Menschen, die weltweit von seltenen Krankheiten betroffen sind, liegt bei etwa 260 bis 450 Millionen. Eine so große und atypische Krankheit macht die oben genannten Probleme zweifellos noch schwieriger.
Darüber hinaus umfasst das globale Gesundheitssystem unterschiedliche Regionen, unterschiedliche Bevölkerungsgruppen und unterschiedliche Sprachen. Für andere Sprachen als Englisch sind die entsprechenden gekennzeichneten Daten meist sehr spärlich oder fehlen sogar vollständig. Daher stellen die begrenzten gekennzeichneten Daten zweifellos eine große Herausforderung für nicht-englischsprachige Trainingssysteme dar, die vorhandene Methoden verwenden. Gleichzeitig erschwert dies den Umgang mit ungewöhnlichen Sprachen mit bestehenden Methoden, was das Ziel der KI-Fairness weiter beeinträchtigt und unterrepräsentierten Gruppen nicht den vollen Nutzen bringt.
auf der anderen Seite,Um Krankheiten effektiv zu klassifizieren, sind aktuelle fortschrittliche Modelle wie BioViL, REFERS, MedKLIP und MRM größtenteils vom Erfolg von CLIP inspiriert, die alle entwickelt wurden, um medizinische multimodale Daten besser zu verstehen. Bei der Implementierung nutzen diese Methoden kontrastives Lernen und trainieren CLIP-Modelle vorab mit medizinischen Daten. Da die meisten Modelle jedoch speziell auf Röntgenaufnahmen des Brustkorbs (CXR) ausgerichtet sind, können sie medizinische Bilder und Texte aus mehreren Domänen und Sprachen in einem einzigen Rahmen im Allgemeinen nicht verarbeiten. Gleichzeitig ist es in früheren Arbeiten auch nicht gelungen, eine Zero-Shot-Krankheitsberichterstattung in verschiedenen Bereichen der Sprache und der Bilder durchzuführen.
* Das CLIP-Modell ist ein von OpenAI entwickeltes kontrastives Sprachbild-Vortrainingsmodell – eine effektive Methode zum Lernen aus der Überwachung natürlicher Sprache. CLIP lernt die Assoziation zwischen Bildern und Text hauptsächlich durch kontrastives Lernen und trainiert vorab anhand umfangreicher Bild-Text-Paare, sodass das Modell Informationen aus verschiedenen Modalitäten verstehen und verknüpfen kann.
In diesem Zusammenhang ist es dringend erforderlich, ein Modell zu entwickeln, das multimodale, multidomänen und mehrsprachige klinische Diagnosen mit wenigen oder keinen Proben durchführen kann.Die in dieser Studie vorgeschlagenen spezifischen Neuerungen sind die folgenden:
* Das vorgeschlagene M³FM ist der erste Versuch, eine multimodale, mehrdomänen- und mehrsprachige klinische Zero-Shot-Diagnose durchzuführen, bei der gekennzeichnete Daten für das Training rar sind oder sogar vollständig fehlen.
* M³FM validiert seine Wirksamkeit anhand von 9 Datensätzen, darunter zwei Bereiche medizinischer Bilddaten, nämlich CXR und CT; zwei verschiedene Sprachen, nämlich Chinesisch und Englisch; zwei klinische Diagnoseaufgaben, nämlich Krankheitsberichterstattung und Krankheitsklassifizierung; und mehrere Krankheiten, darunter zwei Infektionskrankheiten und 14 nicht ansteckende Krankheiten.
M³FM: Zwei Hauptmodule, verifiziert durch mehrere Datensätze
In dieser Studie besteht die Kernidee des vorgeschlagenen M³FM darin, das Modell anhand öffentlicher medizinischer Daten über Modalitäten, Domänen und Sprachen hinweg vorab zu trainieren, um umfassendes Wissen zu erlernen und dieses Wissen dann zu nutzen, um nachgelagerte Aufgaben zu erfüllen, ohne dass gekennzeichnete Daten erforderlich sind. Die Hauptkomponenten des M³FM-Frameworks umfassen 2 Hauptmodule,Das sind MultiMedCLIP und MultiMedLM. Wie in der folgenden Abbildung dargestellt:
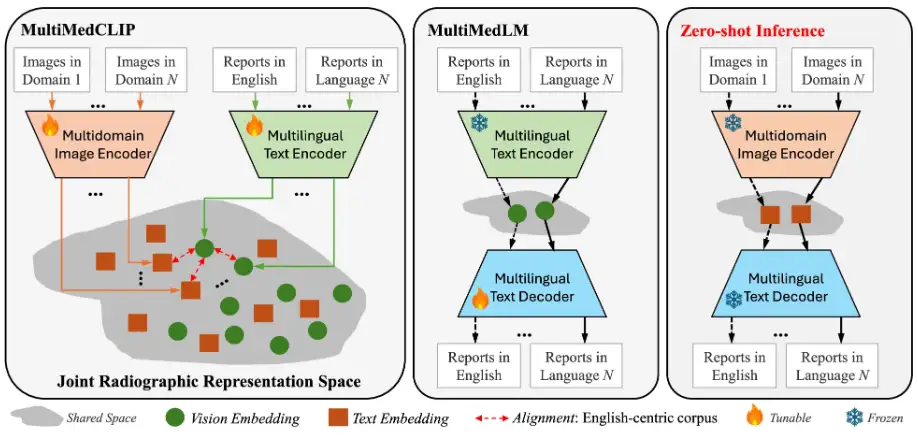
Der Prozess besteht darin, dass MultiMedCLIP verschiedene Sprachen und Bilder in einem gemeinsamen latenten Raum ausrichtet und überbrückt.Anschließend rekonstruiert MultiMedLM den Text basierend auf der Textdarstellung im gemeinsamen latenten Raum und schließlich generiert M³FM mehrsprachige Berichte direkt basierend auf der visuellen Darstellung von Eingabebildern aus verschiedenen Domänen im einheitlichen latenten Raum.
Konkret handelt es sich bei MultiMedCLIP um ein Modul zum Erlernen gemeinsamer Darstellungen, das einen visuellen Multidomänen-Encoder und einen mehrsprachigen Text-Encoder einführt, mit dem Ziel, einen gemeinsamen latenten Raum für die Ausrichtung visueller und textueller Darstellungen aus unterschiedlichen Bereichen der medizinischen Bildgebung und unterschiedlichen Sprachen zu schaffen. Inspiriert durch die Methode des kontrastiven Lernens verwendeten die Forscher den Verlust von InfoNCE (Info Noise Contrastive Estimation) und MSE (mittlerer quadratischer Fehler) als Trainingsziele, um die Ähnlichkeit zwischen positiven Beispielpaaren zu maximieren und die Ähnlichkeit zwischen negativen Beispielpaaren zu minimieren. Dadurch wurde eine Übereinstimmung zwischen visuellen Darstellungen in unterschiedlichen Feldern und Textdarstellungen in unterschiedlichen Sprachen erreicht und eine solide Grundlage für nachgelagertes Zero-Shot-Reasoning geschaffen.
MultiMedLM ist ein Modul zum Erstellen mehrsprachiger Berichte.Es wird ein mehrsprachiger Textdecoder eingeführt, der lernen soll, den endgültigen medizinischen Bericht auf der Grundlage der von MultiMedCLIP extrahierten Darstellungen zu erstellen. Dieser Teil wird durch Rekonstruktion des Eingabetextes trainiert, der chinesischer oder englischer Text sein kann, und verwendet den natürlichen Sprachgenerierungsverlust – XE-Verlust (Cross-Entropy) – als Trainingsziel. Es ist erwähnenswert, dass die Einführung des Rekonstruktionstrainings als unbeaufsichtigtes Training angesehen werden kann, für das zum Training nur unbeschriftete Klartextdaten erforderlich sind. Daher ist es nicht erforderlich, Aufgabenannotationsdaten für nachgelagerte Aufgaben zu trainieren. Um die Stabilität des MultiMedLM-Trainings sicherzustellen, führte das Forschungsteam außerdem zufällige Dropouts und Gaußsches Rauschen ein.
Im Experiment wurde der AdamW-Optimierer verwendet, wobei die Lernrate auf 1e-4 und die Batchgröße auf 32 eingestellt war. Die Experimente wurden auf PyTorch und V100 GPU unter Verwendung eines Trainings mit gemischter Präzision durchgeführt.
In Bezug auf Datensätze,Das Vortraining wurde an den Datensätzen MIMC-CXR und COVID-19-CT-CXR durchgeführt, wobei MIMC-CXR aus 377.110 CXR-Bildern und 227.835 englischen Radiologieberichten besteht, dem bisher größten veröffentlichten Datensatz; COVID-19-CT-CXR umfasst 1.000 CT/CXR-Bilder und entsprechende englische Berichte. Darüber hinaus extrahierten die Forscher die Hälfte des englischen Korpus aus den beiden Datensätzen und verwendeten Google Translator, um ein chinesisch-englisches Trainingsteam aufzubauen. Die Ergebnisse zeigten, dass diese Methode die Ergebnisse maschineller Textübersetzungen verbessern kann.
Während der Evaluierungsphase wurden die folgenden Datensätze verwendet: IU-Xray, COVID-19 CT, COV-CTR, Shenzhen Tuberculosis Dataset, COVID-CXR, NIH ChestX-ray, CheXpert, RSNA Pneumonia und SIIM-ACR Emphysema, was eine umfassende Bewertung der Modellleistung ermöglichte.
* IU-Röntgen:Eingeschlossen waren 7.470 Röntgenbilder des Brustkorbs und 3.955 englische Radiologieberichte. Der Datensatz wird zum Trainieren, Validieren und Testen zufällig in 80% – 10% – 10% aufgeteilt.
* COVID-19 CT:Es enthält 1.104 CT-Bilder und 368 chinesische Radiologieberichte. Ebenso wird der Datensatz zum Trainieren, Validieren und Testen zufällig in 80% – 10% – 10% aufgeteilt.
*COV-CTR:Enthält 726 COVID-19-CT-Bilder, verknüpft mit Berichten auf Chinesisch und Englisch.
* Shenzhen-Tuberkulose-Datensatz:Enthält 662 CXR-Bilder und die Trainings-, Validierungs- und Testsätze sind in 7:1:2 aufgeteilt.
* COVID-CXR:Der Datensatz enthält mehr als 900 CXR-Bilder und ist zum Trainieren, Validieren und Testen zufällig in 80% – 10% – 10% unterteilt.
* NIH-Röntgenaufnahme der Brust:Enthält 112.120 CXR-Bilder, jedes Bild ist mit dem Auftreten von 14 häufigen Strahlenkrankheiten gekennzeichnet und das Verhältnis von Trainings-, Validierungs- und Testsätzen beträgt 7:1:2.
* CheXpert:Enthält über 220.000 CXR-Diagnosebilder. Nach der Vorverarbeitung erhielten wir 218.414 Bilder im Trainingssatz, 5.000 Bilder im Validierungssatz und 234 Bilder im Testsatz.
* RSNA-Pneumonie:Es besteht aus etwa 30.000 radiologischen Bildern mit Trainings-, Validierungs- und Testsatzverhältnissen von 85% – 5% – 10%.
* SIIM-ACR Emphysem:Es umfasst 12.047 CXR-Bilder, wobei das Verhältnis von Trainings-, Validierungs- und Testsätzen 70% – 15% – 15% beträgt.
Experimente zeigen, dass M³FM eine überlegene Leistung aufweist und frühere fortschrittliche Methoden übertrifft.Wie in der Abbildung unten gezeigt. Wie die Ergebnisse der Krankheitsberichterstattung zeigen, sind frühere Methoden nicht in der Lage, die Aufgabe der Krankheitsberichterstattung im Zero-Shot-Setting zu bewältigen, während M³FM in der Lage ist, die Krankheitsberichterstattung in mehreren Sprachen und Domänen gleichzeitig in einem einzigen Framework durchzuführen. Im Few-Shot-Setting erzielt M³FM beim Training mit den nachgelagert gekennzeichneten Daten von 10% hochmoderne Ergebnisse und übertrifft bei der Berichterstellung von CT-zu-Chinesisch sogar den vollständig überwachten Ansatz R2Gen um 1,5%s CIDEr- und 1,2%s ROUGE-L-Werte.Dies zeigt, dass M³FM auch bei knappen gekennzeichneten Daten genaue und gültige mehrsprachige Berichte erstellen kann und daher insbesondere bei seltenen oder neu auftretenden Krankheiten nützlich ist.
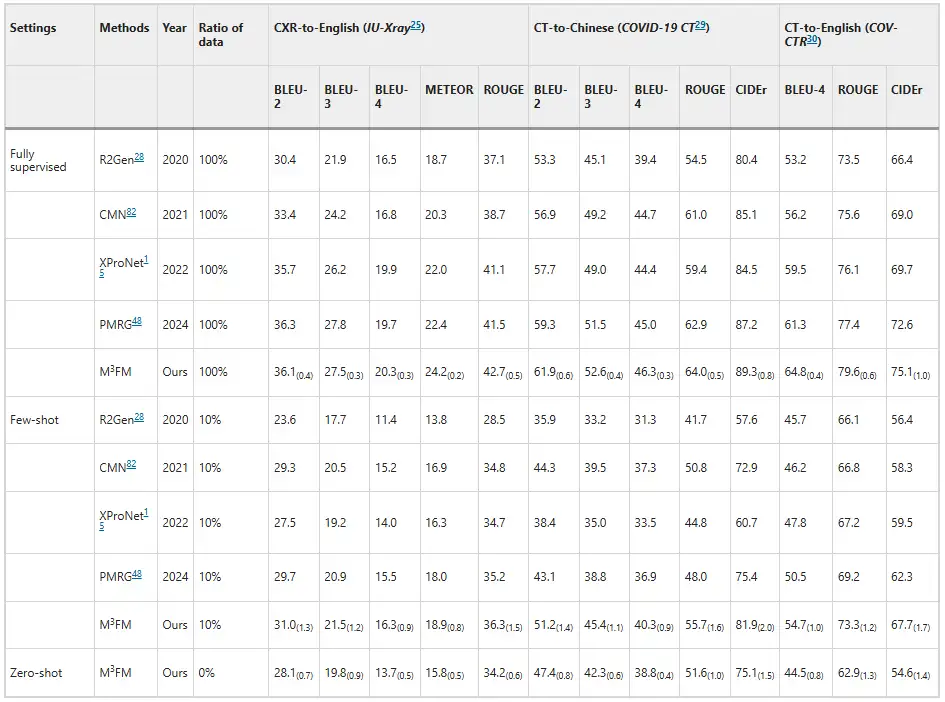
Darüber hinaus luden die Forscher zwei Kliniker ein, das Modell zu bewerten. Die Ergebnisse sind in der folgenden Abbildung dargestellt. Ohne jegliches Training mit gekennzeichneten Daten kann M³FM ideale mehrsprachige und domänenübergreifende Berichte erstellen. wenn nur 10% gekennzeichneter Daten für das Training verwendet werden, kann M³FM bei CXR-zu-Englisch-, CT-zu-Chinesisch- und CT-zu-Englisch-Aufgaben 6%, 8% und 8% höher sein als bei der vollständig überwachten Methode R2Gen; Bei Verwendung vollständiger Trainingsdaten kann M³FM R2Gen in drei Aufgaben um mehr als 20% und um 12%, 10% und 8% höher verbessern als XProNet.Dies zeigt das Potenzial von M³FM, Kliniker von der zeit- und arbeitsintensiven Aufgabe des Berichtschreibens zu befreien.
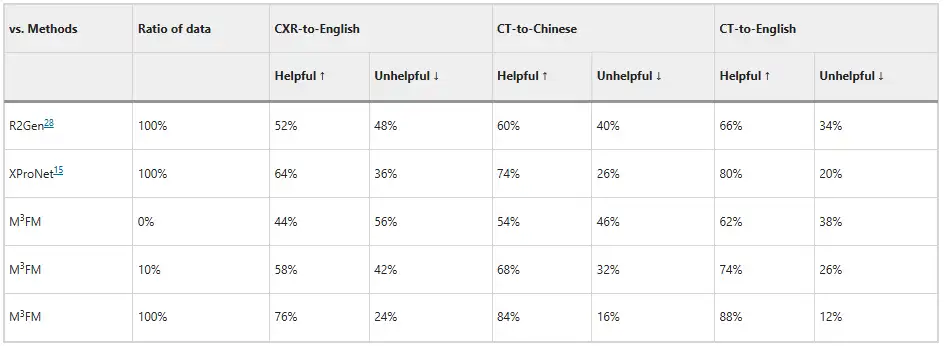
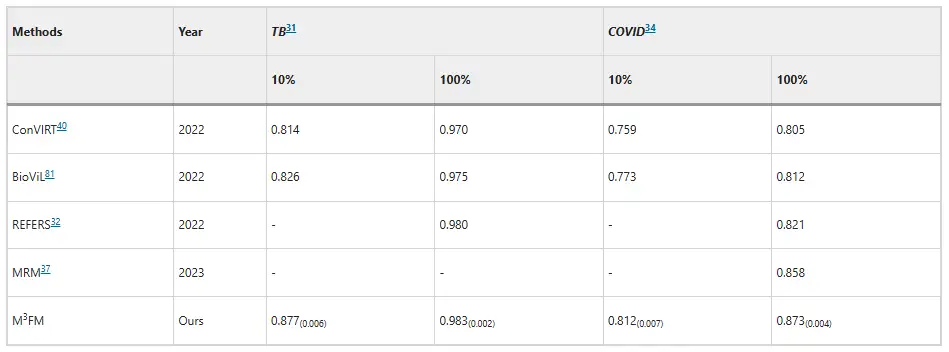
In Bezug auf die Krankheitsklassifizierung hat sich M³FM bei der Diagnose von Infektionskrankheiten als überlegen erwiesen.Im Shenzhen-Tuberkulose-Datensatz und im COVID-CXR-Datensatz liegen die AUC-Werte von M³FM bei Verwendung von 10% Trainingsdaten um 5,1% bzw. 3,9% über den bestehenden Bestergebnissen. Bei vollständiger Nutzung der Trainingsdaten erzielte M³FM bei zwei Infektionskrankheiten die besten Ergebnisse. In Bezug auf nicht übertragbare Krankheiten stammte der Datensatz von NIH ChestX-ray, und M³FM erzielte vergleichbare Ergebnisse mit der vollständig überwachten Methode Model Genesis mit nur 1% Trainingslabels. Bei 10% übertraf M³FM die Basismethoden MRM und REFERS bei der Diagnose mehrerer Krankheiten, was auch die Wirksamkeit und Generalisierungsfähigkeit von M³FM bei der Krankheitsdiagnose bestätigte.
KI führt intelligente Gesundheitsversorgung an, und das Team von Zheng Yefeng übernimmt die Führung
In der Vergangenheit haben sich viele Labore mit diesem Thema befasst und die von ihnen vorgeschlagenen Modelle weisen unterschiedliche Schwerpunkte und Vorteile auf.
Zur automatischen Berichterstellung hat beispielsweise die School of Information Science and Technology der Dalian Maritime University im Fachforum Medical Image Analysis im Bereich der medizinischen und biologischen Bildanalyse eine Forschungsarbeit mit dem Titel „DACG: Dual Attention and Context Guidance model for radiology report generation“ veröffentlicht. In dem Artikel wurde ein Modell mit dualer Aufmerksamkeits- und Kontextführung (DACG) für die automatische Erstellung von Radiologieberichten vorgeschlagen, das die Verzerrung durch visuelle und Textdaten verringern und die Erstellung langer Texte fördern kann.
Papieradresse:
https://www.sciencedirect.com/science/article/abs/pii/S1361841524003025
Es gibt auch Modelle, die für mehrere Sprachen konzipiert sind. Beispielsweise hat das Team von Professor Wang Yanfeng und Professor Xie Weidi von der Shanghai Jiao Tong University ein mehrsprachiges medizinisches Korpus MMedC mit 25,5 Milliarden Token erstellt, einen mehrsprachigen medizinischen Frage-und-Antwort-Bewertungsstandard MMedBench entwickelt, der 6 Sprachen abdeckt, und ein 8B-Basismodell MMed-Llama 3 erstellt, das in mehreren Benchmarktests bestehende Open-Source-Modelle übertraf und für medizinische Anwendungsszenarien besser geeignet ist. Die entsprechenden Forschungsergebnisse wurden in Nature Communications unter dem Titel „Towards building multilingual language model for medicine“ veröffentlicht.
KlickenÜberprüfenAusführlicher Bericht: Der Benchmark-Test im medizinischen Bereich übertrifft Llama 3 und liegt nahe an GPT-4. Das Team der Shanghai Jiaotong University veröffentlichte ein mehrsprachiges medizinisches Modell, das 6 Sprachen umfasst
Im Vergleich dazu wird die herausragende Leistung von M³FM in den Bereichen Multimodalität, Multidomänen, Mehrsprachigkeit und anderen Aspekten der Schnittstelle zwischen künstlicher Intelligenz und Gesundheitswesen zweifellos neue Vitalität verleihen.Wenn wir über diese Forschung sprechen, müssen wir natürlich Dr. Zheng Yefeng erwähnen, einen der Autoren dieses Artikels.
Tatsächlich kann man sagen, dass dieses Papier ein frisch erstelltes Ergebnis darstellt und es kann auch als Zeichen eines Neuanfangs für Dr. Zheng Yefeng gesehen werden. Am 29. Juli 2024 kam der IEEE Fellow, AIMBE Fellow und Medizin-Künstliche-Intelligenz-Wissenschaftler Zheng Yefeng in Vollzeit an die Westlake University, wurde als Professor an der School of Engineering eingestellt und gründete das Medical Artificial Intelligence Laboratory. Zu den Forschungsschwerpunkten des Labors gehören medizinische Bildanalyse, medizinisches natürliches Sprachverständnis, Bioinformatik usw. Dieser Artikel ist eine der wichtigsten Errungenschaften des ersten Jahres des Labors.
Neben dieser Errungenschaft hat das Labor auch mehrere Artikel im Bereich der medizinischen Gesundheit veröffentlicht, wie etwa die Studie mit dem Titel „Unlocking the Potential of Weakly Labeled Data: A Co-Evolutionary Learning Framework for Anomaly Detection and Report Generation“, die ein Framework zur kollaborativen Anomalieerkennung und Berichterstellung (CoE-DG) vorstellt, das vollständig und schwach gekennzeichnete Daten verwendet, um die gemeinsame Entwicklung der beiden Aufgaben der CXR-Anomalienerkennung und Berichterstellung zu fördern. Diese Forschungsergebnisse wurden in IEEE Transactions on Medical Imaging veröffentlicht.
Natürlich verfügt das Labor auch über Forschungsergebnisse zu den derzeit beliebten großen Sprachmodellen, wie beispielsweise die Studie mit dem Titel „Mitigating Hallucinations of Large Language Models in Medical Information Extraction via Contrastive Decoding“, die in EMNLP 2024 veröffentlicht wurde. Dieses Papier bietet eine Lösung für das Phänomen, dass LLMs in medizinischen Szenarien anfällig für „Halluzinationen“ sind, und schlägt eine „Alternate Contrastive Decoding“ (ALCD) vor. Mit dieser Methode können Sie das Auftreten von Fehlern erheblich reduzieren, indem Sie die Erkennungs- und Klassifizierungsfunktionen des Modells trennen und die Gewichte der beiden während des Vorhersageprozesses dynamisch anpassen. Die Technologie eignet sich gut für zahlreiche medizinische Aufgaben.
Heute befinden sich diese Errungenschaften entweder noch im Labor oder müssen erst noch umgesetzt werden, aber letztendlich wird KI den Gesundheitsbereich in Richtung Intelligenz, Intelligenz und Automatisierung führen. Dr. Zheng Yefeng sagte dazu: „Die medizinische künstliche Intelligenz ist ein sich rasant entwickelndes Feld. Ich schätze, dass künstliche Intelligenz in 10 bis 15 Jahren über die Genauigkeit von ärztlichen Diagnosen und Behandlungen verfügen und breit eingesetzt werden kann.“
Quellen:
1.https://www.nature.com/articles/s41746-024-01339-7
2.https://mp.weixin.qq.com/s/pMNXAvzgGRpPwqVtCWjXbA
3.https://mp.weixin.qq.com/s/6hw6EJY6slAIRbGGN9XY9g








