Command Palette
Search for a command to run...
Möglicherweise Werden Neue Krebsbehandlungen Entwickelt! Die Duke University Nutzt PepPrCLIP Zur Lösung Des Problems Der „nicht Medikamentös Behandelbaren“ Krankheit
Im Jahr 2021 veröffentlichte OpenAI das revolutionäre CLIP-Modell (Contrastive Language-Image Pre-training). Durch unüberwachtes Lernen kann CLIP die Beziehung zwischen Bildern und Text effektiv verstehen und zuordnen, ohne dass zusätzliche Anmerkungsinformationen erforderlich sind.
Einige Jahre später ließ sich eine Gruppe biomedizinischer Wissenschaftler davon inspirieren: Da CLIP Bilder und Sprache abgleicht, kann dieselbe Idee auch zum Abgleichen von Peptiden und Proteinen verwendet werden?
Indem wir auf die bahnbrechende Forschung von OpenAI zur Generierung realistischer Bilder durch kontrastives Sprach-Bild-Vortraining zurückgreifen,Ein Forschungsteam der Abteilung für Biomedizintechnik der Duke University hat die PepPrCLIP-Pipeline (Peptide Prioritization by CLIP) entwickelt, mit der kurze Proteine (Peptide) entwickelt werden können, die an bisher nicht medikamentös behandelbare krankheitsverursachende Proteine binden und diese zerstören können.Im Vergleich zu RFDiffusion, einer bestehenden Plattform, die Peptide mithilfe einer 3D-Zielstruktur generiert, ist PepPrCLIP schneller und erstellt Peptide, die fast immer besser mit dem Zielprotein übereinstimmen. Die Forscher haben außerdem durch Experimente bestätigt, dass die von PepPrCLIP ausgewählten „Leitpeptide“ eine robuste und überlegene gezielte Bindung und Regulierung erreichen können, wenn sie als inhibitorische Peptide in vitro verwendet oder mit der E3-Ubiquitinligasedomäne fusioniert werden.
Die entsprechenden Ergebnisse wurden im Januar dieses Jahres in Science Advances unter dem Titel „De novo design of peptide binders to conformationally diverse targets with contrastive language modeling“ veröffentlicht.
Papieradresse:
https://www.science.org/doi/10.1126/sciadv.adr8638
Downloadadresse für zugehörige Datensätze:
https://go.hyper.ai/AT5m9
Das Open-Source-Projekt „awesome-ai4s“ vereint mehr als 200 AI4S-Papierinterpretationen und stellt umfangreiche Datensätze und Tools bereit:
https://github.com/hyperai/awesome-ai4s
Ein neuer Ansatz zur Lösung des „nicht medikamentös behandelbaren“ Problems
Ein Ansatz zur Behandlung der Krankheit besteht in der Entwicklung von Medikamenten, die gezielt die Proteine angreifen und zerstören, die die Krankheit verursachen. Manchmal haben diese Schlüsselproteine genau definierte Strukturen, wie sorgfältig gefaltete Papierkraniche, sodass herkömmliche niedermolekulare Therapeutika leicht an sie binden können.
Das pathogene Protein von mehr als 80% gleicht jedoch eher einem „wirren Durcheinander“, ungeordnet und ineinander verknotet, was es für Standardtherapien fast unmöglich macht, Bindungsstellen auf seiner Oberfläche zu finden und zu wirken. Der Begriff „undruggable“ (nicht medikamentös behandelbar) wird in der traditionellen Arzneimittelentwicklung häufig verwendet, um Proteine zu beschreiben, die aufgrund ihrer strukturellen und funktionellen Eigenschaften nur schwer als Angriffspunkte für Medikamente infrage kommen.
Laut öffentlich zugänglichen Informationen weisen schwer medikamentös behandelbare Zielmoleküle häufig die folgenden Merkmale auf:
* haben eine ausgedehnte und flache funktionelle Schnittstelle, der eine gut definierte Ligandenbindungstasche fehlt;
* Fehlen spezifischer Liganden, die die Funktion des Zielproteins ermöglichen;
* Das Ziel ist ein Krankheitshemmer, der ein Medikament zur Aktivierung der Proteinaktivität benötigt, was die Arzneimittelentwicklung schwieriger macht;
* Nicht medikamentös behandelbare Ziele haben oft komplexe physiologische Funktionen, was die Entwicklung und den Entwurf von Medikamenten erschwert.
* Grenzen von Strategien zur Arzneimittelentwicklung.
Um diese Probleme zu umgehen, haben viele Forscher untersucht, wie man Peptide einsetzen kann, um an krankheitsverursachende Proteine zu binden und diese abzubauen. Da Peptide Miniversionen von Proteinen sind, benötigen sie keine Oberflächentaschen zur Bindung. Stattdessen können Peptide an verschiedene Aminosäuresequenzen in Proteinen binden.
Aber auch diese Ansätze haben ihre Grenzen, da die vorhandenen handelsüblichen Bindemittel nicht dafür ausgelegt sind, an instabilen oder übermäßig verwickelten Proteinstrukturen zu haften. Während Wissenschaftler intensiv an der Entwicklung neuer Bindungsproteine arbeiten, beruhen diese Ansätze noch immer auf der Kartierung der 3D-Strukturinformationen des Zielproteins, die für ungeordnete Ziele nicht verfügbar sind.
Das in diesem Artikel vorgestellte Forschungsteam der Abteilung für Biomedizintechnik der Duke University verfolgte einen anderen Ansatz. Anstatt zu versuchen, die Struktur pathogener Proteine abzubilden, ließen sie sich von großen Sprachmodellen (LLMs) inspirieren und konstruierten PepPrCLIP. Seine erste Komponente, PepPr, verwendet einen generativen Algorithmus, der anhand einer großen Bibliothek natürlicher Proteinsequenzen trainiert wurde, um neue „Leitproteine“ mit spezifischen Merkmalen zu entwerfen. Die zweite Komponente, CLIP, verwendet ein ursprünglich von OpenAI entwickeltes algorithmisches Framework, um zu testen und zu prüfen, ob diese Peptide mit dem Zielprotein übereinstimmen.
Aufbau eines CLIP-basierten Peptidpriorisierungsprozesses - PepPrCLIP
Wie wurde PepPrCLIP konstruiert?
Kurz gesagt verwendeten die Forscher zunächst das Proteinsprachenmodell (pLM) ESM-2, um Gaußsche Rauschstörungen an den Einbettungen realer Peptidbindersequenzen durchzuführen und so Kandidatenpeptidsequenzen mit natürlichen Eigenschaften zu erzeugen. Anschließend wurden diese Kandidatenpeptide im latenten Raum mithilfe einer CLIP-basierten kontrastiven Lernarchitektur gescreent, um ein Modell zu trainieren, das komplementäre Peptid-Protein-Paare gemeinsam kodiert. Schließlich wurde in das konstruierte PepPrCLIP ein Rahmen zur Generationsunterscheidung integriert, um völlig neue Kandidatenpeptidsequenzen herauszufiltern, die in der Lage sind, an die Zielsequenz zu binden.
Die folgende Abbildung zeigt den spezifischen Prozess des PepPrCLIP-Modelltrainings:
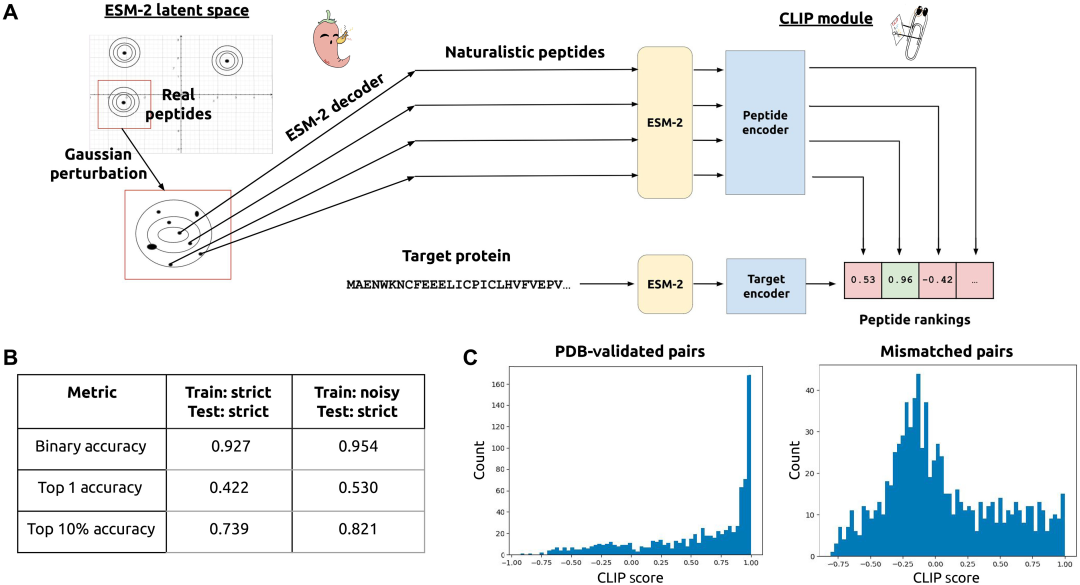
Wie in der Abbildung oben gezeigt, wird das in ESM-2 eingebettete natürliche Peptid abgetastet, um eine Gauß-Verteilung zu erzeugen, die dann wieder in eine Aminosäuresequenz dekodiert wird. Das trainierte CLIP-Modul kodiert gemeinsam die entsprechende Peptid-Protein-Einbettung, durchsucht Tausende von Peptiden und bewertet ihre spezifische Bindungsaktivität an das Ziel, insbesondere:
* CLIP-Architektur und -Schulung
Zunächst wird die Eingabesequenz durch ein eingefrorenes ESM-2-650M-Modell eingebettet, um eine Eingabeeinbettung zu erzeugen. Als nächstes wird die Eingabeeinbettung über die Sequenzlänge gemittelt, um einen Einbettungsvektor zu erhalten, der für Peptide und Proteine geeignet ist; Es werden h MLP-Schichten angewendet und der Einbettungsvektor wird mithilfe der Aktivierungsfunktion der gleichgerichteten linearen Einheit (ReLU) verarbeitet, um die Ausgabeeinbettung zu erhalten. Der CLIP-Score wird durch Berechnung eines Skalarprodukts zwischen den Peptid- und Protein-Vektoreinbettungen mit einem Wert zwischen -1 und 1 ermittelt. Das Modell wird so trainiert, dass Peptid-Protein-Bindungspaare hohe CLIP-Scores aufweisen.
* Generierung von Peptid-Kandidatensequenzen
Kandidatenpeptide werden aus allen Peptiden im Trainingssatz generiert, von denen jedes mithilfe des ESM-2-650M pLM in PyTorch eingebettet wird; für eine gegebene Peptideinbettung wird die Varianz aller Dimensionen der Einbettung berechnet; Für jeden Rest im Quellpeptid wird Rauschen aus einer Standardnormalverteilung abgetastet und mit der Varianz multipliziert, um eine Störung zu erzeugen, die zur Einbettung des entsprechenden Rests hinzugefügt wird. Zum Zeitpunkt der Inferenz wurden Quellpeptide zufällig aus dem Trainingssatz ausgewählt und für jedes Quellpeptid wurden mithilfe der oben beschriebenen Rauschmethode 1.000 Peptide generiert. Schließlich werden diese Peptide (ungefähr 100.000) in das CLIP-Modell eingespeist und basierend auf der vorhergesagten Bindung an die vom Benutzer bereitgestellte Zielsequenz bewertet.
In Computersimulationen verglichen die Forscher die Leistung von PepPrCLIP mit der von RFDiffusion. Die Forscher verglichen die ipTM-Werte der von PepPrCLIP und denen von RFDiffusion generierten Peptide und stellten fest, dass PepPrCLIP bei Peptiden mit 33% am Ziel RFDiffusion übertraf, wie in der Abbildung unten dargestellt. Darüber hinaus kann PepPrCLIP durch die alleinige Verwendung von Sequenzeinbettungen die Geschwindigkeit der Generierung und Priorisierung erheblich steigern, indem es etwa 1.000 Peptide pro Minute generiert und 100.000 Peptide pro Proteinziel in etwa 1 Minute bewertet; Im Vergleich dazu benötigt RFDiffusion etwa 2 Minuten, um ein einzelnes Bindemittel zu entwerfen.Diese Effizienz macht PepPrCLIP besonders vorteilhaft für das Screening großer Peptidbibliotheken, mit oder ohne Strukturinformationen.
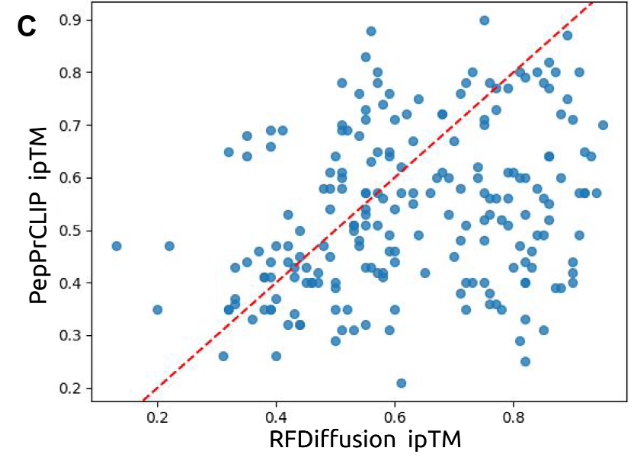
Um die Auswirkungen von PepPrCLIP auf geordnete und ungeordnete Proteinziele weiter zu untersuchen, arbeitete das Forschungsteam auch mit Forschungsteams der Duke University School of Medicine, der Cornell University und des Sanford Burnham Prebys Medical Discovery Institute zusammen, um die Plattform experimentell zu testen.
In den ersten Tests zeigte das Forschungsteam, dassVon PepPrCLIP erzeugte Peptide können effektiv an UltraID, ein relativ einfaches und stabiles Enzymprotein, binden und dessen Aktivität hemmen.
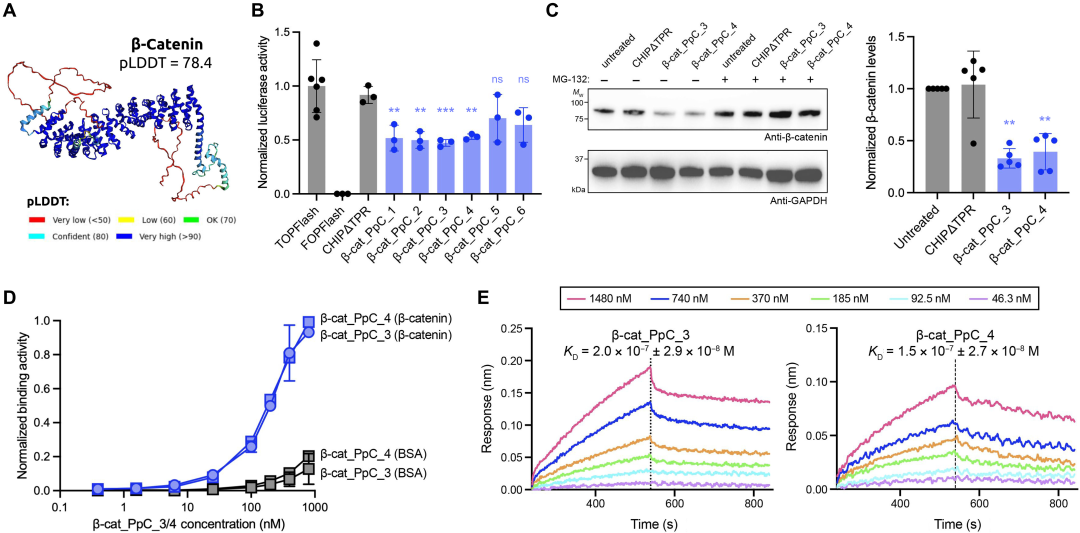
Anschließend verwendeten sie PepPrCLIP, um Peptide zu entwerfen, die an β-Catenin binden können, ein ungeordnetes, komplexes Protein, das an der Signalübertragung bei mehreren verschiedenen Krebsarten beteiligt ist. Wie in der Abbildung unten gezeigt, erzeugte das Team sechs Peptide, die laut CLIP an Proteine binden konnten, und zeigte, dass vier von ihnen effektiv an ihre Ziele binden und diese abbauen konnten. Durch die Zerstörung von Proteinen können sie die Signalübertragung von Krebszellen verlangsamen.
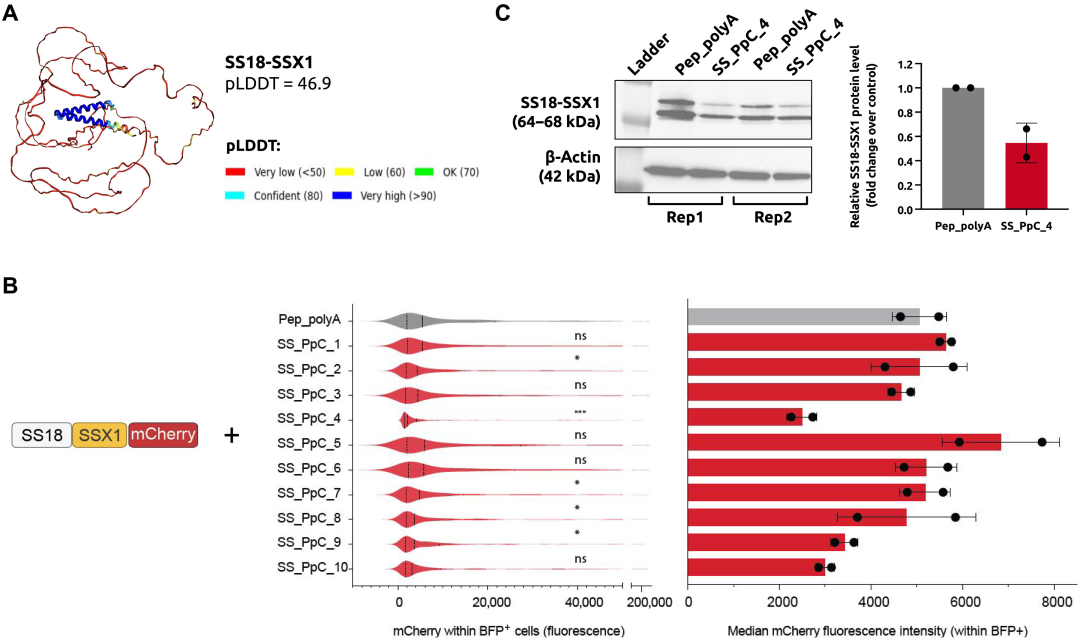
Im komplexesten Test entwickelte das Team Peptide, die an stark ungeordnete Proteine binden können, die mit dem Synovialsarkom in Zusammenhang stehen, einer seltenen, bösartigen Krebsart, die 51 bis 101 TP3T aller Weichteiltumoren ausmacht. Synovialsarkom entwickelt sich in Weichteilen und betrifft hauptsächlich Kinder und junge Erwachsene. Die Krankheit ist durch das Vorhandensein eines einzigartigen, stark ungeordneten onkogenen Fusionsproteins, SS18-SSX, gekennzeichnet.
Das Team brachte die Peptide in Synovialsarkomzellen ein und testete 10 Designs. Wie die Ergebnisse unten zeigen, reduzierte SS_PpC_4 unter den Peptiden, von denen PepPrCLIP vorhersagt, dass sie an SS18-SSX1 binden, die Fluoreszenz von SS18-SSX1-mCherry signifikant. Als nächstes testeten die Forscher auch die Wirkung der Überexpression von SS_PpC_4 auf den Spiegel des endogenen SS18-SSX1-Fusionsproteins. Es ist erwähnenswert, dass die Überexpression des SS_PpC_4-Peptids die SS18-SSX1-Proteinspiegel signifikant reduzierte (>40%).
Mit anderen Worten:PepPrCLIP entwickelt Peptide, die sowohl an Proteine binden als auch diese abbauen können.Wenn es ihnen gelingt, das Protein zu zerstören, haben Forscher die Möglichkeit, eine Therapie für Krebserkrankungen zu entwickeln, die bislang nicht medikamentös behandelbar waren, was viele spannende klinische Möglichkeiten eröffnet.
KI liefert neue Werkzeuge zur Überwindung „nicht behandelbarer“ Krankheiten
Die neueste Übersichtsarbeit, die im September 2023 in einer Fachzeitschrift der Zeitschrift Nature veröffentlicht wurde, stellt umfassend die neuesten Fortschritte bei der Arzneimittelforschung für „nicht medikamentös behandelbare“ Proteine und deren klinische Anwendung vor.Verschiedene Moleküle mit ähnlichen, nicht medikamentös behandelbaren Eigenschaften wurden in die folgenden Kategorien eingeteilt:
① Kleine GTPasen: wie Proteine der RAS-Familie, einschließlich KRAS, HRAS und NRAS, die aufgrund des Fehlens anzielbarer Taschen auf ihrer Oberfläche als nicht medikamentös behandelbar gelten;
2 Phosphatase: Da jede Phosphatase viele strukturelle Ähnlichkeiten aufweist, ist ihre Selektivität gering und es treten unvermeidbare Nebenwirkungen auf, was den Fortschritt der Arzneimittelforschung erheblich behindert.
③ Transkriptionsfaktoren (TFs): Zahlreiche menschliche Erkrankungen stehen mit einer Fehlregulation von TFs in Zusammenhang, die an zahlreichen biologischen Prozessen beteiligt sind. Die meisten dieser TFs können aufgrund ihrer strukturellen Heterogenität und des Fehlens verarbeitbarer Bindungsstellen nicht mit herkömmlichen kleinen Molekülen angesteuert werden.
④ Epigenetische Ziele: Epigenetische Ziele spielen eine entscheidende Rolle bei der Regulierung von Genexpressionsmustern und wirken sich auf verschiedene biologische Prozesse und Krankheiten aus.
⑤ Andere Proteine: Protein-Protein-Interaktionen (PPIs) und ihre Netzwerke sind von großer Bedeutung für biologische Prozesse und die Zellzyklusregulation. Einige PPIs mit flachen Interaktionsoberflächen sind schwieriger anzusteuern als andere PPIs, was sie bis zu einem gewissen Grad „nicht medikamentös behandelbar“ macht.
Angesichts der sogenannten „medikamentös nicht behandelbaren“ Ziele hat die akademische Gemeinschaft heutzutage Dutzende innovativer Methoden entwickelt. Entsprechend dem Mechanismus nicht medikamentös behandelbarer Proteine haben sie modernste Technologien wie die fragmentbasierte Arzneimittelforschung (FBDD), das computergestützte Arzneimitteldesign (CADD), das virtuelle Screening (VS), DNA-kodierte Bibliotheken (DEL) usw. übernommen, um eine systematische Strategie für das Arzneimitteldesign zu entwickeln. Heute stehen durch die Entwicklung künstlicher Intelligenz und die Entstehung großer Proteinsprachenmodelle neue Werkzeuge zur Überwindung dieses Problems zur Verfügung.In den letzten Jahren gab es sowohl in der Industrie als auch in der Wissenschaft wichtige Durchbrüche.
Industrie,Im Dezember 2023 gab die Absci Corporation, ein führendes Unternehmen in der generativen KI-Antikörperforschung, eine Zusammenarbeit mit AstraZeneca bekannt, um KI-basierte Antikörper gegen ein Tumorziel zu entwickeln. Die Zusammenarbeit kombiniert die Integrated Drug Creation-Plattform von Absci mit der onkologischen Expertise von AstraZeneca, um die Entdeckung potenzieller neuer Kandidaten für die Krebsbehandlung zu beschleunigen. Die Integrated Drug Creation-Plattform von Absci generiert proprietäre Daten durch die Messung von Millionen von Protein-Protein-Interaktionen. Diese werden verwendet, um die proprietären KI-Modelle von Absci zu trainieren und in nachfolgenden Iterationen mithilfe neuer KI-Modelle entwickelte Antikörper zu validieren. Die Plattform beschleunigt die Arzneimittelforschung, indem sie die Datenerfassung, das KI-gesteuerte Design und die Laborvalidierung in etwa 6 Wochen abschließt, und hat das Potenzial, das Spektrum der Arzneimittelziele zu erweitern, einschließlich der Entwicklung von Medikamenten für Ziele, die bislang als nicht medikamentös behandelbar galten.
Wissenschaft,Im Januar 2025 kombinierte eine gemeinsam vom führenden Pharmaunternehmen für künstliche Intelligenz (KI) Insilico Medicine und der University of Toronto in Kanada geleitete Studie Quantencomputermodelle mit klassischen Computermodellen und generativer künstlicher Intelligenz, um durch Training, Generierung und Screening riesiger Datensätze ein breiteres Spektrum chemischer Möglichkeiten zu erkunden. Dabei wurden neuartige Moleküle entdeckt, die auf das „nicht medikamentös behandelbare“ Krebstreiberprotein KRAS abzielen.
Die KRAS-Mutation ist eine der häufigsten Mutationen bei Krebs und kommt bei etwa einem Viertel aller menschlichen Tumoren vor. Eine KRAS-Mutation kann zu unkontrollierter Zellvermehrung und damit zu Krebs führen. Um in dieser Studie potenzielle neue KRAS-Inhibitoren zu entwickeln, schlugen die Forscher ein quantenklassisches Hybrid-Framework-Modell vor, das ein quantenvariationales generatives Modell (QCBM) und ein Long Short-Term Memory Network (LSTM) kombiniert und Quantencomputing mit klassischen Computermethoden kombiniert, um neue Moleküle zu entwerfen. Diese Forschung wurde auch von mehreren Forschungseinrichtungen unterstützt, darunter dem St. Jude Children's Research Hospital. Die entsprechenden Forschungsergebnisse wurden in Nature Biotechnology unter dem Titel „Quantum-computing-enhanced algorithm unveils potential KRAS inhibitors“ veröffentlicht.

Mit den Durchbrüchen in den entsprechenden Technologien hat die Menschheit neue Vorstellungsräume und unbegrenzte Möglichkeiten zur Bekämpfung von Krankheiten geschaffen.