Command Palette
Search for a command to run...
Erleben Sie Die Demo Als Erster! Das Genombasierte Modell Evo Ist Auf Dem Cover Von Science Und Ermöglicht Vorhersagen Und Generierungen Von Der Molekularen Bis Zur Genomischen Ebene

In jüngster Zeit wurden im Bereich der Biologie große Fortschritte im Bereich der KI erzielt.Ein Forschungsteam der Stanford University und des Arc Institute in den USA hat ein genombasiertes Modell Evo vorgeschlagen, das bei multimodalen Aufgaben von DNA, RNA und Protein eine Nullprobenvorhersage und hochpräzise Generierung erreichen kann.
Die entsprechende Forschung wurde in Science als Titelartikel mit dem Titel „Sequence modeling and design from molecular to genome scale with Evo“ veröffentlicht.
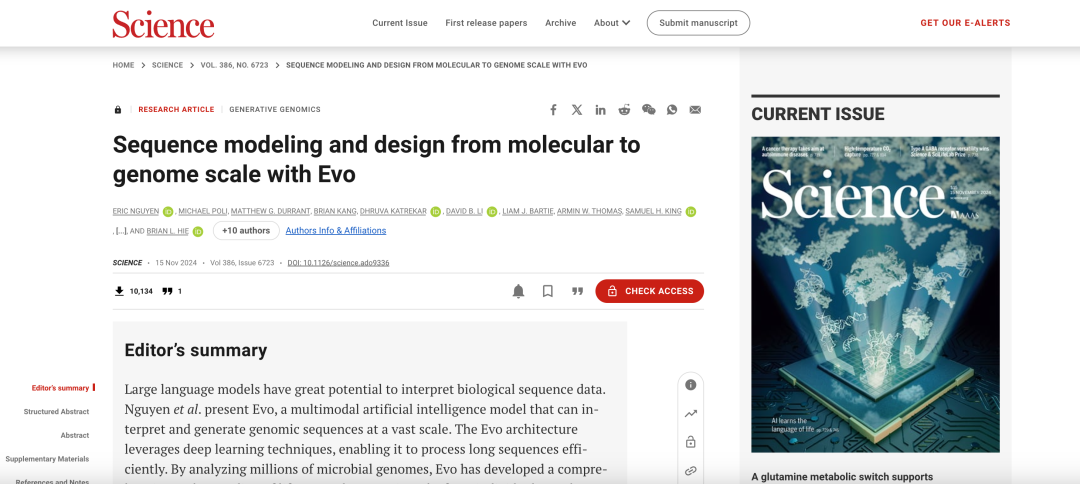
Papieradresse:
https://www.science.org/doi/10.1126/science.ado9336
Folgen Sie dem offiziellen Konto und antworten Sie mit „Evo“, um das vollständige PDF zu erhalten
Als Erstautor des Artikels veröffentlichte Eric Nguyen mehrere Updates, um die Erfolge von Evo vorzustellen, dankte den Teammitgliedern mehrfach und sagte offen: „Es ist eine Ehre, mit einem so großartigen Team zusammenzuarbeiten!“

Der Zeitung zufolge Evo verwendet die StripedHyena-Architektur und wird anhand eines großen Genomdatensatzes trainiert, der mehr als 80.000 Bakterien- und Archaeengenome enthält.und Millionen vorhergesagter Phagen- und Plasmidsequenzen, die 300 Milliarden Nukleotid-Token abdecken und in der Lage sind, DNA-Sequenzen mit einer angemessenen Genomarchitektur von mehr als 1 Megabase Länge zu erzeugen.
Auch,Mit einer Parameterskala von 7 Milliarden und einer maximalen Kontextlänge von 131.072 Token kann Evo die komplizierte Koevolution zwischen codierenden und nichtcodierenden Sequenzen aufdecken und komplexe biologische Systeme entwerfen.Wie CRISPR-Cas-Komplexe und IS200- und IS605-Transposons.
Zusammenfassend lässt sich sagen, dass Evos Fähigkeit, ganze Genomsequenzen vorherzusagen, zu generieren und zu entwerfen, nicht nur neue theoretische Unterstützung für die Biowissenschaften bietet, sondern voraussichtlich auch in der Genomeditierung, Arzneimittelforschung, Krankheitsdiagnose, Landwirtschaft und anderen Bereichen Anwendung finden wird und so dazu beiträgt, in vielen Bereichen bahnbrechende Ergebnisse zu erzielen.
Viele Internetnutzer waren von der Veröffentlichung von Evo schockiert und freuten sich auf die konkrete Anwendung dieses Modells.
Damit jeder die leistungsstarken Funktionen des Evo-Modells zum ersten Mal erleben kann,Der Abschnitt „HyperNeural-Tutorial“ von HyperAI ist jetzt online: „Evo: Vorhersage und Generierung vom molekularen bis zum genomischen Maßstab“.Sie müssen keine Befehle eingeben, sondern können einfach mit einem Klick klonen – für ein schnelles Erlebnis!
Link zum Tutorial:
Demolauf
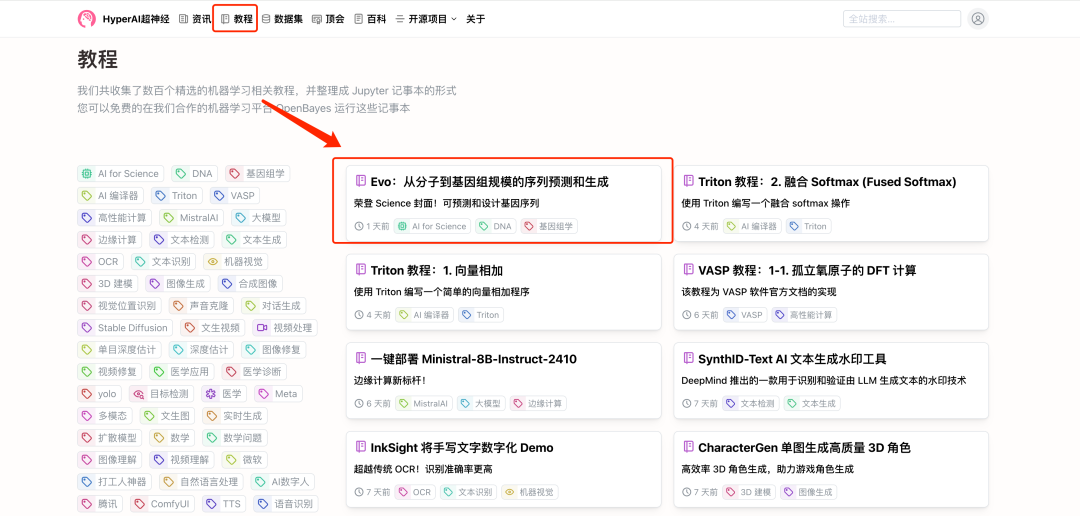
1. Melden Sie sich bei hyper.ai an, wählen Sie auf der Seite „Tutorials“ „Evo: Vorhersage und Generierung vom molekularen bis zum genomischen Maßstab“ und klicken Sie auf „Dieses Tutorial online ausführen“.
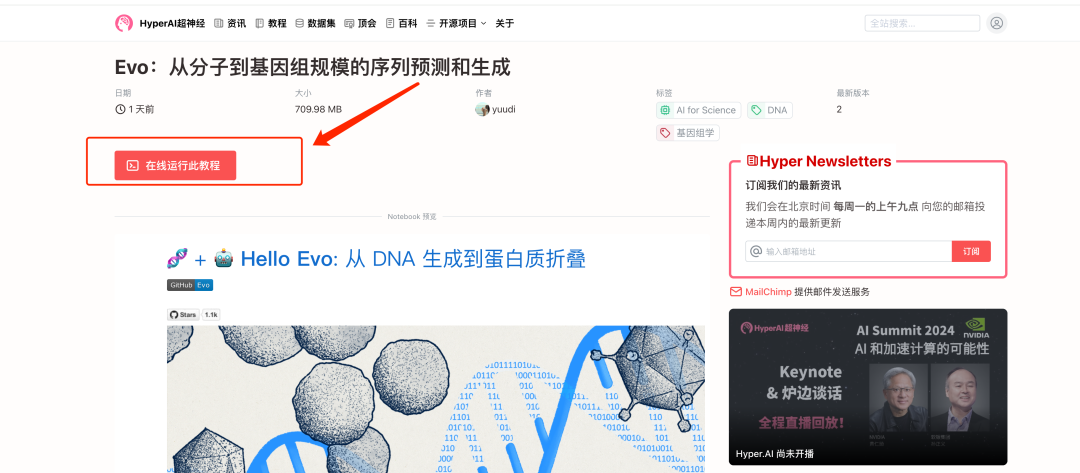
2. Klicken Sie nach dem Seitensprung oben rechts auf „Klonen“, um das Tutorial in Ihren eigenen Container zu klonen.
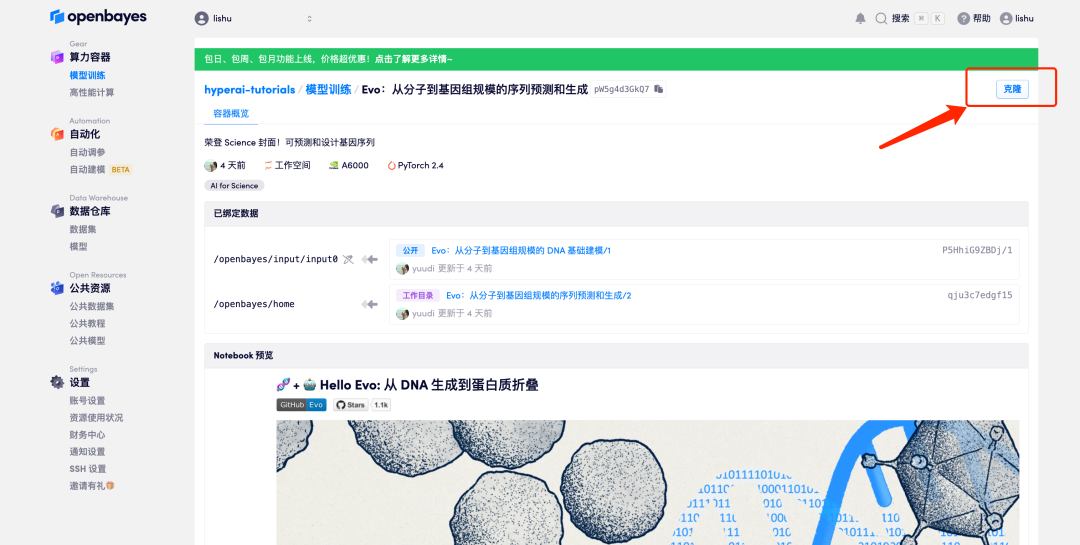
3. Klicken Sie unten rechts auf „Weiter: Hashrate auswählen“.
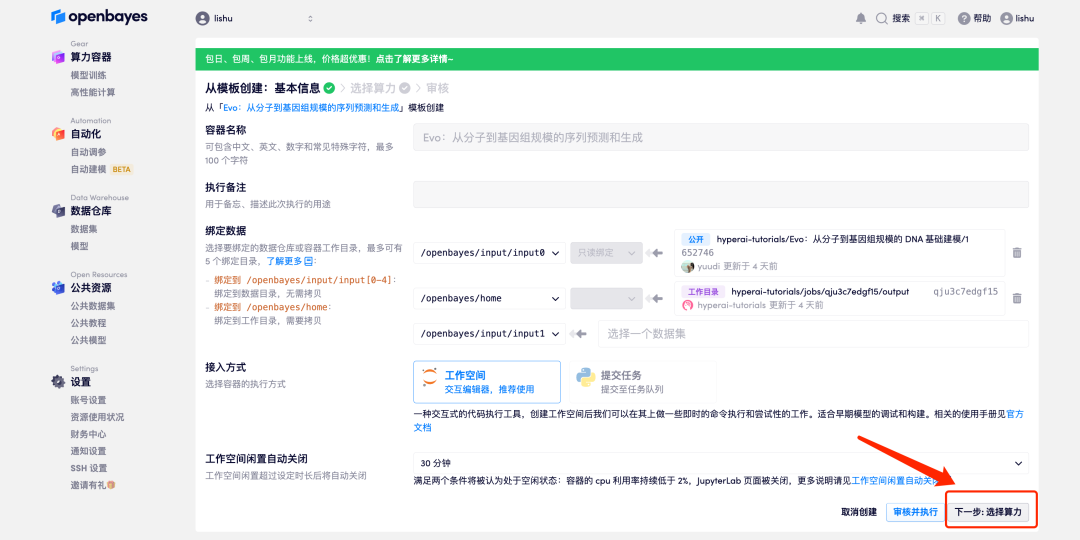
4. Wählen Sie nach dem Seitenwechsel „NVIDIA RTX A6000“ und dann je nach Bedarf „Pay as you go“ oder „Tages-/Wochen-/Monatspaket“. Nachdem Sie das Bild „PyTorch“ ausgewählt haben, klicken Sie auf „Weiter: Überprüfen“. Neue Benutzer können sich über den unten stehenden Einladungslink registrieren, um 4 Stunden RTX 4090 + 5 Stunden CPU-freie Zeit zu erhalten!
Exklusiver Einladungslink von HyperAI (kopieren und im Browser öffnen):
https://openbayes.com/console/signup?r=Ada0322_QZy7
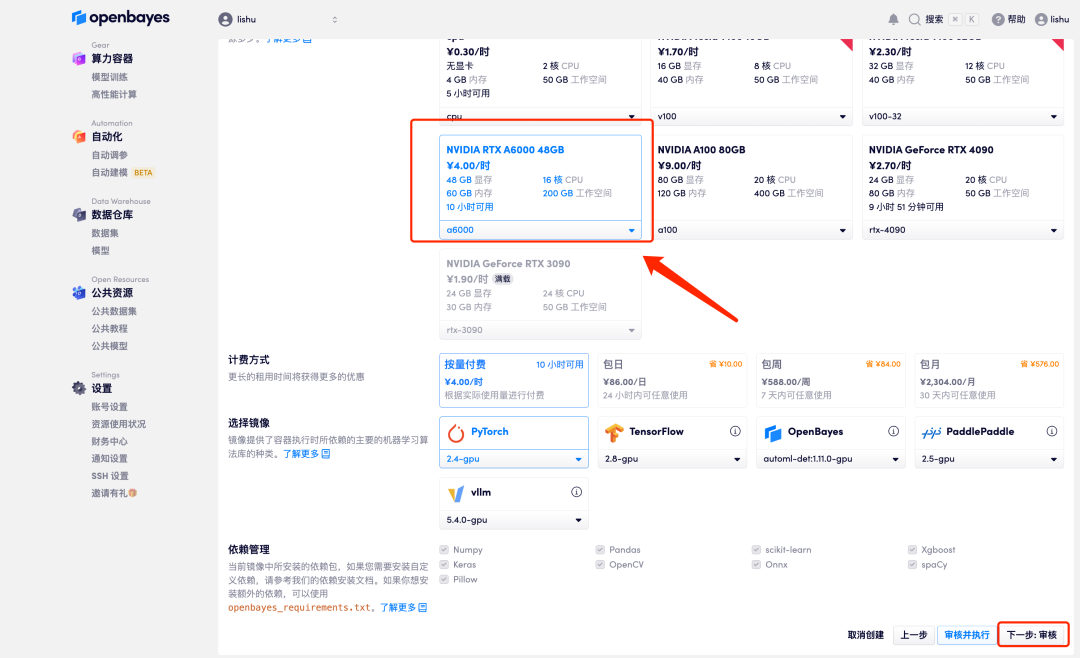
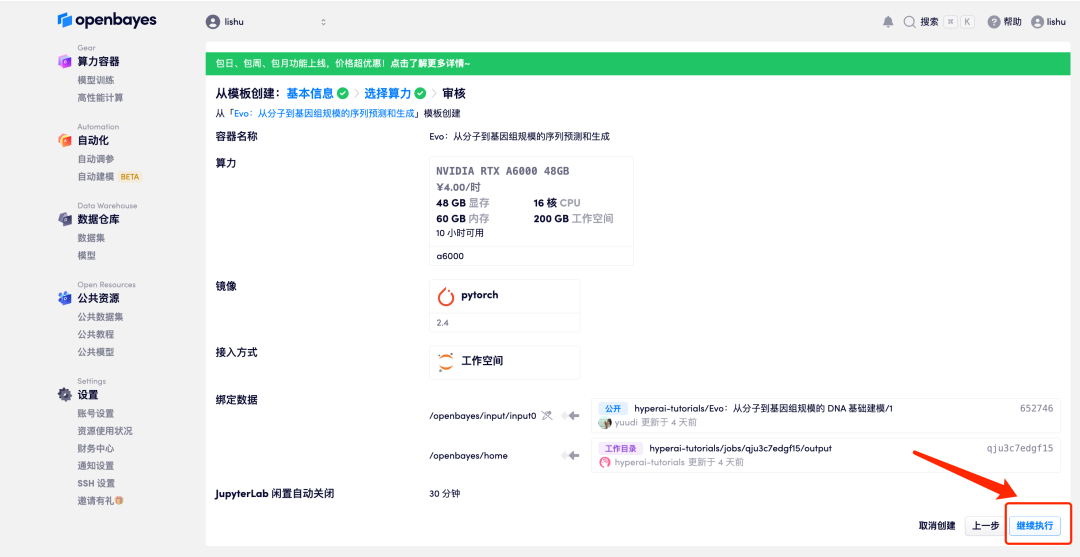
5. Klicken Sie nach der Bestätigung auf „Weiter“ und warten Sie, bis die Ressourcen zugewiesen wurden. Der erste Klonvorgang dauert etwa 2 Minuten.
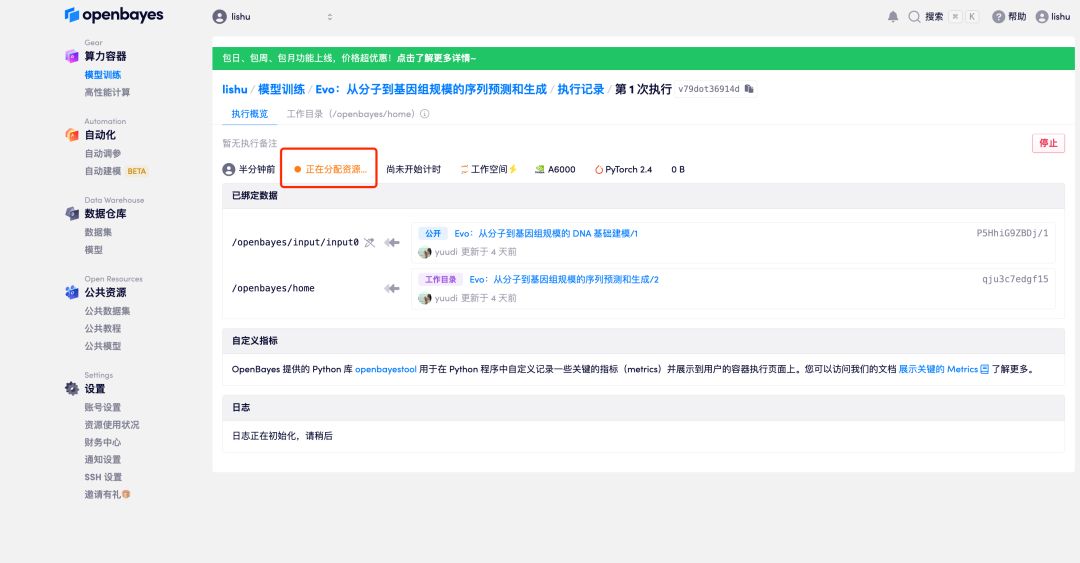
6. Wenn sich der Status in „Wird ausgeführt“ ändert, klicken Sie auf die Option „Arbeitsbereich öffnen“.
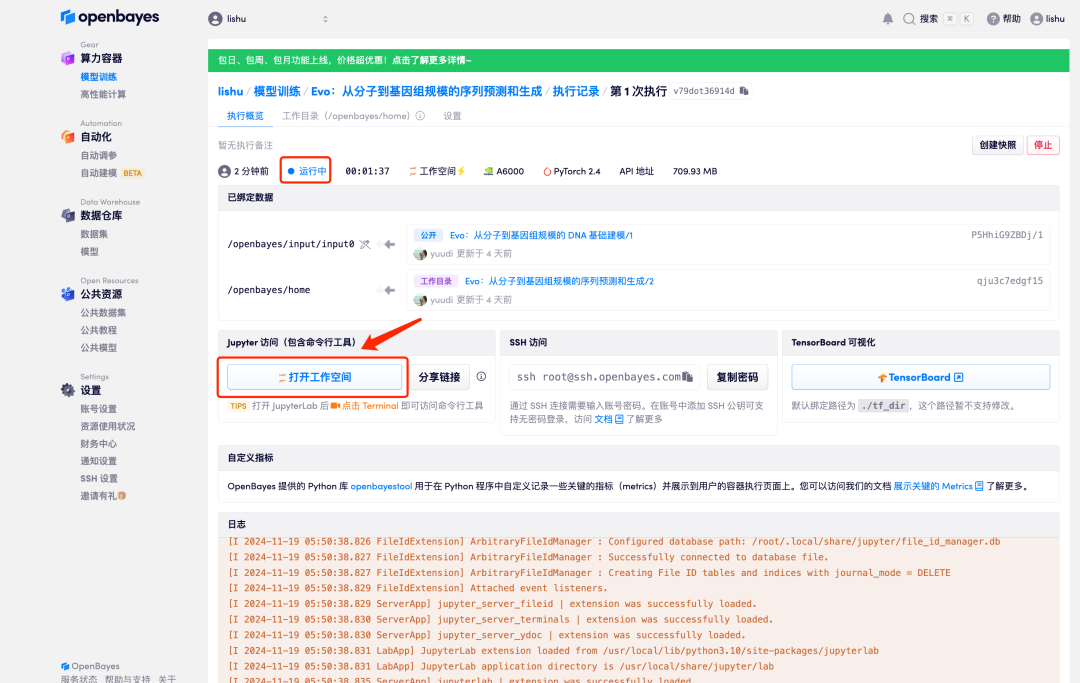
7. Doppelklicken Sie nach dem Aufrufen des Jupyter-Arbeitsbereichs auf die Datei "README", um die Seite zum Ausführen des Evo-Modells offiziell aufzurufen.
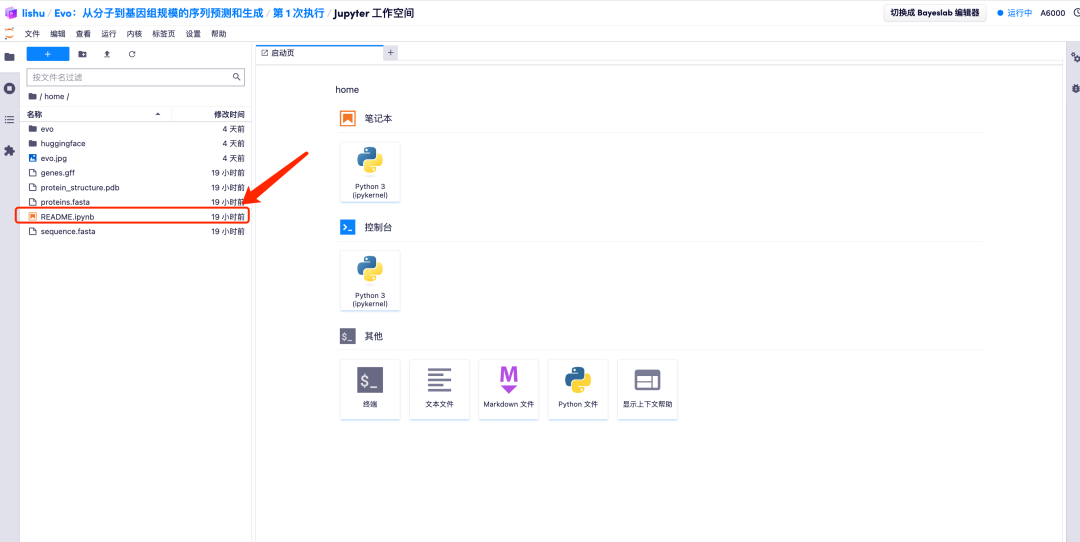
Effektdemonstration
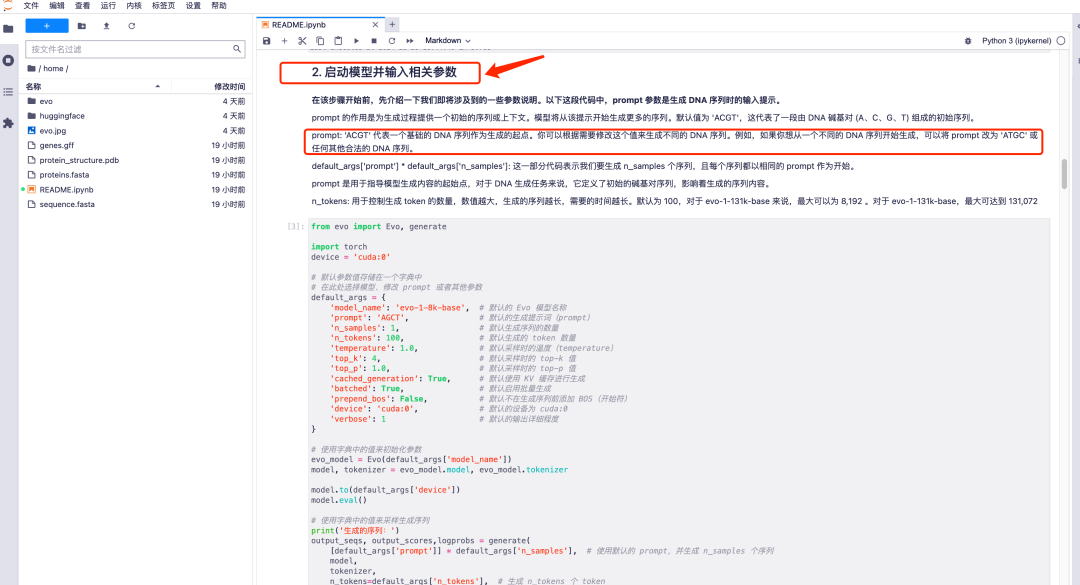
1. Nachdem Sie die Evo-Modell-Laufseite aufgerufen haben, befinden sich alle Parameter im Standardmodus. Scrollen Sie nach unten zu „2. Starten Sie das Modell und geben Sie relevante Parameter ein“ und passen Sie die Eingabeaufforderungsparameterwerte entsprechend Ihren Anforderungen an.Es ist zu beachten, dass der Standardwert der Eingabeaufforderung „ACGT“ ist, was eine Anfangssequenz darstellt, die aus DNA-Basenpaaren (A, C, G, T) besteht.Sie können diesen Wert ändern, um bei Bedarf unterschiedliche DNA-Sequenzen zu generieren.
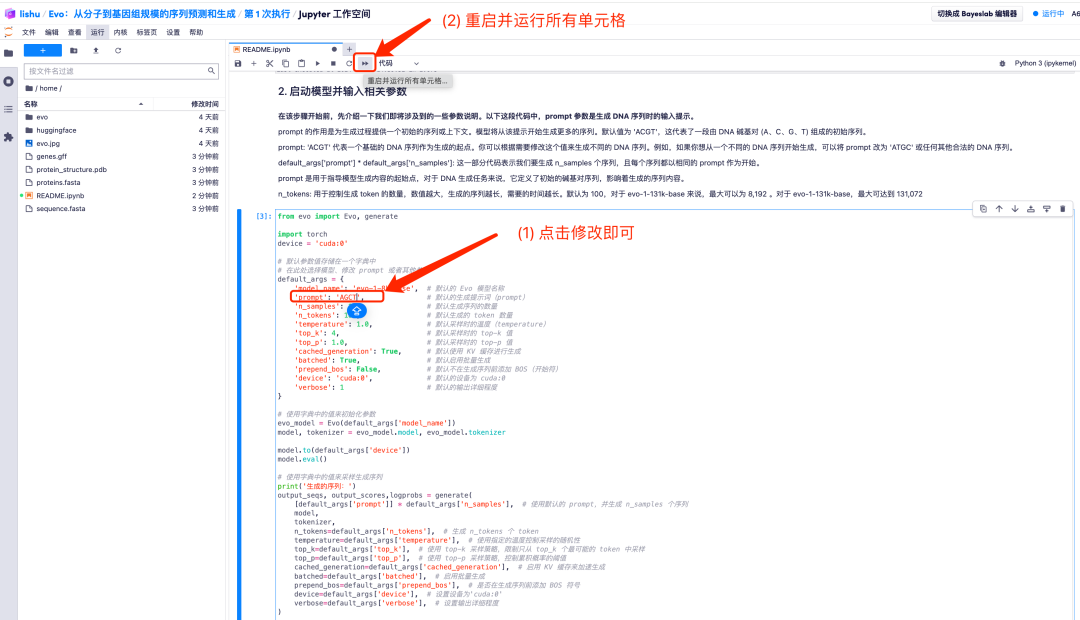
2. Ändern Sie beispielsweise den Standardwert der Eingabeaufforderung in AGCT. Nachdem Sie die Standardparameterwerte angepasst haben, klicken Sie auf die Option „Neu starten und alle Zellen ausführen“ und wählen Sie zum Ausführen „Neu starten“ aus.
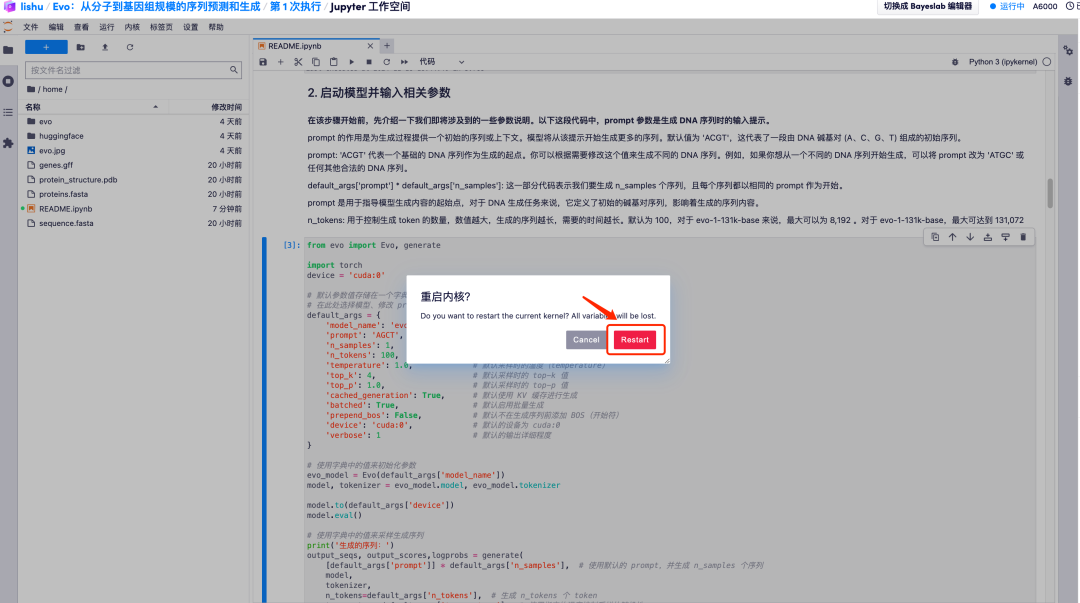
3. Warten Sie einen Moment. Wenn sich das Symbol [*] in eine Zahl ändert, ist der Vorgang abgeschlossen. Unten bei „2. Starten Sie das Modell und geben Sie relevante Parameter ein“ sehen Sie die generierte Sequenz.
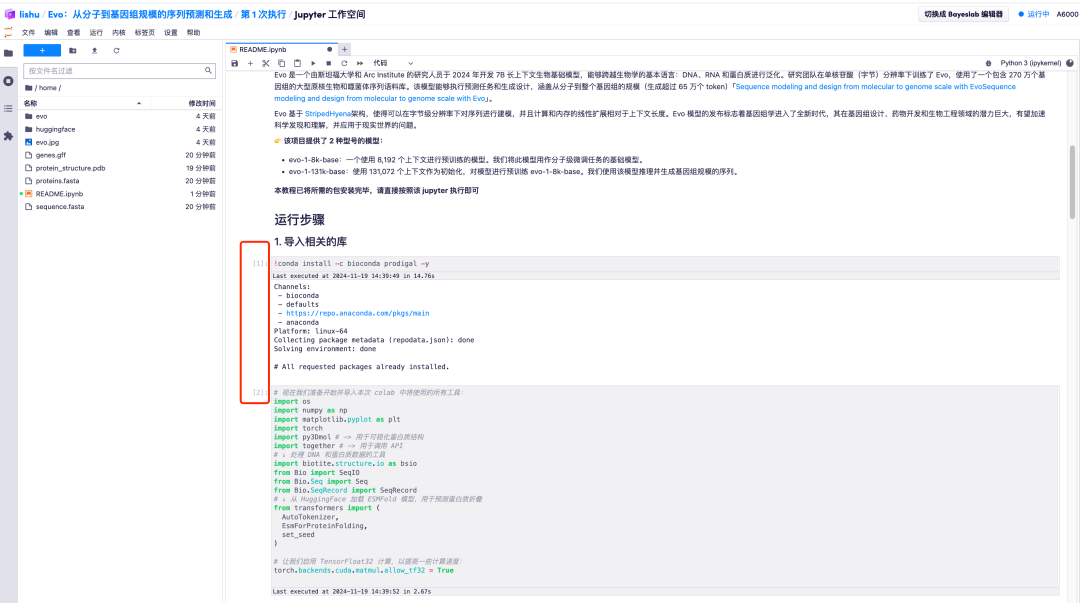
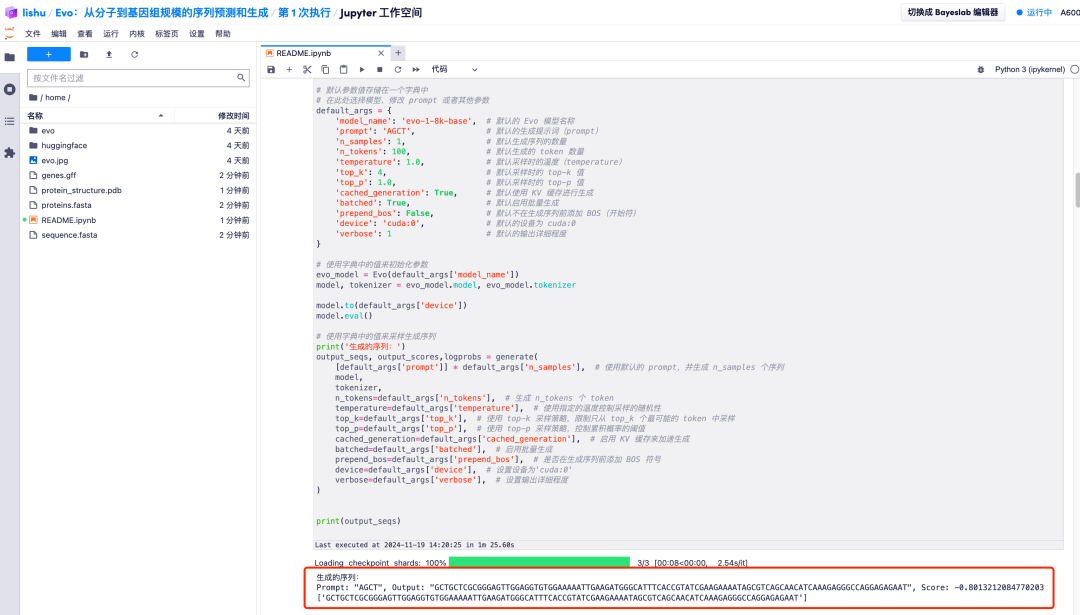
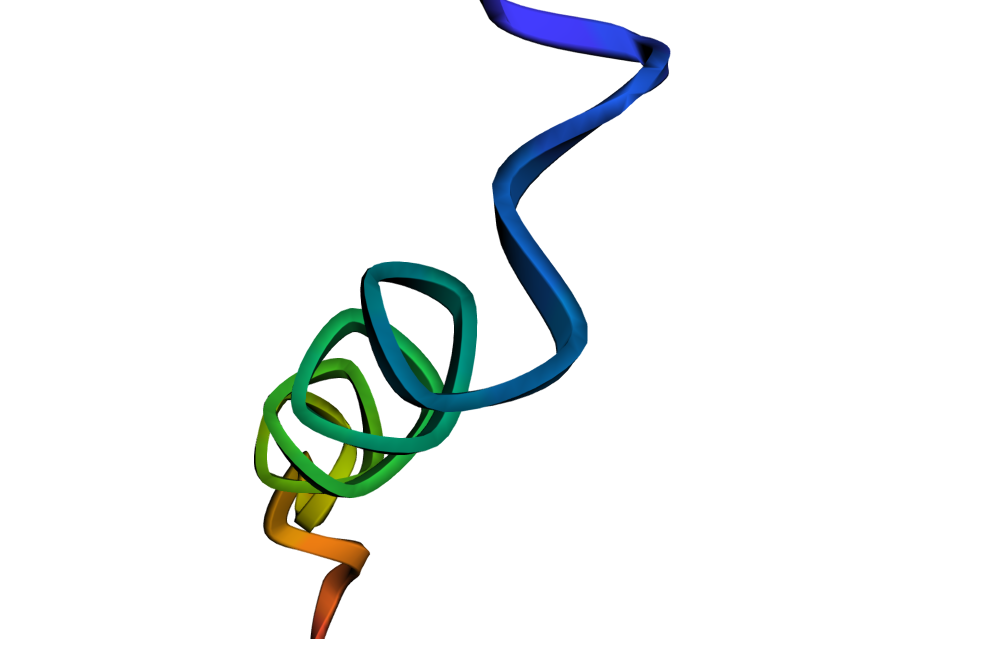
4. Darüber hinaus kann das Evo-Modell auch die generierten DNA-Sequenzen analysieren und die koevolutionäre Beziehung zwischen codierenden und nicht-codierenden Sequenzen erlernen. Darüber hinaus können proteinkodierende Gene aus DNA-Sequenzen vorhergesagt, RNA-Systeme kodiert und entworfen und so die resultierende Proteinfaltungsstruktur vorhergesagt und schließlich in Form eines Bildes dargestellt werden.
Wir haben eine „Stable Diffusion Tutorial Exchange Group“ eingerichtet. Willkommen, Freunde, treten Sie der Gruppe bei, um verschiedene technische Probleme zu diskutieren und Anwendungsergebnisse auszutauschen ~
Scannen Sie den unten stehenden QR-Code, um HyperaiXingXing zu WeChat hinzuzufügen (WeChat-ID: Hyperai01), und notieren Sie „SD Tutorial Exchange Group“, um dem Gruppenchat beizutreten.