Command Palette
Search for a command to run...
Von Der Computervision Bis Zur Medizinischen KI Hat Xie Weidi Von Der Shanghai Jiao Tong University Eine Reihe Von Ergebnissen Veröffentlicht, Die in Nature-Unterzeitschriften/NeurIPS/CVPR usw. Veröffentlicht wurden.

In den letzten Jahren hat sich die Entwicklung der KI für die Wissenschaft beschleunigt, was nicht nur zu innovativen Forschungsideen im Bereich der wissenschaftlichen Forschung geführt, sondern auch die Implementierungskanäle der KI erweitert und ihr anspruchsvollere Anwendungsszenarien beschert hat. In diesem Prozess konzentrieren sich immer mehr KI-Forscher auf traditionelle wissenschaftliche Forschungsfelder wie Medizin, Materialien und Biologie und erforschen die damit verbundenen Forschungsschwierigkeiten und industriellen Herausforderungen.
Xie Weidi, außerordentlicher Professor mit Lehrstuhl an der Shanghai Jiao Tong University, beschäftigt sich intensiv mit dem Gebiet der Computervision. Er kehrte 2022 nach China zurück und widmete sich der Erforschung medizinischer künstlicher Intelligenz.Auf dem von HyperAI koproduzierten Forum „AI for Science“ der COSCon‘24 stellte Professor Xie Weidi unter dem Titel „Towards Developing Generalist Model For Healthcare“ die Erfolge des Teams aus mehreren Perspektiven vor, darunter die Erstellung von Open-Source-Datensätzen und die Entwicklung von Modellen.

HyperAI hat den ausführlichen Austausch organisiert und zusammengefasst, ohne die ursprüngliche Absicht zu verletzen. Nachfolgend finden Sie eine Abschrift der wichtigsten Punkte der Rede.
Medizinische künstliche Intelligenz ist ein unvermeidlicher Trend
Medizinische Forschung ist von entscheidender Bedeutung, da es um das Leben und die Gesundheit aller Menschen geht. Gleichzeitig ist das Problem der ungleichen Verteilung medizinischer Ressourcen seit langem nicht grundlegend gelöst.Daher hoffen wir, eine allgemeine medizinische Versorgung zu fördern und allen Menschen eine qualitativ hochwertige Diagnose und Behandlung zu ermöglichen.
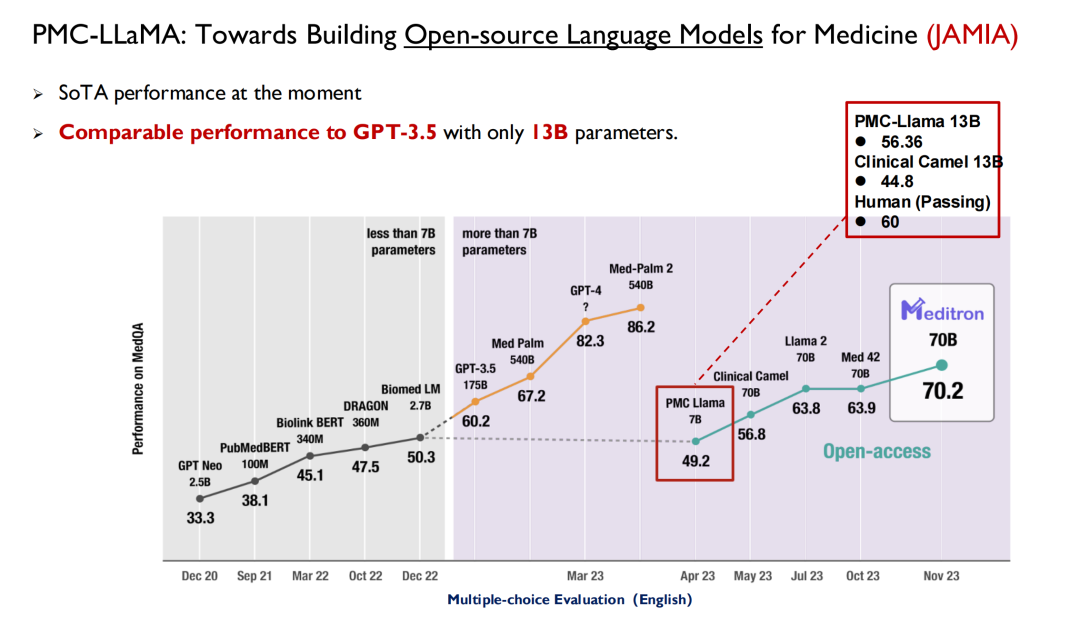
ChatGPT und andere große Modelle, die in den letzten Jahren veröffentlicht wurden, haben alle das Gesundheitswesen als Hauptschlachtfeld für Leistungstests verwendet. Wie in der folgenden Abbildung dargestellt, werden große Modelle bei der US-amerikanischen Zulassungsprüfung für medizinische Zwecke vor 2022 eine Punktzahl von 50 erreichen können, während Menschen 70 erreichen können. Daher hat die KI bei Ärzten nicht viel Aufmerksamkeit erregt.
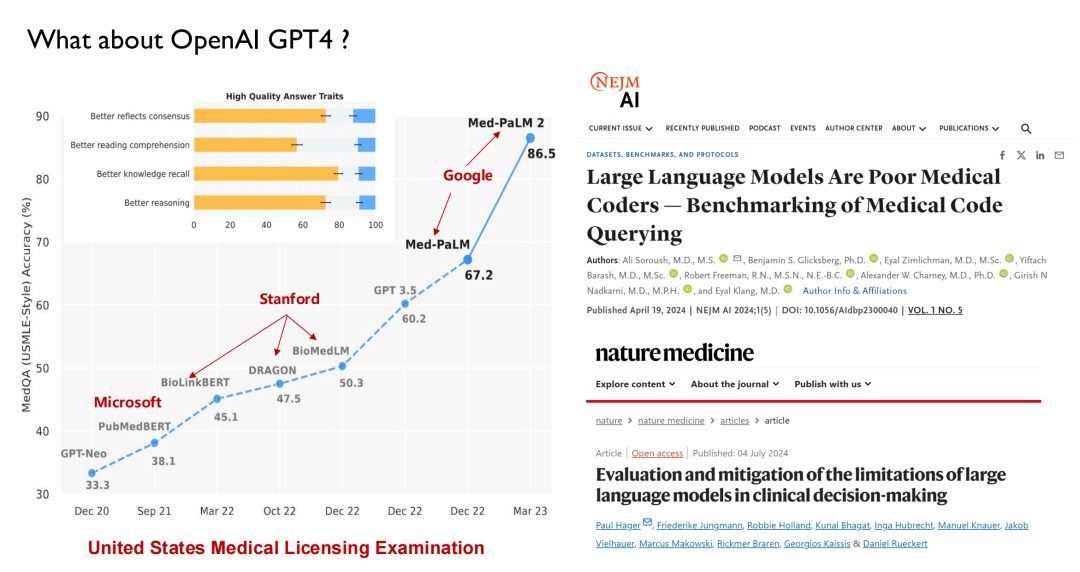
Mit der Veröffentlichung von GPT 3.5 erreichte sein Score 60,2, eine deutliche Verbesserung. Google veröffentlichte daraufhin Med-PaLM und dessen aktualisierte Version, wobei die höchste Punktzahl 86,5 erreichte. Der heutige GPT-4 kann 90 Punkte erreichen. Aufgrund der hohen Leistung und Iterationsgeschwindigkeit beginnen Ärzte, sich für KI zu interessieren.Viele medizinische Fakultäten bieten mittlerweile eine neue Disziplin an: Intelligente Medizin.
Ebenso müssen nicht nur Medizinstudenten etwas über künstliche Intelligenz lernen;Auch KI-Studierende können sich im Abschlussjahr medizinisches Wissen aneignen.Die Harvard University und andere Institutionen haben bereits entsprechende Kurse im Bereich KI eingerichtet.
Andererseits zeigen Studien in Fachzeitschriften wie Nature Medicine, dassDas große Sprachmodell versteht die Medizin eigentlich nicht.Beispielsweise versteht das große Modell derzeit keine ICD-Codes (Diagnosecodes im System der Internationalen Klassifikation der Krankheiten) und kann daher auf Grundlage der Untersuchungsergebnisse des Patienten nur schwer zeitnah medizinische Ratschläge erteilen, wie es ein Arzt kann. Es zeigt sich, dass große Modelle im medizinischen Bereich noch viele Einschränkungen aufweisen.Ich glaube nicht, dass Ärzte dadurch ersetzt werden können. Unser Team möchte dafür sorgen, dass diese Modelle die Ärzte besser unterstützen.
Das Hauptziel des Teams: der Aufbau eines allgemeinen medizinischen künstlichen Intelligenzsystems
Ich bin 2022 nach China zurückgekehrt und habe mit der Forschung zur künstlichen Intelligenz im medizinischen Bereich begonnen. Daher werde ich heute hauptsächlich die Ergebnisse des Teams in den letzten zwei Jahren vorstellen. Die medizinische Industrie deckt ein breites Spektrum an Bereichen ab und wir können nicht behaupten, dass das von uns entwickelte Modell universell ist, hoffen aber, dass es möglichst viele wichtige Aufgaben abdecken kann.
Wie in der Abbildung unten gezeigt,Auf der Eingabeseite hoffen wir, mehrere Modi unterstützen zu können.Beispielsweise Bilder, Audiodateien, Patientenakten usw. Nach der Eingabe in das Multimodale Generalistische Modell für die Medizin können Ärzte damit interagieren.Die Ausgabe des Modells hat mindestens zwei Formen, eine ist visuell,Der Ort der Läsion wird durch Segmentierung und Erkennung ermittelt.Der zweite ist Text (Text),Diagnoseergebnisse (Diagnose) oder Berichte (Bericht) ausgeben.
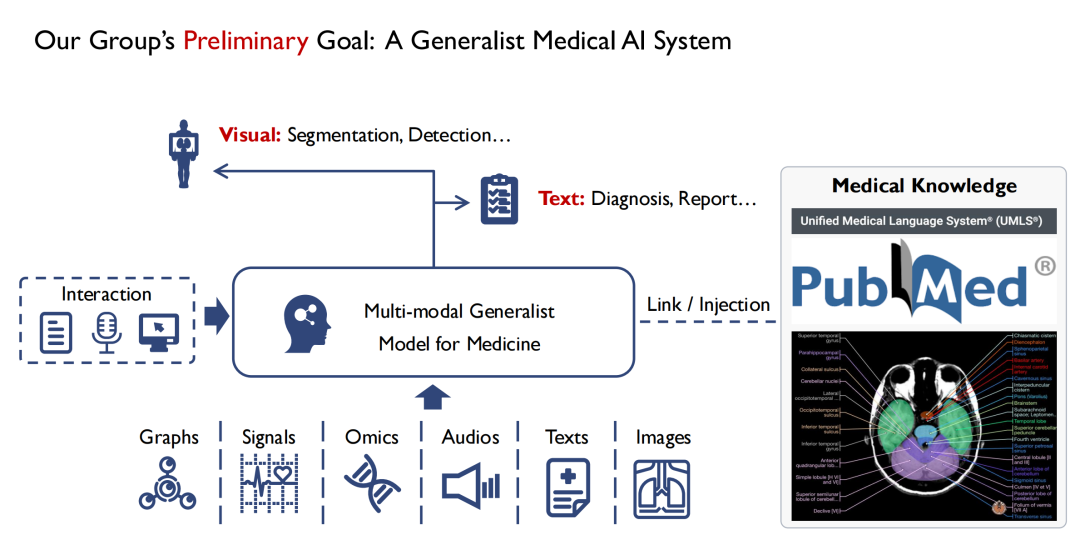
Ich studiere Computer Vision. Meiner Beobachtung nach besteht ein großer Unterschied zwischen der Sehkraft und der Medizin darin, dass das meiste Wissen in der Medizin, insbesondere in der evidenzbasierten Medizin, auf menschlichen Erfahrungen beruht. Wenn ein Anfänger alle medizinischen Bücher durchliest, kann er zumindest in der Theorie ein medizinischer Experte werden. Daher,Während des Modelltrainingsprozesses hoffen wir auch, sämtliche medizinischen Erkenntnisse einfließen zu lassen.Denn wenn dem Modell medizinisches Grundwissen fehlt, wird es schwer, das Vertrauen von Ärzten und Patienten zu gewinnen.
Zusammenfassend lässt sich also sagen:Das Hauptziel unseres Teams besteht darin, ein multimodales universelles medizinisches Modell zu entwickeln und möglichst viel medizinisches Wissen darin einzubringen.
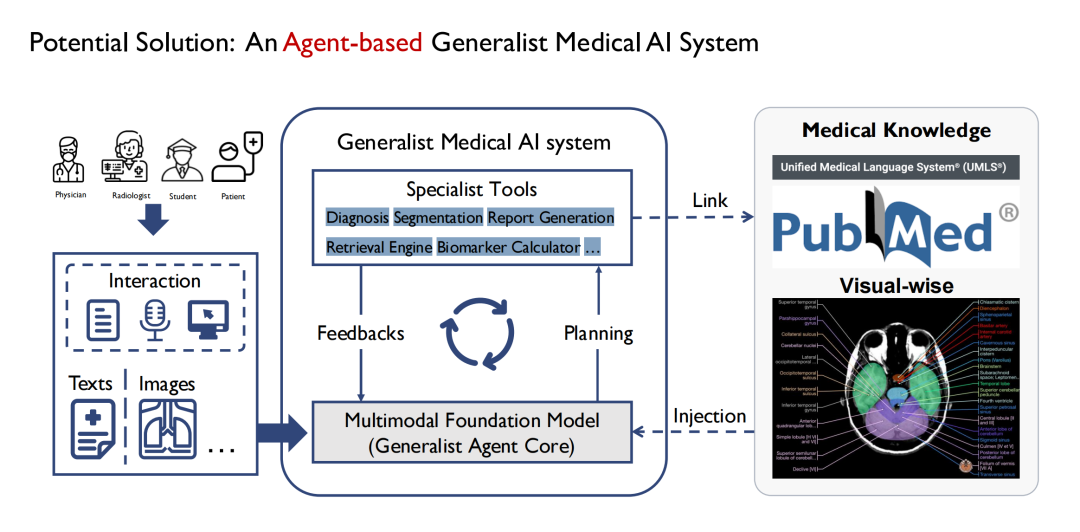
Zunächst begannen wir mit der Definition allgemeiner Modelle und stellten nach und nach fest, dass es nicht realistisch war, ein so allmächtiges medizinisches Modell wie GPT-4 zu entwickeln. Da es im Krankenhaus viele Abteilungen gibt und jede Abteilung unterschiedliche Aufgaben hat, ist es schwierig, alle Aufgaben in einem allgemeinen Modell abzudecken.Daher haben wir uns für die Implementierung über Agent entschieden.Wie in der folgenden Abbildung gezeigt, besteht das allgemeine Modell in der Mitte aus mehreren Untermodellen, und jedes Untermodell ist im Wesentlichen ein Agent, und das allgemeine Modell wird schließlich in Form eines Multi-Agenten erstellt.
Seine Vorteile bestehen darin, dass verschiedene Agenten unterschiedliche Eingaben akzeptieren können, sodass die Eingabeseite des Modells komplexer und vielfältiger sein kann; Mehrere Agenten können beim schrittweisen Bearbeiten verschiedener Aufgaben auch eine Denkkette bilden. Auch die Ausgabe ist umfangreicher. Beispielsweise kann ein Agent die Segmentierung mehrerer Arten medizinischer Bilder wie CT und MRT abschließen. gleichzeitig bietet es auch eine bessere Skalierbarkeit.
Tragen Sie hochwertige Open-Source-Datensätze bei
Mit dem Schwerpunkt auf dem großen Ziel, ein multimodales universelles medizinisches Modell zu entwickeln, werde ich nun die Erfolge des Teams aus verschiedenen Blickwinkeln vorstellen, darunter Open-Source-Datensätze, große Sprachmodelle, Krankheitsdiagnoseagenten usw.
Der erste ist unser Beitrag zu Open-Source-Datensätzen.
Im medizinischen Bereich herrscht kein Mangel an Datensätzen, doch aufgrund von Problemen mit dem eingebauten Datenschutz sind frei verfügbare, qualitativ hochwertige Daten relativ selten. Als akademisches Team hoffen wir, der Branche mehr hochwertige Open-Source-Daten zur Verfügung stellen zu können.Nach meiner Rückkehr nach China begann ich mit dem Aufbau eines umfangreichen medizinischen Datensatzes.
In Bezug auf den Text haben wir mehr als 30.000 medizinische Bücher mit 4 Milliarden Token gesammelt. die gesamte medizinische Literatur in PubMed Central (PMC) durchsucht, darunter 4,8 Millionen Artikel und 75 Milliarden Token; und sammelte medizinische Bücher in acht Sprachen, darunter Chinesisch, Englisch, Russisch und Japanisch, im Internet und wandelte sie in Text um.
Auch,Wir haben auch Superanweisungen im medizinischen Bereich erstellt.Unter Berücksichtigung der Aufgabenvielfalt werden 124 medizinische Aufgaben aufgelistet, die 13,5 Millionen Proben umfassen.

Textdaten sind leichter zu erhalten, Vision-Language (Bild-Text-Paare) sind jedoch schwieriger zu erhalten. Wir haben etwa 200.000 Fälle von der Radiopaedia-Website durchsucht, Bilder und ihre Bildunterschriften aus Zeitungsartikeln sowie mehr als 30.000 Bände mit grundlegenden radiologischen Berichten gesammelt.
Derzeit sind die meisten unserer Daten Open Source.
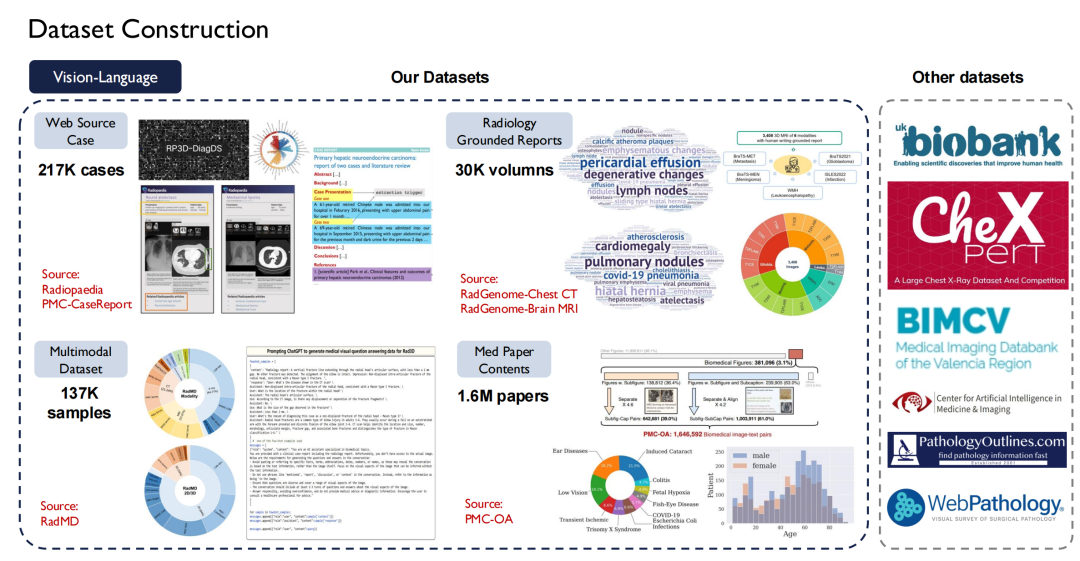
Die rechte Seite der obigen Abbildung zeigt andere öffentliche Datensätze, wie beispielsweise die UK Biobank, für die wir 10 Jahre lang Daten von fast 100.000 Patienten in Großbritannien gekauft haben; Darüber hinaus bietet Pathology Outlines umfassendes Wissen zur Pathologie.
In Bezug auf Erdungsdaten,Dies sind die Segmentierungs- und Erkennungsdaten, die ich gerade erwähnt habe.Wir haben fast 120 auf dem Markt erhältliche öffentliche Datensätze mit radiologischen Bildern in einem Standard vereint, was zu mehr als 35.000 2D/3D-Radiologie-Scanbildern geführt hat.Es deckt vier Modalitäten ab: MR, CT, PET und US, mit 400.000 detaillierten Anmerkungen, und diese Daten decken 500 Organe im Körper ab.Gleichzeitig haben wir die Beschreibung der Läsionen erweitert und alle diese Datensätze als Open Source bereitgestellt.
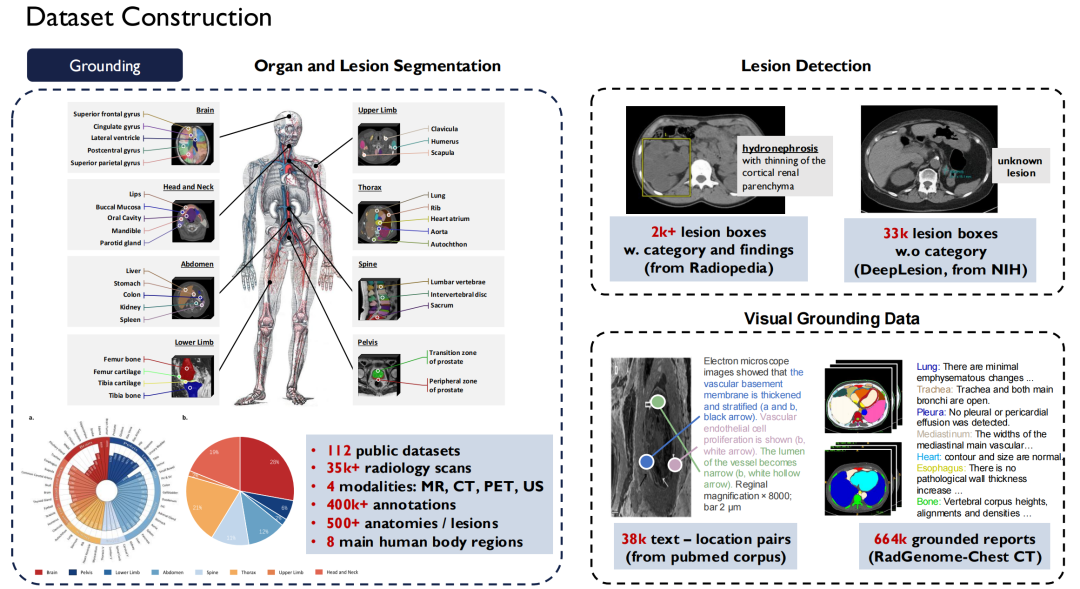
Kontinuierliche Iteration zur Erstellung eines professionellen medizinischen Modells
Sprachmodell
Nur hochwertige Open-Source-Datensätze können Studierenden und Forschern dabei helfen, ein besseres Modelltraining durchzuführen. Als nächstes werde ich die Erfolge des Teams in Bezug auf das Modell vorstellen.
Das erste ist das Sprachmodell, das eine Möglichkeit bietet, menschliches Wissen schnell in das Modell einzuspeisen. Im vergangenen April haben wir ein Modell namens PMC-LLaMA eingeführt und die zugehörige Forschung wurde in JAMIA unter dem Titel „Towards Building Open-source Language Models for Medicine“ veröffentlicht.
Papieradresse:
https://academic.oup.com/jamia/article/31/9/1833/7645318
Dies ist das erste Open-Source-Modell für große Sprachen, das wir im medizinischen Bereich entwickelt haben. Wir haben alle medizinischen Daten und die oben genannten Papierdaten in das Modell eingearbeitet, ein autoregressives Training durchgeführt und dann die Anweisungen zur Umwandlung der Daten in Frage-Antwort-Paare feinabgestimmt.
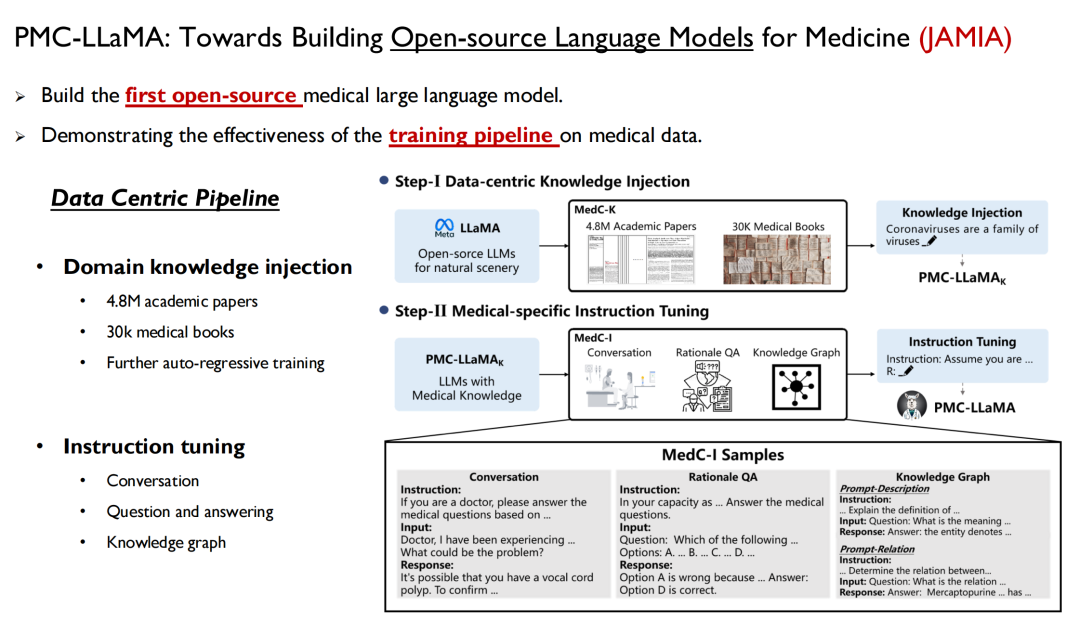
Forscher der Yale University erwähnten in ihrem Artikel: PMC-LLaMA ist das erste Open-Source-Medizinmodell auf diesem Gebiet.Viele Forscher haben es später als Grundlage verwendet, aber meiner Meinung nach besteht immer noch eine Lücke zwischen PMC-LLaMA und Closed-Source-Modellen, daher werden wir dieses Modell auch in Zukunft weiter iterieren und verbessern.
Anschließend veröffentlichten wir einen weiteren Artikel in Nature Communications: „Towards Building Multilingual Language Models for Medicine“.Es wurde ein mehrsprachiges medizinisches Modell eingeführt, das sechs Sprachen abdeckt, darunter Englisch, Chinesisch, Japanisch, Französisch, Russisch und Spanisch, und mit 25 Milliarden medizinbezogenen Token trainiert wurde. Da es keinen einheitlichen mehrsprachigen Standardtestsatz gibt, haben wir auch einen entsprechenden Benchmark erstellt, den jeder testen kann.
In der Praxis haben wir festgestellt, dass sich die Leistung des resultierenden großen medizinischen Modells ebenfalls verbessert, wenn das Basismodell aktualisiert und medizinisches Wissen in das Modell eingebracht wird.
Bei den meisten der oben genannten Aufgaben handelt es sich um „Multiple-Choice-Fragen“. Wir alle wissen jedoch, dass es für Ärzte in ihrer eigentlichen Arbeit unmöglich ist, nur Multiple-Choice-Fragen zu beantworten. Daher hoffen wir, dass das große Sprachmodell in Form von Freitext in den Arbeitsablauf des Arztes eingebettet werden kann. In Anbetracht dessenIn unserer neuen Forschung werden wir uns stärker auf klinische Aufgaben konzentrieren, relevante Datensätze sammeln und die klinische Skalierbarkeit des Modells verbessern.
Das entsprechende Papier wird derzeit noch geprüft.
Visuelles Sprachmodell
Ebenso waren wir eines der ersten Teams, das Forschungen zu visuellen Sprachmodellen im medizinischen Bereich durchführte. Basierend auf den oben genannten Daten,Wir haben 3 Open-Source-Datensätze erstellt:
* 1,6 Millionen großformatige Bildunterschriftenpaare von PubMed Central gesammelt und den PMC-OA-Datensatz erstellt;
* 227.000 medizinische visuelle Frage-Antwort-Paare aus PMC-OA generiert, um PMC-VQA zu bilden;
* Ein Rad3D-Datensatz wurde durch die Erfassung von 53.000 Fällen und 48.000 mehreren Bildunterschriftenpaaren aus der Radiopaedia-Art erstellt.
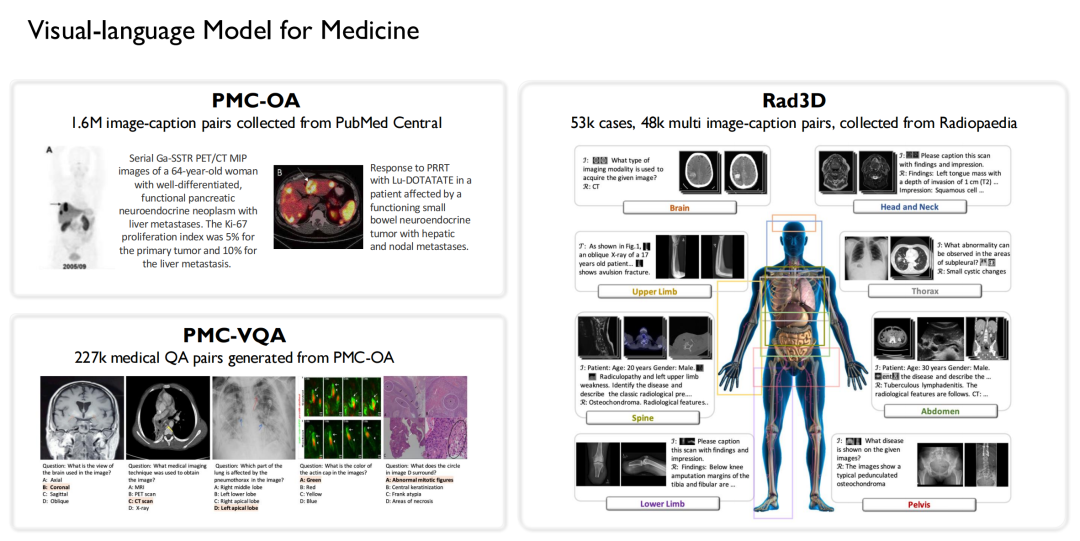
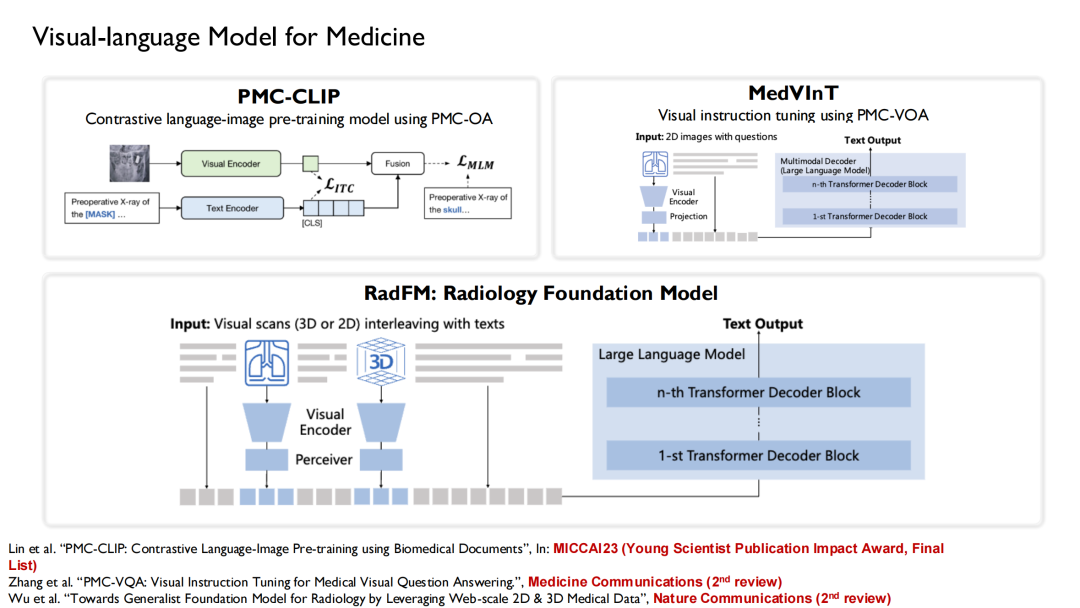
Basierend auf diesen Datensätzen haben wir die trainierten Sprachmodelle kombiniert.Es wurden drei Versionen von Vision-Language-Modellen trainiert: PMC-CLIP, MedVInT und RadFM.
PMC-CLIP ist ein Ergebnis, das wir auf der MICCAI 2023 veröffentlicht haben, der Top-Konferenz im Bereich der medizinischen Bildgebung mit künstlicher Intelligenz.Schließlich wurde er für den „Young Scientist Publication Impact Award, Final List“ ausgewählt.Der Preis wird an drei bis sieben Gewinnerarbeiten verliehen, die aus den in den letzten fünf Jahren veröffentlichten Arbeiten ausgewählt werden.
RadFM (Radiology Foundation Model) erfreut sich mittlerweile großer Beliebtheit und wird von vielen Forschern als Grundlage verwendet. Während des TrainingsprozessesWir geben Text-Bild-Verflechtungen in das Modell ein, wodurch auf Basis von Fragen direkt Antworten generiert werden können.
Erweitern Sie domänenspezifisches Wissen und verbessern Sie die Modellleistung
Das sogenannte wissensbasierte Repräsentationslernen (Knowledge-enhanced Representation Learning) muss das Problem lösen, wie medizinisches Wissen in das Modell eingebracht werden kann. Wir haben zu dieser Herausforderung auch eine Reihe von Untersuchungen durchgeführt.
Zunächst müssen wir das Problem lösen, woher „Wissen“ kommt.Einerseits handelt es sich dabei um allgemeines medizinisches Wissen.Quellen aus dem Internet sowie relevanten Artikeln und Büchern, die von UMLS, dem größten Wissensgraphen im medizinischen Bereich, verkauft werden;Auf der anderen Seite gibt es domänenspezifisches Wissen.Zum Beispiel Fallberichte, radiologische Bilder, Ultraschall usw.; All dies sowie anatomisches Wissen kann auf einigen Websites erworben werden. Natürlich muss hierbei besonders auf urheberrechtliche Aspekte geachtet werden, da die Inhalte mancher Webseiten nicht verwendet werden dürfen.

Nachdem wir dieses „Wissen“ erlangt haben, können wir ein Wissensdiagramm zeichnen.Dabei werden Zusammenhänge zwischen Krankheit und Krankheit, zwischen Arzneimitteln und zwischen Proteinen hergestellt und detailliert beschrieben.
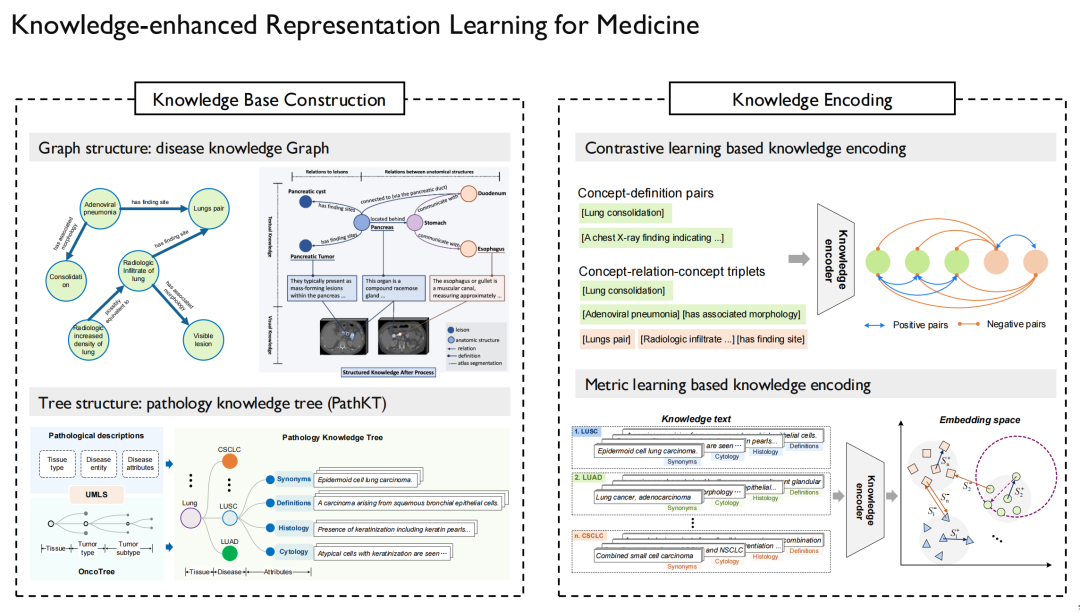
Auf der linken Seite des obigen Bildes sind das von uns erstellte Pathologie-Wissensdiagramm und der Wissensbaum zu sehen.Es wird hauptsächlich zur Krebsdiagnose verwendet, da Krebs in verschiedenen Organen des menschlichen Körpers auftreten kann und in verschiedene Untertypen unterteilt wird, die sich für die Darstellung in einer baumstrukturierten Form eignen. In ähnlicher Weise haben wir neben der multimodalen Pathologie auch verwandte Forschungen zur multimodalen Radiologie und multimodalen Röntgenstrahlen durchgeführt.
Der nächste Schritt besteht darin, dieses Wissen in das Sprachmodell einzufügen, sodass sich das Modell die Beziehung zwischen dem Graphen und den Punkten im Graphen merken kann.Sobald das Sprachmodell trainiert ist, muss das visuelle Modell nur noch an das Sprachmodell angepasst werden.
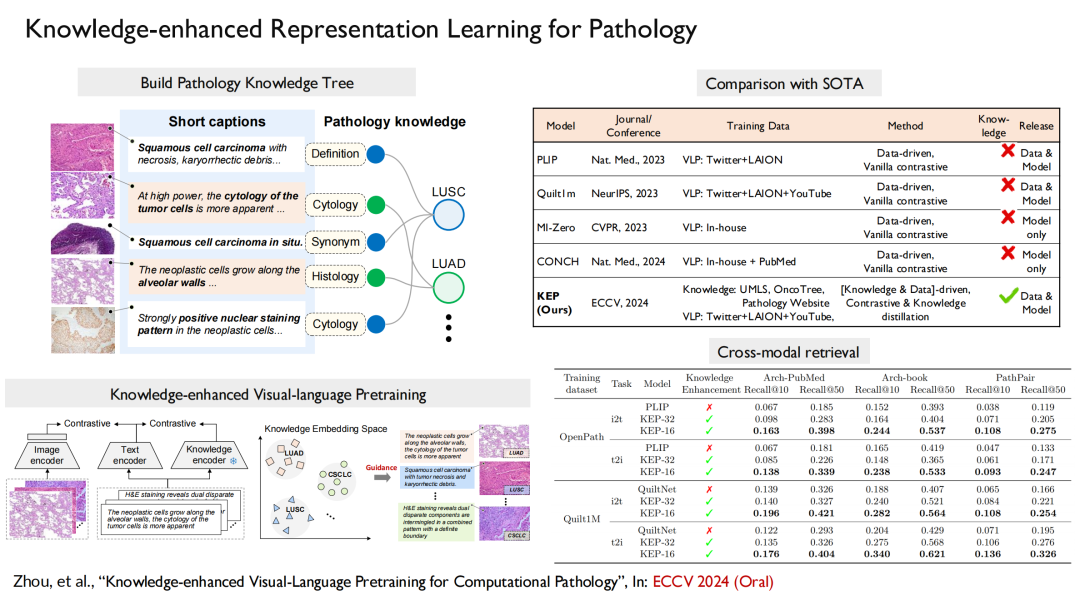
Wir verglichen unsere Ergebnisse mit denen von Microsoft und Stanford und die Ergebnisse zeigten, dassDas Modell mit zusätzlichem Domänenwissen weist eine viel höhere Leistung auf als andere Modelle ohne Domänenwissen.
Für die Pathologie wurde unser Beitrag „Knowledge-enhanced Visual-Language Pretraining for Computational Pathology“ für die führende Konferenz zum maschinellen Lernen ECCV 2024 (mündlich) ausgewählt. In dieser Arbeit erstellen wir einen Wissensbaum, fügen ihn in das Modelltraining ein und richten dann Vision und Sprache aus.
Darüber hinaus haben wir dieselbe Methode verwendet, um ein multimodales radiologisches Bildgebungsmodell zu erstellen. Die Ergebnisse wurden in Nature Communications unter dem Titel „Large-scale long-tailed disease diagnosis on radiology images“ veröffentlicht.Das Modell kann anhand der radiologischen Bilder des Patienten direkt die entsprechenden Symptome ausgeben.
Zusammenfassend:Unsere Arbeit umfasste einen vollständigen Prozess: Zunächst erstellten wir den größten Open-Source-Datensatz radiologischer Bilder, der 200.000 Bilder, 41.000 Patientenbilder, 930 Krankheiten usw. umfasst; zweitens haben wir ein multimodales, mehrsprachiges Modell entwickelt, um das Wissen in bestimmten Bereichen zu erweitern; Schließlich haben wir einen entsprechenden Benchmark erstellt.
Über Professor Xie Weidi
Er ist außerordentlicher Professor mit Lehrstuhl an der Shanghai Jiao Tong University und Stipendiat des National (Overseas) High-level Young Talent Program, des Shanghai Overseas High-level Talent Program und des Shanghai Morning Star Program. Er ist außerdem der junge Projektleiter des Großprojekts „Wissenschaftliche und technologische Innovation 2030 – Künstliche Intelligenz der neuen Generation“ des Ministeriums für Wissenschaft und Technologie und der Projektleiter der National Natural Science Foundation of China.
Er erhielt seinen Ph.D. von der Visual Geometry Group (VGG) an der Universität Oxford, wo er bei Professor Andrew Zisserman und Professor Alison Noble studierte. Er ist einer der ersten Empfänger des Google-DeepMind-Vollstipendiums, des China-Oxford-Stipendiums und des Outstanding Award der Ingenieurabteilung der Universität Oxford.
Seine Hauptforschungsgebiete sind Computer Vision und medizinische künstliche Intelligenz. Er hat mehr als 60 Artikel veröffentlicht, darunter CVPR, ICCV, NeurIPS, ICML, IJCV, Nature Communications usw., mit mehr als 12.500 Zitaten bei Google Scholar. Er hat bei führenden internationalen Konferenzen und Seminaren mehrfach den Best Paper Award, den Best Poster Award und den Best Journal Paper Award gewonnen und ist Finalist des MICCAI Young Scientist Publication Impact Award. Er ist Sondergutachter für Nature Medicine und Nature Communications und leitet die Fachgebiete CVPR, NeurIPS und ECCV, die wichtigsten Konferenzen im Bereich Computer Vision und künstliche Intelligenz.
* Persönliche Homepage:










