Command Palette
Search for a command to run...
Im Nature Journal Veröffentlicht! Der Erstautor Des Papiers Erläutert Ausführlich Die Methode Des Lernens Kleiner Stichproben Des Proteinsprachenmodells, Um Das Problem Des Mangels an Nassen Experimentellen Daten Zu Lösen

In der dritten Folge der Serie „Meet AI4S“ haben wir die Ehre, Zhou Ziyi, Postdoktorand am Institut für Naturwissenschaften der Shanghai Jiao Tong University und dem Shanghai National Center for Applied Mathematics, einzuladen.Seine Forschungsgruppe an der Shanghai Jiao Tong University, die Gruppe von Hong Liang, konzentriert sich auf KI-Protein- und Arzneimitteldesign sowie molekulare Biophysik. Die Forschungsgruppe hat fruchtbare Ergebnisse erzielt. Bis heute haben sie 77 Forschungsarbeiten veröffentlicht, viele davon in Nature-Zeitschriften.
In dieser gemeinsamen Sitzung stellte Dr. Zhou Ziyi die neuesten Forschungsergebnisse des Teams unter dem Titel „Small Sample Learning Method for Protein Language Model“ vor und erkundete neue Ideen für die KI-gestützte gerichtete Evolution.
Forschungshintergrund des Protein Language Model (PLM)
Protein und Protein-Engineering
Protein ist der Hauptträger biologischer Funktionen und der Ausführende von Lebensaktivitäten. Die natürliche Aminosäure Ammoniak durchläuft eine Dehydratationskondensationsreaktion, um die Restsequenz des Proteins zu bilden, das dann in eine Tertiärstruktur gefaltet wird. Eine Änderung des Aminosäureprofils eines Proteins kann dessen Struktur und Funktion beeinträchtigen.
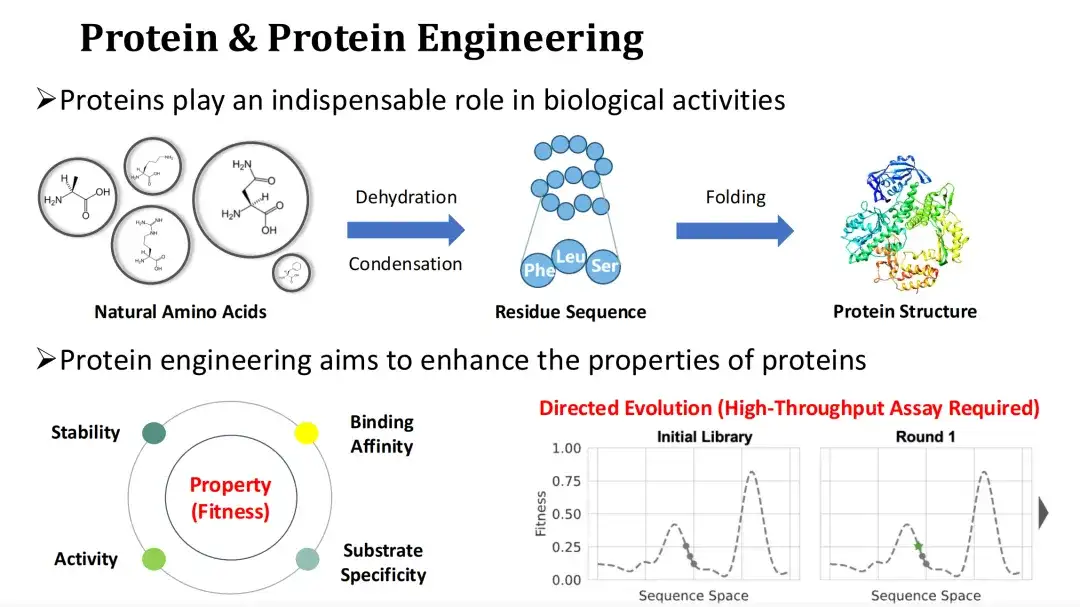
Da es oft schwierig ist, den industriellen oder medizinischen Anforderungen natürlicher Proteine gerecht zu werden, hofft man beim Protein-Engineering, die funktionellen Eigenschaften von Proteinen, wie etwa katalytische Aktivität, Stabilität, Bindungsfähigkeit usw., durch Mutation zu verbessern.
Die Quantifizierung der funktionellen Eigenschaften von Proteinen bezeichnen wir üblicherweise als Fitness. Die gerichtete Evolution ist heute die gängige Methode des Protein-Engineerings.Es beruht auf zufälliger Mutagenese und Hochdurchsatzexperimenten, um Mutanten mit hoher Fitness zu finden, aber die experimentellen Kosten sind hoch. In Anbetracht dessenDas Thema, über das ich heute sprechen werde, ist, wie man KI-Methoden zur Vorhersage der Fitness einsetzen und so die Versuchskosten senken kann.
PLM-Architektur
Wir wissen, dass die von ChatGPT dargestellten Sprachmodelle sehr leistungsstark sind und ein hochwertiges Textverständnis und eine hochwertige Textgenerierung ermöglichen. Diese Sprachmodelle werden anhand riesiger Textmengen vortrainiert und sind in der Lage, die statistischen Gesetze von Texten zu erlernen und die grundlegende Grammatik sowie die Semantik von Wörtern im Kontext zu beherrschen. Ist es also möglich, Proteinsprachenmodelle auf ähnliche Weise anhand riesiger Proteinsequenzen zu trainieren? Die Antwort ist ja.

Das Proteinsprachenmodell PLM hat drei Hauptfunktionen. Erstens kann PLM die koevolutionären Informationen von Proteinsequenzen modellieren und die gegenseitigen Abhängigkeiten und evolutionären Einschränkungen zwischen Resten erlernen.Genau wie eine natürliche Sprache kann LM die Grammatik eines Textes lernen. PLM kann diese Fähigkeit nutzen, um abzuschätzen, welche Mutationen schädlich oder nützlich sind, und so die Fitness der Mutation vorherzusagen.
Zweitens kann PLM zusätzlich zur Fitnessvorhersage auch die Vektordarstellung von Proteinen berechnen.Diese Darstellungen können zur Strukturvorhersage oder zum Protein-Mining verwendet werden und können nach Feinabstimmung auch zur Funktionsvorhersage verwendet werden.
Schließlich kann PLM eine bedingte Proteingenerierung wie ChatGPT durchführen, um ein De-novo-Proteindesign zu erreichen.
Die Architektur von PLM ähnelt der von LM in natürlicher Sprache, das in ein autoregressives Modell und ein maskiertes Modell unterteilt ist.Die Netzwerkstruktur dieser beiden Modelle verwendet beide Transformer, das aus einem Selbstaufmerksamkeitsmechanismus und einer vollständig verbundenen Schicht besteht. Der Hauptunterschied liegt in den Zielen vor dem Training.
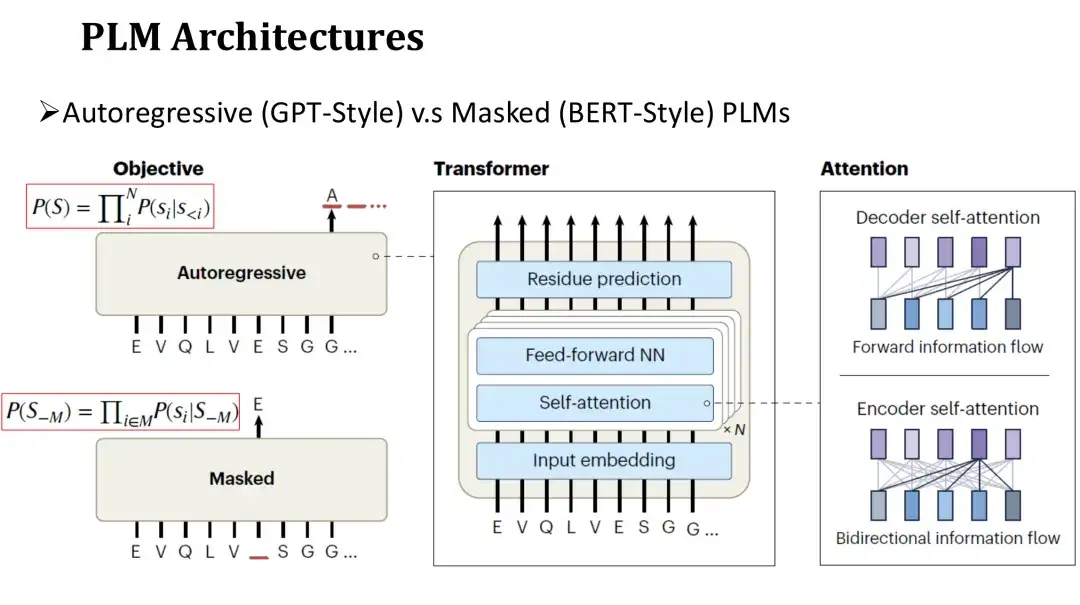
Das Vortrainingsziel des autoregressiven Modells besteht darin, die nächste Aminosäure in der Sequenz von links nach rechts zu generieren.Das Ziel des Maskierungsmodells besteht darin, zufällig maskierte Aminosäuren wiederherzustellen, ähnlich dem Ausfüllen der Lücken. Da sich das autoregressive Modell bei der Vorhersage jeder Aminosäure nur auf die generierte Sequenz auf der linken Seite verlassen kann, ist seine Aufmerksamkeit unidirektional.Das Maskierungsmodell kann während der Vorhersage die Aminosäuren auf beiden Seiten der maskierten Position sehen.Daher ist seine Aufmerksamkeit zweiseitig.
Zwei heiße Forschungsrichtungen von PLM
Derzeit sind die Forschungsschwerpunkte des PLM hauptsächlich in zwei Richtungen unterteilt. Das erste ist Retrieval-Augmented PLM.Während des Trainings oder der Vorhersage verwendet dieser Modelltyp die Multiple Sequence Alignment (MSA) des aktuellen Proteins als zusätzlichen Input und verbessert die Vorhersageleistung durch die abgerufenen Informationen. Typische Modelle dieser Art sind beispielsweise MSA Transformer und Tranception.
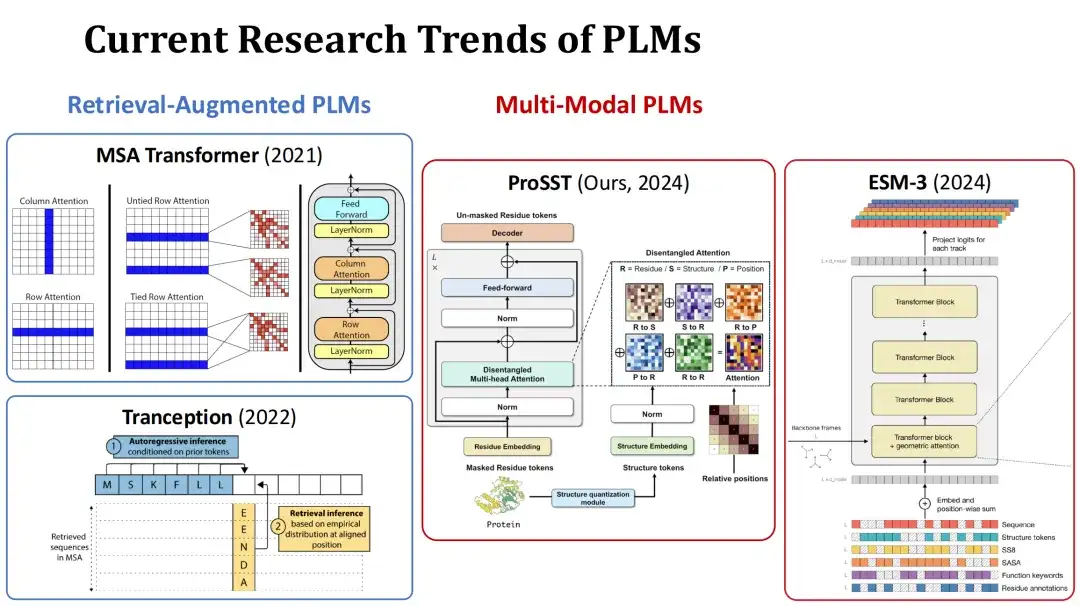
Das zweite ist Multimodales PLM.Zusätzlich zu den Proteinsequenzen verwendet dieser Modelltyp auch die Proteinstruktur oder andere Informationen als zusätzliche Eingabe, um die Darstellungsfähigkeit des Modells zu verbessern. Beispielsweise quantifiziert das von unserer Gruppe in diesem Jahr eingereichte ProSST-Modell die Proteinstruktur in eine strukturelle Token-Sequenz und gibt sie zusammen mit der Aminosäuresequenz in das Transformer-Modell ein, wobei diese beiden Arten von Informationen durch einen separaten Aufmerksamkeitsmechanismus zusammengeführt werden. Ein weiteres Beispiel ist das moderne Modell ESM-3, das umfangreichere Informationen berücksichtigt, darunter Aminosäuretyp, vollständige Tertiärstruktur, Tertiärstruktur-Token, Sekundärstruktur, lösungsmittelzugängliche Oberfläche (SASA) und Funktionsbeschreibung von Proteinen und Rückständen, insgesamt 7 Eingaben.
Unüberwachte und überwachte Fitnessvorhersage
Als nächstes besprechen wir das Problem der Fitnessvorhersage.Da PLM die Wahrscheinlichkeitsverteilung von Proteinsequenzen modellieren kann, kann es direkt zur Fitnessvorhersage von Mutationen ohne gekennzeichnete Daten verwendet werden. Diese Methode wird als Zero-Shot-Vorhersage oder unüberwachte Vorhersage bezeichnet.

Insbesondere bewertet PLM Mutationen, indem es das Log-Likelihood-Verhältnis zwischen der Mutante und dem Wildtyp berechnet. Beim autoregressiven Modell ist die Wahrscheinlichkeit der Sequenz P das Produkt der Wahrscheinlichkeiten für die Erzeugung jeder Aminosäure. Der Mutationswert kann durch Subtraktion des Wildtyp-logP vom Mutanten-logP ermittelt werden. Intuitiv geht es darum, die Wahrscheinlichkeit des Auftretens einer Mutation im Verhältnis zum Wildtyp zu vergleichen und dann die Auswirkungen der Mutation zu bewerten. Es handelt sich hierbei um eine empirische Auswertungsmethode.
Für das Maskierungsmodell ist es unmöglich, die Wahrscheinlichkeit der gesamten Sequenz direkt zu berechnen, aber es kann zuerst einen bestimmten Punkt maskieren und dann die Wahrscheinlichkeitsverteilung der Aminosäuren an diesem Punkt schätzen. Daher kann für jede Mutationsposition der logP der Wildtyp-Aminosäure vom logP der vorhergesagten mutierten Aminosäure nach der Maskierung subtrahiert werden. Anschließend kann die Differenz an allen Positionen addiert werden, um den Score des Mutanten zu erhalten.
Da PLMs zudem Vektordarstellungen von Proteinsequenzen bereitstellen, können sie auch feinabgestimmt werden, um eine überwachte Fitnessvorhersage zu erreichen, wenn ausreichend experimentelle Daten vorliegen.
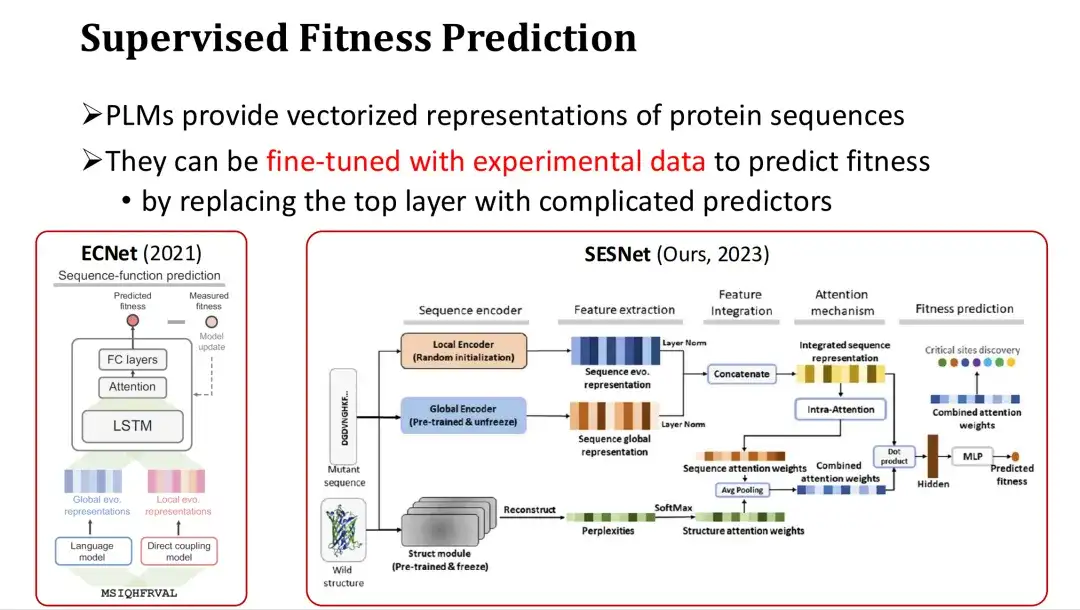
Der spezifische Ansatz besteht darin, nach der letzten Feature-Ebene von PLM eine Ausgabeebene zur Vorhersage der Fitness (wie etwa einen Aufmerksamkeitsmechanismus oder ein mehrschichtiges Perzeptron MLP) hinzuzufügen und das Fitness-Label für das vollständige oder teilweise Training zu verwenden. Beispielsweise fügt ECNet MSA-Funktionen basierend auf den großen Modellfunktionen hinzu, integriert sie über LSTM und führt ein überwachtes Training durch. Das im letzten Jahr von unserer Forschungsgruppe entwickelte SESNet-Modell kombiniert die Sequenzmerkmale von ESM-1b, die Strukturmerkmale von ESM-IF und die MSA-Merkmale, um eine überwachte Fitnessvorhersage durchzuführen.
Einführung in FSFP: Eine kleine Beispiel-Lernmethode für PLM
Bedeutung des Lernens kleiner Stichproben für die Fitnessvorhersage
Bevor die FSFP-Methode vorgestellt wird, muss die Bedeutung des Lernens anhand kleiner Stichproben bei der Fitnessvorhersage geklärt werden. Obwohl für das Training unbeaufsichtigter Methoden keine gekennzeichneten Daten erforderlich sind, ist ihre Zero-Shot-Wertungsgenauigkeit geringer. Da die auf Log-Likelihood-Verhältnissen basierenden Werte zudem nur bestimmte Naturgesetze von Proteinen widerspiegeln können, ist es auch schwierig, die nicht-natürlichen Eigenschaften von Proteinen effektiv vorherzusagen.

Obwohl überwachte Lernmethoden präzise sind, sind aufgrund der großen Anzahl an PLM-Parametern umfangreiche experimentelle Daten für das Training erforderlich, um die Leistung deutlich zu verbessern. Die Auswertung von überwachten Lernmodellen beinhaltet im Allgemeinen die Aufteilung des vorhandenen Hochdurchsatzdatensatzes im Verhältnis 8:2, während der Trainingssatz von 80% bereits Zehntausende von Daten enthalten kann, deren Beschaffung in der Praxis sehr teuer ist.
Um dieses Problem zu lösen, schlagen wir die FSFP-Methode vor, einen Small-Shot-Learning-Ansatz für PLM. Diese Methode kann die Fitnessvorhersageleistung von PLM mithilfe einer kleinen Anzahl von Trainingsbeispielen (Dutzende) erheblich verbessern. Gleichzeitig ist die FSFP-Methode sehr flexibel und kann auf verschiedene PLMs angewendet werden.
FSFP-Methode: Ranking-Lernen für Fitness
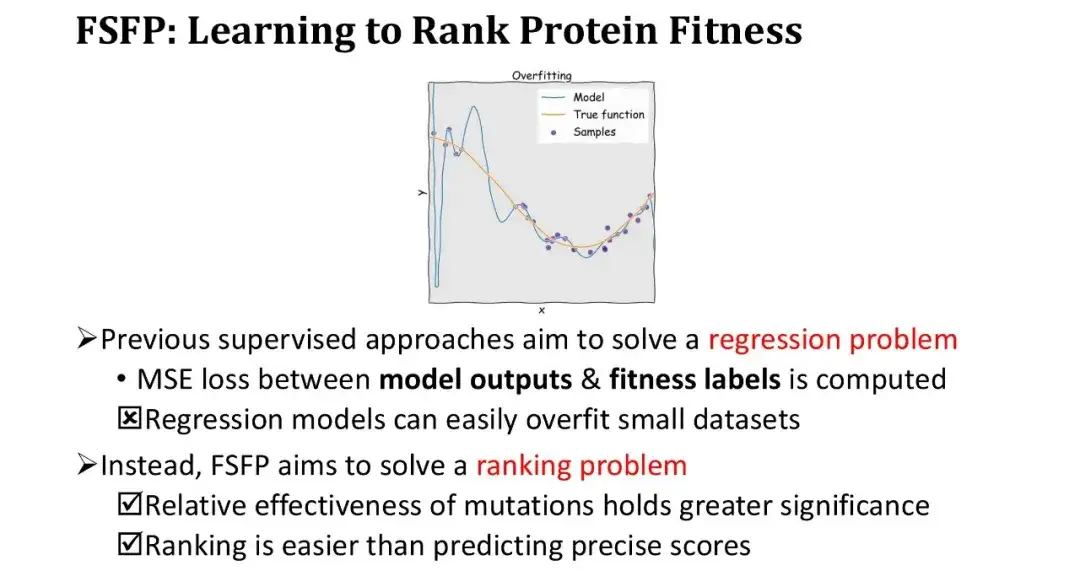
Bisherige Methoden des überwachten Lernens betrachteten die Fitnessvorhersage als ein Regressionsproblem, d. h. die Optimierung des Modells durch Berechnung des mittleren quadratischen Fehlers (MSE) zwischen der Modellausgabe und dem Fitnesslabel. Unter kleinen Stichprobenbedingungen kommt es jedoch sehr leicht zu einer Überanpassung des Regressionsmodells und der Trainingsverlust sinkt sehr schnell. Deshalb haben wir unsere Denkweise geändert und keine Regression mehr durchgeführt, sondern stattdessen Ranking-Learning eingesetzt, das nur eine genaue Sortierung erfordert und keine präzise Anpassung numerischer Werte erfordert.
Dieser Ansatz hat zwei große Vorteile. Erstens erfüllt die Sequenzierung selbst die grundlegenden Anforderungen des Protein-Engineerings, bei dem lediglich die relative Wirksamkeit von Mutationen gemessen werden muss. Zweitens ist die Rangfolgeaufgabe einfacher als die Vorhersage absoluter Werte.
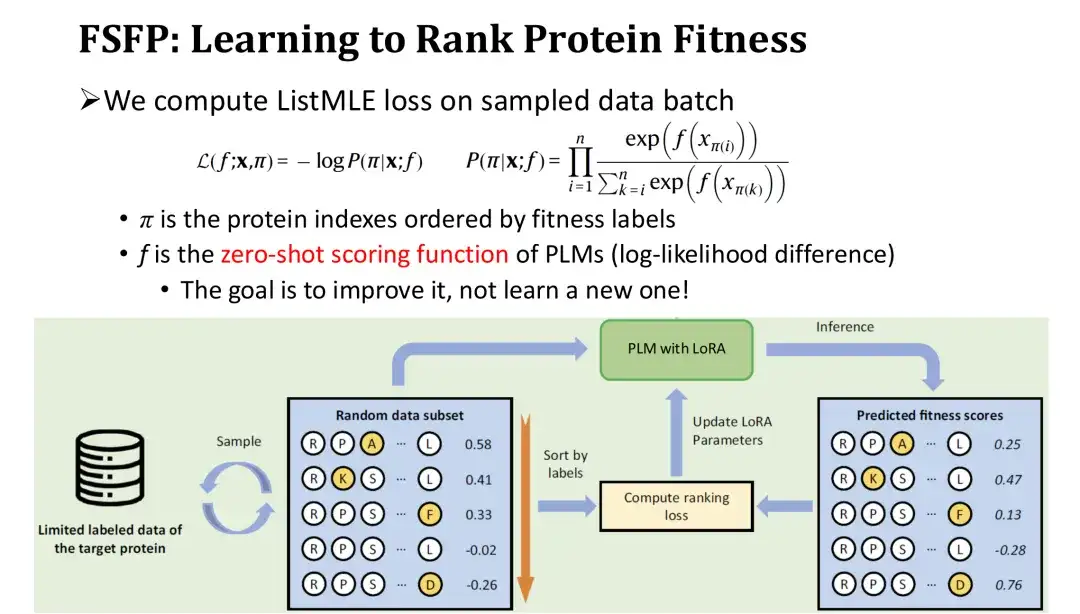
In der Trainingsiteration sortieren wir den abgetasteten Satz von Mutanten in umgekehrter Reihenfolge entsprechend ihrer Bezeichnungen und berechnen dann den Ranking-Verlust - ListMLE basierend auf den Vorhersagewerten des Modells für diese Mutanten.Je näher die Rangfolge der vom Modell vorhergesagten Werte an der wahren Rangfolge liegt, desto geringer ist der Verlust. Darunter verwenden wir die Zero-Shot-Bewertungsfunktion basierend auf dem Log-Likelihood-Verhältnis als Bewertungsfunktion f für das Modell zur Mutation. Der Zweck besteht darin, die Zero-Shot-Wertung als Ausgangspunkt zu verwenden und sie schrittweise mit Trainingsdaten zu korrigieren, um die Leistung zu verbessern, ohne ein Modul neu zu initialisieren und so den Trainingsaufwand zu verringern.
FSFP-Methode: Parametereffiziente Feinabstimmung von PLM
Da die Anzahl der Parameter in einem PLM normalerweise mehrere Hundert Millionen beträgt, führt die Feinabstimmung des gesamten Modells mit sehr wenigen Daten zwangsläufig zu einer Überanpassung.Daher haben wir eine zweite Technik, LoRA, eingeführt, um die Anzahl der trainierbaren Parameter des Modells zu begrenzen.
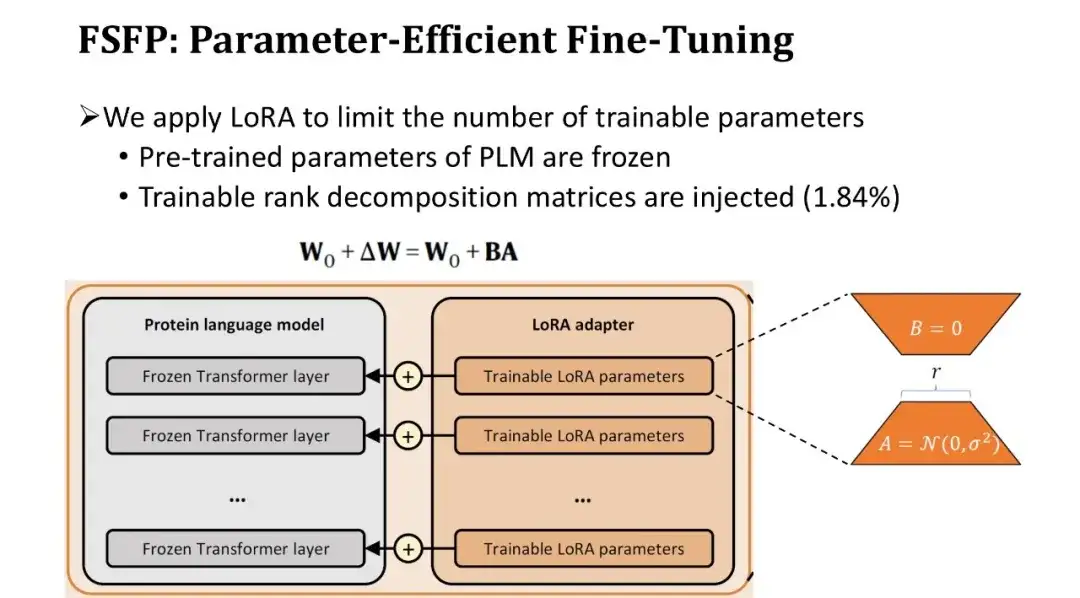
LoRA fügt ein Paar trainierbarer Rangzerlegungsmatrizen in die vollständig verbundene Schicht jedes Transformer-Blocks ein und lässt die vortrainierten Parameter unverändert. Da die Rangzerlegungsmatrix sehr klein ist, kann die Anzahl der trainierbaren Parameter auf die ursprünglichen 1,84% reduziert werden. Obwohl die Anzahl der trainierbaren Parameter reduziert ist, ist die Lernfähigkeit des Modells weiterhin gewährleistet, da jede Schicht des Transformers fein abgestimmt ist.
FSFP-Methode: Anwendung von Meta-Learning zur Fitnessvorhersage
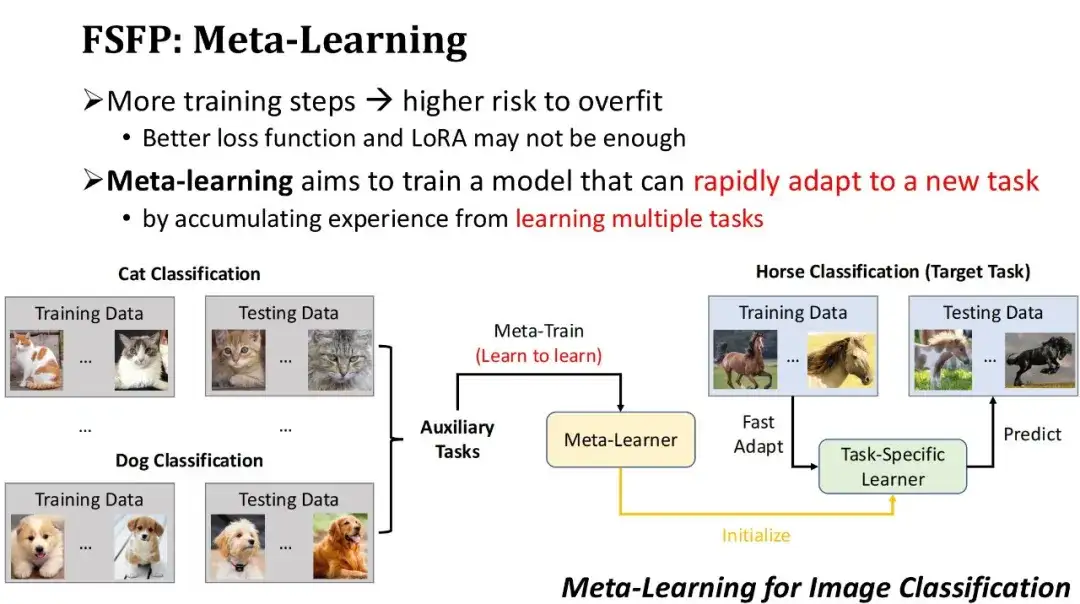
Um eine Überanpassung zu vermeiden, haben wir nicht nur eine bessere Verlustfunktion verwendet, sondern auch die Menge der trainierbaren Parameter durch die LoRA-Technologie begrenzt. Dennoch besteht immer noch die Gefahr einer Überanpassung, wenn zu viele Trainingsiterationen mit einer kleinen Stichprobe von Trainingsdaten durchgeführt werden. Daher hoffen wir, die Modellleistung mit weniger Trainingsiterationen schnell verbessern zu können.Aufgrund dieses Bedarfs haben wir die dritte Technologie übernommen – Meta-Learning. Die Grundidee des Meta-Lernens besteht darin, das Modell zunächst bei einigen Hilfsaufgaben Erfahrungen sammeln zu lassen, um ein erstes Modell zu erhalten, und dann das erste Modell zu verwenden, um sich schnell an neue Aufgaben anzupassen.
Wie in der folgenden Abbildung gezeigt, ist dies ein Beispiel für eine Bildklassifizierung basierend auf Meta-Learning. Angenommen, die Zielaufgabe besteht darin, ein Modell zur Klassifizierung von Pferden zu trainieren, es liegen jedoch relativ wenige gekennzeichnete Daten über Pferde vor. Daher können wir zunächst einige Hilfsaufgaben mit einer großen Datenmenge finden, z. B. die Klassifizierung von Katzen, Hunden usw., und Meta-Lernalgorithmen verwenden, um diese Hilfsaufgaben zu trainieren, das Erlernen neuer Aufgaben zu erlernen und einen Meta-Lerner zu erhalten. Wenn Sie dann diesen Meta-Lerner als Ausgangsmodell verwenden und ihn mehrere Schritte lang mit einer kleinen Menge gekennzeichneter Pferdedaten trainieren, können Sie schnell einen Pferdeklassifizierer erhalten. Voraussetzung für das Funktionieren des Meta-Lernens ist natürlich, dass die verwendeten Hilfsaufgaben den Zielaufgaben ausreichend nahe kommen.
Wie lässt sich Meta-Learning auf das Szenario der Fitnessvorhersage anwenden?Zunächst besteht unser Ziel darin, die Mutationen des Zielproteins nach Fitness zu ordnen, und das zu trainierende Modell ist PLM unter Verwendung der LoRA-Technologie.
Wir wenden zwei Strategien zum Erstellen von Hilfsaufgaben an. Die erste besteht darin, in der vorhandenen DMS-Datenbank nach Mutationsexperiment-Datensätzen ähnlicher Proteine zu suchen, basierend auf ihrer Ähnlichkeit mit dem Zielprotein, und die ersten beiden Datensätze als zwei Hilfsaufgaben auszuwählen.Ausgangspunkt hierfür ist die Überlegung, dass die Fitnesslandschaft ähnlicher Proteine ebenfalls ähnlich ist.
Die zweite Strategie besteht darin, mithilfe des MSA-Modells mögliche Mutationen des Zielproteins zu bewerten, um einen Pseudo-Label-Datensatz zu bilden und ihn als dritte Hilfsaufgabe zu verwenden.Der Grund, warum wir uns für das MSA-Modell entscheiden, besteht darin, dass der Mutationsvorhersageeffekt des MSA-Modells dem des PLM im Allgemeinen nicht unterlegen ist. Wir hoffen, durch MSA eine Datenverbesserung durchführen zu können, um die Darstellungsfähigkeit des PLM voll auszuschöpfen.
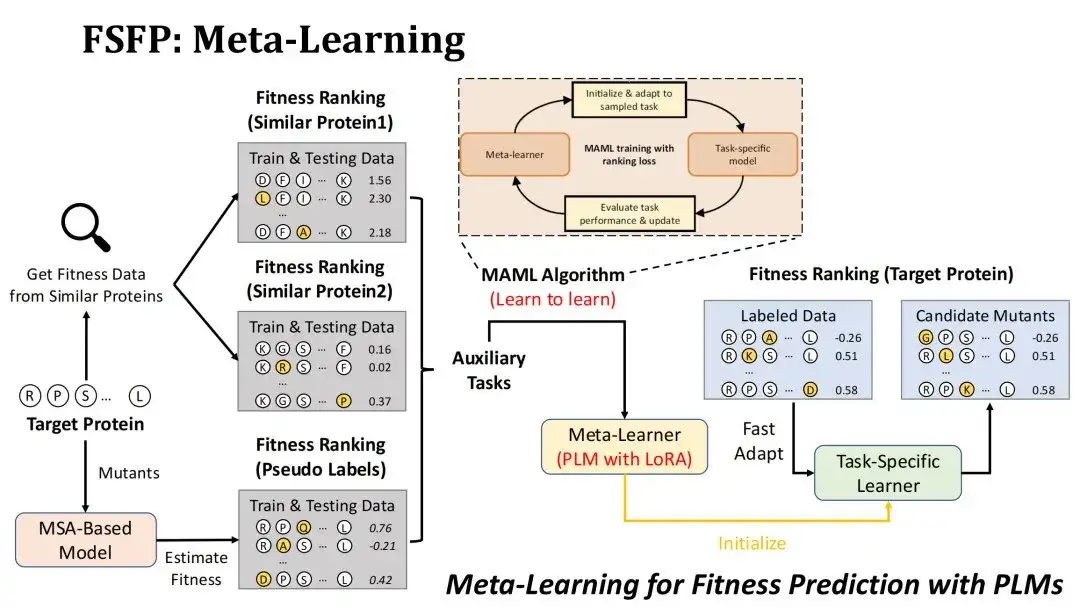
Der von uns verwendete Meta-Learning-Algorithmus ist MAML, dessen Trainingsziel darin besteht, den Testverlust des Meta-Lerners nach k-Schritten Feintuning mit den Trainingsdaten einer Hilfsaufgabe möglichst gering zu halten, sodass dieser nach k-Schritten Feintuning grob auf die Zielaufgabe konvergieren kann.
Leistungsbewertung der FSFP-Methode bei der Vorhersage der Proteinfitness
Benchmark-Erstellung
Unsere Benchmark-Daten stammen von ProteinGym, das ursprünglich 87 DMS-Datensätze enthielt und jetzt auf 217 aktualisiert wurde.Die den 87 DMS entsprechenden Proteine werden grob in vier Kategorien unterteilt: Eukaryoten, Prokaryoten, Menschen und Viren, und decken insgesamt etwa 15 Millionen Mutationen und die entsprechende Fitness ab.
Für jeden Datensatz wählten wir zufällig 20, 40, 60, 80 und 100 Einzelpunktmutationen als kleine Stichproben-Trainingssätze aus und die verbleibenden Mutationen wurden als Testsätze verwendet. Es ist zu beachten, dass wir für das frühzeitige Stoppen keinen zusätzlichen Validierungssatz verwendet haben, sondern stattdessen die Anzahl der Trainingsschritte durch Kreuzvalidierung des Trainingssatzes geschätzt haben.

Es wurde bereits erwähnt, dass für das Meta-Lernen drei Hilfsaufgaben erforderlich sind, von denen zwei aufgrund ihrer Ähnlichkeit mit dem Zielprotein aus der DMS-Datenbank abgerufen werden.Wenn wir mit einem Datensatz trainieren, rufen wir ihn aus den restlichen Datensätzen in ProteinGym ab, vorausgesetzt, es handelt sich um die Datenbank.
Wie in der Abbildung rechts unten gezeigt, wird jedes Protein in ProteinGym als Abfrage verwendet und die Ähnlichkeitsverteilung der ähnlichsten Proteine wird jeweils über MMseqs2 und FoldSeek abgerufen. Es ist ersichtlich, dass die durchschnittliche Sequenz- oder Strukturähnlichkeit der ähnlichsten Proteine bei etwa 0,5 liegt. Die dritte Zusatzaufgabe besteht darin, Mutationen mithilfe des MSA-Modells zu bewerten. Wir haben uns für das GEMME-Modell entschieden, das einen auf MSA basierenden Evolutionsbaum erstellt und die Erhaltung jedes Punkts auf dem Evolutionsbaum berechnet, um die Mutation zu bewerten.
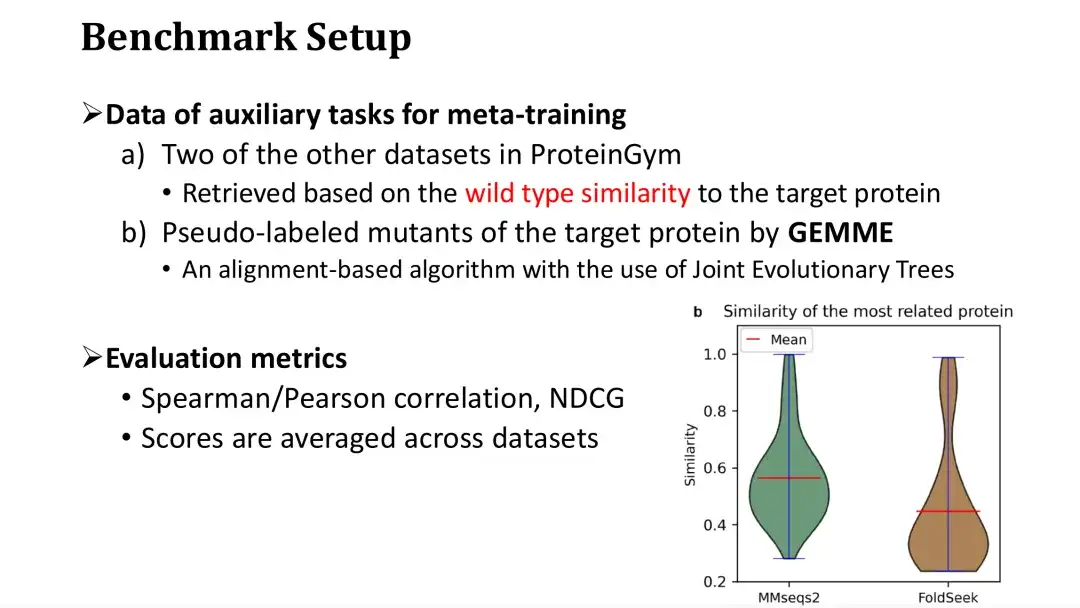
Als Bewertungsindikatoren werden der Spearman/Pearson-Koeffizient und der NDCG verwendet, die gängige Bewertungskriterien bei Fitnessvorhersageaufgaben sind. Das endgültige Bewertungsergebnis ist die Durchschnittsbewertung der 87 Datensätze.
Ablationsexperiment von FSFP auf ESM-2
Wie in der folgenden Abbildung gezeigt, stellt die x-Achse in der linken Abbildung die Größe des Trainingssatzes dar, die y-Achse stellt den Spearman-Koeffizienten dar und jede Linie entspricht einer anderen Modellkonfiguration. Die oberste Zeile stellt das vollständige FSFP-Modell dar; Die zweite Zeile stellt das Ersetzen der dritten Hilfsaufgabe des Meta-Lernens durch DMS-Daten ähnlicher Proteine ohne Verwendung von MSA dar. Es ist ersichtlich, dass die Modellleistung nach dem Entfernen der MSA-Informationen abnimmt. Die dritte Zeile stellt dar, dass kein Meta-Learning verwendet wird und man sich nur auf Ranking-Learning und LoRA verlässt, und der Spearman-Koeffizient nimmt weiter ab.
Die grüne Linie stellt das zuvor in NBT veröffentlichte Ridge-Regressionsmodell dar, das eines der wenigen Basismodelle ist, das derzeit für Szenarien mit kleinen Stichproben geeignet ist. die graue gepunktete Linie stellt den Nullschuss-Score von ESM-2 dar; Die unteren beiden Zeilen stellen die Ergebnisse des Trainings von ESM-2 mit herkömmlichen Regressionsmethoden dar.
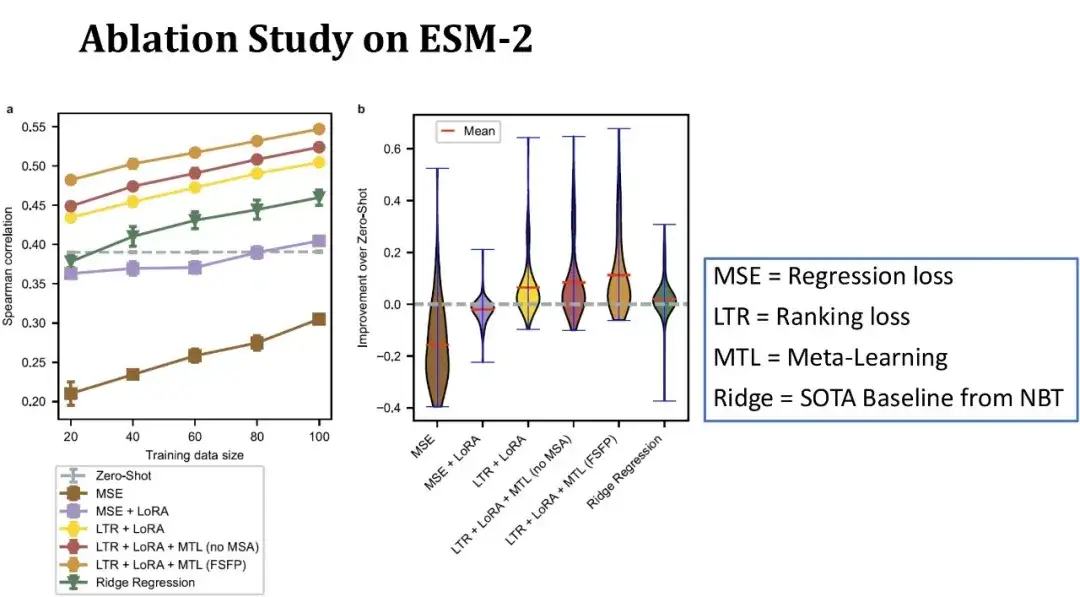
Insgesamt verbessert unsere Methode den Spearman bei nur 20 Trainingsbeispielen um 10 Punkte im Vergleich zum Zero-Shot, und jedes Modul spielt eine positive Rolle für die Modellleistung. Die Abbildung rechts zeigt die Verteilung der Leistungsverbesserung im Vergleich zum Zero-Shot auf 87 Datensätzen mit einer Trainingssatzgröße von 40 Stichproben.Es ist ersichtlich, dass unsere Methode die Modellleistung bei den meisten Datensätzen verbessern kann und die Verbesserung bei einigen Datensätzen sogar 40 Punkte übersteigt, was eine stabilere Leistung als die Basislinie zeigt.
Wirksamkeit von Meta-Lernen
Das Ziel des Meta-Learning besteht darin, PLM in die Lage zu versetzen, mit einer geringen Anzahl von Iterationen schnell auf die Zielaufgabe zu konvergieren.Im Folgenden finden Sie einige Beispiele, die dies veranschaulichen.
Die folgenden 3 Diagramme zeigen die Trainingskurven der Feinabstimmung auf 3 Datensätzen mit 40 Trainingsbeispielen. Die x-Achse stellt die Anzahl der Trainingsschritte dar und die y-Achse stellt den Spearman-Koeffizienten im Testsatz dar. Die orangefarbenen und roten Linien oben sind beides mit Meta-Learning trainierte Modelle. Ersteres verwendet MSA zum Erstellen von Hilfsaufgaben, letzteres nicht. Die gelbe Linie stellt das Modell dar, das nur Learning to Ranking und LoRA ohne Meta-Learning verwendet.
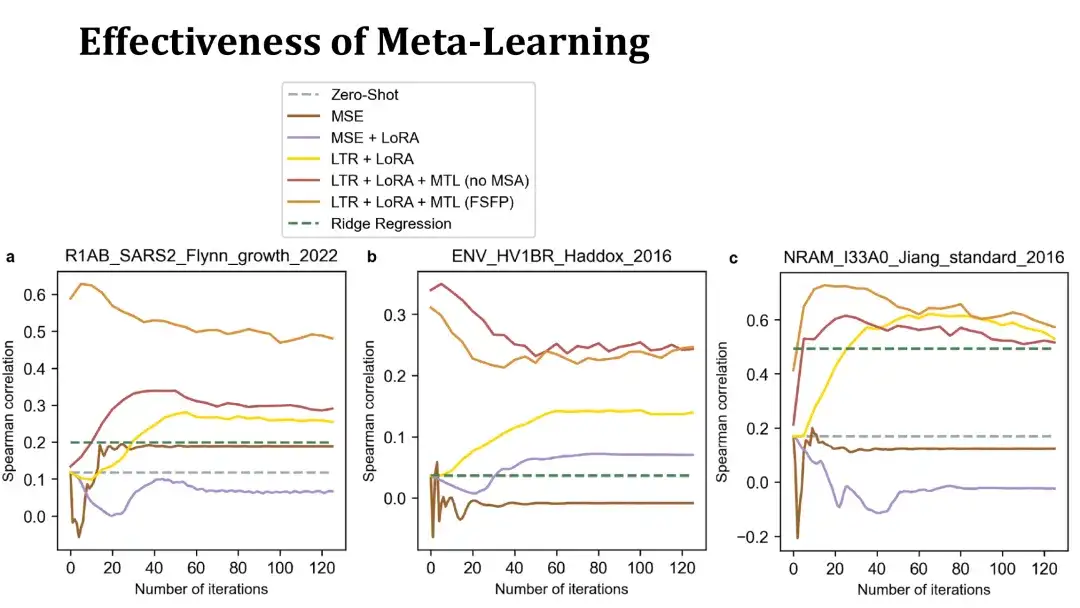
Wie Sie sehen können,Mit Meta-Learning trainierte Modelle können die Leistung bei Zielproteinen schneller verbessern und innerhalb von 20 Schritten höhere Punktzahlen erreichen. Manchmal übertreffen sie sogar das ursprüngliche Modell ohne Feinabstimmung. Dies deutet darauf hin, dass Meta-Lernen ein wirksames Ausgangsmodell hervorbringt.Das folgende MSE-basierte Modell weist eine schlechte Leistung auf und ist schnell überangepasst, sodass es schwierig ist, die Zero-Shot-Methode zu übertreffen.
Ergebnisse der Anwendung von FSFP auf verschiedene PLMs
Wir haben drei typische PLMs ausgewählt, nämlich ESM-1v, ESM-2 und SaProt.Die ersten beiden Modelle verwenden nur Proteinsequenzinformationen, während SaProt Protein-Tertiärstruktur-Token kombiniert.
Das Liniendiagramm links zeigt den Spearman-Score zur Vorhersage der Auswirkung einer einzelnen Punktmutation bei unterschiedlichen Trainingssatzgrößen. Dieselbe Farbe steht für dasselbe Modell und unterschiedliche Punktformen stehen für unterschiedliche Trainingsmethoden. Die Punkte oben stellen die FSFP-Methode dar, das umgekehrte Dreieck unten stellt die Ridge-Regression dar und die gepunktete Linie stellt die Zero-Shot-Leistung des Modells dar. Die violette Linie stellt das GEMME-Modell dar, das kein PLM ist, mit dem aber die Ridge-Regressionsmethode kombiniert werden kann.Es ist ersichtlich, dass die FSFP-Methode die Leistung jedes PLM stetig verbessern kann und viel besser ist als die Ridge-Regression und der Zero-Shot des entsprechenden Modells.
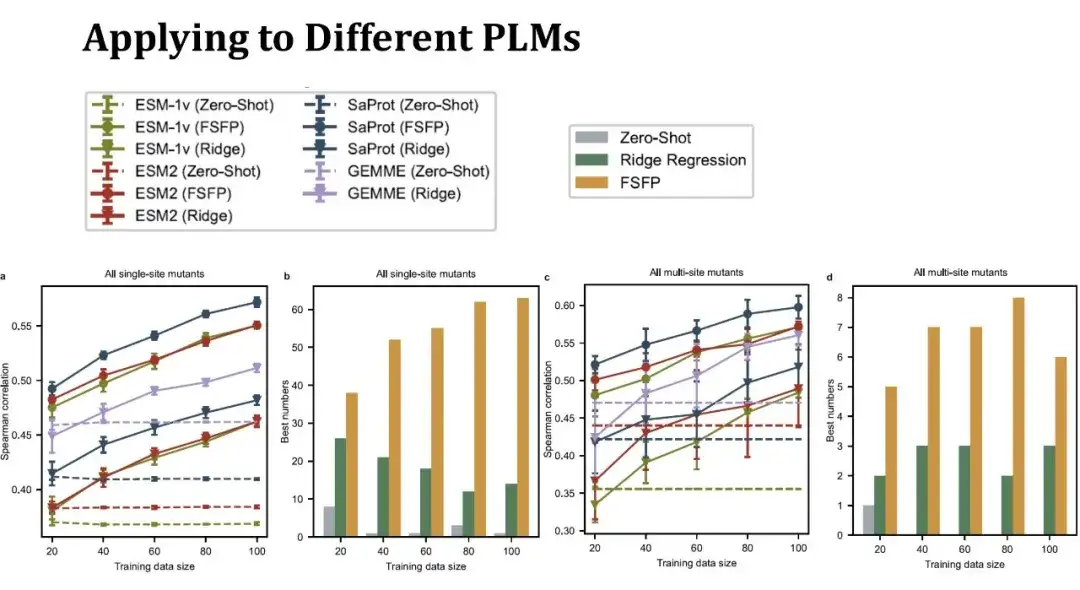
Das zweite Balkendiagramm zeigt die Anzahl der höchsten Punktzahlen, die mit den drei Strategien (Zero-Shot, Ridge-Regression und FSFP) in verschiedenen Datensätzen erzielt wurden. FSFP liefert in den meisten Datensätzen die beste Leistung.Die beiden Abbildungen rechts zeigen die Leistung bei der Vorhersage von Mehrfachpunktmutationen. Es handelt sich um 11 Datensätze mit Mehrfachpunktmutationen und die erhaltenen Schlussfolgerungen ähneln denen bei Einzelpunktmutationen. Allerdings weist das Ridge-Regressionsmodell hier eine größere Varianz auf, was darauf hindeutet, dass es empfindlich auf die Datenaufteilung reagiert.
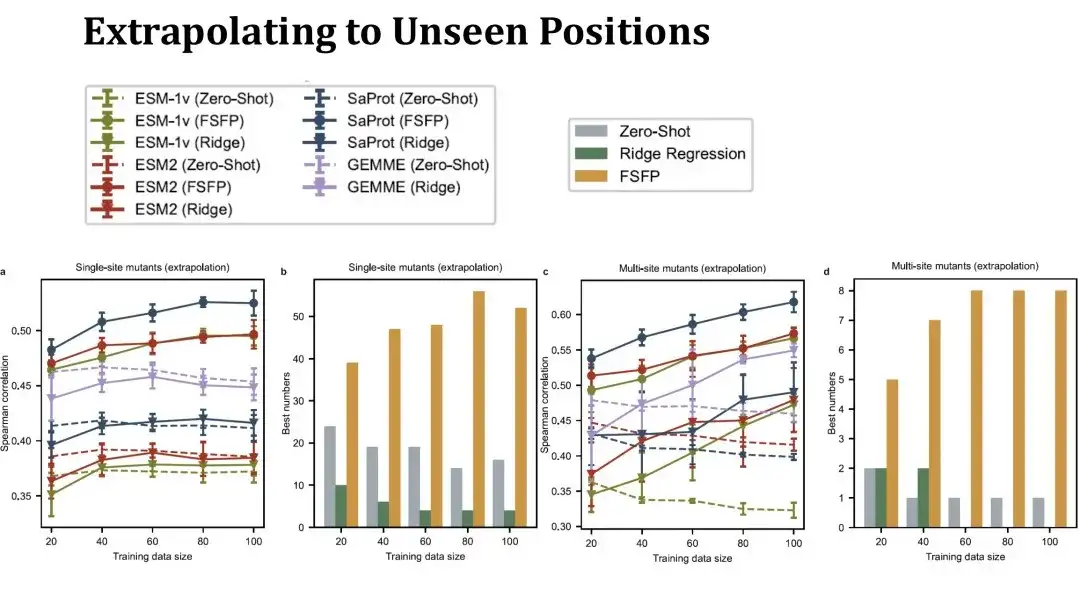
Anschließend haben wir die Extrapolationsleistung von FSFP ausgewertet und dabei insbesondere die Vorhersageleistung an Mutationsstellen bewertet, die im Trainingssatz nicht vorhanden waren.. In diesem Fall ist der Testsatz viel kleiner als zuvor und ändert sich erheblich, wenn der Trainingssatz größer wird, sodass die Zero-Shot-Leistung in der Tabelle keine gerade Linie mehr darstellt. Diese Einstellung ist anspruchsvoller. Wir können sehen, dass die Leistung der Einzelpunktmutations-Ridge-Regression auf der linken Seite kaum über Null hinausgehen kann, FSFP die Leistung jedoch immer noch stetig verbessern kann. Die Testergebnisse der Multipunktmutationen auf der rechten Seite zeigen auch, dass unsere Trainingsmethode über eine gute Generalisierungsfähigkeit verfügt.
Transformation von Phi29 mit FSFP
Darüber hinaus haben wir FSFP verwendet, um eine Fallstudie zur Proteinmodifikation durchzuführen.Das Zielprotein ist Phi29, eine DNA-Polymerase, und wir hoffen, sein Tm durch eine Punktmutation zu verbessern.
Der experimentelle Ablauf ist wie folgt: Verwenden Sie zunächst ESM-1v, um eine Zero-Shot-Bewertung gesättigter Einzelpunktmutationen durchzuführen, wählen Sie die 20 besten Mutationen aus und führen Sie Nassexperimente durch, um Tm zu messen. Verwenden Sie dann diese 20 experimentellen Daten als Trainingssatz, verwenden Sie FSFP, um ESM-1v zu trainieren, verwenden Sie das trainierte Modell, um die gesättigten Einzelpunktmutationen erneut zu bewerten, und wählen Sie die 20 besten Mutationen erneut zum Testen aus.
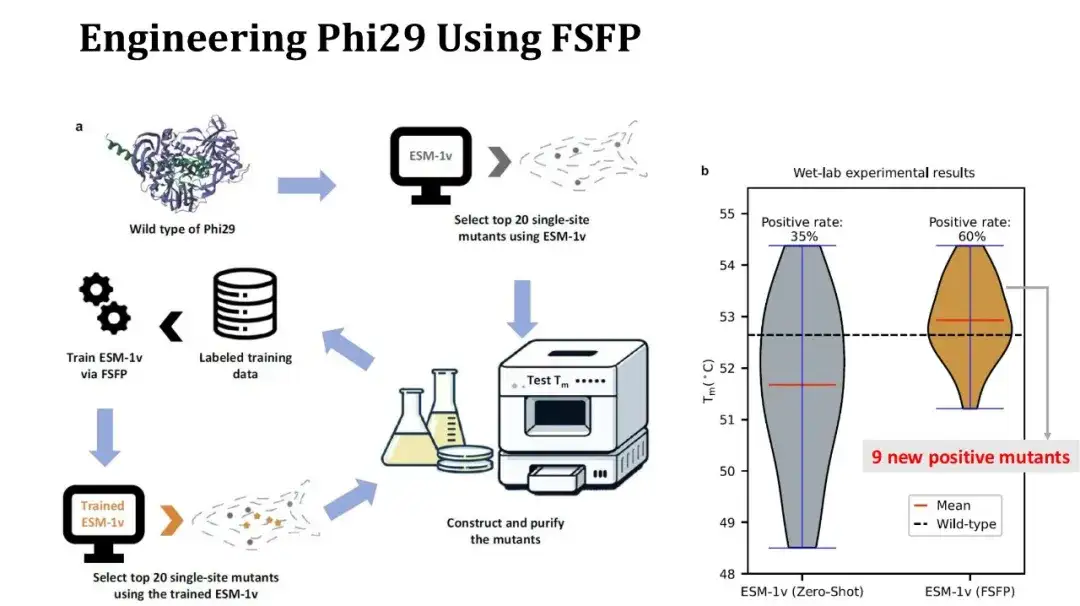
Die Abbildung rechts zeigt den Vergleich der Tm-Verteilung vor und nach den beiden Versuchsrunden. Sieben von 20 Mutationen in der ersten Runde waren positiv, in der zweiten Runde stieg diese Zahl auf 12, und der durchschnittliche Tm-Wert erhöhte sich um 1 Grad. Unter ihnen waren 9 der in der zweiten Runde gefundenen positiven Mutationen neu. Obwohl sich die Positivrate und die durchschnittliche Tm verbesserten, stieg die höchste Tm leider nicht an, da die Mutation mit der höchsten in der zweiten Runde erzielten Tm in den Ergebnissen der ersten Runde noch vorhanden war. Da jedoch mehr positive Einzelpunktmutationen erzielt wurden, können wir versuchen, diese Stellen zu kombinieren, um Experimente mit vielen Punktmutationen durchzuführen und so Tm weiter zu verbessern.
Zusammenfassung der FSFP-Methode und zukünftige Forschungsaussichten
FSFP ist eine Lernstrategie für PLM mit kleinen Stichproben, die die Leistung von PLM bei der Vorhersage von Mutationseffekten mithilfe einer kleinen Anzahl (Dutzende) gekennzeichneter Trainingsstichproben erheblich verbessern kann und flexibel auf eine Vielzahl verschiedener PLMs angewendet werden kann.Experimente zeigen, dass das Design von FSFP sinnvoll ist:
* Ranking-Lernen erfüllt die Grundanforderungen des Mutations-Rankings im Protein-Engineering und reduziert den Trainingsaufwand.
* LoRA reduziert das Risiko einer Überanpassung, indem es die Menge der trainierbaren Parameter von PLM kontrolliert;
* Meta-Learning kann gute Anfangsparameter für das Modell bereitstellen, sodass das Modell schnell zur Zielaufgabe migrieren kann.

Abschließend diskutieren wir die zukünftigen Richtungen der KI-gestützten gerichteten Evolution. Der allgemeine Prozess der KI-gestützten gerichteten Evolution besteht darin, mit einer Reihe anfänglicher Mutationen zu beginnen, ihre Fitness-Labels durch Nassexperimente zu erhalten und die aus den Experimenten zurückgemeldeten gekennzeichneten Daten zum Trainieren des maschinellen Lernmodells zu verwenden. Anschließend wird die nächste Runde der zu testenden Mutationen basierend auf den Vorhersagen des Modells ausgewählt und der Vorgang wiederholt.
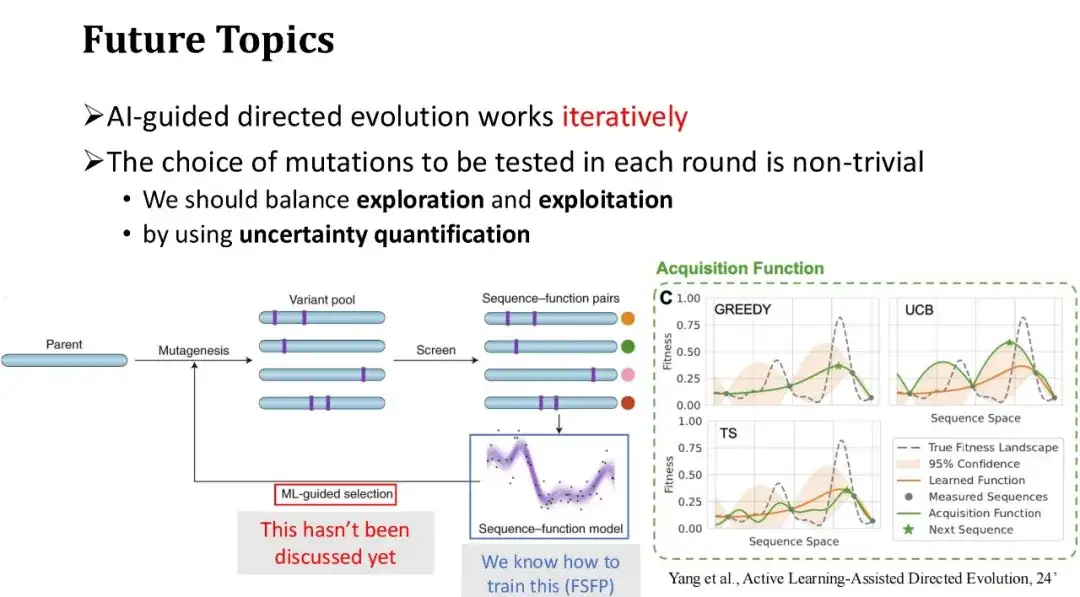
FSFP löst hauptsächlich das Problem des Trainings des Modells mit kleinen Stichproben in jeder Runde der experimentellen Iteration und verbessert die Vorhersagegenauigkeit des Modells.Wir haben jedoch noch nicht besprochen, wie die in der nächsten Runde zu testenden Mutationen, d. h. die neuen Trainingsbeispiele, die in der nächsten Runde hinzugefügt werden sollen, effektiv ausgewählt werden können. Im vorherigen Beispiel der Phi29-Proteinmodifikation haben wir direkt die 20 Mutationen mit den höchsten Modellwerten ausgewählt. In einem iterativen Szenario mit mehreren Runden ist die Strategie der gierigen Auswahl jedoch nicht unbedingt die beste Methode, da sie dazu neigt, in die lokale Optimalität zu fallen. Daher muss ein Gleichgewicht zwischen Exploration und Ausbeutung gefunden werden.
Tatsächlich handelt es sich bei dem Prozess der iterativen Auswahl von Testproben zur Kennzeichnung und der schrittweisen Erweiterung der Trainingsdaten um ein aktives Lernproblem, das im Bereich des Protein-Engineerings zu einigen Forschungsfortschritten geführt hat. Frances H. Arnold, eine Autorität auf dem Gebiet der gerichteten Evolution, diskutiert beispielsweise verwandte Themen in ihrem Artikel „Active Learning-Assisted Directed Evolution“.
Papieradresse:
https://www.biorxiv.org/content/10.1101/2024.07.27.605457v1.full.pdf
Wir können Techniken zur Quantifizierung der Unsicherheit verwenden, um die Unsicherheit des Modellwerts für jeden Mutanten zu bewerten. Aufgrund dieser Unsicherheiten wird die Auswahlstrategie für Testproben vielfältiger ausfallen.. Eine häufig verwendete Strategie ist die UCB-Methode, bei der Mutationsproben mit der höchsten Modellvorhersageunsicherheit für die nächste Annotationsrunde ausgewählt werden, d. h. Proben mit der größten Vorhersagevarianz werden bevorzugt. Dies ähnelt dem menschlichen Lernprozess: Wenn wir bestimmte Wissenspunkte nicht gut beherrschen oder uns diesbezüglich unsicher sind, konzentrieren wir uns darauf, unseren Lernfortschritt zu festigen.








