Command Palette
Search for a command to run...
Werden Die Neuen Ergebnisse Von DeepMind Kritisiert, Weil Sie Wie Werbung Aussehen? AlphaProteo Kann Effizient Zielproteinbinder Mit 300-fach Erhöhter Affinität Entwickeln
AlphaFold ist auf dem Gebiet der Proteinvorhersage weit voraus und AlphaFold 3 hat die Beschränkungen durchbrochen und die Vorhersage aller Lebensmoleküle erreicht. Bei der Veröffentlichung hieß es, dass die strukturierte Vorhersage und Generierung von AlphaFold 3 die Entwicklung von AIDD (Artificial Intelligence-driven Drug Design) beschleunigen werde.
Jetzt hat DeepMind einen weiteren Versuch in der Entwicklung von Medikamenten auf Basis künstlicher Intelligenz unternommen und AlphaProteo für die Entwicklung neuer Proteine herausgebracht.
Dieses neue Mitglied der Alpha-Familie zeigte auch eine revolutionäre Verbesserung der Fähigkeiten. Bei den sieben im Experiment getesteten Zielproteinen erzielte AlphaProteo nicht nur eine höhere experimentelle Erfolgsrate,Die Bindungsaffinität ist außerdem 3- bis 300-mal höher als bei den besten bestehenden Methoden.Gleichzeitig wandten die Forscher es auch auf das Zielprotein VEGF-A an, das mit Krebs und diabetischen Komplikationen in Zusammenhang steht.Ein Durchbruch wurde bei der Verwendung von KI-Tools beim Design von VEGF-A-Proteinkonjugaten erzielt.
Einstufiges Mitteldurchsatz-Screening generiert gebrauchsfertige Proteinbinder
Am 5. September Ortszeit schlugen Forscher von DeepMind AlphaProteo vor, ein maschinelles Lernmodell für das Proteindesign, das Zielproteinbinder mit hoher Erfolgsrate und hoher Affinität entwerfen kann und keine komplexen menschlichen Eingriffe erfordert. AlphaProteo erfordert nur eine Runde Mitteldurchsatz-Screening und erfordert keine weitere Optimierung, um „gebrauchsfertige“ Proteinbinder für viele Forschungsanwendungen zu erzeugen.
Modellstruktur: Kombination generativer Modelle mit Filtern zur effizienten Generierung von Zielproteinbindern
AlphaProteo besteht aus 2 Komponenten, wie in der folgenden Abbildung dargestellt.
Ein generatives Modell (Generator),Trainiert anhand der Struktur- und Sequenzdaten der Protein Data Bank (PDB) und des Destillationssatzes der von AlphaFold vorhergesagten Strukturen, um zu lernen, wie Moleküle aneinander binden;Ein Filter,Wird verwendet, um die generierten Designs zu bewerten und vorherzusagen, ob der Binder im Experiment erfolgreich an das Zielprotein binden kann.
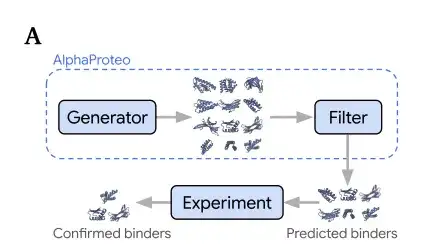
Um Zielproteinbinder zu entwerfen, geben die Forscher zunächst die Struktur des Zielproteins ein und wählen die „Hotspot“-Reste des Zielprotein-Epitops aus, um die bevorzugte Bindungsposition abzuleiten. Das generative Modell gibt die Kandidaten-Binderstruktur und -sequenz für das Zielprotein aus. Der Filter filtert diese vorhergesagten Bindemittel vor der experimentellen Prüfung auf eine kleinere Menge heraus. In Benchmarktests mit Computersimulationen schnitt das generative Modell besser ab als die besten bestehenden Methoden.
Experimentelles Fazit: Übertrifft die besten bestehenden Lösungen, mit hoher Erfolgsquote und starker Affinität
Die Forscher verwendeten AlphaProteo, um Binder für acht Zielproteine mit unterschiedlichen Strukturen zu entwerfen und sie experimentell zu testen. Darunter befinden sich zwei virale Proteine, die mit Infektionen in Zusammenhang stehen, BHRF1 und die SARS-CoV-2-Spike-Protein-Rezeptor-Bindungsdomäne SC2RBD, sowie fünf Proteine, die mit Krebs, Entzündungen und Autoimmunerkrankungen in Zusammenhang stehen, IL-7Rɑ, PD-L1, TrkA, IL-17A und VEGF-A, und ein Protein TNF, das mit Autoimmunerkrankungen wie rheumatoider Arthritis in Zusammenhang steht.
Wie in der Abbildung unten gezeigt,Die sieben von AlphaProteo erzeugten Zielproteinbinder konnten bei experimentellen Tests fest an das Zielprotein binden.Für das achte Ziel, TNF, gelang es jedoch nicht, erfolgreiche Bindemittel zu entwickeln.
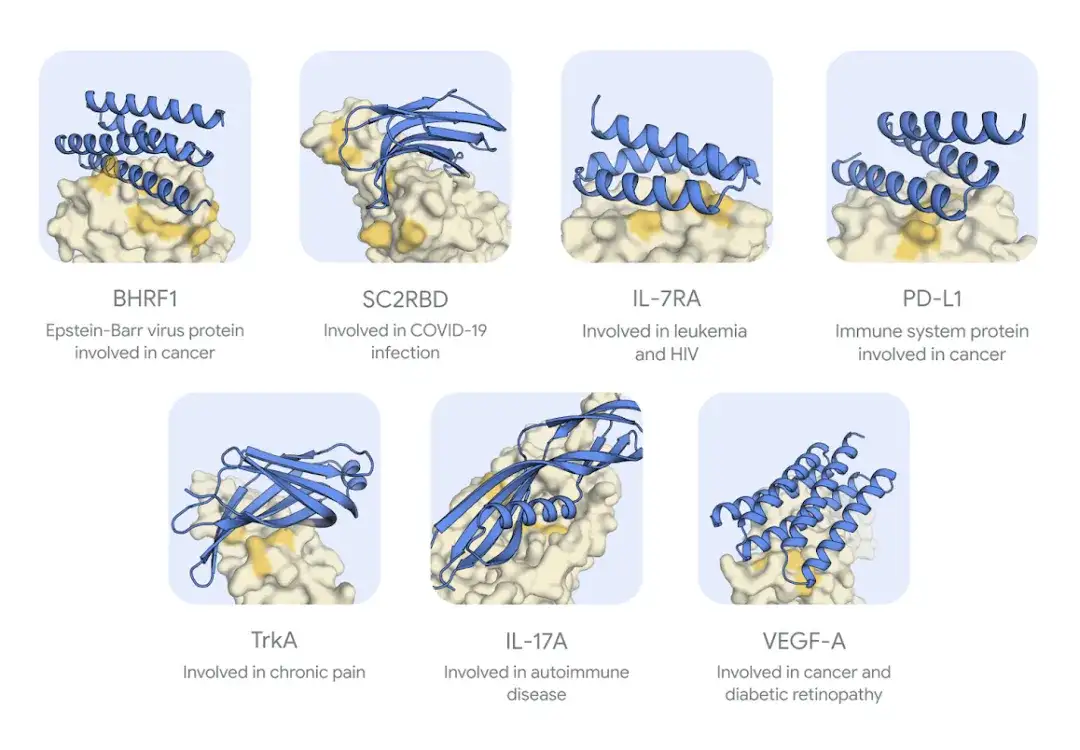
* Blau zeigt den Binder an, Gelb zeigt das Proteinziel an, Dunkelgelb ist die erwartete Bindungsregion
Darüber hinaus verglichen die Forscher, wie in der Abbildung unten gezeigt, AlphaProteo mit anderen Designmethoden, um die In-vitro-Erfolgsrate zu vergleichen. sich herausstellen,Bei Nasslabortests konnten die 9%-88%-Bindemittelkandidaten in 5- bis 100-mal höheren Konzentrationen erfolgreich an das Zielprotein binden als andere Methoden.
Unter ihnen weisen die von AlphaProteo generierten Kandidatenmoleküle für das virale Protein BHRF1 eine Bindungserfolgsrate von 88% mit dem Zielprotein auf, und die Bindungsstärke ist zehnmal so hoch wie bei der bestehenden besten Designmethode. Für das Protein TrkA ist der neue Binder stärker als der am besten konzipierte Binder, der nach mehreren Versuchsrunden für dieses Protein optimiert wurde.
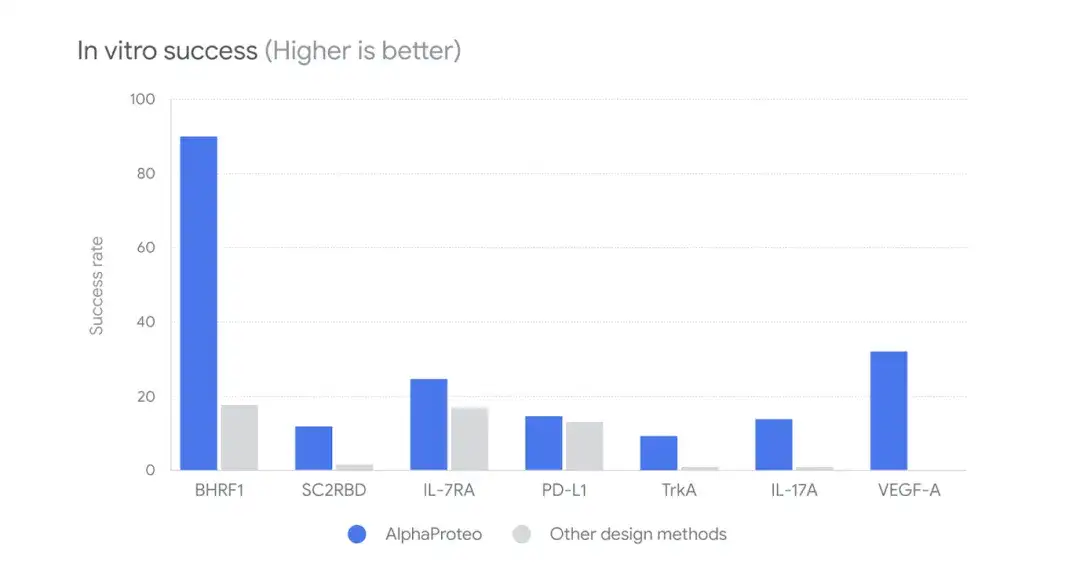
Darüber hinaus untersuchten die Forscher die beste Affinität der von AlphaProteo entwickelten Bindemittel zu Zielproteinen im Vergleich zu anderen Designmethoden ohne experimentelle Optimierung. sich herausstellen,Von AlphaProteo erzeugte Bindemittel weisen Bindungsaffinitäten auf, die etwa 300- bis 300-mal höher sind als die von Bindemitteln, die mit den besten verfügbaren Methoden entwickelt wurden.
* Je größer die Affinität, desto stärker die Bindung
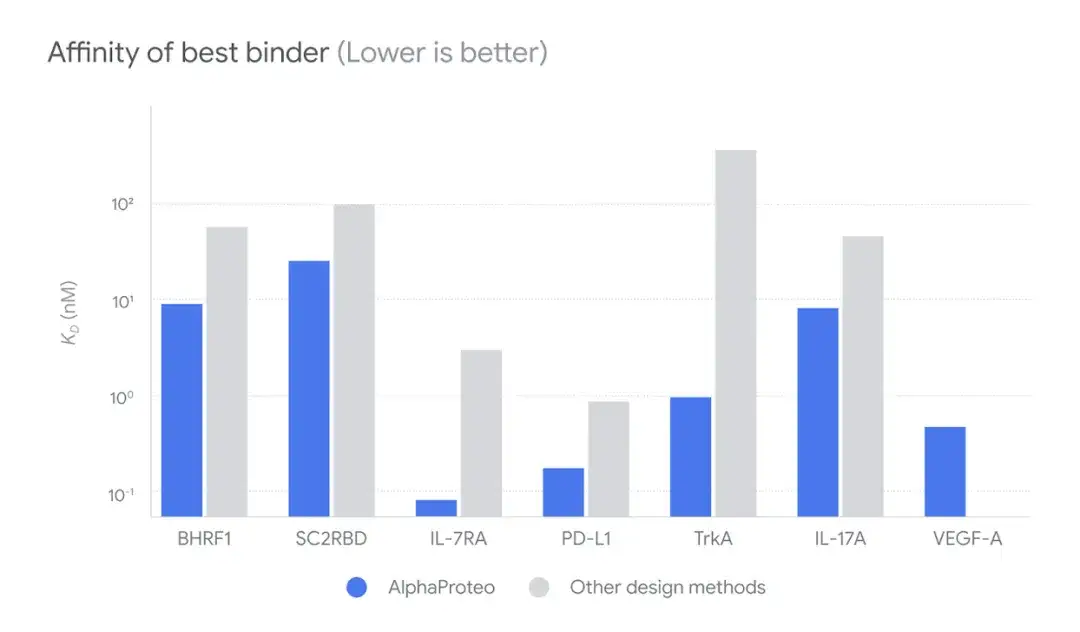
Die Forscher untersuchten weiter die Ähnlichkeiten zwischen stärkeren Bindern von SC2RBD und VEGF-A-Protein und denen von AlphaProteo und fanden heraus, dassDie Bindungsinteraktionen dieser stärkeren Bindemittel ähnelten den von AlphaProteo vorhergesagten, was auf die Zuverlässigkeit von AlphaProteo hinweist.
Darüber hinaus bestätigten die Forscher auch, dass diese Bindemittel tatsächliche biologische Funktionen haben. Beispielsweise wurde gezeigt, dass einige SC2RBD-Binder SARS-CoV-2 und seine Varianten daran hindern, Zellen zu infizieren. Noch wichtiger ist, dass AlphaProteo breit anwendbar ist und kein Hochdurchsatz-Screening oder Affinitätsoptimierungsexperimente erfordert, was die anfängliche Versuchszeit für die Entwicklung von Proteinbindern erheblich verkürzen kann.
Zusammenarbeit mit Europas größtem biomedizinischen Forschungslabor
Zu den ersten Experimenten, die das neue Proteindesignsystem AlphaProteo testen, gehören drei Teams des Francis Crick Institute.
Das Francis Crick Institute befindet sich in London, England. Es ist derzeit das größte biomedizinische Forschungslabor in Europa und ein Forschungszentrum in Großbritannien. Es wurde gemeinsam vom National Institute for Medical Research (NIMR) und Cancer Research UK initiiert und kostete etwa 700 Millionen Pfund.
Bis heute beschäftigt das Crick Institute über 2.000 Forscher und über 100 Forschungsgruppen. Darüber hinaus ist das Institut eine einzigartige Partnerschaft zwischen sechs der weltweit führenden biomedizinischen Forschungsorganisationen, darunter dem Medical Research Council, Cancer Research UK, dem Wellcome Trust, dem University College London, dem Imperial College London und dem King’s College London.
Offizielle Website-Adresse des Francis Crick Institute:
* https://www.crick.ac.uk/
Die neuesten Ergebnisse von Deepmind stoßen auf gemischte Kritiken
Forschungsbedeutung vs. Open Source
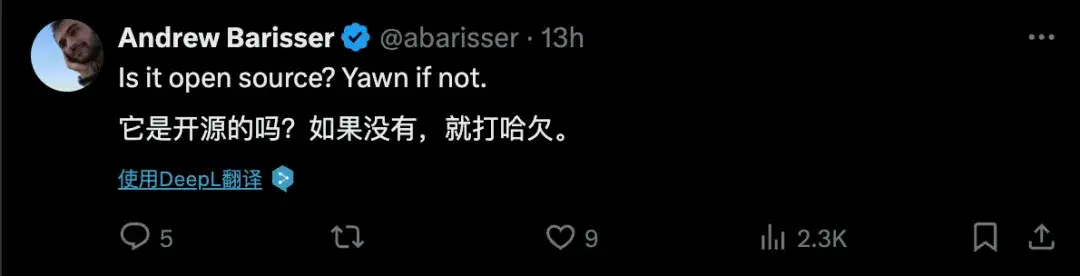
Obwohl der hohe Wert von AlphaProteo in der praktischen Forschung für alle offensichtlich ist, sind viele Internetnutzer eher daran interessiert, ob dieses Tool Open Source ist. Ein Internetnutzer meinte: „Ist es Open Source? Wenn nicht, wäre es zu langweilig.“
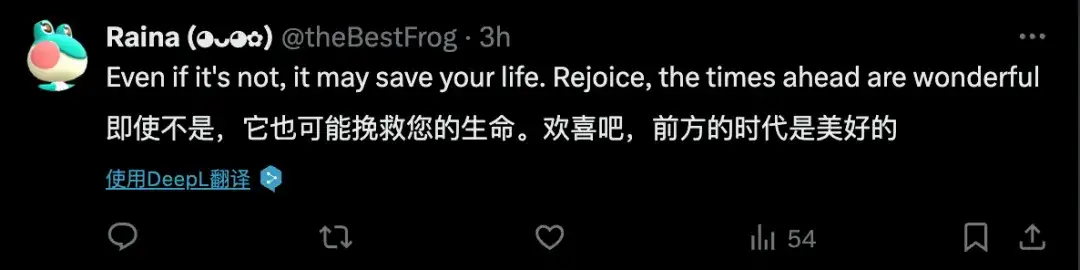
Einige andere Internetnutzer sagten:Für die Rettung von Leben ist AlphaProteo wichtiger als die Frage, ob es Open Source ist.Und äußerte sich optimistisch hinsichtlich seines Entwicklungspotenzials.
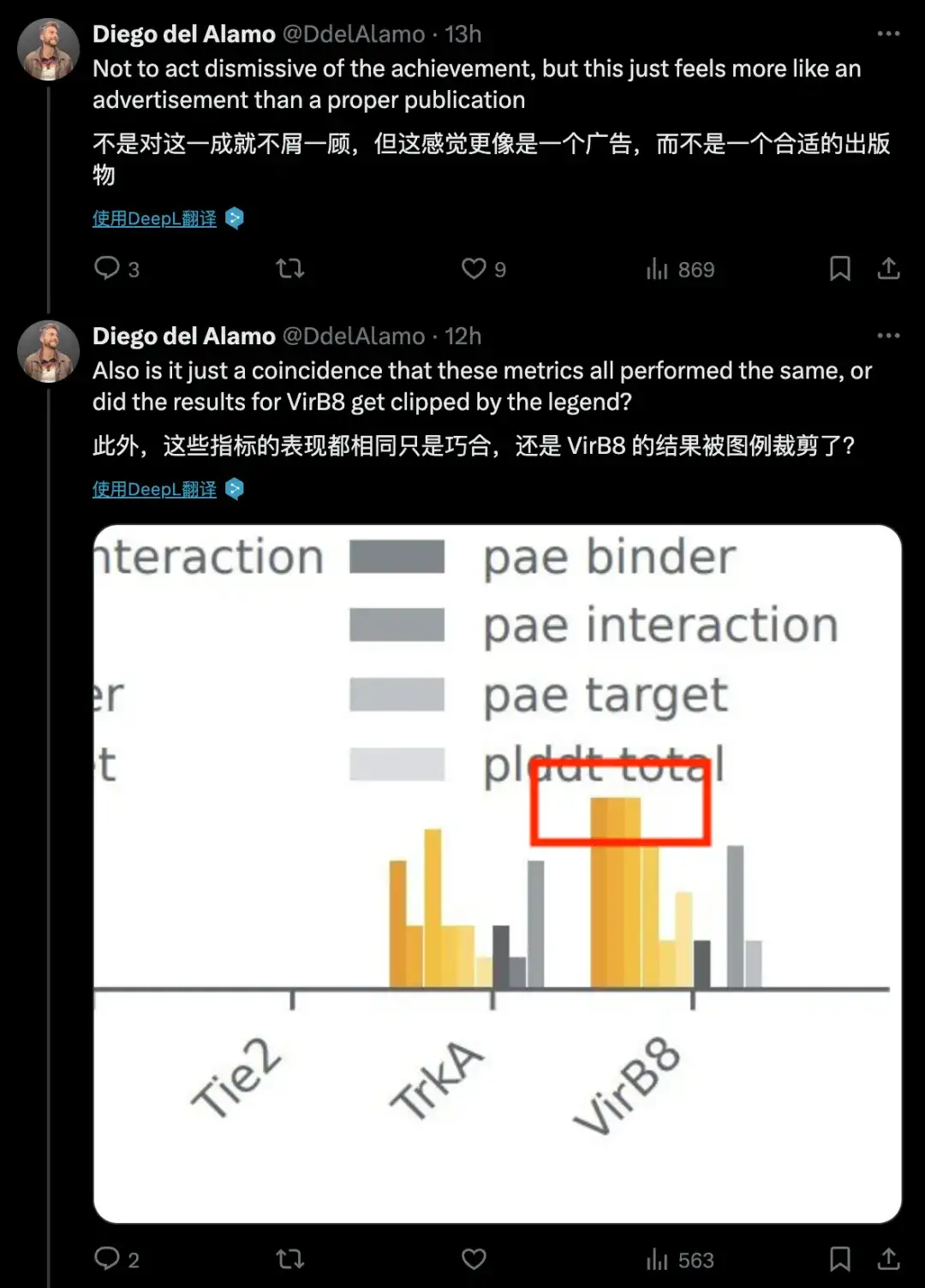
Die methodischen Details sind zu spärlich, es sieht eher aus wie eine Anzeige als wie ein Papier
Das DeepMind-Team hat ein Dokument für das Modell verfasst, das jedoch nur sehr wenige oder sogar fast keine Details zu den Methoden des Modells enthält. Ein Wissenschaftler eines weltweit führenden Pharma- und Gesundheitsunternehmens veröffentlichte drei Updates hintereinander und sagte: „Dieses Whitepaper enthält zu wenige methodologische Details“ und „Es wirkt eher wie eine Werbung als eine richtige Veröffentlichung“, und stellte dieselben Legendenangaben im Papier in Frage.
Ein anderer promovierter Biologe vom MIT betonte nicht nur die Bedeutung der Open-Source-Wissenschaft, sondern kritisierte auch den Schritt von DeepMind: „DeepMind ist zu weit gegangen und ich möchte wirklich wissen, wie es funktioniert.“
DeepMind eröffnet ein neues Schlachtfeld und ergänzt AlphaFold
DeepMinds „Upgrade und Monster-Kampf“ für die biopharmazeutische Industrie hat ein neues Schlachtfeld eröffnet. Kann es ein Anstoß sein, die Umsetzung der KI-basierten Arzneimittelentwicklung zu beschleunigen?
Wie wir alle wissen, ist AlphaFold 3 bereits in der Lage, die Strukturen und Wechselwirkungen aller Lebensmoleküle (Proteine, DNA, RNA, Liganden usw.) mit höchster Präzision vorherzusagen. Was die zukünftige Iterationsrichtung angeht, prognostizieren Branchenkenner, dass es zu Verbesserungen bei der Vorhersage kinetischer Informationen kommen könnte.Das diesmal erschienene AlphaProteo kann sich in gewissem Maße mit AlphaFold ergänzen und gegenseitig fördern.
einerseits,Beim Proteindesign muss man von der Funktion ausgehen und neue oder verbesserte Proteinstrukturen entwerfen, um bestimmte biologische oder chemische Funktionen zu erreichen. Daher ist es in hohem Maße vom Verständnis der Beziehung zwischen Proteinstruktur und -funktion abhängig.auf der anderen Seite,Die Proteinvorhersage kann wichtige Strukturinformationen liefern und als Leitfaden für das Design dienen. Gleichzeitig können die entworfenen Proteine auch dazu beitragen, die Vorhersagemodelle zu verbessern und die Genauigkeit der Vorhersagen in Aspekten wie Proteinfaltung und -dynamik zu erhöhen.
DeepMind erwähnte in seiner Einführung zu AlphaProteo, dass diese Innovation das Verständnis der Menschen für biologische Prozesse beschleunigen und bei der Entdeckung neuer Medikamente, der Entwicklung von Biosensoren und vielem mehr helfen könnte. In Zukunft wird das Unternehmen mit der wissenschaftlichen Gemeinschaft zusammenarbeiten, um mithilfe von AlphaProteo weitreichende biologische Fragen zu beantworten und seine Grenzen zu verstehen. Gleichzeitig hat DeepMind mit Isomorphic Labs auch seine Anwendungen im Arzneimitteldesign erkundet.
Isomorphic Labs ist aus DeepMind hervorgegangen und konzentriert sich auf den Bereich der KI-Medizin. Seine Mission besteht darin, künstliche Intelligenz und Methoden des maschinellen Lernens zu nutzen, um den Prozess der Arzneimittelentdeckung zu beschleunigen und zu verbessern und so Behandlungsmöglichkeiten für einige der verheerendsten Krankheiten der Menschheit zu finden.
Isomorphic Labs ist gewissermaßen auch der Tentakel von DeepMind im Bereich der KI-Medizin, der erstmals die Anwendung seiner neuesten Ergebnisse im Arzneimitteldesign gezielt erforschen kann. Das Team war auch an der Forschung zu AlphaFold 3 beteiligt. Wir erwarten, dass es mit der Unterstützung von DeepMind gute Neuigkeiten für die Umsetzung der KI-basierten Arzneimittelentwicklung bringen wird.
Abschließend empfehle ich eine akademische Austauschaktivität!
Zur dritten Live-Übertragung von Meet AI4S war Zhou Ziyi eingeladen, ein Postdoktorand am Institut für Naturwissenschaften der Shanghai Jiao Tong University und dem Shanghai National Center for Applied Mathematics. Am 25. September wird Dr. Zhou Ziyi in Form einer Online-Liveübertragung die Methode zum Lernen kleiner Stichproben des Proteinsprachenmodells mit allen teilen und neue Ideen für die gerichtete Evolution mit Unterstützung durch KI erkunden. Klicken Sie hier, um einen Termin für die Live-Übertragung zu vereinbaren!








