Command Palette
Search for a command to run...
Die Neueste Anwendung Von SAM 2 Ist Jetzt Verfügbar! Das Team Der Universität Oxford Veröffentlicht Medical SAM 2 Und Aktualisiert Damit Die SOTA-Liste Der Medizinischen Bildsegmentierung
Im April 2023 veröffentlichte Meta das Segment Anything Model (SAM) und behauptete, „alles segmentieren“ zu können. Es war wie eine Bombe, die das gesamte Feld der Computervision erschütterte und von vielen sogar als eine Forschung angesehen wurde, die traditionelle CV-Aufgaben unterwanderte.
Nach mehr als einem JahrMeta hat ein weiteres Meilenstein-Update veröffentlicht – SAM 2, das eine suggestive Objektsegmentierung in Echtzeit für statische Bilder und dynamische Videoinhalte ermöglicht und Bild- und Videosegmentierungsfunktionen in dasselbe System integriert.Wie Sie sich vorstellen können, hat diese starke Position es der Branche ermöglicht, die Erforschung der SAM-Anwendungen in verschiedenen Bereichen zu beschleunigen, insbesondere im Bereich der medizinischen Bildsegmentierung. Viele Labore und akademische Forschungsteams betrachten es bereits als die einzige Wahl für medizinische Bildsegmentierungsmodelle.
Bei der sogenannten medizinischen Bildsegmentierung geht es darum, die Teile mit besonderer Bedeutung in medizinischen Bildern zu segmentieren und relevante Merkmale zu extrahieren, wodurch eine zuverlässige Grundlage für klinische Diagnosen, pathologische Forschung usw. geschaffen wird.
In den letzten Jahren hat sich die Segmentierung auf Basis neuronaler Netzwerkmodelle mit der kontinuierlichen Weiterentwicklung der Deep-Learning-Technologie allmählich zur gängigen Methode der medizinischen Bildsegmentierung entwickelt, und automatisierte Segmentierungsmethoden haben die Effizienz und Genauigkeit erheblich verbessert. Jedoch,Angesichts der Besonderheiten des Bereichs der medizinischen Bildsegmentierung gibt es noch einige Herausforderungen, die bewältigt werden müssen.
Das erste ist die Modellgeneralisierung.Für bestimmte Ziele (wie Organe oder Gewebe) trainierte Modelle lassen sich nur schwer an andere Ziele anpassen. Daher ist es häufig erforderlich, entsprechende Modelle für unterschiedliche Segmentierungsziele neu zu entwickeln.Der zweite Grund sind Datenunterschiede.Viele für die Computervision entwickelte Standard-Deep-Learning-Frameworks sind für 2D-Bilder konzipiert, in der medizinischen Bildgebung liegen die Daten jedoch normalerweise im 3D-Format vor, wie etwa bei CT-, MRT- und Ultraschallbildern. Dieser Unterschied verursacht zweifellos große Probleme beim Modelltraining.
Um die oben genannten Probleme zu lösen,Das Team der Universität Oxford entwickelte ein medizinisches Bildsegmentierungsmodell namens Medical SAM 2 (MedSAM-2).Das Modell basiert auf dem SAM 2-Framework und behandelt medizinische Bilder wie Videos. Es eignet sich nicht nur gut für die Segmentierung medizinischer 3D-Bilder, sondern bietet auch eine neue Funktion zur Einzeleingabeaufforderungssegmentierung. Der Benutzer muss lediglich einen Hinweis für ein neues bestimmtes Objekt geben, und die Segmentierung ähnlicher Objekte in nachfolgenden Bildern kann vom Modell ohne weitere Eingaben automatisch abgeschlossen werden.
Die entsprechenden Arbeiten und Ergebnisse wurden auf der Preprint-Plattform arXiv unter dem Titel „Medical SAM 2: Segment medical images as video via Segment Anything Model 2“ veröffentlicht.
Forschungshighlights:
* Das Team war Pionier des medizinischen Bildsegmentierungsmodells MedSAM-2 basierend auf SAM 2
* Das Team hat ein neuartiges Konzept „medizinische Bilder als Videos“ übernommen und damit die „Single-Prompt-Segmentierungsfunktion“ freigeschaltet.

Papieradresse:
https://arxiv.org/pdf/2408.00874
Direkter Download des SA-V-Videosegmentierungsdatensatzes:
Beispieldatensatz für medizinische Segmentierung von Medical SAM 2:
Das Open-Source-Projekt „awesome-ai4s“ vereint mehr als 100 AI4S-Papierinterpretationen und stellt umfangreiche Datensätze und Tools bereit:
https://github.com/hyperai/awesome-ai4s
Datensatz: Klassifikationsdesign, umfassende Auswertung
Das Team führte Experimente mit fünf verschiedenen medizinischen Bildsegmentierungsdatensätzen unter Verwendung automatisch generierter Maskenhinweise durch, die in zwei Kategorien unterteilt wurden:
Die erste Kategorie zielt darauf ab, die allgemeine Segmentierungsleistung zu bewerten,Das Team entschied sich für die Aufgabe der abdominalen Multiorgansegmentierung und wählte den BTCV-Datensatz aus, der 12 anatomische Strukturen enthält.
Die zweite Kategorie zielt darauf ab, die Generalisierungsfähigkeit des Modells über verschiedene Bildgebungsmodalitäten hinweg zu bewerten.Die Forscher verwendeten den REFUGE2-Datensatz zur Bildsegmentierung der Papille und des Augenbechers. der BraTs 2021-Datensatz zur Segmentierung von Hirntumoren in MRT-Scans; Der TNMIX-Benchmark wurde verwendet, um Schilddrüsenknoten in Ultraschallbildern zu segmentieren, die aus 4.554 Bildern von TNSCUI und 637 Bildern von DDTI bestehen. und der ISIC-Datensatz 2019 wurde verwendet, um Hautläsionen in Melanome oder Nävus zu segmentieren.
Darüber hinaus richtete das Team zehn weitere 2D-Bildsegmentierungsaufgaben ein, um die Einzelhinweis-Segmentierungsfähigkeit des Modells durch die Verwendung unterschiedlicher Hinweistypen weiter zu bewerten. Insbesondere verwenden die Datensätze KiTS23, ATLAS23, TDSC und WBC die Point-Hinting-Technologie. die Datensätze SegRap, CrossM23 und REFUGE verwenden BBox-Hinweise (Bounding Box); Die Datensätze CadVidSet, STAR und ToothFairy verwenden Maskenhinweise.
Modellarchitektur: Effektive Segmentierungsverarbeitung für medizinische Bilder unterschiedlicher Dimensionen
Die Architektur von MedSAM-2 ähnelt grundsätzlich der von SAM 2, das Forschungsteam hat jedoch zusätzlich ein einzigartiges und effizientes Verarbeitungsmodul und eine Pipeline dafür entwickelt, kombiniert mit einer Vertrauensspeicherbank und einer gewichteten Aufnahmestrategie, um die Fähigkeiten des Modells technisch zu garantieren.
Speziell,Die Architektur von MedSAM-2 ist in der folgenden Abbildung dargestellt.Enthalten:
* Image Encoder, abstrahiert die Eingabe in Einbettung
* Memory Attention, das in der Speicherbank gespeicherte Erinnerungen nutzt, um die Eingabeeinbettung anzupassen
* Memory Decoder, der die vorhergesagte Frame-Einbettung abstrahiert
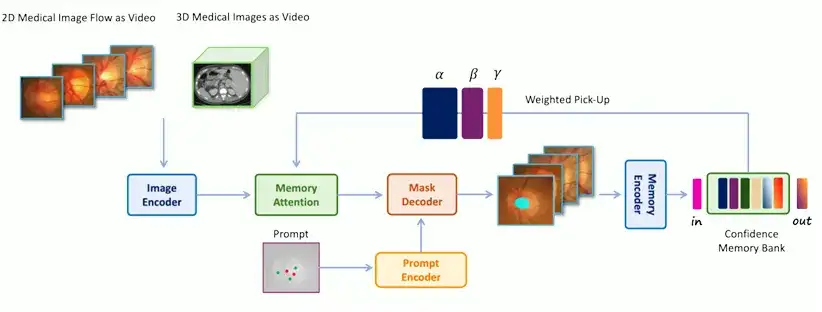
Der Encoder und Decoder im Netzwerk ähneln denen in SAM. Der Encoder besteht aus einem hierarchischen visuellen Transformator, und der Decoder enthält einen leichten bidirektionalen Transformator, der Prompt-Embedding und Image-Embedding integriert, wobei das Prompt-Embedding vom Prompt-Encoder generiert wird. Die Memory-Attention-Komponente besteht aus einer Reihe gestapelter Aufmerksamkeitsblöcke, von denen jeder einen Self-Attention-Block und einen Cross-Attention-Mechanismus enthält.
Es ist erwähnenswert, dassEine wichtige Neuerung von MedSAM-2 besteht darin, die medizinische Bildverarbeitung als Videosegmentierung zu behandeln.Dies ist der Schlüssel zur Verbesserung der Leistung der 3D-Segmentierung medizinischer Bilder und zur Freischaltung der „Single-Prompt-Segmentierungsfunktion“. Zu diesem Zweck hat das Team auch zwei verschiedene Betriebsprozesse für 2D- und 3D-medizinische Bilder entwickelt, um eine effektive Segmentierungsverarbeitung für medizinische Bilder unterschiedlicher Abmessungen durchzuführen.
Für die medizinische 3D-BildverarbeitungDa in medizinischen 3D-Bildern eine starke zeitliche Korrelation zwischen benachbarten Schichten besteht, ähnelt die Verarbeitungsmethode der von Videodaten. Das ursprüngliche Speichersystem von SAM 2 wird verwendet, um vorherige Scheiben und ihre entsprechenden Vorhersagen für die kontinuierliche Scheibensegmentierung abzurufen. Die Einbettung des Eingabebildes wird dann durch den Memory Attention-Mechanismus verbessert und die Segmentierungsergebnisse werden wieder zum Speicherbereich hinzugefügt, um die Segmentierung nachfolgender Slices zu unterstützen.
Für die 2D-Bildverarbeitung im medizinischen Bereich,Die Verarbeitungsmethode unterscheidet sich von der zeitlichen First-In-First-Out-Warteschlange, die in SAM 2 verwendet wird. Stattdessen wird eine Gruppe medizinischer Bilder, die dasselbe Organ oder Gewebe enthalten, in einem „medizinischen Bildstrom“ zusammengefasst und ein „Confidence-First“-Speicherbereich wird zum Speichern der Vorlagen des Modells verwendet. Die Konfidenz wird auf Grundlage der vom Modell vorhergesagten Wahrscheinlichkeit berechnet und es werden Bilddiversitätsbeschränkungen implementiert. Beim Zusammenführen der eingebetteten Eingabebilder und der Speicherbereichsinformationen wird eine gewichtete Auswahlstrategie angewendet. Während der Trainingsphase wird ein Kalibrierungskopf verwendet, um sicherzustellen, dass die Modellvorhersagen genauer sind. Letztendlich können wir eine automatische Segmentierung von Zielen mit nur einer Beispielaufforderung ohne jegliche zeitliche Zuordnung erreichen.
Experimentelle Ergebnisse: MedSAM-2 führt bei Leistung und Generalisierungsfähigkeit
Das Forschungsteam verwendete IoU (Intersection over Union) und Dice Score, um die Leistung des Modells bei der medizinischen Bildsegmentierung zu bewerten, und führte die Metrik Hausdorff Distance (HD95) ein, um die Genauigkeit der Leistungsbewertung sicherzustellen.
*LoU, auch als Jaccard-Index bekannt, ist eine Metrik, die zur Bewertung der Genauigkeit eines Objektdetektors anhand eines bestimmten Datensatzes verwendet wird.
* Der Dice Score, auch als Dice Coefficient bekannt, ist ein statistisches Tool zum Vergleichen der Ähnlichkeiten zwischen zwei Stichproben.
* Die Hausdorff-Distanz (HD95) ist eine Metrik, die verwendet wird, um den Grad der Differenz zwischen zwei Punktsätzen zu bestimmen. Es wird häufig verwendet, um die Genauigkeit von Objektgrenzen bei Bildsegmentierungsaufgaben zu bewerten und ist besonders effektiv bei der Quantifizierung des Worst-Case-Abstands zwischen der vorhergesagten Segmentierung und der tatsächlichen Grenze.
Zunächst hat das Team MedSAM-2 mit einer Reihe von SOTA-Methoden zur Segmentierung medizinischer Bilder verglichen, darunter auch Segmentierungsaufgaben für 2D- und 3D-medizinische Bilder. Bei medizinischen 3D-Bildern wird der Hinweis zufällig mit einer Wahrscheinlichkeit von 0,25 bereitgestellt; Bei 2D-medizinischen Bildern beträgt die Wahrscheinlichkeit 0,3.
Um die allgemeine Leistung des vorgeschlagenen Modells bei medizinischen 3D-Bildern zu bewerten,Das Team verglich MedSAM-2 mit fortschrittlichen Segmentierungsmethoden, die auf dem BTCV-Datensatz zur Multiorgansegmentierung basieren, darunter die bekannten Modelle nnUNET, TransUNet, UNetr, Swin-UNetr und diffusionsbasierte Modelle (wie EnsDiff, SegDiff und MedSegDiff). Darüber hinaus führte das Team auch vergleichende Bewertungen interaktiver Segmentierungsmodelle durch, beispielsweise des ursprünglichen SAM, des vollständig optimierten MedSAM, SAMed, SAM-Med2D, SAM-U, VMN und FCFI. Die Leistung wird mithilfe des Dice Score quantifiziert und die Ergebnisse sind in der folgenden Abbildung dargestellt:
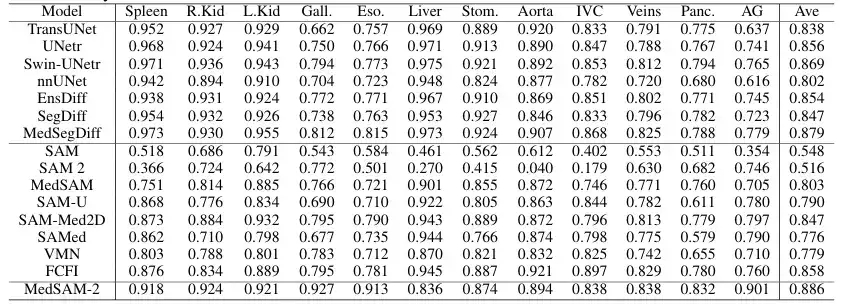
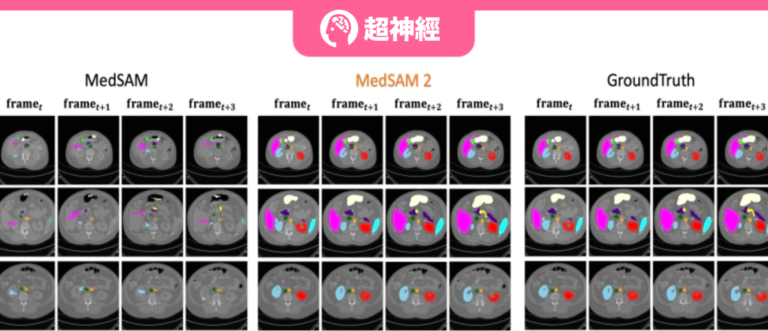
Die Ergebnisse zeigten, dass MedSAM-2 eine deutliche Verbesserung gegenüber früheren SAM- und MedSAM-Modellen darstellte. Im BTCV-Datensatz erzielt MedSAM-2 bei der Multiorgan-Segmentierungsaufgabe eine hervorragende Leistung und erreicht einen endgültigen Dice-Score von 88,57%. Unter den interaktiven Modellen behauptet MedSAM-2 seine führende Position und übertrifft das bisher führende interaktive Modell Med-SA um 2,78%. Alle diese interaktiven Modelle erfordern Eingabeaufforderungen für jeden Frame, während MedSAM-2 mit weniger Eingabeaufforderungen bessere Ergebnisse erzielt.
Bei der 2D-Segmentierung medizinischer BilderDas Team verglich MedSAM-2 mit Methoden, die auf bestimmte Aufgaben in verschiedenen Bildmodalitäten zugeschnitten sind. Insbesondere wurde es für die Segmentierung des Augenbechers mit ResUnet und BEAL verglichen. zur Segmentierung von Hirntumoren wurde es mit TransBTS und SwinBTS verglichen; für die Segmentierung von Schilddrüsenknoten wurde es mit MTSeg und UltraUNet verglichen; Zur Segmentierung von Hautläsionen wurde es mit FAT-Net und BAT verglichen. Darüber hinaus hat das Team auch das interaktive Modell einem Benchmarking unterzogen. Die Ergebnisse sind in der folgenden Abbildung dargestellt:
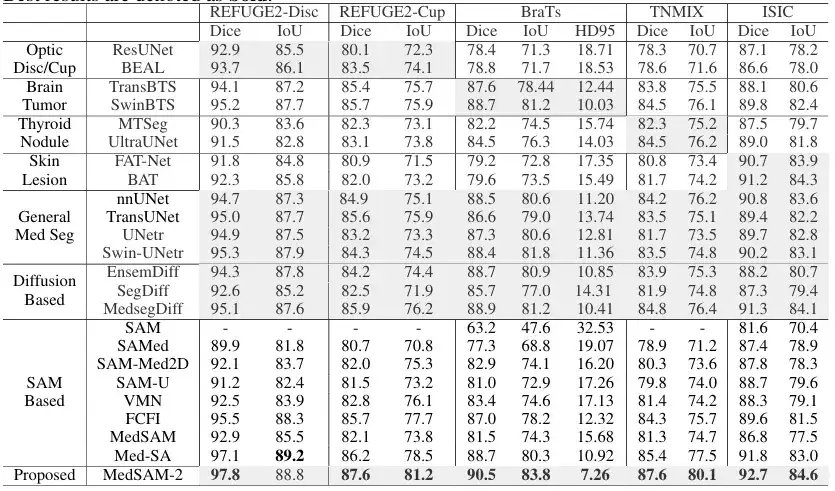
Die Ergebnisse zeigen, dass MedSAM-2 bei fünf verschiedenen Aufgaben allen anderen Methoden überlegen ist und seine hervorragende Generalisierungsfähigkeit bei verschiedenen medizinischen Bildsegmentierungsaufgaben unter Beweis stellt. Insbesondere erreichte MedSAM-2 eine Verbesserung von 2,01 TP3T bei Augenbechern, 1,61 TP3T bei Hirntumoren und 2,81 TP3T bei Schilddrüsenknoten. Im interaktiven Modellvergleich behält MedSAM-2 weiterhin seine Spitzenleistung.
endlich,Das Team bewertete auch die Leistung von MedSAM-2 bei nur einem einzigen Hinweis.Und es gibt keine klare Verbindung zwischen aufeinanderfolgenden Bildern, was die Fähigkeit von MedSAM-2 zur Einzelbild-Cue-Segmentierung weiter bestätigt. Insbesondere verglich das Team MedSAM-2 mit PANet, ALPNeu, SENet und UniverSeg, die alle mit nur einer einzigen Eingabeaufforderung getestet wurden. Darüber hinaus verglich das Team MedSAM-2 mit Einzellinsenmodellen wie DAT, ProbONE, HyperSegNas und One-prompt.
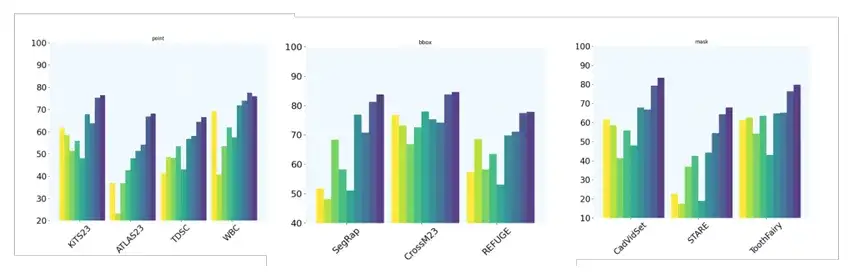
Die Ergebnisse zeigen, dass MedSAM-2 über eine Vielzahl von Aufgaben hinweg robuste Generalisierungsfähigkeiten aufweist und sogar im Vergleich zum intensiv trainierten One-Prompt gut abschneidet und nur bei einer der zehn Aufgaben zurückfällt. Darüber hinaus zeigt MedSAM-2 in Szenarien, in denen alle Methoden Masken bereitstellen, einen deutlicheren Vorteil und übertrifft den zweiten Platz häufig im Durchschnitt um 3,1%, was den größten Abstand zwischen allen Eingabeaufforderungseinstellungen darstellt.
SAM unterstützt die Forschung zur Segmentierung medizinischer Bilder auf Hochtouren
Die Veröffentlichung dieses Artikels kann als eine weitere eingehende Untersuchung des Potenzials von SAM und SAM 2 im medizinischen Bereich angesehen werden. Es bietet eine neue Idee und Methode für den Bereich der medizinischen Bildsegmentierung und zeigt insbesondere großes Potenzial und Wert in klinischen Anwendungen. Es kann den Arbeitsaufwand bei der Segmentierung medizinischer Bilder erheblich reduzieren und die Effizienz und Genauigkeit der Segmentierung medizinischer Bilder verbessern.
Erwähnenswerter ist, dass, wie am Anfang des Artikels erwähnt,Viele Labore und akademische Teams erforschen das Potenzial von SAM.Auf dem Gebiet der medizinischen Bildsegmentierung gibt es mehr als nur das in diesem Artikel erwähnte Team der Universität Oxford.
Zufälligerweise startete das Team von Professor Ni Dong von der School of Biomedical Engineering der School of Medicine der Universität Shenzhen kurz nach der Veröffentlichung von SAM in Zusammenarbeit mit der Universität Oxford, der ETH Zürich, der Zhejiang-Universität, dem Shenzhen People’s Hospital und Duying Medical umfassende Experimente und Bewertungen aus mehreren Blickwinkeln zur Anwendung von SAM bei medizinischen Bildgebungsaufgaben. Die entsprechenden Arbeiten und Ergebnisse wurden im internationalen Top-Journal im Bereich der medizinischen Bildanalyse, „Medical Image Analysis“, unter dem Titel „Segment Anything Model for Medical Images?“ veröffentlicht.

Im Rahmen der Recherche für dieses Dokument hat das zuständige Team schließlich einen ultragroßen medizinischen Bildsegmentierungsdatensatz COSMOS 1050K erstellt, der 18 Bildgebungsmodalitäten, 84 biomedizinische Segmentierungsziele, 1050.000 2D-Bilder und 6033.000 Segmentierungsmasken umfasst. Auf der Grundlage dieses Datensatzes führten die Forscher eine umfassende Evaluierung von SAM durch und untersuchten Möglichkeiten zur Verbesserung der Fähigkeiten von SAM bei der medizinischen Zielwahrnehmung.
Direkter Download des COSMOS 1050K-Datensatzes zur Segmentierung medizinischer Bilder:
Darüber hinaus haben Teams der School of Big Data der Fudan University und der School of Biomedical Engineering der Shanghai Jiao Tong University eine Reihe von Studien zu SAM im Bereich der medizinischen Bildsegmentierung durchgeführt. Das entsprechende Papier trägt den Titel „Segment anything model for medical image segmentation: Current applications and future directions“ und ist auf bekannten akademischen Websites und in Zeitschriften wie arXiv und Computer in Biology and Medicine enthalten.

In diesem Artikel geht es um die mögliche Anwendung von SAM, das bemerkenswerte Erfolge bei der natürlichen Bildsegmentierung erzielt hat, im Bereich der medizinischen Bildsegmentierung. Außerdem werden die Feinabstimmung des SAM-Moduls und die Umschulung ähnlicher Architekturen zur Anpassung an die medizinische Bildsegmentierung untersucht.
Papieradresse:
https://www.sciencedirect.com/science/article/abs/pii/S0010482524003226
Zusammenfassend lässt sich sagen, dass Wissenschaftler, wie in den oben genannten Artikeln erörtert, durch die Erforschung des Potenzials von SAM die Verarbeitung und Analyse medizinischer Bilder einfacher und effizienter gemacht haben, was für die Wissenschaft, die medizinische Gemeinschaft und sogar die Patienten ein erfreuliches Ergebnis darstellt. Gleichzeitig hat die Veröffentlichung allgemeiner Bildsegmentierungsmodelle wie SAM auch eine magische Tür zu verschiedenen Bereichen geöffnet.Ich glaube, dass nicht nur der Bereich der medizinischen Bildgebung, sondern auch autonomes Fahren, neue Medien, AR/VR usw. in Zukunft stark davon profitieren könnten.
Buchverlosung

HyperAI und der Verlag der Elektronikindustrie haben Ihnen gemeinsam kostenlose Bücher gebracht! Wir haben 5 äußerst nützliche populärwissenschaftliche Bücher „KI für die Wissenschaft: Künstliche Intelligenz treibt wissenschaftliche Innovation voran“ vorbereitet. Kommen Sie und nehmen Sie an der Verlosung teil~
So können Sie teilnehmen
Folgen Sie dem offiziellen WeChat-Konto von HyperAI, antworten Sie im Hintergrund mit „AI4S-Gratisbuch“ und klicken Sie auf die Verlosungsseite, um an der Verlosung teilzunehmen. Wir haben 5 Bücher für Sie vorbereitet, die Ihnen per Expressversand zugestellt werden. Komm und mach mit!
Bucheinführung
Von der Vorhersage der Proteinstruktur bis hin zur Ableitung der Pathogenität von Genmutationen hat uns das neue, von der KI geprägte Paradigma neue Möglichkeiten in zahlreichen wissenschaftlichen Bereichen, einschließlich der Biowissenschaften, eröffnet.
Das Buch „KI für die Wissenschaft: Künstliche Intelligenz fördert wissenschaftliche Innovationen“ konzentriert sich auf die bereichsübergreifende Integration künstlicher Intelligenz in fünf große Bereiche: Materialwissenschaften, Biowissenschaften, Elektronik, Energiewissenschaften und Umweltwissenschaften. Es verwendet eine leicht verständliche Sprache, um grundlegende Konzepte, technische Prinzipien und Anwendungsszenarien umfassend vorzustellen und ermöglicht es den Lesern, sich schnell die Grundkenntnisse der KI für die Wissenschaft anzueignen. Darüber hinaus bietet das Buch für jeden bereichsübergreifenden Bereich eine detaillierte Einführung anhand von Fallbeispielen, gliedert die Branchenübersicht und vermittelt relevante politische Erkenntnisse.








