Command Palette
Search for a command to run...
Ausgewählt Für ACL 2024! Um Eine Modalübergreifende Interpretation Von Proteindaten Und Textinformationen Zu Erreichen, Schlug Das Team Von Wang Xiang Vom USTC Das Protein-Text-Generierungsframework ProtT3 Vor

Die Erforschung der Geheimnisse der dynamischen Struktur von Proteinen ist nicht nur ein entscheidender Schritt zur Förderung der Entwicklung neuer Medikamente, sondern auch ein wichtiger Grundstein für das Verständnis von Lebensprozessen. Aufgrund der Komplexität von Proteinen ist es jedoch schwierig, ihre tiefen Strukturinformationen direkt zu erfassen und zu analysieren. Die Umwandlung komplexer biologischer Daten in intuitive und leicht verständliche Ausdrücke war im Bereich der wissenschaftlichen Forschung schon immer eine große Herausforderung.
Mit der rasanten Entwicklung von Sprachmodellen (LM) entstand eine innovative Idee:Da Sprachmodelle Textinformationen aus großen Datenmengen lernen und extrahieren können, stellt sich die Frage, ob sie auch lernen können, Proteininformationen aus Proteindaten zu „lesen“ und dynamische Proteinstrukturinformationen direkt in für Menschen leicht verständliche Textbeschreibungen umzuwandeln?
Diese vielversprechende Idee ist in der praktischen Anwendung auf viele Herausforderungen gestoßen. Beispielsweise wird das Sprachmodell anhand eines Textkorpus aus Proteinsequenzen vortrainiert. Obwohl es über starke Textverarbeitungsfähigkeiten verfügt, ist es nicht in der Lage, die nicht-menschliche „Sprache“ der Proteinstruktur zu verstehen. Im Gegensatz dazu sind Proteinsprachenmodelle (PLMs) anhand von Proteinsequenzkorpora vortrainiert und verfügen über ausgezeichnete Fähigkeiten zum Verständnis und zur Generierung von Proteinen.Die Einschränkung ist jedoch ebenso gravierend: Es fehlen Funktionen zur Textverarbeitung.
Wenn wir die Vorteile von PLMs und LM kombinieren können, um eine neue Modellarchitektur zu erstellen, die nicht nur die Proteinstruktur gründlich verstehen, sondern auch Textinformationen nahtlos verknüpfen kann, wird dies tiefgreifende Auswirkungen auf die Arzneimittelentwicklung, die Vorhersage von Proteineigenschaften, das Moleküldesign und andere Bereiche haben. Jedoch,Proteinstrukturen und Texte in menschlicher Sprache gehören zu unterschiedlichen Datenmodalitäten und es ist nicht einfach, die Barrieren zu durchbrechen und sie zusammenzuführen.
In diesem ZusammenhangWang Xiang von der University of Science and Technology of China hat zusammen mit dem Team von Liu Zhiyuan von der National University of Singapore und dem Forschungsteam der Universität Hokkaido ein neues Protein-Text-Modellierungsframework namens ProtT3 vorgeschlagen.Das Framework kombiniert PLM und LM mit Modalitätsunterschieden durch einen kreuzmodalen Projektor, wobei PLM zum Proteinverständnis und LM zur Textverarbeitung verwendet wird. Um eine effiziente Feinabstimmung zu erreichen, haben die Forscher LoRA in LM integriert, um den Prozess der Protein-zu-Text-Generierung effektiv zu regulieren.
Darüber hinaus haben die Forscher auch quantitative Bewertungsaufgaben für Protein-Text-Modellierungsaufgaben erstellt, darunter Protein-Beschriftung, Protein-Fragen-Beantwortung (Protein-QA) und Protein-Text-Retrieval. ProtT3 erzielte bei allen drei Aufgaben eine hervorragende Leistung.
Die Forschung mit dem Titel „ProtT3: Protein-to-Text Generation for Text-based Protein Understanding“ wurde für die Top-Konferenz ACL 2024 ausgewählt.
Forschungshighlights:
* Das ProtT3-Framework kann die Modalitätslücke zwischen Text und Protein schließen und die Genauigkeit der Proteinsequenzanalyse verbessern
* Bei der Protein-Captioning-Aufgabe liegt der BLEU-2-Score von ProtT3 in den Datensätzen Swiss-Prot und ProteinKG25 um mehr als 10 Punkte über dem Basiswert
* Bei der Protein-Frage-Antwort-Aufgabe verbesserte sich die exakte Übereinstimmungsleistung von ProtT3 im PDB-QA-Datensatz um 2,5%
* Bei der Protein-Text-Retrieval-Aufgabe ist die Abrufgenauigkeit von ProtT3 in den Datensätzen Swiss-Prot und ProteinKG25 um mehr als 14% höher als der Basiswert
Papieradresse:
https://arxiv.org/abs/2405.12564
Adresse zum Herunterladen des Datensatzes:
https://go.hyper.ai/j0wvp
Das Open-Source-Projekt „awesome-ai4s“ vereint mehr als 100 AI4S-Papierinterpretationen und stellt umfangreiche Datensätze und Tools bereit:
https://github.com/hyperai/awesome-ai4s
Aufbau und Optimierung dreier wichtiger Datensätze für die Proteinforschung
Die Forscher wählten drei Datensätze aus: Swiss-Prot, ProteinKG25 und PDB-QA.
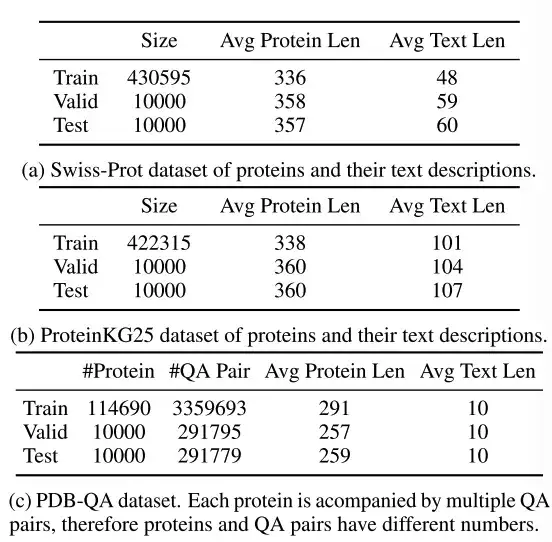
Wie in der obigen Tabelle gezeigt,Swiss-Prot ist eine Proteinsequenzdatenbank mit Textanmerkungen.Die Forscher verarbeiteten den Datensatz und schlossen Proteinnamen aus den Textanmerkungen aus, um Informationslecks zu verhindern. Die generierte Textbeschreibung verknüpft Anmerkungen zur Proteinfunktion, zum Standort und zur Familie.
ProteinKG25 ist ein Wissensgraph, der aus der Gene Ontology-Datenbank abgeleitet ist.Die Forscher aggregierten zunächst Tripletts desselben Proteins und füllten dann die Proteininformationen in eine vordefinierte Textvorlage, um die Tripletts in freien Text umzuwandeln.
PDB-QA ist ein Protein-Single-Turn-Frage-Antwort-Datensatz, der aus RCSB PDB2 abgeleitet ist.Enthält 30 Fragenvorlagen zur Proteinstruktur, zu Proteineigenschaften und zu ergänzenden Informationen. Wie in der folgenden Tabelle gezeigt, haben die Forscher die Fragen zur detaillierten Auswertung in vier Kategorien unterteilt, basierend auf dem Antwortformat (Zeichenfolge oder Zahl) und dem inhaltlichen Schwerpunkt (Struktur/Attribut oder ergänzende Informationen).

ProtT3: Eine innovative Modellarchitektur zur Protein-zu-Text-Generierung
Wie in Abbildung a unten gezeigt,ProtT3 besteht aus einem Proteinsprachenmodell (PLM), einem kreuzmodalen Projektor (Cross-ModalProjector), einem Sprachmodell (LM) und einem LoRA-Modul.Regulieren Sie effektiv den Protein-zu-Text-Generierungsprozess.
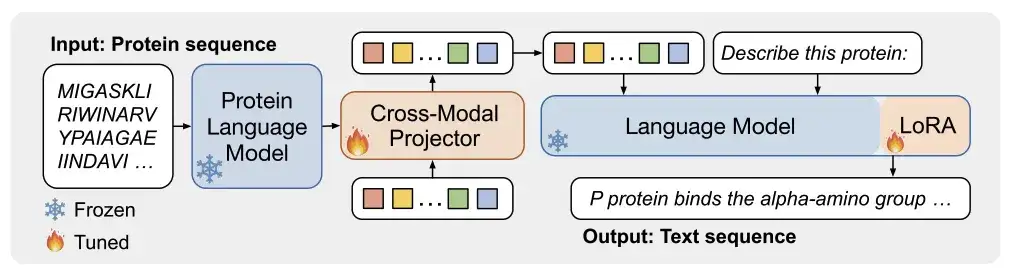
Unter ihnen ist das von den Forschern ausgewählte Proteinsprachenmodell ESM-2150M, das zum Verständnis von Proteinen verwendet wird; Der ausgewählte kreuzmodale Projektor ist Q-Former, der verwendet wird, um die modalen Unterschiede zwischen PLM und dem Sprachmodell LM zu überbrücken und dann die Proteindarstellung auf den Textraum von LM abzubilden. Das ausgewählte Sprachmodell ist Galactica1.3B, das für die Textverarbeitung verwendet wird. Um die Effizienz der nachgelagerten Anpassung aufrechtzuerhalten, haben die Forscher auch LoRA in das Sprachmodell integriert, um eine effiziente Feinabstimmung zu erreichen.
Wie in Abbildung b gezeigt,ProtT3 verwendet zwei Trainingsphasen, um die effektive Modellierung von Proteintexten zu verbessern.Dabei handelt es sich um Protein-Text Retrieval Training und Protein-to-Text Generation Training.
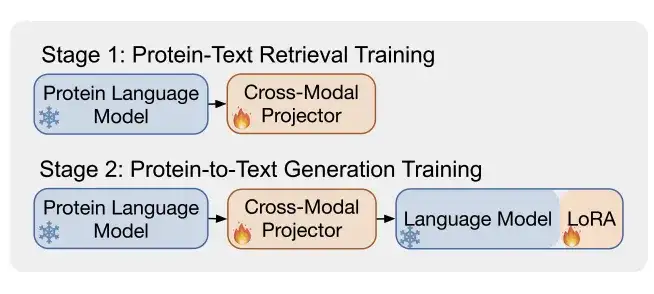
* Phase 1: Protein-Text-Retrieval-Training
Wie in Abbildung a unten dargestellt, besteht der kreuzmodale Projektor Q-Former aus zwei Transformatoren: Proteintransformatoren für die Proteinkodierung und Texttransformatoren für die Textverarbeitung. Zwei Transformatoren teilen sich die Selbstaufmerksamkeit, um die Interaktion zwischen Proteinen und Text zu ermöglichen.
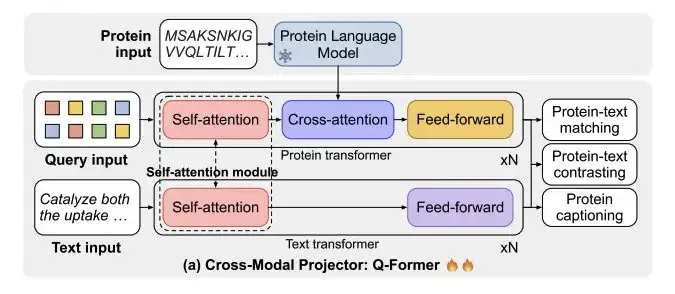
Die Forscher trainierten ProtT3 anhand eines kombinierten Datensatzes aus Swiss-Prot und ProteinKG25 für die Proteintextsuche.Es umfasst drei Aufgaben: Protein-Text-Kontrastierung, Protein-Text-Matching (PTM) und Protein-Untertitelung (PCap).
* Phase 2: Training zur Protein-zu-Text-Generierung
Die Forscher verbanden den Cross-Modal-Projektor mit einem Sprachmodell (LM) und speisten die Proteindarstellung Z in das LM ein, um den Textgenerierungsprozess durch Proteininformationen zu konditionieren. Unter anderem verwendeten die Forscher eine lineare Schicht, um Z auf dieselbe Dimension der Sprachmodelleingabe zu projizieren, trainierten ProtT3 für jeden generierten Datensatz separat und fügten nach der Proteindarstellung verschiedene Textaufforderungen hinzu, um den Generierungsprozess weiter zu steuern.
Darüber hinaus führten die Forscher LoRA ein und optimierten es individuell anhand von drei Datensätzen für die Aufgabe der Protein-zu-Text-Generierung.
Ein Allrounder im Proteinbereich, der die Leistung von ProtT3 in drei Hauptaufgaben bewertet
Um die Leistung von ProtT3 zu bewerten,Die Forscher testeten das System in drei Aufgaben: Proteinbeschriftung, Protein-Qualitätssicherung und Proteintextabruf.
ProtT3 kommt der wahren Beschreibung von Proteinen näher und hat eine höhere Genauigkeit
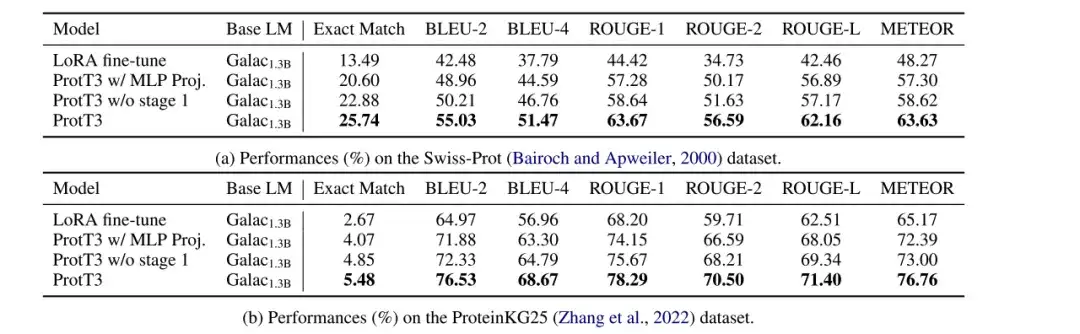
Die Forscher bewerteten die Leistung der mit LoRA feinabgestimmten Modelle Galactica1.3B, ProtT3 mit MLP Proj., ProtT3 ohne Stufe 1 und ProtT3 bei Protein-Captioning-Aufgaben in den Datensätzen Swiss-Prot und ProteinKG25 und verwendeten BLEU, ROUGE und METEOR als Bewertungsmetriken.
* ProtT3 mit MLP-Projektion: Eine Variante von ProtT3, die den Cross-Modal-Projektor von ProtT3 durch MLP ersetzt
* ProtT3 ohne Stufe 1: Eine Variante von ProtT3, die die Trainingsstufe 1 von ProtT3 überspringt
Wie in der Abbildung unten gezeigt, im Vergleich mit LoRA fein abgestimmtem Galactica1.3B,ProtT3 verbessert den BLEU-2-Score um mehr als 10 Punkte.Die Bedeutung der Einführung eines Proteinsprachenmodells und die Wirksamkeit von ProtT3 beim Verständnis des Protein-Inputs werden intuitiv demonstriert. Darüber hinaus übertrifft ProtT3 seine beiden Varianten in verschiedenen Metriken, was den Vorteil der Verwendung des Q-Former-Projektors und der Trainingsphase 1 zeigt.
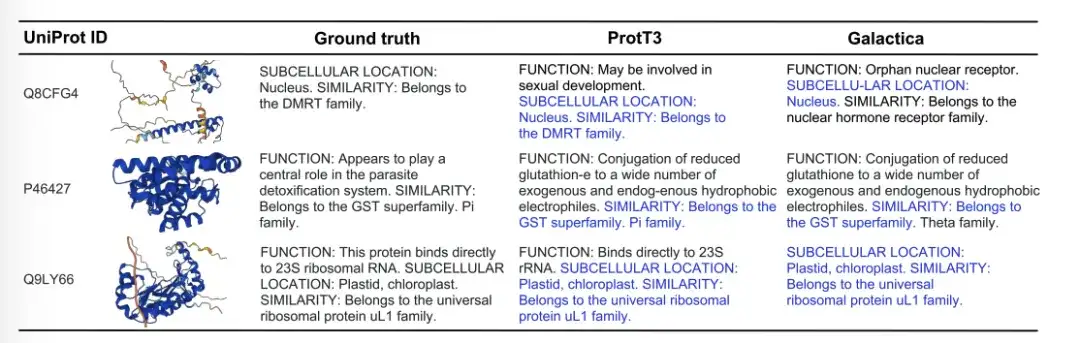
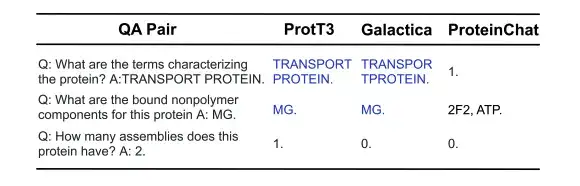
Die folgende Abbildung zeigt drei Beispiele für die Generierung von Proteinuntertiteln von Ground Truth, ProtT3 und Galactica. Im Beispiel Q8CFG4 identifizierte der Annotationsinhalt von ProtT3 die DMRT-Familie genauer, während dies bei Galactica nicht der Fall war. Im Fall von P46427 konnten beide Modelle die Funktion des Proteins nicht identifizieren, ProtT3 lieferte jedoch eine genauere Vorhersage der Proteinfamilie. Im Fall von Q9LY66 konnten beide Modelle den subzellulären Ort und die Proteinfamilie erfolgreich vorhersagen. ProtT3 geht bei der Vorhersage der Funktion von Proteinen einen Schritt weiter und kommt der wahren Beschreibung näher.
Die Genauigkeit ist 141 % höher als beim Basismodell. TP3T, ProtT3 hat eine bessere Fähigkeit zur Proteintext-Abrufung
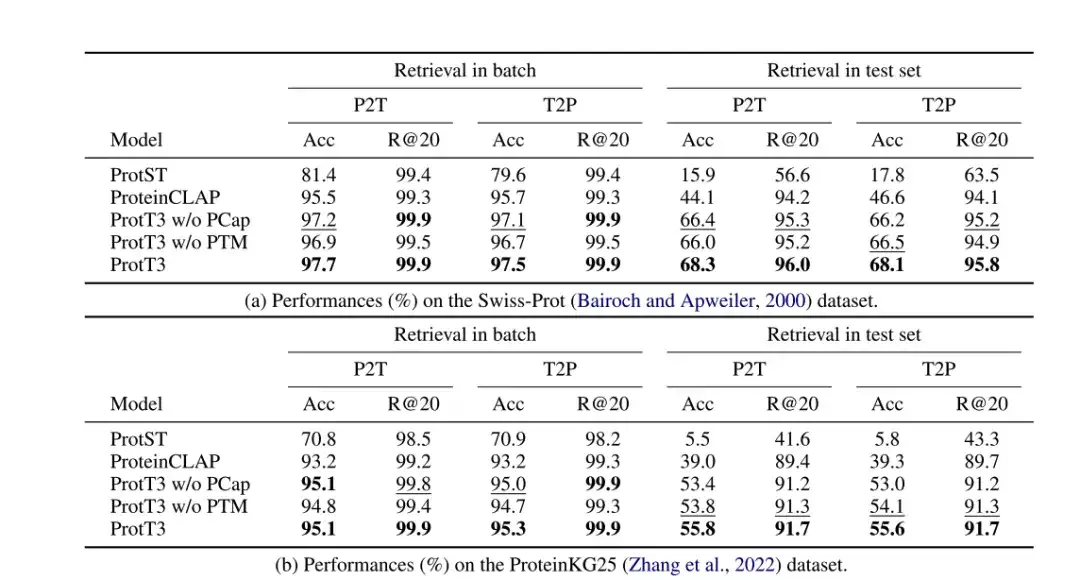
Die Forscher bewerteten die Leistung von ProtT3 bei der Proteintextsuche anhand der Datensätze Swiss-Prot und ProteinKG25. Dabei verwendeten sie Genauigkeit und Recall@20 als Bewertungsmaßstäbe und übernahmen ProtST und ProteinCLAP als Basismodelle.
Wie in der folgenden Tabelle gezeigt,Die Genauigkeit von ProtT3 ist mehr als 14% höher als die des Basismodells.Dies deutet darauf hin, dass ProtT3 bei der Zuordnung von Proteinen zu ihren entsprechenden Textbeschreibungen überlegen ist. Auch,Protein-Text-Matching (PTM) verbesserte die Genauigkeit von ProtT3 um 1%-2%,Dies liegt daran, dass PTM die Interaktion von Protein- und Textinformationen in den frühen Schichten von Q-Former ermöglicht und so ein feineres Maß für die Protein-Text-Ähnlichkeit erreicht.Protein Captioning (PCap) verbessert die Abrufgenauigkeit von ProtT3 um etwa 2%.Dies liegt daran, dass PCap Abfrage-Token dazu anregt, die für die Texteingabe relevantesten Proteininformationen zu extrahieren, was die Protein-Text-Ausrichtung unterstützt.
* ProtT3 ohne PTM: Überspringen Sie die PTM-Phase von ProtT3
* ProtT3 ohne PCap: Überspringen Sie die PCap-Phase von ProtT3
ProtT3 kann Proteinstruktur und -eigenschaften vorhersagen und verfügt über bessere Fähigkeiten zur Frage- und Antwortbeantwortung
Die Forscher bewerteten die Leistung von ProtT3 bei der Beantwortung von Proteinfragen anhand des PDB-QA-Datensatzes, wobei sie die exakte Übereinstimmung als Bewertungsmaßstab wählten und Galactica1.3B, feinabgestimmt durch LoRA, als Basismodell (LoRA ft) verwendeten.
Wie in der Abbildung unten gezeigt,Die exakte Übereinstimmungsleistung von ProtT3 ist 2,51-mal höher als die des Basis-TP3T.Es übertrifft die Basislinie bei der Vorhersage der Proteinstruktur und -eigenschaften durchweg und zeigt, dass ProtT3 über hervorragende multimodale Fähigkeiten zum Verständnis von Protein- und Textfragen verfügt.
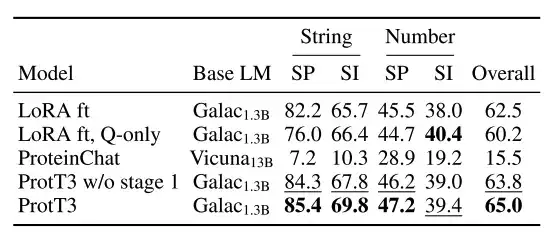
Wie in der Abbildung unten gezeigt, haben ProtT3 und Galactica in den folgenden drei Beispielen zum Beantworten von Fragen zu Proteinen die ersten beiden Fragen zu Proteineigenschaften/-struktur richtig beantwortet, konnten jedoch die dritte Frage, die eine numerische Antwort erforderte, nicht beantworten. ProteinChat hatte bei allen drei Fragen Schwierigkeiten und konnte keine davon beantworten.
Die Sprache der Proteine entschlüsseln, LLMs bahnbrechende Forschung in den Biowissenschaften
Die Forschung der Forscher auf dem Gebiet der Protein-zu-Text-Generierung kann es dem Menschen ermöglichen, komplexe biologische Phänomene auf eine für den Menschen verständliche Weise zu entschlüsseln. Das Sprachmodell in der obigen Studie zeigt nicht nur ein tiefes Verständnis des „latenten Raums“ von Proteinen, sondern dient auch als Brücke zwischen biomedizinischen Aufgaben und der Verarbeitung natürlicher Sprache und eröffnet neue Wege für die Forschung, beispielsweise in der Arzneimittelentwicklung und der Vorhersage von Proteinfunktionen. Weiter,Wenn große Sprachmodelle mit Milliarden oder mehr Parametern zur Verarbeitung komplexerer Sprachstrukturen verwendet werden, dürfte dies die zukünftige Erforschung der Biowissenschaften auf mehreren Ebenen voranbringen.
Zum Beispiel,Das von Zhang Qiang und Chen Huajun von der Zhejiang-Universität geleitete Team schlug ein innovatives großes Sprachmodell namens InstructProtein vor.Das Modell kann sowohl menschliche Sprache als auch Proteinsprache in beide Richtungen generieren: (i) Es nimmt eine Proteinsequenz als Eingabe und sagt ihre textuelle Funktionsbeschreibung voraus; und (ii) die Verwendung natürlicher Sprache zur Förderung der Generierung von Protein-Sequenzen.

Konkret trainierten die Forscher LLM anhand von Protein- und natürlichen Sprachkorpora vor und setzten dann eine überwachte Befehlsoptimierung ein, um die Ausrichtung der beiden unterschiedlichen Sprachen zu erleichtern. InstructProtein eignet sich gut für eine große Anzahl von Aufgaben zur bidirektionalen Proteintextgenerierung. Es handelt sich um einen bahnbrechenden Schritt in der textbasierten Vorhersage von Proteinfunktionen und im Sequenzdesign, wodurch die Lücke zwischen dem Verständnis von Proteinen und der menschlichen Sprache effektiv verringert wird.
Das Papier mit dem Titel „InstructProtein: Aligning Human and Protein Language via Knowledge Instruction“ wurde für ACL 2024 ausgewählt.
* Originalarbeit:https://arxiv.org/pdf/2310.03269
Auch,Das Team der University of Technology Sydney hat sich außerdem mit dem Forschungsteam der Zhejiang-Universität zusammengeschlossen, um gemeinsam das große Sprachmodell ProtChatGPT zu starten.Das Modell lernt und versteht die Proteinstruktur, sodass Benutzer proteinbezogene Fragen hochladen und an interaktiven Gesprächen teilnehmen können, um letztendlich umfassende Antworten zu generieren.
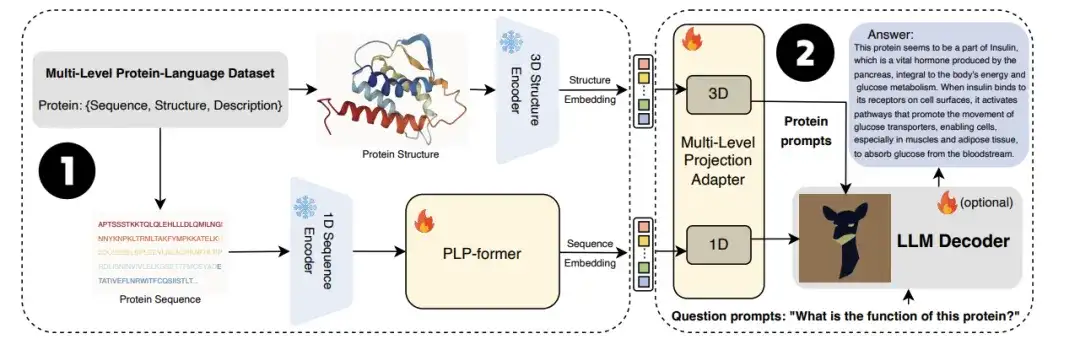
Insbesondere durchlaufen Proteine zunächst Protein-Encoder und einen vortrainierten Proteinsprachen-Transformator (PLP-Former), um Proteineinbettungen zu erzeugen, und dann werden diese Einbettungen über den Projektionsadapter auf LLM projiziert. Schließlich kombiniert LLM Benutzerfragen mit projizierten Einbettungen, um informative Antworten zu generieren. Experimente zeigen, dass ProtChatGPT professionelle Antworten auf Proteine und die entsprechenden Fragen generieren kann und so der eingehenden Erforschung und Anwendungserweiterung der Proteinforschung neue Vitalität verleiht.
* Originalarbeit:https://arxiv.org/abs/2402.09649
Wenn große Sprachmodelle in der Zukunft in der Lage sind, aus riesigen und umfangreichen Datenmengen Rückschlüsse auf die zugrunde liegenden Gesetze oder Tiefenstrukturen von Proteinen zu ziehen, die weit über die Grenzen der menschlichen Wahrnehmung hinausgehen, wird ihr Potenzial enorm freigesetzt. Wir gehen davon aus, dass große Sprachmodelle mit der kontinuierlichen Weiterentwicklung der Technologie die Proteinforschung in eine bessere Zukunft führen werden.








