Command Palette
Search for a command to run...
Das Niveau Ist Fast so Gut Wie Das Der Leitenden Pathologen! Tsinghua-Team Schlägt KI-basiertes Modell ROAM Vor, Um Eine Genaue Diagnose Von Gliomen Zu Erreichen
Ein Gliom ist ein Tumor, der aus Gliazellen im Gehirn entsteht. Er macht 40 bis 60 % aller primären Tumoren des zentralen Nervensystems aus und gilt als der häufigste primäre intrakraniale Tumor bei Erwachsenen. Die histopathologische Klassifizierung von Gliomen ist sehr komplex und wird üblicherweise in drei Untertypen unterteilt: Astrozytome, Oligodendrogliome und Ependymome, die jeweils wiederum in mehrere Grade unterteilt werden können. daher,Eine genaue Klassifizierung und Einstufung ist für die Prognose und Behandlung von Gliomen von entscheidender Bedeutung.
Die Diagnose eines Glioms wird in der Regel von erfahrenen Pathologen durch die Untersuchung von Gewebeschnitten gestellt. Allerdings ist diese Methode mit Herausforderungen verbunden, wie beispielsweise dem Mangel an Pathologen, der subjektiven Diagnose und dem zeitaufwändigen Diagnoseprozess.Es ist schwierig, den aktuellen diagnostischen Bedarf für Gliome zu decken.
Jüngste Fortschritte in der digitalen Pathologie und im maschinellen Lernen haben die Digitalisierung histologischer Schnitte ermöglicht, die in Gigapixel-Ganzkörperbilder (WSIs) umgewandelt werden, die umfangreiche Kontextdaten mit großem Potenzial für Diagnose, Prognose und molekulare Charakterisierung enthalten. Diese Methoden analysieren jedoch nur die von Pathologen ausgewählten Bereiche von Interesse (ROI) in Pathologiebildern.Eine automatisierte Analyse des gesamten Objektträgers ist nicht möglich.
In diesem ZusammenhangAssoziierter Forscher Lu Hairong, Professor Jiang Rui, Professor Zhang Xuegong vom Life Basic Model Laboratory der Abteilung für Automatisierung an der Tsinghua-Universität und Professor Hu Zhongliang vom Xiangya-Krankenhaus der Central South University,Für die Diagnose auf klinischer Ebene und die Entdeckung molekularer Marker bei Gliomen wird ein präzises KI-Basismodell ROAM zur pathologischen Diagnose vorgeschlagen, das auf großen Interessenbereichen und einem Pyramidentransformator basiert und auf die pathologische Diagnose anderer Tumorarten ausgeweitet werden kann.
Die entsprechenden Forschungsergebnisse wurden in Nature Machine Intelligence unter dem Titel „A transformer-based weakly supervised computational pathology method for clinical-grade diagnosis and molecular marker discovery of gliomas“ veröffentlicht.
ROAM kann aus pathologischen Bildern effektiv umfangreiche Informationen in mehreren Maßstäben extrahieren und eine genaue Diagnose für verschiedene Klassifizierungsaufgaben wie die Erkennung von Gliomtumoren, die Klassifizierung/Einstufung von Subtypen und die Vorhersage molekularer Merkmale erreichen.Anhand interner Daten zeigte ROAM eine hervorragende Diagnoseleistung, indem es automatisch wichtige morphologische Merkmale erfasste, die mit der Erfahrung der Pathologen übereinstimmten, und eine genaue, zuverlässige und anpassbare Diagnose von Gliomen auf klinischem Niveau lieferte.
Darüber hinaus kann ROAM auf unabhängige externe Daten erweitert werden und verfügt über hervorragende Generalisierungsmöglichkeiten. Durch die Visualisierung und Erklärung von Diagnosen kann ROAM Pathologen dabei helfen, die Zuverlässigkeit der diagnostischen Grundlage des Modells zu überprüfen, wertvolle Informationen zu extrahieren und Zusatzdiagnosen zur Verbesserung der medizinischen Versorgung zu fördern. Am wichtigsten ist jedoch, dass ROAM die Entdeckung molekularer und morphologischer Biomarker erleichtert und so neue Erkenntnisse für die Diagnose und Behandlung von Gliomen liefert.
Forschungshighlights:
* ROAM ermöglicht eine effiziente Extraktion visueller Darstellungen von Ganzschnittbildern der Gewebepathologie durch großformatige Bildausschnitte und mehrskalige Feature-Learning-Module
* ROAM kann auf unabhängige externe Daten erweitert werden und verfügt über eine hervorragende Generalisierungsfähigkeit
* ROAM erleichtert die Entdeckung molekularer und morphologischer Biomarker und liefert neue Erkenntnisse für die Diagnose und Behandlung von Gliomen

Papieradresse:
https://www.nature.com/articles/s42256-024-00868-w
Datensatz-Download:
Das Open-Source-Projekt „awesome-ai4s“ vereint mehr als 100 AI4S-Papierinterpretationen und stellt umfangreiche Datensätze und Tools bereit:
https://github.com/hyperai/awesome-ai4s
Datensatz: Zwei wichtige digitale Objektträger-Datensätze zur Gewebepathologie
In dieser Studie wurden zwei groß angelegte digitale Gewebepathologie-Datensätze (WSI) für die Gliomforschung gesammelt.
1. Xiangya Glioma WSI-Datensatz
Dieser Datensatz stammt aus Gliomschnitten des Xiangya-Krankenhauses der Central South University.Wie in der Abbildung unten gezeigt, enthält es 1.109 WSIs, die bei 40-facher Vergrößerung in der gleichen Anzahl unterschiedlicher Fälle verarbeitet wurden und Diagnoseaufgaben wie Gliomerkennung, Subtypklassifizierung, Graduierung und Vorhersage molekularer Merkmale abdecken, und für jeden Fall wurde die gleiche Anzahl von Schnitten gesammelt.

Der Datensatz enthält nur Anmerkungen auf Scheibenebene, die den Subtyp und den Grad von 530 Astrogliomen, 224 Oligodendrogliomen und 213 Ependymomen angeben. Darüber hinaus wurden in 634 Fällen mit IDH-Mutation und 641 Fällen mit MGMT-Promoter-Methylierung molekulare Tests durchgeführt.
Der Datensatz ist zufällig in zwei Teile aufgeteilt:Einer ist ein interner Trainingsdatensatz mit 736 WSIs, der für das Modelltraining verwendet wird; Der andere ist ein interner Testdatensatz mit 373 WSIs, der zur Modellbewertung und für arztbezogene Experimente verwendet wird. Der Anteil der Klassen in beiden Datensätzen ist derselbe wie im gesamten Datensatz.
2. TCGA Glioma WSI-Datensatz
Ein weiterer WSI-Datensatz zur Gliom-Histopathologie stammt aus dem Brain Low-Grade Gliomas and Glioblastoma Project.Es gibt insgesamt 860 Glioblastomabschnitte (aus 389 Fällen) und 844 niedriggradige Gliomabschnitte (aus 491 Fällen). Die Diagnosekriterien unterscheiden sich von denen im Xiangya-Datensatz, daher werden nur die Anmerkungen auf Slice-Ebene in den Daten beibehalten. Zwei Pathologen, die an der Annotation des Xiangya-Datensatzes beteiligt waren, wurden gebeten, die Diagnoseergebnisse dieser Abschnitte gemäß der Version 2016 der Diagnoserichtlinien zu überprüfen und zu überarbeiten.
Schließlich umfasst der überprüfte Datensatz 618 WSIs mit Vergrößerungen von 40-fach und 20-fach und deckt 4 Aufgaben ab, die mit dem Xiangya-Datensatz übereinstimmen. Dieser Datensatz dient als externer Testdatensatz für die Klassifizierung von Gliom-Subtypen, den Grad und die Vorhersage molekularer Merkmale.
Modellarchitektur: Basierend auf großem regionalen Interesse und Pyramidentransformator
ROAM ist eine schwach überwachte Methode der rechnergestützten PathologieDiese Methode verwendet Multi-Instance-Lernen als Grundgerüst, große Gewebebildblöcke als grundlegende Forschungseinheit und verwendet Pyramid Transformer, um die intra- und interskaligen Korrelationsmerkmale jedes Gewebeblocks systematisch zu erlernen und so eine effektive Extraktion visueller Darstellungen ganzer Gewebeschnittbilder zu erreichen.
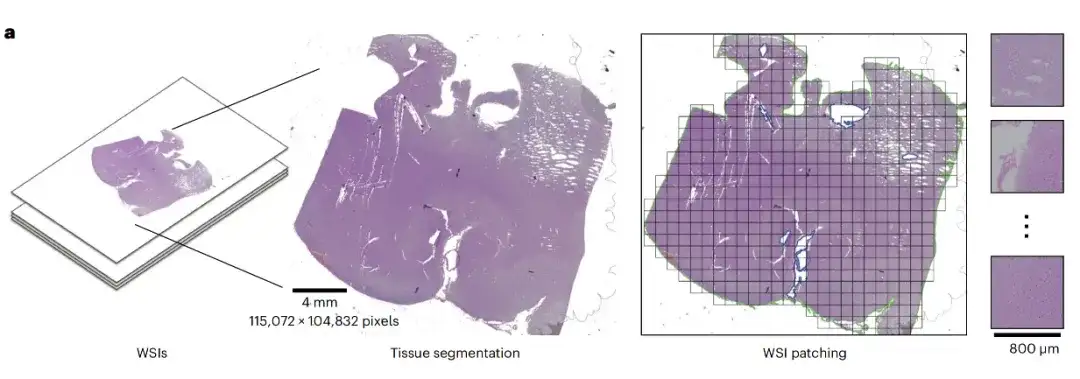
Erste,ROAM führt eine Gewebesegmentierung für jedes vollständige Schnittbild durch und extrahiert daraus großformatige Gewebebildausschnitte (2048 × 2048) als Grundeinheit für die nachfolgende Analyse, nämlich ROI, wie in der folgenden Abbildung WSI-Ausschnitt dargestellt:
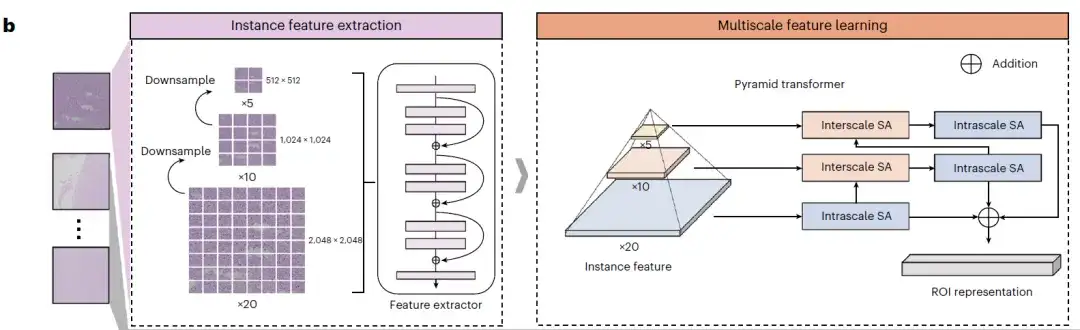
Zweitens,Jeder ROI wurde zweimal kontinuierlich heruntergesampelt, um drei Bilder mit unterschiedlichen Vergrößerungen zu erzeugen. Jedes Bild wird dann in kleine Bildausschnitte segmentiert, die durch ein vorab trainiertes Convolutional Neural Network kodiert werden, um ihre visuellen Darstellungen zu extrahieren, die als Eingabe des MIL-Modells verwendet werden, wie in der Instanzmerkmalsextraktion auf der linken Seite von Abbildung b unten gezeigt; Ein mehrskaliges Self-Attention-Modul (SA) und ein Aufmerksamkeitsnetzwerk werden verwendet, um Darstellungen auf Instanzebene zu generieren, und diese Informationen werden in Darstellungen auf Slice-Ebene aggregiert, wie in der mehrskaligen Merkmalsextraktion auf der rechten Seite von Abbildung b unten gezeigt.
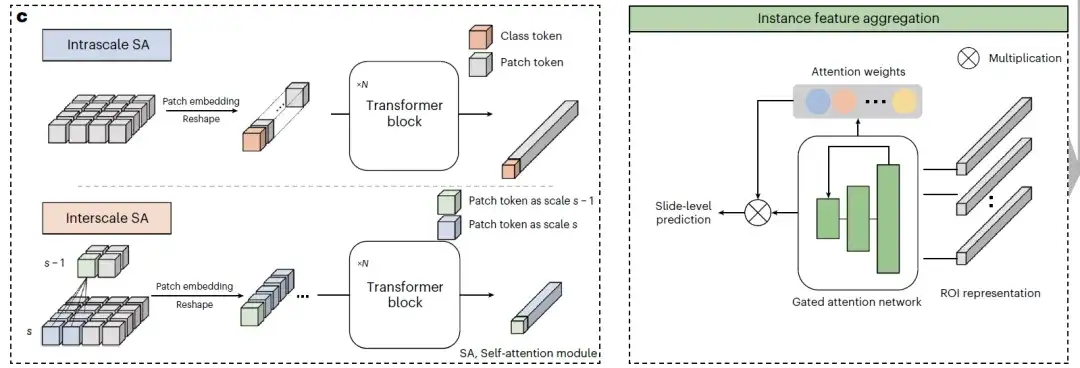
endlich,Wie in Abbildung c (Instanzmerkmalsaggregation) gezeigt, verwenden zwei verschiedene Arten von SA-Modulen die Pyramidentransformatorarchitektur, um mehrskalige Merkmale schrittweise von hoher zu niedriger Vergrößerung zu verschmelzen, um eine mehrskalige visuelle Darstellung des Gewebeblocks zu erhalten. Das Intra-Scale-SA-Modul und das Inter-Scale-SA-Modul lernen jeweils die Intra-Scale- und Inter-Scale-bezogenen Funktionen von ROI. Beide Module enthalten mehrere Multi-Head-SA-Schichten und Feedforward-Schichten.
Forschungsergebnisse: ROAM ermöglicht präzise Gliomdiagnose
ROAM ermöglicht präzise Gliomdiagnose
Die Forscher bewerteten die Klassifizierungsleistung von ROAM anhand interner Datensätze und externer TCGA-Datensätze.
Wie in Abbildung a unten gezeigt, übertrifft ROAM fünf Methoden, darunter CLAM, TransMIL, GTP, TEA-Graph und H(2)MIL, und übertrifft andere Basismethoden bei Aufgaben im Zusammenhang mit der Gliomdiagnose anhand interner Datensätze. Bei der Erkennung von drei Gliomtypen, nämlich normalen Gliomen, Gliose- und Tumorgliomen, betrug der makrodurchschnittliche Eins-zu-eins-ROC-Kurvenbereich (AUC) von ROAM 0,990 ± 0,002.
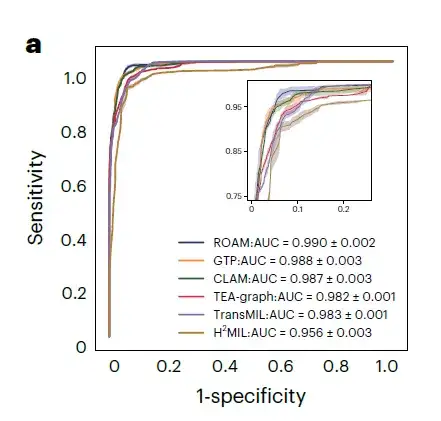
Für die Klassifizierung von drei Gliom-Subtypen, nämlich Astrozytom, Oligodendrogliom und Ependymom, betrug die AUC von ROAM 0,950±0,003, wie in Abbildung b unten dargestellt.
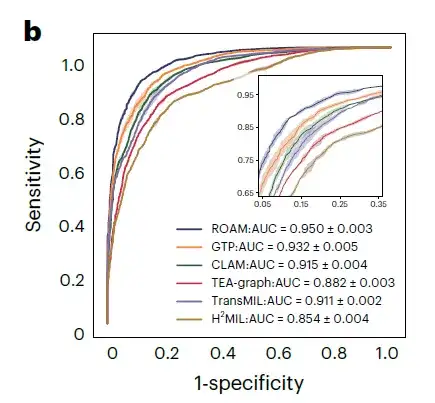
Bei diesen Gliomdiagnoseaufgaben erreichte ROAM die höchste AUC aller Basismethoden.Die Wirksamkeit und hohe Leistungsfähigkeit des ROAM-Modells bei der Gliomdiagnose wurden nachgewiesen.
gleichzeitig,ROAM verfügt außerdem über gute Generalisierungseigenschaften.Obwohl ROAM nur mit internen Datensätzen trainiert und an externen TCGA-Datensätzen getestet wurde, übertrifft es insgesamt immer noch andere Basismethoden. Auch,Die Visualisierungsergebnisse der ROAM-Vorhersagen zeigen auch, dass die mit dieser Methode zusammengefasste Diagnosegrundlage sehr gut mit den klinischen Diagnosekriterien übereinstimmt.
ROAM bringt die klinische Zusatzdiagnostik von Gliomen deutlich voran
Die Forscher führten eine umfassende klinische Bewertung von ROAM durch.Die Leistung von ROAM bei der Zusatzdiagnose von Gliomen wurde untersucht: 3 Gruppen mit je 5 Pathologen wurden zur Teilnahme an der Studie eingeladen, eine Gruppe bestand aus jungen Pathologen mit weniger als 5 Jahren klinischer Erfahrung, zwei Gruppen bestanden aus fortgeschrittenen Pathologen mit 5 bis 15 Jahren klinischer Erfahrung und die anderen beiden Gruppen bestanden aus erfahrenen Pathologen mit mehr als 15 Jahren klinischer Erfahrung.
Wie in der Abbildung unten gezeigt, schnitt das vorgeschlagene System bei den fünf Aufgaben der Gliomkaskadendiagnose gut ab, übertraf dabei vier von fünf Pathologen und war mit dem leistungsstärksten leitenden Pathologen (Senior 1) vergleichbar. Insbesondere bei der Erkennung von Gliomen,Das neue System übertraf die Leistung aller Pathologen deutlich.Einschließlich der Pathologen mit der besten Leistung, die 21.30% überschreiten, wie in Abbildung f unten dargestellt.
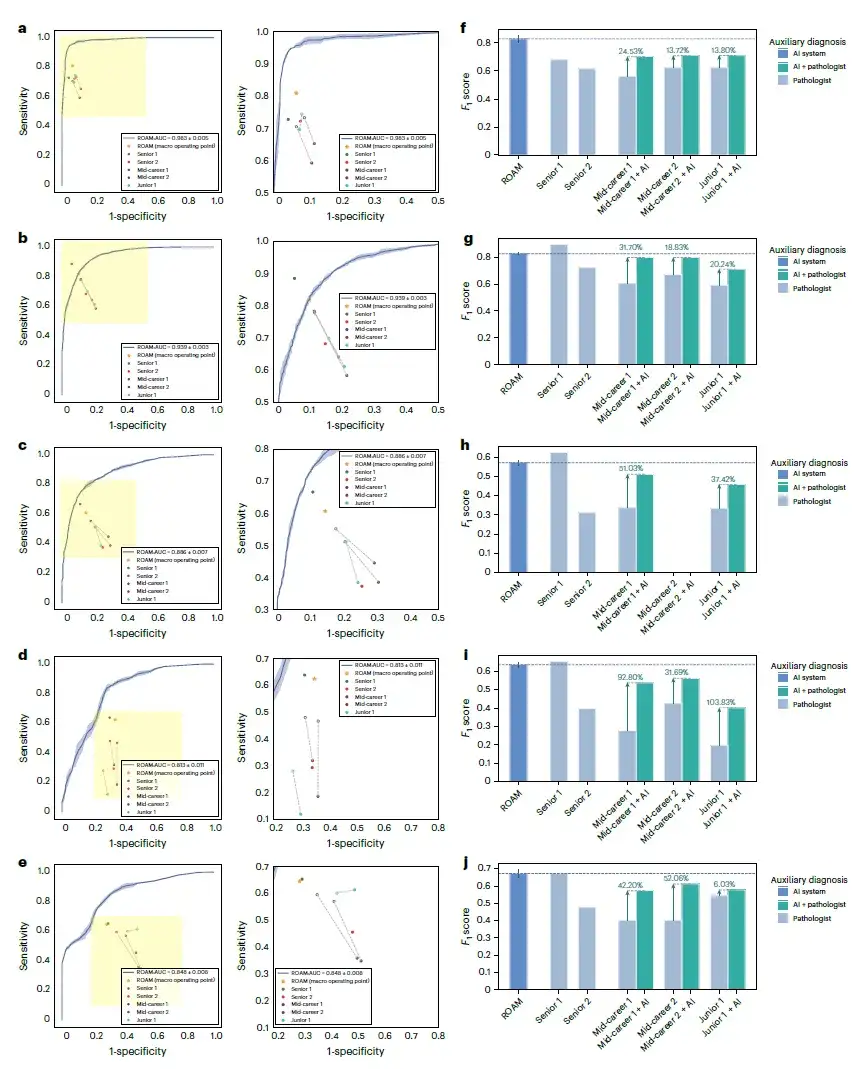
Anschließend baten die Forscher drei Pathologen auf Junior- und mittlerer Ebene, mithilfe von ROAM Diagnosen durchzuführen, um zu untersuchen, ob sich ihre diagnostische Leistung verbesserte. Die Ergebnisse zeigen, dass sich die diagnostische Genauigkeit der drei Pathologen mit Hilfe von ROAM in allen Aufgaben durchschnittlich um 7,27% (Primär 1), 12,87% (Mittelstufe 1) und 9,96% (Mittelstufe 2) verbesserte.Dies spiegelt den großen klinischen Anwendungswert von ROAM wider.
ROAM fördert die Entdeckung molekularmorphologischer Marker von Gliomen
Mithilfe von ROAM untersuchten die Forscher die morphologischen Manifestationen wichtiger molekularer Merkmale im Zusammenhang mit der Gliomdiagnose und stellten fest, dass ROAM bei der molekularen Merkmalsaufgabe der Vorhersage von Isocitrat-Dehydrogenase (IDH)-Mutationen gute Ergebnisse erzielte. Sie führten eine vollständige visuelle Analyse der Vorhersageergebnisse von ROAM in dieser Aufgabe durch und analysierten und fassten die gewebemorphologischen Merkmale der Schlüsselbereiche mit hoher Aufmerksamkeit, auf die sich ROAM konzentrierte, zusammen. Sie stellten fest, dass Eosinophilie, gleichmäßiges Zytoplasma und dunkle Färbung des Zellkerns in pathologischen Bildern von IDH-Mutationen häufig vorkommen.
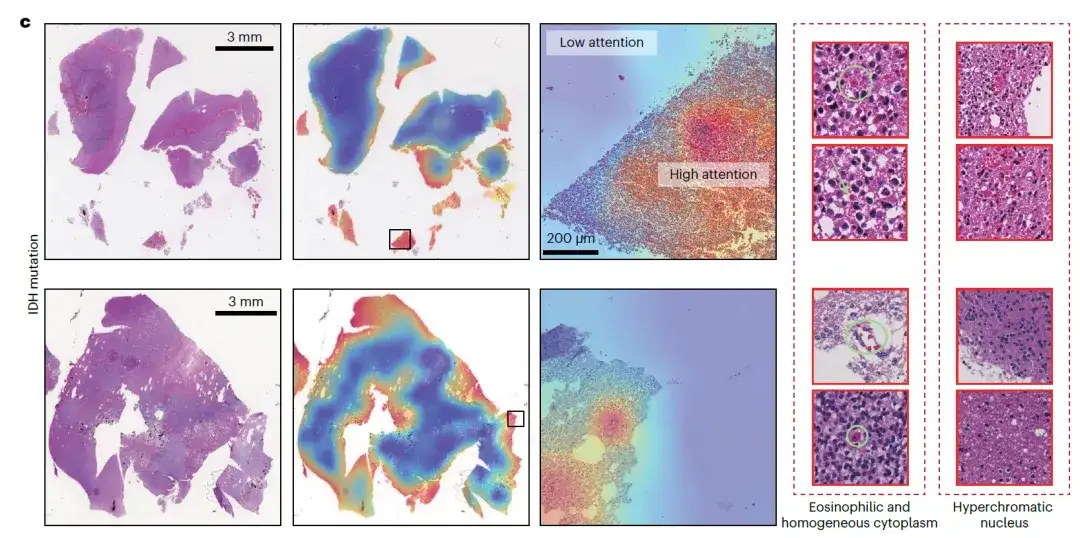
Diese wichtige Entdeckung hilft Ärzten dabei, vorläufige Vorhersagen zum IDH-Status ohne die Hilfe molekularer Tests zu treffen und wird die Optimierung und Verbesserung klinischer Diagnosestandards für Gliome erheblich vorantreiben.
Das Labor für grundlegende Modelle der Abteilung für Automatisierung im Lebensbereich der Tsinghua-Universität fördert weiterhin KI, um die biowissenschaftliche Forschung zu stärken
In diesem ArtikelDie korrespondierenden Autoren sind der assoziierte Forscher Lü Hairong, Professor Zhang Xuegong und Professor Hu Zhongliang vom Xiangya-Krankenhaus der Central South University.Professor Jiang Rui und Masterstudent Yin Xiaoxu von der Tsinghua-Universität, Yang Pengshuai vom China Mobile Research Institute und Cheng Lingchao vom Xiangya-Krankenhaus sind die Co-Erstautoren des Artikels. Hu Jun, Yang Jiao, Wang Ying, Fu Xiaodan, Shang Li, Li Liling, Lin Wei und Zhou Huan waren an der Datenerfassung und Kommentierung für diese Studie beteiligt. Chen Fuxun und das Fuzhou Data Technology Research Institute leisteten Forschungs- und Entwicklungsunterstützung für die Online-Softwareplattform.
Als einer der Hauptverantwortlichen für diese StudieDas Life Basic Model Laboratory der Abteilung für Automatisierung an der Tsinghua-Universität erforscht ständig, wie die biowissenschaftliche Forschung durch fortschrittliche Technologien der künstlichen Intelligenz gestärkt werden kann.
Im Juni dieses Jahres trafen sich Professor Zhang Xuegong, Direktor des Life Basic Model Laboratory der Abteilung für Automatisierung der Tsinghua-Universität, Professor Ma Jianzhu von der Abteilung für Elektronik/AIR und Dr. Song Le von der Abteilung für Biotechnologie.Es wurde ein Großzellenmodell namens scFoundation eingerichtet.Das Modell wurde anhand von Genexpressionsdaten von 50 Millionen Zellen trainiert, verfügt über 100 Millionen Parameter und kann ungefähr 20.000 Gene gleichzeitig verarbeiten. Als Basismodell hat es bei einer Vielzahl biomedizinischer Downstream-Aufgaben, wie beispielsweise „virtuellen Arzneimittelstudien“, hervorragende Leistungsverbesserungen gezeigt.Bietet ein neues Paradigma für künstliche Intelligenz in der Einzelzellforschung.
Die Forschungsergebnisse wurden in Nature Methods unter dem Titel „Large-scale foundation model on single-cell transcriptomics“ veröffentlicht.Klicken Sie hier, um den vollständigen Bericht anzuzeigen: „Ein Zellmodell mit 100 Millionen Parametern ist da! Veröffentlicht im Nature-Subjournal: Das Team der Tsinghua-Universität veröffentlicht scFoundation: Simultane Modellierung von 20.000 Genen.“
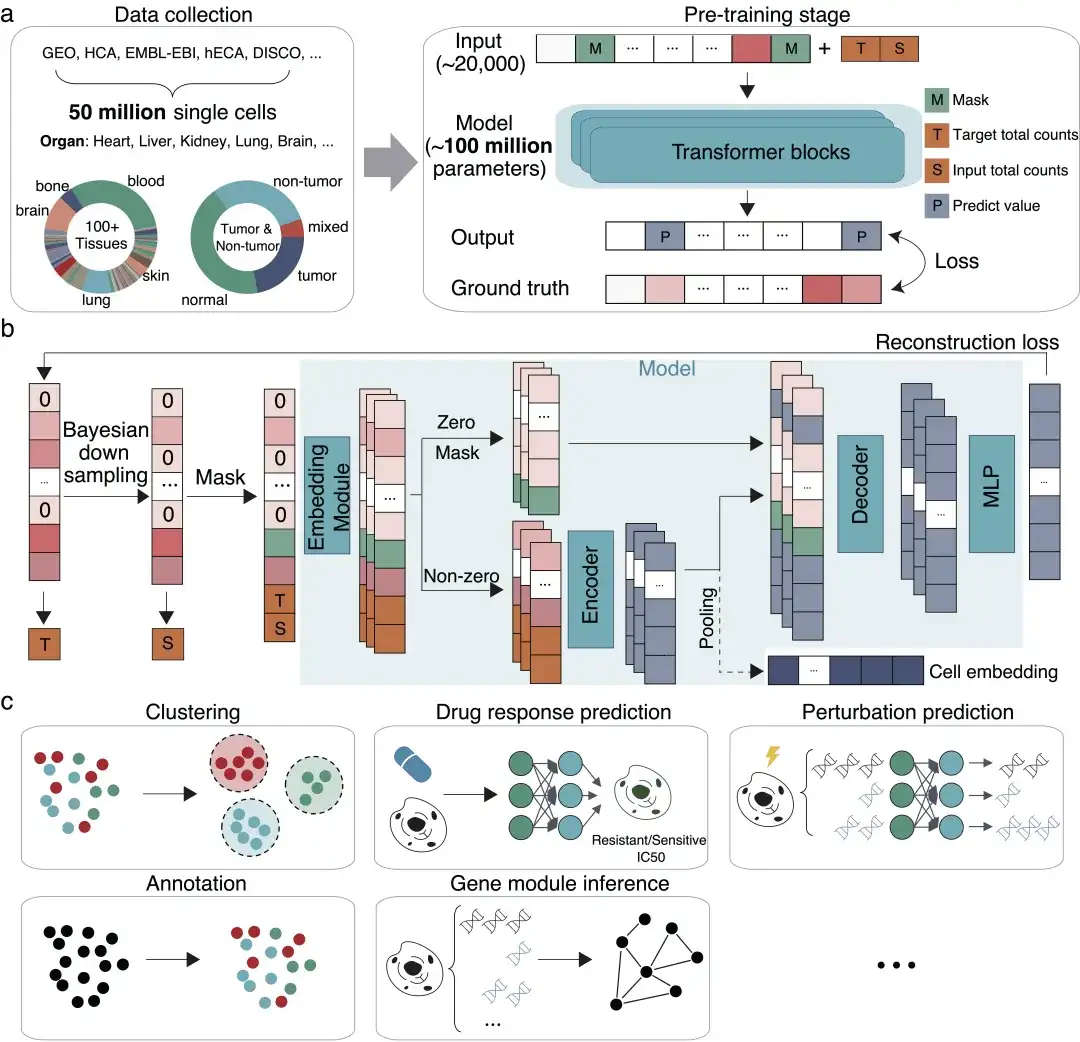
Das scFoundation-Modell bietet innovative Methoden und Werkzeuge für die Grundlagenforschung in den Biowissenschaften, die Vorhersage von Zellstörungsreaktionen, die Entdeckung von Arzneimittelzielen und anderen Bereichen. Darüber hinaus bietet es neue Ideen und Methoden für die groß angelegte Zellmodellforschung hinsichtlich Modellarchitektur, Trainingsrahmen und nachgelagertem Demonstrationsanwendungssystem. Es hat die Grenzen grundlegender Modelle im Einzelzellbereich erfolgreich erweitert und den Grundstein für zukünftige Forschungen wie virtuelle Arzneimittelexperimente im digitalen Raum gelegt.
Mit Blick auf die Zukunft wird das Life Basic Model Laboratory der Abteilung für Automatisierung der Tsinghua-Universität weiterhin im interdisziplinären Bereich der künstlichen Intelligenz und Biowissenschaften forschen. Mit der kontinuierlichen Weiterentwicklung und Verbesserung der KI-Technologie wird die Anwendung künstlicher Intelligenz im Bereich der Biowissenschaften den Fortschritt der Medizintechnik erheblich vorantreiben, die Genauigkeit von Diagnose und Behandlung verbessern, die medizinischen Kosten senken und letztendlich die Gesundheit und Lebensqualität der Menschen verbessern.
Quellen:
1.https://www.nature.com/articles/s42256-024-00868-w
2.https://mp.weixin.qq.com/s/oB3kTgcgObawPKU-75KsHQ
3.https://mp.weixin.qq.com/s/nflI4PVTJB3xVPXuA5zbZQ
Aufruf zum Handeln
HyperAI (hyper.ai) ist Chinas größte Suchmaschine im Bereich Datenwissenschaft. Es konzentriert sich seit langem auf die neuesten Forschungsergebnisse der KI für die Wissenschaft und hat mehr als 100 wissenschaftliche Arbeiten in Top-Zeitschriften interpretiert.
Forschungsgruppen und Teams, die Forschung und Erkundung im Bereich KI für die Wissenschaft betreiben, können sich gerne an uns wenden, um ihre neuesten Forschungsergebnisse mitzuteilen, ausführliche Interpretationsartikel beizusteuern und an der Live-Übertragungskolumne „Meet AI4S“ teilzunehmen. Weitere Möglichkeiten zur Förderung von AI4S warten darauf, von uns gemeinsam erkundet zu werden!
WeChat hinzufügen: PH (WeChat-ID: G18539589505)








