Command Palette
Search for a command to run...
3,5-fache Erhöhung Der Katalytischen Kapazität! Das Team Der Chinesischen Akademie Der Wissenschaften Entwickelte Eine P450-Enzym-De-novo-Designmethode Basierend Auf Dem Diffusionsmodell P450Diffusion

Cytochrom-P450-Enzyme sind in fast allen Organismen allgegenwärtig und spielen eine wichtige Rolle bei verschiedenen Stoffwechselprozessen im Zusammenhang mit Wachstum und Entwicklung von Lebewesen. Als vielseitigster Biokatalysator in der Natur können P450-Enzyme nicht nur mehr als 951 bekannte Redoxreaktionen katalysieren, sondern auch inerte Kohlenstoff-Wasserstoff-Bindungen unter milden Bedingungen selektiv oxidieren.In industriellen Anwendungen ist er als „Universalkatalysator“ bekannt.
Heutzutage wird die gerichtete Evolution häufig eingesetzt, um neue P450-Enzyme mit besserer Leistung zu entwickeln. Allerdings erfordern herkömmliche Methoden üblicherweise mehrere Runden zufälliger Mutagenese und Hochdurchsatz-Screenings, was es schwierig macht, den potenziellen Proteinraum im Detail zu erforschen, sei es durch die Durchführung tatsächlicher Experimente oder durch Computersimulationsberechnungen.
Obwohl Deep Learning bemerkenswerte Erfolge bei der Vorhersage von Proteinstrukturen erzielt hat, bleibt das ideale Funktionsdesign eine enorme Herausforderung. Beim Entwerfen von Proteinfunktionen ist es schwierig, genügend hochwertige Funktionsdaten zu sammeln und ein komplexes Modell zu trainieren, um Sequenzen mit den gewünschten Funktionen zu erstellen.Durch die Kombination wissensbasierter Techniken mit leistungsstarken Deep-Learning-Modellen wird der natürliche Proteinsequenzraum erweitert.Es könnte eine geeignete Methode für die Entwicklung neuer P450-Enzyme sein.
Kürzlich analysierten Jiang Huifeng, Cheng Jian und andere Forscher vom Tianjin Institute of Industrial Biotechnology der Chinesischen Akademie der Wissenschaften die Taschendesignprinzipien des P450-Enzyms Flavonoid-6-Hydroxylase (F6H).Entwickelte eine De-novo-Designmethode für das P450-Enzym basierend auf dem Diffusionsmodell und den Pocket-Designprinzipien, P450Diffusion.Die entsprechende Forschung wurde in Research unter dem Titel „Cytochrome P450 Enzyme Design by Constraining the Catalytic Pocket in a Diffusion Model“ veröffentlicht.
Basierend auf der P450-Diffusion wurde in dieser Studie ein neues Enzym mit besserer Aktivität und höherer Stabilität als natürliche P450-Enzyme entwickelt. Im Vergleich zur natürlichen Flavonoid-6-Hydroxylase war die katalytische Kapazität des neuen Enzyms um das 1,3- bis 3,5-fache erhöht.
Forschungshighlights:
* Diese Studie analysierte den Ursprungsmechanismus neuer Funktionen in der Evolution von P450-Enzymen und schlug das „Dreipunktfixierungsprinzip“ der Substratbindung von P450-Enzymen vor
* Die katalytische Kapazität des durch P450-Diffusion erzeugten neuen Enzyms wird um das 1,3- bis 3,5-fache erhöht
* Diese Studie liefert neue Ideen für die Entwicklung neuer funktioneller P450-Enzyme im Rahmen eines Deep-Learning-Diffusionsmodells. Man geht davon aus, dass diese Methode in Zukunft in Bereichen wie der Biotechnik und der industriellen Katalyse eine Rolle spielen und die Entwicklung und Anwendung neuer Enzyme fördern wird.

Papieradresse:
https://spj.science.org/doi/10.34133/research.0413
Das Open-Source-Projekt „awesome-ai4s“ vereint mehr als 100 AI4S-Papierinterpretationen und stellt umfangreiche Datensätze und Tools bereit:
https://github.com/hyperai/awesome-ai4s
Datensatz: Sammeln und kodieren Sie den Datensatz
Um P450Diffusion zu konstruieren, durchsuchten und analysierten die Forscher alle potenziellen P450-Enzyme aus veröffentlichten P450-Enzymdatenbanken und öffentlichen Datenbanken, filterten Sequenzen mit einer Länge von mehr als 560 heraus und erhielten 226.509 Sequenzen als Trainingsdatensätze.
Anschließend kodierten die Forscher den Trainingsdatensatz, wobei jede Aminosäure in der Proteinsequenz als 8-dimensionaler Vektor kodiert wurde und jeder Stapel von Proteinsequenzen als 64×1×560×8-Vektor kodiert wurde – wobei 64 die Stapelgröße ist, die der Anzahl der Proben in den Trainingsdaten entspricht; 1 steht für die Kanalgröße; 560 stellt die maximale Länge der Proteinsequenz dar; und 8 stellt den VHSE8-Kodierungsvektor für jede Aminosäure in der Proteinsequenz dar.
War die Proteinsequenz kürzer als 560 Ziffern, fügten die Forscher Lücken hinzu, bis eine Länge von 560 Ziffern erreicht war. In diesem Fall wird als Code für die Lücke ein Vektor aus 8 Nullen zugewiesen.
Modellarchitektur: P450-Diffusion, eine De-novo-Designmethode für P450-Enzyme
Als Beispiel diente den Forschern eine Flavonoid-6-Hydroxylase (CYP706X1) aus Erigeron breviscapus. Dieses Enzym gehört zur CYP706X-Unterfamilie und wandelt Apigenin im Breviscapin-Biosyntheseweg in Breviscapin um.
Erste,Durch die Rekonstruktion der ursprünglichen Sequenz, Rückmutationsexperimente, progressive Vorwärtsakkumulation und kristallographische Analyse identifizierten die Forscher die Gründungsreste, aus denen die katalytische Tasche besteht und die für die Innovation der Genfunktion des P450-Enzyms verantwortlich sind.Zweitens,Durch eine eingehende Strukturanalyse erläuterten die Forscher die Konstruktionsprinzipien der funktionell innovativen Katalysatortasche.endlich,Es kombiniert das Prinzip des katalytischen Taschendesigns mit dem Rauschunterdrückungs-Diffusionswahrscheinlichkeitsmodell, das bei der Bilderzeugung gute Ergebnisse liefert, und entwirft ein künstliches P450-Enzymerzeugungsmodell P450Diffusion.
Schritt eins: Identifizieren Sie die grundlegenden Reste, die die katalytische Tasche bilden und für die Innovation der Genfunktion des P450-Enzyms verantwortlich sind.
Unter den identifizierten P450-Enzymen der CYP706-Familie sind nur die P450-Enzyme der CYP706X-Unterfamilie in der Lage, Flavonoidsubstrate zu katalysieren. Dies lässt darauf schließen, dass die Funktion des P450-Enzyms Flavon-6-Hydroxylase (F6H) möglicherweise im Vorgänger der CYP706X-Unterfamilie neu entwickelt wurde.
Um den molekularen Mechanismus der Bildung der katalytischen Tasche mit F6H-Funktion aufzuklären, schlugen die Forscher vor, die Änderungen in der Restzusammensetzung zwischen den katalytischen Taschen des nicht-funktionalen ancXY (dem gemeinsamen Vorfahren der Unterfamilien CYP706X und CYP706Y) und des funktionellen ancX (dem gemeinsamen Vorfahren der Unterfamilie CYP706X) zu analysieren. Innerhalb von 8 Å vom aktiven Zentrum unterscheiden sich 16 der 48 Reste.
Wie in Abbildung A unten gezeigt, sind die Reste von ancX und ancXY jeweils Cyan und Magenta gefärbt; und wenn alle diese 16 Reste durch die entsprechenden Reste in ancX ersetzt werden, erhält der Mutant (genannt ancXY-16) die F6H-Funktion, wie in Abbildung B unten gezeigt.
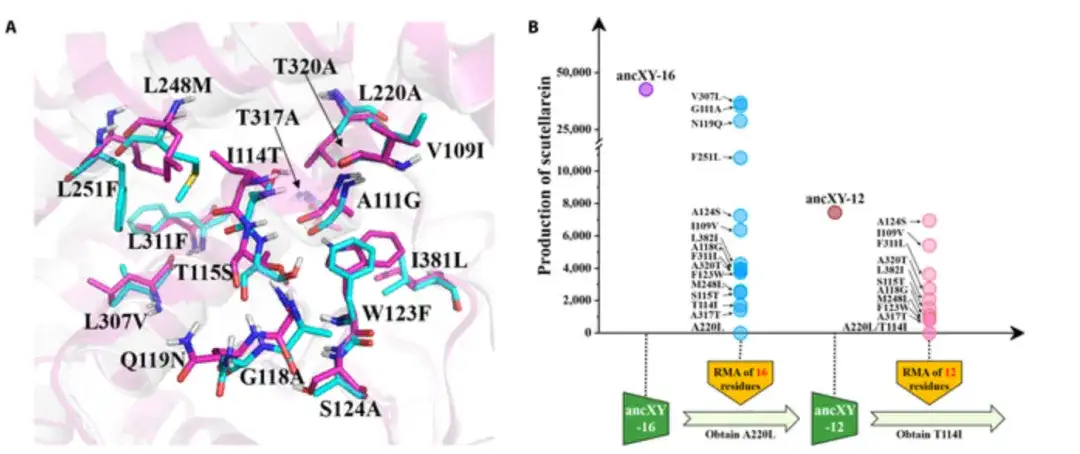
Da die Positionen der Reste in der katalytischen Tasche im dreidimensionalen Raum unterschiedlich sind, tragen nicht alle Reste wesentlich zur Substraterkennung und -bindung bei. Daher versuchten die Forscher, den ursprünglichen Rest in der katalytischen Tasche durch einen Rückmutationstest (RMA: Bewertung des Mutationseffekts jedes Rests in der katalytischen Tasche von ancXY-16 durch Rückführung in seinen ursprünglichen Typ) zu identifizieren. Um die Gründungsreste schneller zu identifizieren, verwendeten die Forscher außerdem eine progressive Vorwärtsakkumulationsstrategie (PFA), um ancXY schrittweise wichtige Mutationen hinzuzufügen, bis der Mutant die F6H-Funktion erlangte.
Schließlich ergab das Experiment, dass die Mutation von 5 Aminosäuren (L220A/I114T/T317A/W123F/L248M) eine Gründerrolle (Gründungsrest) im funktionellen Innovationsprozess von F6H von ancXY zu ancX spielte.
Schritt 2: Erläutern Sie die Designprinzipien der katalytischen Tasche mit funktionaler Innovation.
Durch eine eingehende Analyse des Apigenin-Bindungsmodells in ancXY-5 interpretierten die Forscher den potenziellen Mechanismus der fünf an der funktionellen Innovation beteiligten Gründungsreste weiter. Basierend auf Mutationen der fünf Gründungsreste scheint die katalytische Tasche dem Prinzip der „Dreipunktfixierung“ zu folgen.
„Dreipunktfixierung“ bezieht sich auf die Schlüsselinteraktionen mit den drei Knotenpunkten im Apigeninmolekül.Hierzu gehören: Das 4'-OH (erster Knotenpunkt) im Apigeninmolekül wird durch Wasserstoffbrücken fixiert, die von T114 bereitgestellt werden, der „B“-Ring von Apigenin (zweiter Knotenpunkt) wird durch π-Stapelwechselwirkungen zwischen F123 und M248 fixiert und das 7-OH von Apigenin (dritter Knotenpunkt) wird durch Wasserstoffbrücken mit der Eisenoxylgruppe von CpdI fixiert.
Das Modell hält das Substrat Apigenin in einer Near-Reaction-Konformation (NAC) und behält die relative Ausrichtung zwischen der Apigenin-Reaktionsstelle und der CpdI-Eisenoxylgruppe in einem günstigen Abstand und Winkel (3,6 Å und 155°) bei, wodurch die 6-Hydroxylierungsreaktion von Apigenin während des katalytischen Prozesses eingeleitet wird.
Die Forscher schlugen vor, dass die „Dreipunktfixierung“ als katalytisches Taschendesignprinzip für die natürliche Funktionsinnovation von F6H verwendet werden kann, was auch neue Ideen für die Entwicklung von P450-Enzymen mit gewünschten Funktionen liefert.
Schritt 3: Kombinieren Sie das Designprinzip der katalytischen Tasche mit dem Rauschunterdrückungs-Diffusionswahrscheinlichkeitsmodell, das bei der Bilderzeugung gute Ergebnisse liefert, um ein künstliches P450-Enzymerzeugungsmodell P450Diffusion zu entwerfen.
Die Forscher kombinierten das Diffusionsmodell mit den Designprinzipien der katalytischen Tasche F6H, um ein P450-Enzym mit der gewünschten Funktion von Grund auf zu entwerfen, wie in der folgenden Abbildung dargestellt. Der Designprozess neuer P450-Enzyme umfasst die Konstruktion des P450-Diffusionsmodells, die Sequenzgenerierung, das Screening und die experimentelle Verifizierung.
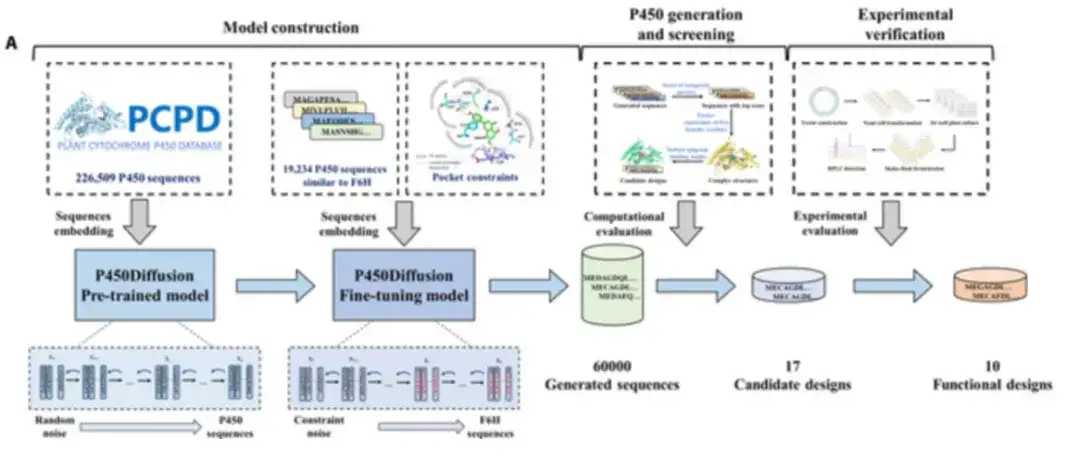
P450Diffusion besteht hauptsächlich aus zwei Modellen, nämlich dem vortrainierten Modell und dem Feinabstimmungsmodell.
Erste,226.509 natürliche P450-Enzymsequenzen wurden gesammelt, um das vortrainierte P450-Sequenzdiffusionsmodell zu trainieren.
Das vortrainierte Modell besteht aus zwei Teilprozessen: einem Vorwärtsdiffusions-Teilprozess, der der Darstellung der P450-Enzymsequenz schrittweise Gaußsches Rauschen hinzufügt, bis es zu zufälligem Rauschen wird; und ein Unterprozess zur umgekehrten Generierung, der mit zufälligem Rauschen beginnt und die Darstellung der P450-Enzymsequenz schrittweise entrauscht, um neue P450-Sequenzen zu generieren. Nach 150.547 Trainingsrunden kann das vortrainierte Diffusionsmodell eine große Vielfalt an Sequenzen mit Ähnlichkeiten zu natürlichen Sequenzen im Bereich von 20% bis 50% generieren.
Zweitens,Das vorab trainierte Diffusionsmodell wurde mithilfe von 19.202 P450-Enzymsequenzen mit signifikanter Ähnlichkeit zur CYP706X-Unterfamilie feinabgestimmt, um sicherzustellen, dass die generierten Sequenzen ein ähnliches strukturelles Rückgrat wie F6H hatten.
Darüber hinaus wurden den fünf Gründungsresten T114, F123, A220, M248 und A317 Beschränkungen auferlegt, um sicherzustellen, dass das Designprinzip der „Dreipunktfixierung“ in den neu generierten Sequenzen reproduziert werden konnte. Das Modell, das die Feinabstimmung des Trainingssatzes mit der eingeschränkten Generierung kombiniert, wird als Feinabstimmungsdiffusionsmodell bezeichnet.
Dann,Um die Erfolgsrate der experimentellen Validierung zu verbessern, führten die Forscher ein virtuelles Screening von 60.000 generierten Sequenzen anhand von drei Kriterien durch: dem berechneten Score eines umfassenden Indikators zur Bewertung der Qualität der generierten Sequenzen, den dreidimensionalen Taschenbeschränkungen der fünf Gründungsreste und der Robustheit des Apigenin-Bindungsmodus.
Nach dem virtuellen ScreeningDie Forscher wählten sorgfältig 17 vielversprechende nicht-natürliche P450-Enzyme für die weitere Erforschung aus.
Forschungsergebnisse: Katalytische Kapazität um das 1,3- bis 3,5-fache erhöht
Die Forscher testeten experimentell, ob die durch P450Diffusion erzeugten Sequenzen authentische P450-Enzyme waren und F6H-Funktionen erfüllten.
Nach dem virtuellen Screening synthetisierten die Forscher die ausgewählten 17 Designs und drückten sie in einem Hefe-Expressionssystem aus. Im Vergleich zu CYP706X1Diese Designs zeigten Sequenzidentität von 70% bis 87%,Dies unterstreicht ihr Potenzial als neue Katalysatoren.
Durch die Fütterung von Apigenin als Substrat, die Kultivierung der rekombinanten Hefe für 4 Tage und die Durchführung einer HPLC-Analyse fanden die Forscher 10 Designs mit signifikanter F6H-Aktivität, wie in der Abbildung unten dargestellt.
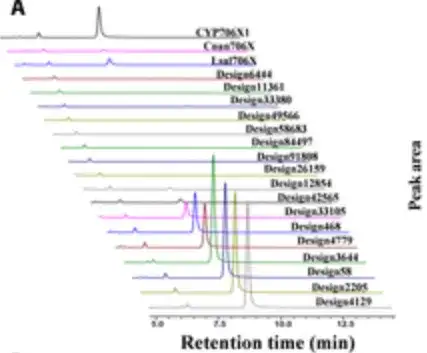
Überraschenderweise,Sechs Designs zeigten eine 1,3- bis 3,5-fache Steigerung der katalytischen Kapazität für die Oleandrinproduktion im Vergleich zu CYP706X1.Wie in den 6 Balken auf der rechten Seite der Abbildung unten gezeigt, zeigten die verbleibenden 4 aktiven Designs auch Aktivitäten, die mit denen anderer natürlicher F6H-Enzyme (d. h. Cnan706X und Lsal706X) vergleichbar sind.
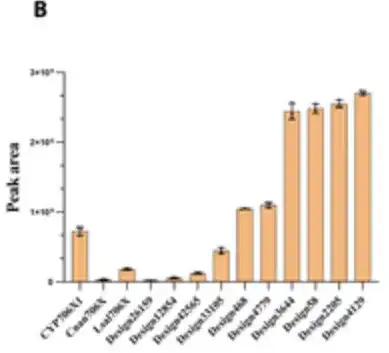
Die Ergebnisse zeigten, dass P450Diffusion nicht nur die grundlegenden Designprinzipien der F6H-Katalysetasche erfassen und effektiv P450-Enzymsequenzen mit F6H-Aktivität erzeugen kann,Es ist auch möglich, P450-Enzyme herauszufiltern, die besser sind als natürliche Sequenzen aus dem P450-Enzymsequenzraum.
Datenbasiertes maschinelles Lernen beschleunigt die Enzymentwicklung
In der Natur vorkommende Enzyme haben vielfältige Funktionen und werden in der industriellen Produktion und der akademischen Forschung eingesetzt. Allerdings können die Eigenschaften und Funktionen vieler dieser Enzyme den Anforderungen der Anwendung nicht vollständig gerecht werden. Die Verbesserung bestimmter Eigenschaften dieser Enzyme durch Modifikation ist eine wichtige Aufgabe des Enzym-Engineerings.
In,Durch die gerichtete Evolution kann die Geschwindigkeit der Enzymevolution erhöht werden, indem der Evolutionsprozess in der Natur simuliert wird.Es hat sich zu einer Schlüsseltechnologie für die Modifikation von Enzymmolekülen entwickelt. Die gerichtete Evolution spielt eine wichtige Rolle in der Biokatalyse und der Arzneimittelentwicklung, doch die enorme Zahl der durch zufällige Mutationen entstehenden Mutanten stellt eine enorme Herausforderung für die experimentellen Screening-Möglichkeiten dar. In den letzten Jahren haben sich neue Technologien wie künstliche Intelligenz und Big Data-Verarbeitung zu wichtigen Forschungsmethoden im Bereich der Biokatalyse entwickelt. Unter anderem ermöglicht maschinelles Lernen die datengesteuerte Zuordnung von Sequenz/Struktur zur Enzymfunktion und trägt so zur Verbesserung der Effizienz der Enzymtechnik bei.
Obwohl die Gene, die die Enzyme kodieren, leicht identifiziert werden können, ist in den allermeisten Fällen (mehr als 991 TP3T) die genaue Funktion der Synthetase unbekannt, da die experimentelle Charakterisierung der Enzymfunktion – das heißt, welche Startermoleküle durch ein bestimmtes Enzym in welche spezifischen Endmoleküle umgewandelt werden – extrem zeitaufwändig ist.
Um diese Herausforderung zu bewältigen, entwickelten Forscher der Universität Düsseldorf (HHU) ein allgemeines maschinelles Lernmodell ESP zur Vorhersage von Enzym-Substrat-Paaren.Übertrifft 91% in der Genauigkeit anhand unabhängiger und vielfältiger Testdaten. ESP kann erfolgreich auf die sehr unterschiedlichen Enzyme und die breite Palette an Metaboliten angewendet werden, die in den Trainingsdaten enthalten sind, und übertrifft dabei Modelle, die für einzelne, gut untersuchte Enzymfamilien entwickelt wurden.
Die Studie wurde im Mai 2023 in Nature Communications unter dem Titel „Ein allgemeines Modell zur Vorhersage kleiner Molekülsubstrate von Enzymen basierend auf maschinellem und Deep Learning“ veröffentlicht.
Link zum Artikel:
https://www.nature.com/articles/s41467-023-38347-2
Das De-novo-Enzymdesign ist zwar spannend, wird aber durch die Komplexität der Enzymkatalyse auch herausgefordert. Forscher des Unternehmens Enzymit, das sich auf zellfreies Enzym-Engineering spezialisiert hat, stellen CoSaNN (Conformational Sampling using Neural Networks) vor, eine neue Strategie für das Enzymdesign.Nutzung der Fortschritte im Deep Learning zur Strukturvorhersage und Sequenzoptimierung.Durch die Kontrolle der Konformation des Enzyms können Forscher den chemischen Raum über den Rahmen einfacher Mutagenese hinaus erweitern.
Darüber hinaus entwickelte das Team SolvIT weiter,Dies ist ein Graph-Neural-Netzwerk, das darauf trainiert ist, die Proteinlöslichkeit in E. coli vorherzusagen.Als zusätzliche Optimierungsebene zur Herstellung hochexprimierter Enzyme. Mithilfe dieses Ansatzes entwickelten die Forscher neue Enzyme mit höheren Expressionsniveaus. 54% wurde für die Expression in E. coli und eine verbesserte thermische Stabilität konzipiert, während mehr als 30% einen höheren Tm-Wert als das Vorlageenzym aufweisen sollte.
Die Studie mit dem Titel „Context-Dependent Design of Induced-fit Enzymes using Deep Learning Generates Well Expressed, Thermally Stable and Active Enzymes“ wurde im August 2023 auf der Preprint-Plattform bioRxiv veröffentlicht.
Link zum Artikel:
https://www.biorxiv.org/content/10.1101/2023.07.27.550799v3

Daten zeigen, dass der weltweite Markt für Industrieenzyme allein bis 2023 einen Wert von 7,4 Milliarden US-Dollar erreichen wird. Indem Forscher künftig die Möglichkeiten künstlicher Intelligenz nutzen und charakteristische Informationen über die Zusammensetzung und Evolution von Proteinen gewinnen, können sie zahlreiche Probleme der Enzymtechnik lösen, etwa Mutationen mit positiver Wirkung vorhersagen, die Proteinstabilität optimieren, die katalytische Aktivität verbessern usw. Dadurch werden die Kosten der Bioproduktion weiter gesenkt und der kommerzielle Wert gesteigert.
Quellen:
1.https://spj.science.org/doi/10.34133/research.0413
2.https://www.cas.cn/syky/202407/t20240718_5026250.shtml
3.https://biotech.aiijournal.com/CN/10.13560/j.cnki.biotech.bull.1985.2022-0724
4.https://www.jiqizhixin.com/articles/2023-06-25-12
5.https://www.jiqizhixin.com/articles