Command Palette
Search for a command to run...
20 Experimentelle Daten Schaffen Einen Meilenstein Für KI-Proteine! Die Shanghai Jiao Tong University Und Das Shanghai AI Lab Haben Gemeinsam FSFP Veröffentlicht, Um Protein-Vortrainingsmodelle Effektiv Zu Optimieren

Proteine, diese winzigen, aber leistungsstarken biologischen Moleküle, sind die Grundlage aller Lebensaktivitäten und spielen in Organismen vielfältige Rollen. Allerdings ist die präzise Abstimmung und Optimierung von Proteinfunktionen zur Erfüllung spezifischer industrieller oder medizinischer Anforderungen eine äußerst anspruchsvolle Aufgabe. Traditionell verlassen sich Wissenschaftler auf Nasslabormethoden, um die Geheimnisse der Proteine zu erforschen, aber dieser Ansatz ist zeitaufwändig und teuer.
Glücklicherweise steht uns mit der rasanten Entwicklung der künstlichen Intelligenz ein neues Tool zur Verfügung: vortrainierte Proteinsprachenmodelle (PLMs), die uns helfen, das Verhalten von Proteinen auf beispiellose Weise zu verstehen und vorherzusagen. PLMs erlernen die Verteilungsmerkmale von Aminosäuresequenzen in Millionen von Proteinen auf unbeaufsichtigte Weise und zeigen großes Potenzial bei der Aufdeckung der impliziten Beziehung zwischen Proteinsequenzen und ihren Funktionen, wodurch sie dazu beitragen, einen großen Designraum effizient zu erkunden. Jetzt,Vortrainierte PLMs haben trotz fehlender experimenteller Daten erhebliche Fortschritte erzielt, ihre Genauigkeit und Interpretierbarkeit müssen jedoch noch verbessert werden.Darüber hinaus erfordern herkömmliche überwachte Lernmodelle eine große Anzahl gekennzeichneter Trainingsbeispiele, was in praktischen Anwendungen ebenfalls ein schwer zu überwindendes Hindernis darstellt.
Um die oben genannten Probleme zu lösen,Die Forschungsgruppe von Professor Hong Liang von der School of Natural Sciences/School of Physics and Astronomy/Zhangjiang Institute for Advanced Studies/School of Pharmacy der Shanghai Jiao Tong University, in Zusammenarbeit mit Tan Pan, einem jungen Forscher am Shanghai Artificial Intelligence Laboratory,Durch die umfassende Nutzung von Meta-Transfer-Learning (MTL), Learning to Rank (LTR) und parametereffizienter Feinabstimmung (PEFT)Wir haben eine Trainingsstrategie namens FSFP entwickelt, mit der Proteinsprachenmodelle auch bei extremer Datenknappheit effektiv optimiert werden können.Es kann zum Lernen der Proteinanpassungsfähigkeit anhand kleiner Stichproben verwendet werden. Es verbessert die Wirkung des herkömmlichen Vortrainings großer Proteinmodelle bei der Vorhersage von Mutationseigenschaften erheblich, wenn nur sehr wenige nasse experimentelle Daten verwendet werden, und zeigt auch großes Potenzial in praktischen Anwendungen.
Die entsprechende Forschungsarbeit wurde in Nature Communications, einem Tochterunternehmen von Nature, unter dem Titel „Enhancing efficiency of protein language models with minimal wet-lab data through few-shot learning“ veröffentlicht.

Papieradresse:
https://doi.org/10.1038/s41467-024-49798-6
Downloadadresse für den ProteinGym-Proteinmutationsdatensatz:
Das Open-Source-Projekt „awesome-ai4s“ vereint mehr als 100 AI4S-Papierinterpretationen und stellt umfangreiche Datensätze und Tools bereit:
https://github.com/hyperai/awesome-ai4s
FSFP optimiert Proteinsprachenmodell, um das Problem des Datenmangels zu lösen
Der FSFP-Ansatz besteht aus drei Phasen:Erstellen Sie Hilfsaufgaben für das Metatraining, trainieren Sie PLMs anhand der Hilfsaufgaben meta und übertragen Sie PLMs per LTR an die Zielaufgabe.
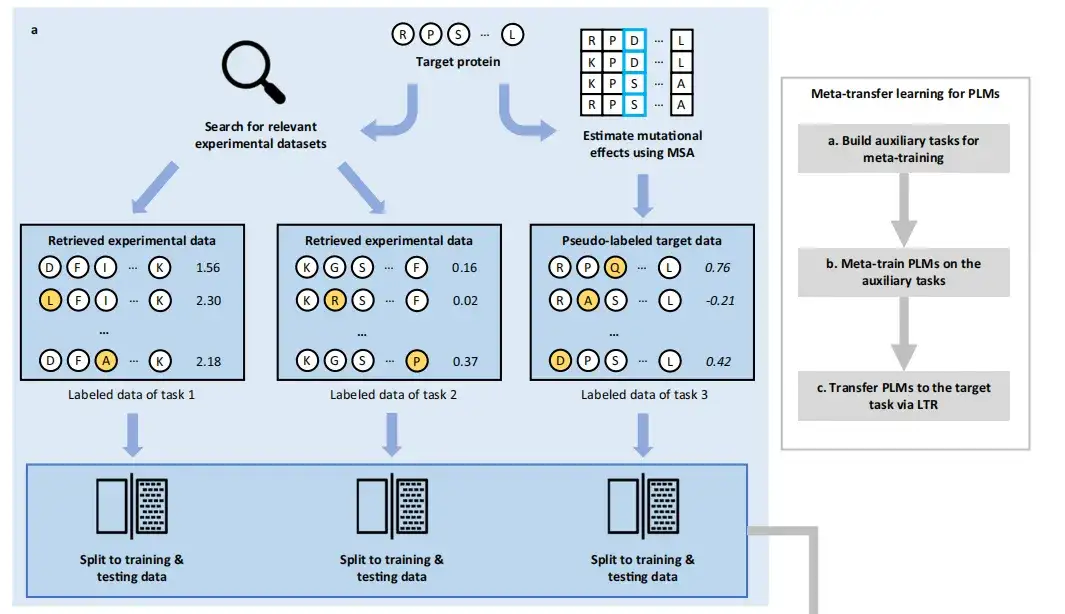
In,Ziel des Meta-Learnings ist es, Erfahrungen aus mehreren Lernaufgaben zu sammeln, um mit nur einer kleinen Anzahl von Trainingsbeispielen und Iterationen ein Modell zu trainieren, das sich schnell an neue Aufgaben anpassen kann.. Daher wurden in dieser Studie zunächst PLMs verwendet, um die Wildtypsequenz oder -struktur des Zielproteins und die Sequenz oder Struktur in der Datenbank in einen eingebetteten Vektor zu kodieren.
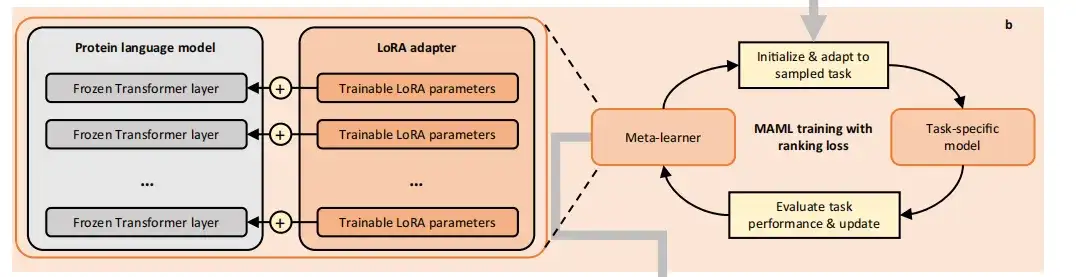
Auch,In der Studie wurde eine gradientenbasierte Meta-Lernmethode namens Model-Agnostic Meta-Learning (MAML) verwendet.Meta-Trainieren Sie PLMs anhand der erstellten Aufgaben. MAML ist in der Lage, optimale anfängliche Modellparameter zu finden, sodass selbst kleine Änderungen an diesen zu erheblichen Verbesserungen der Zielaufgabe führen können. In jeder Iteration besteht der Meta-Trainingsprozess aus zwei Optimierungsebenen und wandelt die PLMs schließlich in initialisierte Meta-Lerner um.
Bei der internen Optimierung verwenden wir den aktuellen Meta-Learner, um einen temporären Basis-Learner zu initialisieren, der dann durch Abtasten der Trainingsdaten der Aufgabe auf ein aufgabenspezifisches Modell aktualisiert wird. Bei der externen Optimierung verwenden wir den Testverlust eines aufgabenspezifischen Modells für die Aufgabe, um den Meta-Lerner zu optimieren.
Um katastrophales Overfitting aufgrund zu geringer Trainingsdaten zu vermeiden,FSFP verwendet Low Rank Adaptation (LoRA), um trainierbare Rangfaktorisierungsmatrizen in PLMs einzufügen.Ihre ursprünglichen vortrainierten Parameter werden eingefroren und alle Modellaktualisierungen werden auf eine kleine Anzahl trainierbarer Parameter beschränkt.
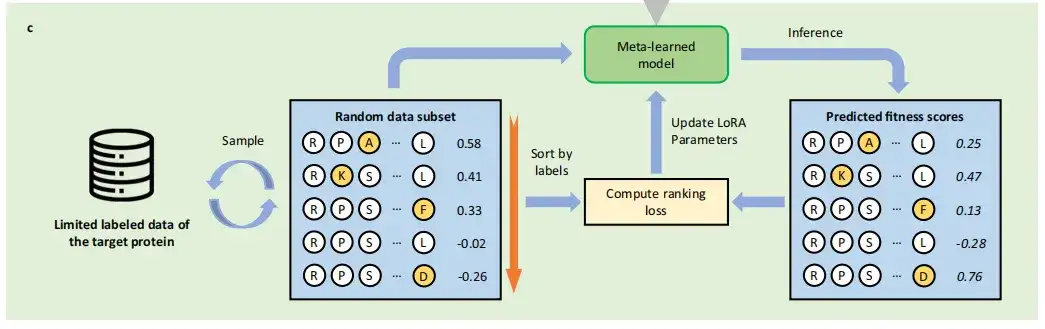
Nach dem Metatraining kann die Studie die Initialisierung basierend auf LoRA-Parametern erhalten und schließlich die metatrainierten PLMs auf die Ziellernaufgabe mit kleinen Stichproben übertragen, d. h. lernen, den Mutationseffekt des Zielproteins mit begrenzten markierten Daten vorherzusagen. Anders als bei herkömmlichen überwachten Lernmethoden zur Vorhersage von Proteinmutationen,FSFP behandelt es als Sortierproblem und nutzt die LTR-Technologie.
Insbesondere lernt FSFP, die Mutationsfitness durch Berechnung des ListMLE-Verlusts zu bewerten. In jeder Iteration trainiert die Studie das Modell, sodass seine Vorhersagen für eine oder mehrere Teilmengen der abgetasteten Daten in Richtung der Grundwahrheit konvergieren. Diese Trainingsschemata werden gleichzeitig zur internen Optimierung in der Transferlernphase unter Verwendung der Zieltrainingsdaten und in der Metatrainingsphase unter Verwendung der Trainingsdaten der Hilfsaufgabe angewendet.
ProteinGym-Benchmark basierend auf 87 Hochdurchsatz-Mutationsdatensätzen
Um die für das Meta-Lernen erforderlichen Trainingsaufgaben zu konstruieren,Diese Methode ruft zunächst vorhandene markierte Mutantendatensätze ab, ruft die Mutationsdatensätze der ersten beiden Proteine ab, die dem Zielprotein am nächsten sind, aus der größten öffentlichen Sammlung von DMS-Datensätzen, ProteinGym, und verwendet die MSA-basierte GEMME-Pseudomarkierungsmethode, um die Mutationsinformationen des Zielproteins zu bewerten und den Datensatz für die dritte Aufgabe zu erstellen. Diese Datensätze können hilfreich sein, um die Auswirkungen von Varianten auf Zielproteine vorherzusagen. Die gekennzeichneten Daten für diese Aufgaben werden zufällig in Trainingsdaten und Testdaten aufgeteilt.
Um die Leistung des Modells zu bewerten,Für diese Studie wurde der Proteinmutationsdatensatz (ProteinGym) als Benchmarkdatensatz ausgewählt. Der Datensatz enthält insgesamt etwa 1,5 Millionen Missense-Varianten aus 87 DMS-Sequenzierungsexperimenten. Da die maximale Eingabelänge von ESM-1v 1.024 beträgt, wurden in dieser Studie Proteine mit mehr als 1.024 Aminosäuren gekürzt und sichergestellt, dass die meisten ihrer Mutationen in den entsprechenden Datensätzen innerhalb des generierten Intervalls auftraten.
Anschließend wählte die Studie zufällig 20 Einzelpunktmutationen als anfänglichen Trainingssatz aus und fügte dann weitere 20 Einzelpunktmutationen hinzu, um die Trainingssatzgröße auf 40 zu erweitern, sowie ähnlich aufgebaute Trainingssätze von 60, 80 und 100. Nach 5 zufälligen DatenaufteilungsprozessenMit dieser Studie kann die Modellleistung auf verschiedenen Partitionen einer bestimmten Trainingsskala gemittelt werden.
FSFP wird erfolgreich auf drei Basismodelle angewendet und bietet erhebliche Vorteile bei Lernaufgaben mit kleinen Stichproben
Theoretisch kann FSFP auf jedes Proteinsprachenmodell angewendet werden, das auf Gradientenabstiegsoptimierung basiert.Um seine Universalität zu überprüfen,Für diese Studie wurden drei repräsentative PLMs – ESM-1v, ESM-2 und SaPro-t – als Basismodelle für das Training ausgewählt und die 650M-Version zur Evaluierung ausgewählt.
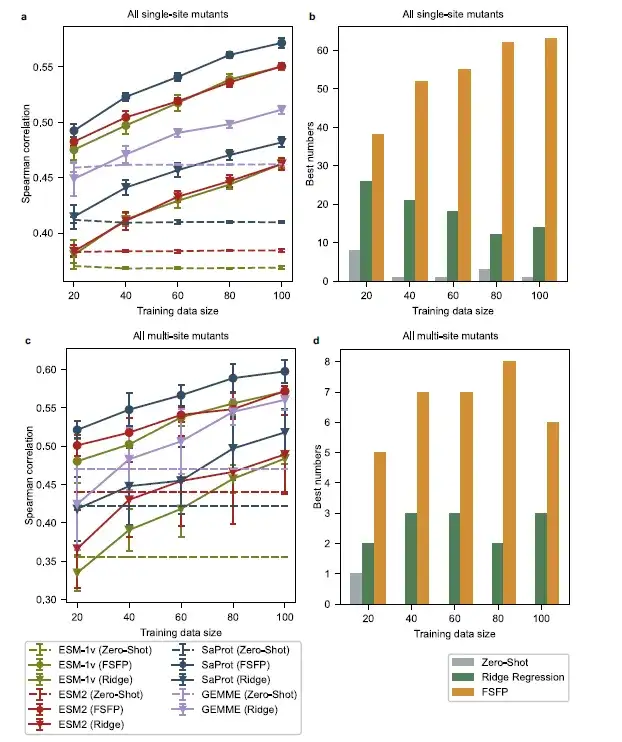
Was die durchschnittliche Leistung betrifft,Mit FSFP trainierte PLMs übertreffen bei allen Trainingsdatengrößen durchweg andere Baselines. Unter ihnen schnitt SaProt (FSFP) am besten ab, während ESM-1v (FSFP) und ESM-2 (FSFP) gleich gut abschnitten. Darüber hinaus erreichten die mit FSFP trainierten PLMs bei den meisten Datensätzen von ProteinGym die beste Spearman-Korrelation. Im Vergleich zur Zero-Shot-Vorhersage verbessert FSFP die Leistung von PLMs bei einzelnen Mutanten um fast 0,1, indem es die Spearman-Korrelation einzelner Mutanten mithilfe von nur 20 Trainingsbeispielen verbessert, und diese Lücke wird noch größer, wenn mehrere Mutanten beteiligt sind. Diese Verbesserungen nehmen mit zunehmendem Trainingsdatensatz zu, was mit den Ergebnissen des Ablationsexperiments dieser Studie übereinstimmt.
Das Modell mit FSFP erzielt gegenüber GEMME und seiner erweiterten Version der Ridge-Regression bei allen Trainingsbeispielen erhebliche Verbesserungen. Dies deutet darauf hin, dass FSFP nicht nur das Wissen über die Ausrichtung mehrerer Sequenzen in GEMME auf PLM überträgt, sondern es durch Multitasking-Lernen auch erfolgreich mit den Überwachungsinformationen aus den Zieltrainingsdaten kombiniert.Dies bestätigt erneut den Vorteil von FSFP bei Lernaufgaben mit wenigen Stichproben.
Extrapolationsleistungsbewertung, Spearman-Korrelationsbewertung von FSFP-trainierten PLMs ist besser
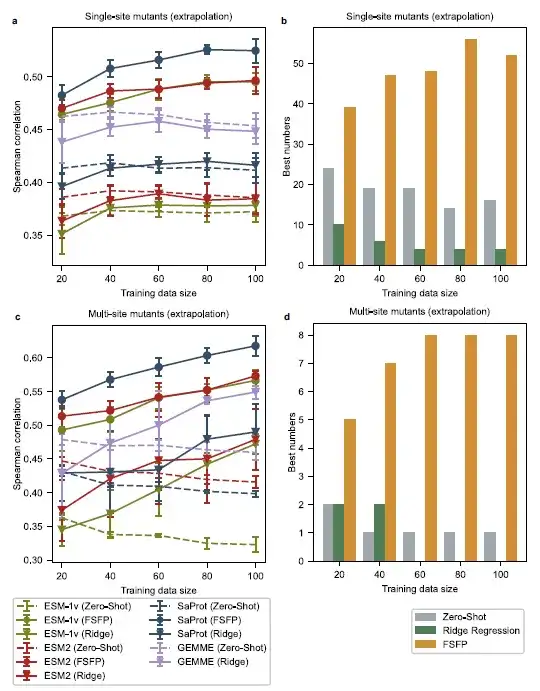
Die Forscher wählten aus jedem ursprünglichen Testsatz alle Einzelpunktmutanten aus, deren Mutationsstellen sich von den Trainingsbeispielen unterschieden, und erhielten so einen Testsatz von Einzelpunktmutanten, die sich von den Trainingsbeispielen unterschieden. Anschließend wählten die Forscher Multipunktmutanten aus, deren einzelne Mutationen sich nicht mit denen in den Trainingsdaten überschnitten, was zu einem weiteren anspruchsvollen Testsatz führte. In dieser Einstellung stellen wir fest, dass die Zero-Shot-Leistung des Basismodells erheblich mit der Größe des Trainingssatzes skaliert.
Bei einzelnen Punktmutationen an unterschiedlichen Positionen zeigt das durch Ridge-Regression verbesserte Modell auch bei 100 Trainingsbeispielen keine bessere Leistung als das Basismodell. Bei Mehrpunktmutationen kann die Ridge-Regressionsmethode die Leistung von GEMME und ESM-2 nicht effektiv verbessern, wenn die Trainingsgröße weniger als 60 beträgt. Im Gegensatz dazu wiesen mit FSFP trainierte PLMs im Vergleich zu allen Basismodellen bei allen Trainingsgrößen höhere Spearman-Korrelationswerte auf. Auch,Die leistungsstärksten Modelle für die meisten Datensätze sind diejenigen, die mit FSFP trainiert wurden.
Umfassender Vergleich von 4 Proteinen, FSFP bietet größere Vorteile beim Training mit kleinen Datensätzen
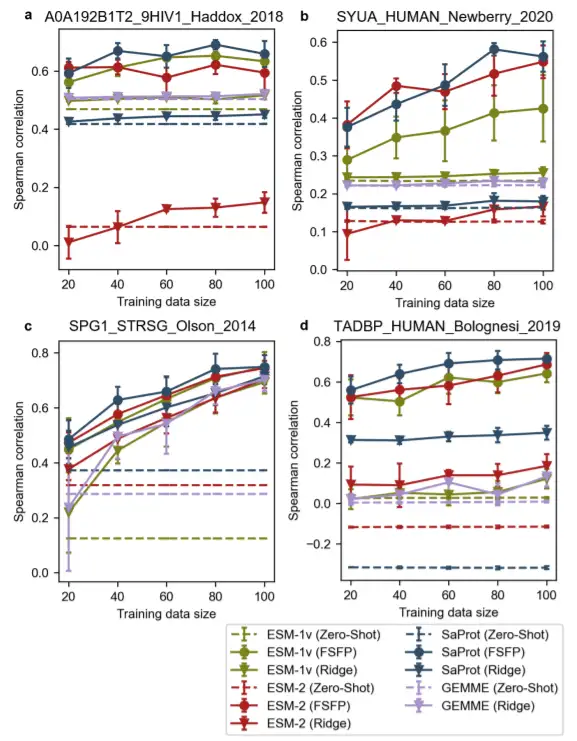
Um die Anwendbarkeit und Generalisierung von FSFP weiter zu demonstrieren,Die Studie zeigte auch die Vergleichsergebnisse zwischen verschiedenen Methoden für vier Proteine: das Hüllprotein Env von HIV, das menschliche α-Synuclein, Protein G (GB1), das menschliche TAR-DNA-bindende Protein 43 (TDP-43). In mehreren dieser Fälle wiesen ein oder mehrere unbeaufsichtigte Modelle eine schlechte Leistung auf.
Bemerkenswerterweise liegen bei TDP-43 alle Spearman-Korrelationen für Nullstichprobenvorhersagen nahe Null. Mit Ausnahme von GB1 erzielen die meisten durch Ridge-Regression verbesserten Modelle auch bei größeren Trainingsdatensätzen keine signifikanten Leistungsverbesserungen. Im Gegensatz dazu können vortrainierte Modelle erhebliche Fortschritte erzielen, wenn sie mit FSFP an kleinen Datensätzen trainiert werden.
Durch die Verwendung von FSFP zur Entwicklung der Phi29 DNA-Polymerase erhöhte sich die positive Rate um 25%
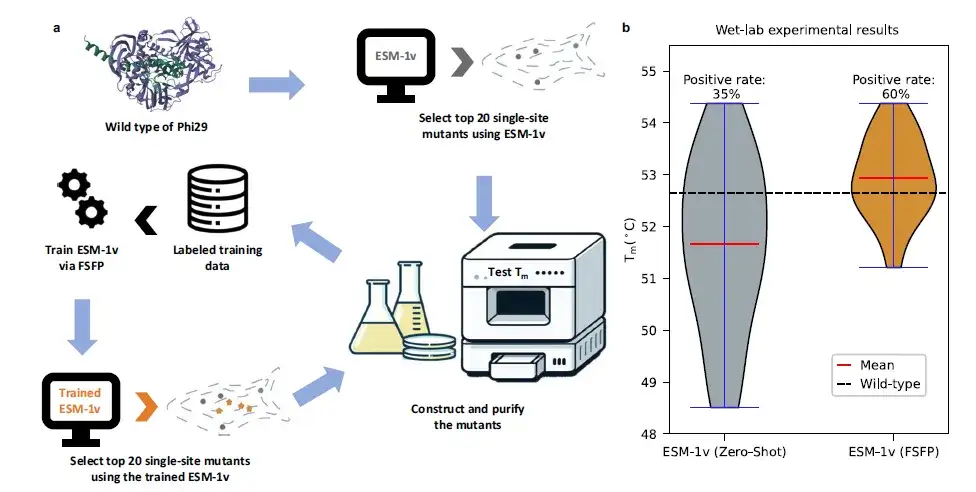
Die Studie untersuchte auch einen spezifischen Fall einer Modifikation des Proteins Phi29.Es wurde eine Nasstestüberprüfung durchgeführt.Basierend auf einem begrenzten Satz nasser experimenteller Daten wurde in dieser Studie FSFP zum Trainieren von ESM-1v verwendet, um neue Einzelstellenmutanten zu finden und eine experimentelle Verifizierung durchzuführen. Beim Vergleich der 20 besten Vorhersageergebnisse von ESM-1v vor und nach dem FSFP-Training stieg der durchschnittliche Tm-Wert um mehr als 1 °C und die Positivrate um 25%.
Insbesondere wurden die besten Mutanten (d. h. die Mutanten mit den höchsten Tm-Werten), die von ESM-1v (FSFP) gefunden wurden, auch von ESM-1v (Zero-Shot) empfohlen. Unter den von ESM-1v (FSFP) vorhergesagten positiven Mutanten tauchten jedoch 9 nicht in den Trainingsdaten auf, was darauf hindeutet, dass FSFP es PLMs ermöglichen kann, mehr Proteinvarianten zu identifizieren.Diese Ergebnisse bestätigen das Potenzial von FSFP, iterative Zyklen des Designs und der Tests von Protein-Engineering zu beschleunigen.Dies könnte dazu beitragen, Proteine mit verbesserten funktionellen Eigenschaften zu entwickeln.
Ein typischer Vertreter der KI für Bioengineering, eine starke Allianz steht an der Spitze der Zeit
Heute, wo KI und wissenschaftliche Forschung eng miteinander verzahnt sind, stehen wir vor einer historischen Chance. Professor Hong Liang ist der Ansicht, dass Chinas biopharmazeutische Industrie zwar über eine starke Stärke verfügt, ihr Gewinnanteil in der internationalen Industriekette jedoch noch steigerungsfähig ist. Durch KI haben wir die Möglichkeit, „die Spur zu wechseln und zu überholen“ und die Leistungsfähigkeit der künstlichen Intelligenz direkt zu nutzen, um die Branchenentwicklung voranzutreiben. Auf der Grundlage dieses Konzepts haben Professor Hong Liang und der Forscher Tan Pan endlose Erkundungen im Bereich der KI für die Biotechnik gestartet.
Dr. Tan Pan konzentriert sich auf Molekularbiophysik, funktionales Proteindesign mithilfe künstlicher Intelligenz und Arzneimittelmoleküldesign.Veröffentlichte 15 SCI-Artikel in Zeitschriften wie Nature Communications, PRL, Journal of Cheminformatics, PCCP usw. Entwickelte eine Vielzahl von KI-gestützten Algorithmen für Proteindesign und -modifikation. Durch die Kombination der Fachkompetenz von Professor Hong Liang mit den KI-Algorithmen von Dr. Tan Pan hat die gemeinsame Forschung der beiden Parteien wiederholt Erfolge hervorgebracht.
Im Laufe der Jahre haben sich die beiden Parteien auf innovative Forschung zur allgemeinen künstlichen Intelligenz im Bereich des Protein-Engineerings konzentriert und erfolgreich die Pro-Serie der allgemeinen künstlichen Intelligenz für das Protein-Engineering entwickelt. Ähnlich wie ChatGPT die menschliche Sprache versteht, verwendet die Pro-Serie große Modelle, um die Aminosäureanordnung von Proteinen in der Natur zu verstehen und Proteinprodukte mit überlegener Leistung zu entwickeln. Darunter befinden sich zwei Meilensteinprodukte in der industriellen Anwendung:
* Extrem alkaliresistenter Einzeldomänen-Antikörper:Das weltweit erste großmaßstäblich modellbasierte Proteinprodukt, das gemeinsam mit Jinsai Pharmaceuticals entwickelt wurde, hat eine industrielle Produktion von 5.000 Litern erreicht und bietet eine neue Lösung für die Reinigung biologischer Makromoleküle.
* Glykosyltransferase:In Zusammenarbeit mit Hanhai New Enzyme haben wir das Enzym zur Herstellung von EPS-G7 entwickelt, dem Kernmaterial für das Pankreatitis-Screening. Dadurch haben wir das langjährige ausländische Monopol gebrochen und die Kosten erheblich gesenkt.
Bei diesen beiden Fällen handelt es sich um den weltweit ersten und zweiten groß angelegten Modellentwurf sowie die erfolgreiche Ausweitung der Produktion von Proteinprodukten in die Industrialisierungsphase. Aufgrund seiner umfassenden Kenntnisse im Bereich des KI-Proteindesigns gründete Professor Hong Liang im Jahr 2021 die Shanghai Tianwu Technology Co., Ltd. In nur drei Jahren schloss das Unternehmen nicht nur mehrere Proteindesignprojekte ab, sondern erhielt in einer Pre-A-Finanzierungsrunde auch mehrere zehn Millionen Yuan. Zu den Investoren zählen namhafte Institutionen wie Glory Ventures und GSR Ventures.
Derzeit decken die Dienstleistungen des Unternehmens zahlreiche Bereiche ab, beispielsweise innovative Arzneimittel, In-vitro-Diagnostik, synthetische Biologie usw. Das Unternehmen ist aktiv auf der Suche nach Kooperationen mit weiteren wissenschaftlichen Forschungsinstituten und Unternehmen und hat sich zum Ziel gesetzt, im Bereich des Protein-Engineerings nationale und sogar globale Maßstäbe zu setzen.
Im hart umkämpften Bereich des Protein-Engineerings ist die Vision von Professor Hong Liang klar:Wir müssen nicht nur eine nationale Führungsrolle übernehmen, sondern auch eine globale Führungsrolle.Auf ihrem zukünftigen Weg in die wissenschaftliche Forschung streben Professor Hong Liang und sein Team danach, die Zusammenarbeit mit wissenschaftlichen Forschungseinrichtungen und Unternehmen weltweit intensiv auszubauen, die unendlichen Möglichkeiten des Proteindesigns kontinuierlich zu erforschen und auf diesem Gebiet technologische Durchbrüche und Anwendungsinnovationen anzustreben, um im Inland Maßstäbe zu setzen und international Spitzenleistungen zu demonstrieren.
Abschließend empfehle ich eine Online-Aktivität zum wissenschaftlichen Austausch. Interessierte Freunde können den QR-Code scannen, um teilzunehmen!









