Command Palette
Search for a command to run...
Beste Studentenarbeit Bei CVPR! Ein Großer Datensatz Mit 10 Millionen Bildern Und Über 450.000 Arten: Das Multimodale Modell BioCLIP Ermöglicht Zero-Shot-Learning

Anders als im traditionellen akademischen Bereich, wo großen Wert auf die Veröffentlichung von Fachzeitschriften gelegt wird, sind in der Computerwelt, insbesondere in den Bereichen maschinelles Lernen, Computersehen, künstliche Intelligenz usw., Top-Konferenzen das Maß aller Dinge. Von hier aus werden zahlreiche „heiße Forschungsrichtungen“ und „innovative Methoden“ hervorgehen.
Als eine der drei akademisch einflussreichsten Konferenzen im Bereich Computer Vision und sogar künstliche Intelligenz brach die diesjährige International Conference on Computer Vision and Pattern Recognition (CVPR) bisherige Rekorde hinsichtlich Konferenzgröße und Anzahl angenommener Beiträge.

Laut der jüngsten offiziellen Ankündigung von CVPR,CVPR 2024 ist die größte und am besten besuchte Konferenz in der Geschichte der Konferenz geworden.Seit dem 19. Juni liegt die Zahl der Teilnehmer vor Ort bei über 12.000.

Darüber hinaus präsentiert die CVPR als führende Veranstaltung zum Thema Computer Vision jedes Jahr die neuesten Forschungsergebnisse im aktuellen visuellen Bereich. Von den 11.532 gültigen Beiträgen, die in diesem Jahr eingereicht wurden, wurden im Vergleich zum CVPR 2023 2.719 Beiträge angenommen.Die Anzahl der eingegangenen Beiträge stieg um 20,61 TP3T, während die Annahmequote um 2,21 TP3T sank.Diese Daten zeigen, dass die Popularität, der Wettbewerb und die Qualität der Gewinnerarbeiten des CVPR 2024 zugenommen haben.

Am frühen Morgen des 20. Juni (Peking-Zeit) gab CVPR 2024 offiziell die besten Arbeiten und andere Auszeichnungen dieser Sitzung bekannt. Laut Statistik wurden insgesamt 10 Arbeiten ausgezeichnet.Darunter befinden sich 2 Auszeichnungen für die besten Arbeiten, 2 Auszeichnungen für die besten studentischen Arbeiten, 2 Nominierungen für die besten Arbeiten und 4 Nominierungen für die besten studentischen Arbeiten.

In,Als beste studentische Arbeit wurde „BIoCLIP: A Vision Foundation Model for the Tree of Life“ ausgezeichnet.In diesem Zusammenhang kommentierte Sara Beery, Assistenzprofessorin am Computer Science and Artificial Intelligence Laboratory des MIT, dass die Autoren und das Team „wohlverdiente“ Gewinner seien, und der Erstautor des Papiers, Samuel Stevens, war der erste, der auf der Plattform seinen Dank ausdrückte.
HyperAI wird „BIoCLIP: Ein Vision Foundation Model für den Baum des Lebens“ unter den Aspekten Datensatz, Modellarchitektur, Modellleistung usw. umfassend interpretieren und weitere Errungenschaften von Sam Stevens für alle zusammenfassen.
Downloadadresse:
https://arxiv.org/pdf/2311.18803
Erstellen des größten und vielfältigsten biologischen Bilddatensatzes
Der derzeit größte biologische Bilddatensatz für maschinelles Lernen ist iNat21, der 2,7 Millionen Bilder enthält und 10.000 Arten abdeckt. Obwohl die Klassifizierungsbreite von iNat21 im Vergleich zu allgemeinen Domänendatensätzen wie ImageNet-1k stark verbessert wurde, sind 10.000 Arten in der Biologie immer noch selten. Die Internationale Union zur Bewahrung der Natur (IUCN) meldete im Jahr 2022 mehr als 2 Millionen bekannte Arten, darunter allein mehr als 10.000 Vogel- und Reptilienarten.
Um das Problem der Artenkategoriebeschränkungen in biologischen Bilddatensätzen zu lösen,Die Forscher erstellten einen Datensatz namens TreeOfLife-10M, der 10 Millionen Bilder enthält.Mit einer Erfassung von über 450.000 Arten wurde ein revolutionärer Durchbruch hinsichtlich der Datensatzgröße und Artenvielfalt erzielt.
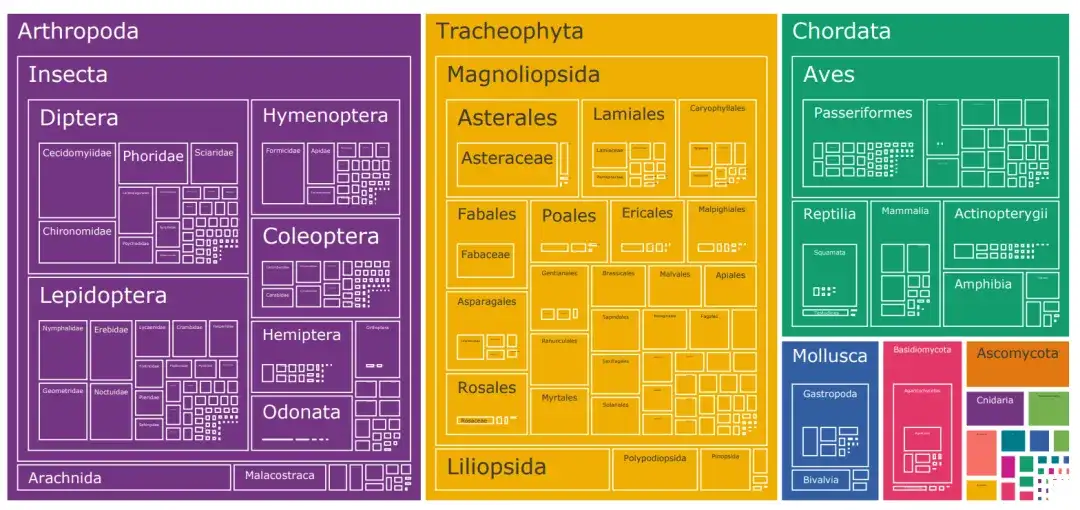
Der Datensatz kombiniert biologische Bilder von iNaturalist, BIOSCAN-1M und der Encyclopedia of Life (EOL).
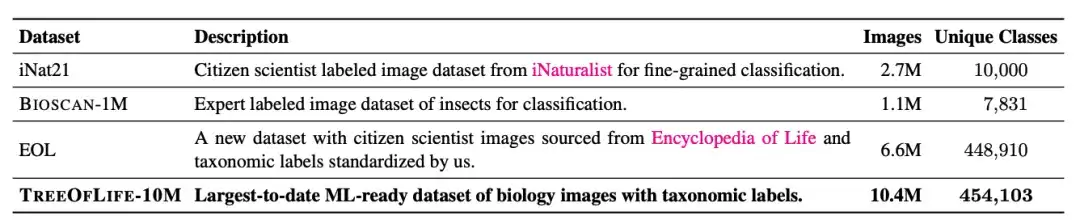
Zusätzlich zu den 10.000 Artenkategorien, die von iNat21 abgedeckt werden, luden die Forscher 6,6 Millionen Bilder von EOL herunter, um TreeOfLife-10M auf weitere 440.000 Taxa zu erweitern. Um dem Basismodell dabei zu helfen, extrem detaillierte visuelle Darstellungen von Insekten zu erlernen, integrierten die Forscher gleichzeitig auch BIOSCAN-1M, einen aktuellen Datensatz mit 1 Million Laborbildern von Insekten, die 494 verschiedene Familien und 7.831 Artenklassifizierungen abdecken.
TreeOfLife-10M Download-Adresse:
https://go.hyper.ai/Gliol
Multimodales Modell BioCLIP: Verbesserung der Generalisierungsfähigkeit basierend auf CLIP
Im Vergleich zu allgemeinen Aufgaben ist der Beschriftungsraum der biologischen Computervision umfangreicher. Es gibt nicht nur eine riesige Anzahl an Klassifizierungsanmerkungen, die Annotationen sind auch im hierarchischen Klassifizierungssystem miteinander verknüpft. Dies bringt zweifellos enorme Herausforderungen für das Training von Basismodellen mit hoher Artenabdeckung und starken Generalisierungsfähigkeiten mit sich.
Die Forscher stützen sich auf Hunderte von Jahren Erfahrung in der biologischen Forschung und sind überzeugt, dass das Modell, wenn es die Struktur des Annotationsraums erfolgreich kodieren kann, möglicherweise auch dann, wenn eine bestimmte Art nicht beobachtet wurde, in der Lage ist, die zugehörige Gattung oder Familie zu identifizieren und eine entsprechende Darstellung bereitzustellen. Diese hierarchische Darstellung wird dazu beitragen, das Lernen neuer Taxa mit wenigen oder sogar gar keinen Versuchen zu erreichen.
Auf dieser Grundlage entschieden sich die Forscher für CLIP, eine von OpenAI entwickelte multimodale Modellarchitektur.Und nutzen Sie das multimodale kontrastive Lernziel von CLIP, um kontinuierlich auf TREEOFLIFE-10M vorzutrainieren.
Insbesondere trainiert CLIP zwei unimodale Einbettungsmodelle, den visuellen Encoder und den Textencoder, um die Merkmalsähnlichkeit zwischen positiven Paaren zu maximieren und die Merkmalsähnlichkeit zwischen negativen Paaren zu minimieren, wobei die positiven Paare aus den Trainingsdaten stammen und die negativen Paare alle anderen möglichen Paare im Stapel sind.

Ein wichtiger Vorteil von CLIP besteht außerdem darin, dass sein Textencoder Freitext akzeptiert, der mit den verschiedenen Klassennamenformaten im biologischen Bereich zurechtkommt. Hinsichtlich der Textform berücksichtigten die Forscher in dieser Studie vor allem:
* Taxonomischer Name:Die standardmäßige biologische Klassifizierung erfolgt in sieben Stufen, von oben nach unten: Reich, Stamm, Klasse, Ordnung, Familie, Gattung und Art. Für jede Art wird die Taxonomie „abgeflacht“, indem alle Bezeichnungen von der Wurzel bis zu den Blättern zu einer einzigen Zeichenfolge zusammengefasst werden, die den taxonomischen Namen darstellt.
* Wissenschaftlicher Name:Besteht aus Gattung und Art.
* Allgemeiner Name:Klassennamen sind normalerweise in Latein, was in allgemeinen Bild-Text-Vortrainingsdatensätzen nicht üblich ist. Stattdessen sind gebräuchliche Namen wie „Schwarzschnabelelster“ häufiger. Es ist zu beachten, dass es möglicherweise keine Eins-zu-eins-Zuordnung zwischen gebräuchlichen Namen und Taxa gibt. Eine Art kann mehrere gebräuchliche Namen haben oder derselbe gebräuchliche Name kann sich auf mehrere Arten beziehen.
In praktischen Anwendungen gibt es möglicherweise nur eine Art von Anmerkungseingabe. Um die Flexibilität beim Denken zu erhöhen,Die Forscher schlugen eine Trainingsstrategie für gemischte Texttypen vor.Das heißt, bei jedem Trainingsschritt wird jedes Eingabebild mit einem Text gepaart, der zufällig aus allen verfügbaren Textarten ausgewählt wird. Experimente zeigen, dass diese Trainingsstrategie nicht nur den Generalisierungsvorteil von Klassifikationsnamen beibehält, sondern auch mehr Flexibilität beim Denken bietet.
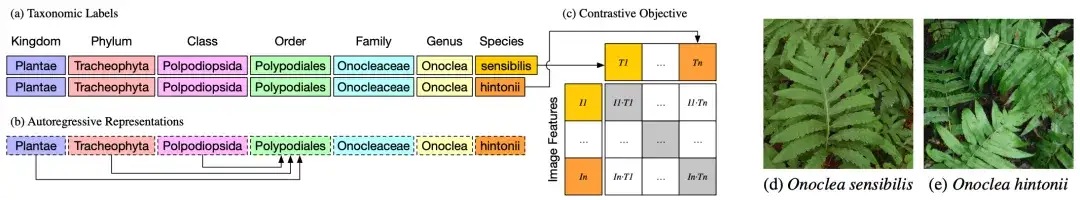
Wie in Abbildung a oben gezeigt, sind die taxonomischen Gruppen oder Klassifizierungsbezeichnungen zweier verschiedener Pflanzen, Onoclea sensibilis (d) und Onoclea hintonii (e), bis auf die Art genau gleich.
Wie in Abbildung 2b oben gezeigt, ist der Textcodierer ein autoregressives Sprachmodell, das die hierarchischen Darstellungen der Taxonomie auf natürliche Weise codieren kann, wobei die Ordnungsdarstellung Polypodiales nur von höheren Ordnungen abhängen und Informationen aus Reich-, Stamm- und Klassentoken aufnehmen kann. Diese hierarchischen Darstellungen taxonomischer Bezeichnungen werden in ein standardmäßiges kontrastives Vortrainingsziel eingespeist und mit den Bilddarstellungen (d) und (e) abgeglichen.
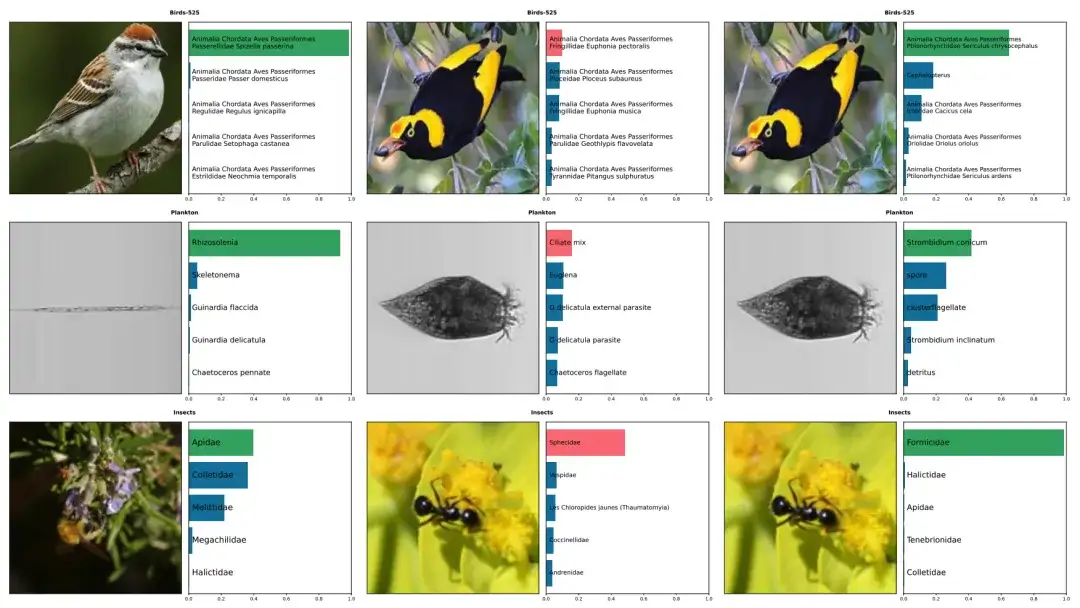
Die obige Abbildung zeigt ein Beispiel für die von BioCLIP und CLIP erstellten Vorhersagen für fünf Arten, darunter Vögel 525, Plankton und Insekten. Die richtigen sind grün markiert, die falschen rot. Die linke Spalte zeigt die korrekten Vorhersagen von BioCLIP. In der Mitte und rechts sind Bilder, die von CLIP falsch, von BioCLIP jedoch richtig annotiert wurden.
BioCLIP bietet gute Leistung bei Zero-Shot- und Few-Shot-Aufgaben
Die Forscher verglichen BioCLIP mit einem allgemeinen Sehmodell. ErgebnisseBioCLIP schneidet sowohl bei Zero-Shot- als auch bei Few-Shot-Aufgaben gut ab und übertrifft CLIP deutlich und OpenCLIP,Die durchschnittliche absolute Verbesserung bei Zero-Shot- und Few-Shot-Aufgaben beträgt 17% bzw. 16%. Die intrinsische Analyse zeigte außerdem, dass BioCLIP eine feinkörnigere hierarchische Darstellung lernte, die mit dem Baum des Lebens übereinstimmt, was seine überlegene Generalisierungsfähigkeit erklärt.
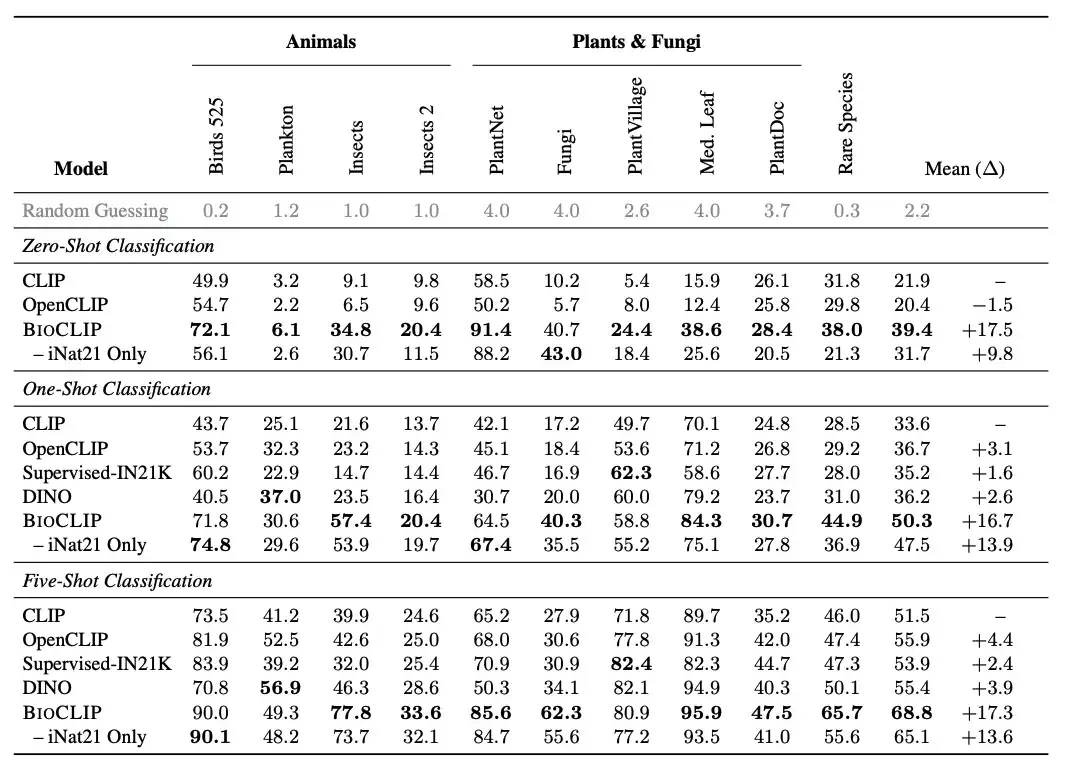
Konkret führten die Forscher eine neue Bewertungsaufgabe mit dem Titel „SELTENE ARTEN“ ein, bei der etwa 25.000 Arten aus der Roten Liste der IUCN erfasst wurden, die als potenziell gefährdet, gefährdet, stark gefährdet, vom Aussterben bedroht oder in der Wildnis ausgestorben eingestuft sind. Die Forscher wählten 400 solcher Arten mit mindestens 30 Bildern im EOL-Datensatz aus und entfernten sie aus TreeOfLife-10M.Ein neues Testset seltener Arten wurde erstellt,Es gibt 30 Bilder für jede Art.
Wie in der Abbildung oben gezeigt, übertrifft BioCLIP das CLIP-Basismodell und das mit iNat21 trainierte CLIP-Modell bei der Zero-Shot-Klassifizierung deutlich, insbesondere bei ungesehenen Klassifizierungen (siehe Spalte „Seltene Arten“).
Fruchtbare Ergebnisse, Erkundung der wissenschaftlichen Forschung hinter dem besten BioCLIP
„BioCLlP: A Vision Foundation Model for the Tree of Life“ wurde gemeinsam von der Ohio State University, Microsoft Research, der University of California, Irvine und dem Rensselaer Polytechnic Institute veröffentlicht.Der Erstautor des Artikels, Dr. Samuel Stevens, und der korrespondierende Autor, Jiaman Wu, sind beide von der Ohio State University.
Obwohl Samuel Stevens sich auf seiner persönlichen Website bescheiden als „eine Person, die sich selbst nicht besonders ernst nimmt“ beschreibt, ist er, gemessen an seinen fruchtbaren wissenschaftlichen Forschungsergebnissen und unermüdlichen Bemühungen der letzten Jahre, offensichtlich jemand, der die wissenschaftliche Forschung ernst nimmt.
Es wird davon ausgegangen, dass Samuel Stevens seit 2017 mit Computern arbeitet. Das multimodale Modell BioCLlP ist ein Forschungsergebnis, das er im Dezember 2023 veröffentlichte und das im Februar 2024 von CVPR 2024 angenommen wurde.
Tatsächlich ist Computer-Vision-Arbeit wie BioCLlP nur eine seiner Forschungsrichtungen. Er hat ein breites Spektrum an Interessen und hat eine Reihe von Forschungsarbeiten in Bereichen wie KI für Krypto und verschiedenen LLM-Projekten durchgeführt.
So beteiligte er sich beispielsweise am „MMMU: A Massive Multi-discipline Multimodal Understanding and Reasoning Benchmark for Expert AGI“.Ein neuer Benchmark MMMU (Massive Multi-Task Language Understanding) wird vorgeschlagen.Als einer der einflussreichsten Benchmarks für die Bewertung großer Modelle in der Branche konzentriert sich MMMU auf die Nutzung von Wissen in bestimmten Bereichen (Wissenschaft, Gesundheit und Medizin, Geisteswissenschaften usw.) für fortgeschrittene Wahrnehmung und Argumentation, wobei multimodale Modelle in der Lage sein müssen, ähnliche Aufgaben wie Experten zu erfüllen.
Die Forscher verwendeten es, um 14 Open-Source-LMMs und das proprietäre GPT-4V (ision) zu bewerten und stellten fest, dass selbst das fortschrittliche GPT-4V nur eine Genauigkeit von 56% erreichte, was zeigt, dass im Modell noch viel Raum für Verbesserungen besteht. Als Reaktion darauf äußerten die Forscher die Hoffnung, dass der Benchmark die Community dazu inspirieren würde, die nächste Generation multimodaler Basismodelle zu entwickeln, um künstliche allgemeine Intelligenz auf Expertenniveau zu erreichen.
MMMU: https://mmmu-benchmark.github.io
Natürlich sind auch seine Leidenschaft für die wissenschaftliche Forschung und seine offene Einstellung wichtige Faktoren für seinen Erfolg. Gestern kam die Nachricht heraus, dass BioCLlP zur besten studentischen Arbeit gekürt wurde, und Dr. Samuel Stevens äußerte seine Ansichten sofort über soziale Plattformen nach außen: „Wenn Sie über Computer Vision von Tieren, multimodale Basismodelle oder KI für die Wissenschaft sprechen möchten, senden Sie mir bitte eine private Nachricht!“

Erwähnenswert ist, dass Dr. Samuel Stevens nicht nur die wissenschaftliche Forschung vorantreibt, sondern auch nie vergisst, die jüngere Generation zu unterstützen. Auf seiner persönlichen Website gibt er auch Tipps für Anfänger: „Wenn Sie in maschinelles Lernen und künstliche Intelligenz einsteigen möchten, sollten Sie mit dem Kurs „Machine Learning“ von Coursera und „Neural Networks: Zero to Hero“ von Andrej Karpathy beginnen. Beide Kurse sind von sehr hoher Qualität und sollten im Vergleich zu anderen kostenlosen Ressourcen einen großen Mehrwert bieten.“
Quellen:
1. https://samuelstevens.me/#news
Zum Schluss empfehle ich noch eine Aktivität!
Scannen Sie den QR-Code, um sich für das 5. Offline-Treffen des Technologie-Salons „Meet AI Compiler“ anzumelden ↓









