Command Palette
Search for a command to run...
Neue Errungenschaft Des Fudan Institute of Brain Science: Verwendung Der Semantischen Segmentierung Als Referenz Zur Entwicklung Eines Räumlichen Transkriptom-semantischen Annotationstools Pianno

Seit der Wahl zur Technologie des Jahres 2020 durch Nature MethodsDie „räumliche Transkriptomik“ hat sich heute zu einer der angesagtesten revolutionären Technologien im Bereich der Biowissenschaften entwickelt.Einfach ausgedrückt kann diese Technologie räumliche Gewebeinformationen und Transkriptomdaten gewinnen, Genexpressionsmuster in Geweben sowie biologische Eigenschaften wie die räumliche Positionsbeziehung von Zellpopulationen aus der Zeit- und Raumdimension genau analysieren. Es ist von großem Wert für die Forschung in den Bereichen Krankheitsforschung, Wachstum und Entwicklung, Organstruktur und Artenentwicklung.
Da die räumliche Transkriptomik in der akademischen Forschung weiterhin beliebt ist, sind auch räumliche Transkriptomik-Technologien wie 10x Visium, Slide-seq und Stereo-seq entstanden. Diese neuesten Errungenschaften und Fortschritte verändern die menschliche Forschung zu Genexpressionsmustern in Geweben völlig. Um die Komplexität biologischer Systeme vollständig zu verstehen, reicht es jedoch nicht aus, lediglich das Genexpressionsprofil an bestimmten physikalischen Koordinaten innerhalb eines Gewebes zu erhalten. Um die Komplexität vollständig zu verstehen, ist es notwendig, die biologische Identität jedes räumlichen Punkts innerhalb des Gewebes zu identifizieren.
Derzeit werden auf maschinellem Lernen basierende Methoden häufig eingesetzt, um Cluster räumlicher Punkte zu identifizieren und ihre biologische Identität mithilfe von Markergenen zu interpretieren.Allerdings sind diese Methoden oft dadurch eingeschränkt, dass sie nicht in der Lage sind, explizite Verbindungen zu bekannten Strukturen innerhalb des Clusters herzustellen.Darüber hinaus wird häufig eine manuelle Annotation verwendet, um die Identifizierung bekannter Strukturen zu unterstützen. Diese Methode ist jedoch häufig durch die Fachkenntnisse und das subjektive Urteil des Forschers eingeschränkt und kann nicht auf groß angelegte Analysen angewendet werden.
Als Reaktion auf die oben genannten Herausforderungen veröffentlichte das Team von Zhu Ying vom Institute of Brain Science der Universität Fudan kürzlich in „Nature Communications“ ein Forschungsergebnis mit dem Titel „Pianno: ein probabilistisches Framework zur Automatisierung der semantischen Annotation für die räumliche Transkriptomik“.Das Forschungsteam übernahm die Idee der „semantischen Segmentierung“ aus der Computervision, schlug das Konzept der „räumlichen Transkriptom-semantischen Annotation“ vor und entwickelte das räumliche Transkriptom-semantische Annotationstool Pianno.Durch die Fähigkeit, Strukturen oder Zelltypen für räumliche Punkte innerhalb von Geweben automatisch zu definieren, können Informationen aus mehreren Dimensionen kombiniert werden, um die Interpretation komplexer biologischer Systeme zu verbessern.
Forschungshighlights:
* Pianno verfügt über einen einzigartigen automatischen Beschriftungsmodus, der auf Daten anwendbar ist, die von verschiedenen räumlichen Transkriptomik-Technologien generiert werden
* Pianno zeigt im Vergleich zu modernsten räumlichen Clustering-Methoden eine überlegene Leistung und bietet neue Einblicke in räumliche Transkriptomik-Daten

Papieradresse:
https://doi.org/10.1038/s41467-024-47152-4
Datensatz: Öffentliche Daten, strenge Berechnungen
Bei den in dieser Studie verwendeten Datensätzen handelt es sich hauptsächlich um öffentliche Datensätze von verschiedenen räumlichen Technologieplattformen, darunter der Datensatz dlPFC zum dorsolateralen präfrontalen Kortex des Menschen, der Datensatz Stereo-seq zum Coronalschnitt der Großhirnhemisphäre einer erwachsenen Maus, der Datensatz Slide-seqV2 zur Vorverarbeitung des Hippocampus einer Maus, der Datensatz ST zum duktalen Adenokarzinom des menschlichen Pankreas, der Datensatz Visium zum Brustkrebs des Menschen, der Datensatz scRNA-seq zum primären visuellen Kortex einer Maus, die snRNA-seq-Datensätze mehrerer menschlicher Kortexregionen und die DAPI-Färbungsbilder des Riechkolbens einer Maus.
Um die Zerstörung der ursprünglichen biologischen Merkmale durch Bildverarbeitungstechnologien wie Rauschunterdrückung, Glättung und Schärfung zu vermeiden, wurden in der StudieDas Forschungsteam erstellte einen Bayes-Klassifikator auf Grundlage der Rohzählungen, um die anfänglichen Anmerkungen zu verfeinern.Gleichzeitig wandte das Forschungsteam ein Markov-Random-Field-Prior-Modell höherer Ordnung (MRF) an. Da im Kontext der räumlichen Transkriptomik die Genexpression und die räumliche Position jeder Stelle zusammen betrachtet werden müssen, übernahm das Forschungsteam auch das Modell des räumlichen Poisson-Punktprozesses (sPPP).
Pianno: Innovatives neues Tool zur automatisierten räumlichen semantischen Annotation des Transkriptoms
Das Forschungsteam schlug ein neues Tool namens Pianno vor, das auf dem Bayes-Framework basiert.Das Tool kombiniert Markov-Zufallsfelder (MRFs) mit räumlichen Poisson-Punktprozessen (sPPPs) und nutzt die Fähigkeit von sPPPs voll aus, die Verteilung von RNA-Sequenzzähldaten zu modellieren und gleichzeitig die Standortinformationen räumlicher Punkte zu berücksichtigen. Es kann die biologische Identität jedes Punkts in räumlichen Transkriptomdaten automatisch anhand einer vordefinierten Liste von Markergenen kommentieren.
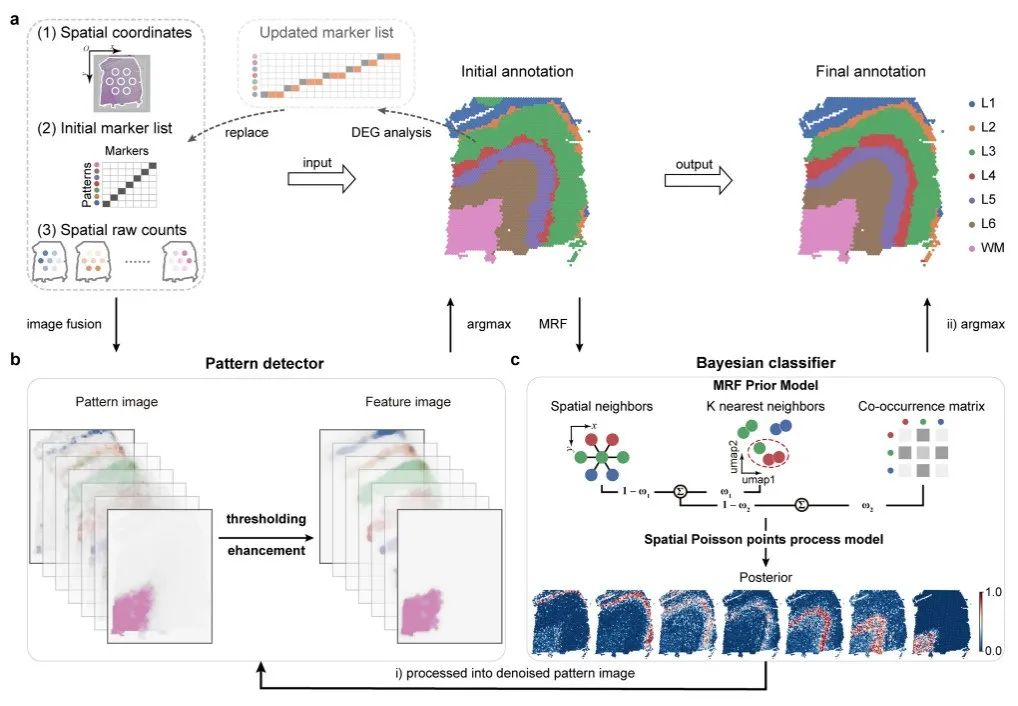
Die von Pianno eingegebenen räumlichen Transkriptomdaten bestehen aus räumlichen Koordinaten, einer anfänglichen Markergenliste und Rohgenzählungen.Jedes Muster liefert mindestens ein bekanntes Token.
Der Annotationsprozess besteht aus einem ersten Segmentierungsschritt und einem Verfeinerungsschritt:
Im ersten SegmentierungsschrittDer räumliche Ausdruck jedes Gens wurde in ein Graustufenbild umgewandelt. Für jedes Zielmuster wird ein Musterbild erstellt, indem die Graustufenbilder der mit diesem Muster verknüpften Markergene aggregiert werden. Anschließend werden zusätzliche Kandidatenmarkergene für jedes Muster ermittelt, um die ursprüngliche Markerliste zu aktualisieren. Die aktualisierten Markierungslisten werden in nachfolgende Verfeinerungsschritte integriert, wobei ihre einzigartigen Expressionsmuster in den ursprünglich annotierten Strukturen berücksichtigt werden.
Im VerfeinerungsschrittEin Bayes-Klassifikator wird erstellt, um die Posterior-Wahrscheinlichkeit zu bewerten, dass jeder räumliche Punkt zu unterschiedlichen Modi gehört. Anschließend wird die Annotation basierend auf der Posterior-Wahrscheinlichkeit aktualisiert.
Pianno bietet zwei Methoden zum Aktualisieren von Anmerkungen:
* Für kontinuierliche Muster in der semantischen Annotation wird empfohlen, die Wahrscheinlichkeitsverteilung als Musterbild zu verwenden und sie zur Aktualisierung der Annotation an den Musterdetektor zurückzugeben. * Bei verstreuten oder scharfen Bildmustern wird empfohlen, die Beschriftung direkt basierend auf dem Wahrscheinlichkeitswert zu aktualisieren, da dadurch detaillierte Informationen gespeichert werden können.
Im Allgemeinen,Pianno vereinfachte den Annotationsprozess und wählte einen heuristischen Ansatz, um anhand eines anfänglichen einzelnen Markergens zusätzliche Markergene zu identifizieren, wodurch die Eingabe der Anzahl bekannter Marker minimiert werden kann.
Forschungsergebnisse: hervorragende Leistung und hohe Anwendbarkeit
In dieser Studie überprüfte das Forschungsteam die Leistung, Genauigkeit und Anpassungsfähigkeit von Pianno und demonstrierte die Fähigkeiten von Pianno durch einen Vergleich mit bestehenden Methoden.
In einem Vergleich mit clusterbasierten Tools zur Annotation anatomischer Strukturen bewertete das Forschungsteam die Leistung von Pianno anhand von 12 Proben aus dem dlPFC-Datensatz und verglich es mit CellAssign, einer anderen Annotationsmethode, die auf Markern, aber ohne räumliche Informationen basiert. Darüber hinaus wurden im Bewertungsprozess auch die unüberwachte Clustering-Methode Leiden-Algorithmus und fünf räumliche Clustering-Methoden (SpaGCN, SEDR, BayesSpace, DeepST und STAGATE) berücksichtigt.
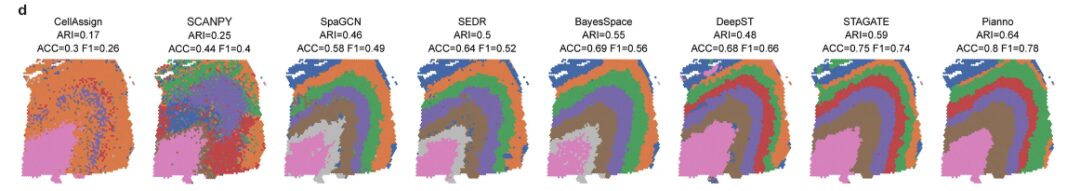
Die Bewertung ergab, dassPiannos Leistung erreichte die höchste Übereinstimmung mit manuellen Anmerkungen erfahrener Forscher auf der Grundlage morphologischer Merkmale und Markierungen.Elf der zwölf Proben schnitten besser ab als die anderen Testmethoden.
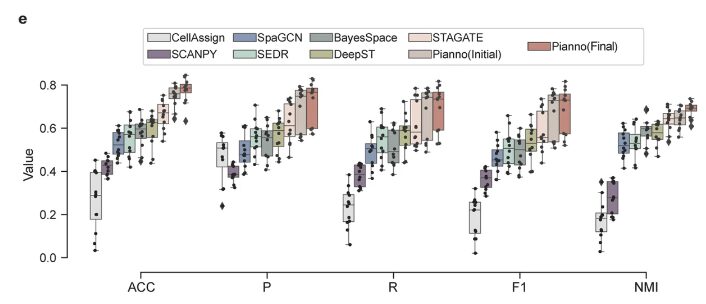
Darüber hinaus bewertete das Forschungsteam die überlegene Leistung von Pianno umfassend anhand anderer Klassifizierungsindikatoren wie Genauigkeit (ACC), Makro-Durchschnittspräzision (P), Makro-Durchschnitts-Recall (R), Makro-Durchschnitts-F1-Score (F1) und normalisierte gegenseitige Information (NMI), wie in Abbildung e oben dargestellt.Die zugehörigen Indikatoren von Piano liegen alle auf einem hohen Niveau.
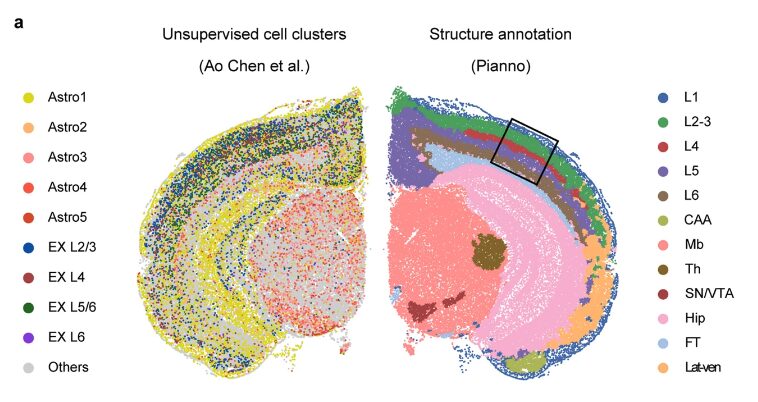
Anschließend bewertete das Forschungsteam Pianos Fähigkeit, die räumliche Verteilung von Zelltypen vorherzusagen. In dieser Validierungsrunde verwendete das Forschungsteam einen Stereosequenz-Datensatz von Coronalschnitten des Hemihirns erwachsener Mäuse und verglich die Ergebnisse mit der Zelltypverteilung, die durch verschiedene Strategien abgeleitet wurde, darunter unüberwachtes Clustering nach Zellsegmentierung und drei räumliche Dekonvolutionstools, die auf der Integration räumlicher und Einzelzell-Transkriptomik basieren.
Die Studie ergab, dass Piannos Vorhersagen für die Verteilung der Subtypen erregender Neuronen Muster zeigten, die mit denen von Tangram und RCTD vergleichbar waren, und in hohem Maße mit ihren bekannten Positionen in jeder Schicht übereinstimmten. Im Allgemeinen,Diese Ergebnisse zeigen die Robustheit und Genauigkeit von Pianno bei der Vorhersage komplexer Zelltypverteilungen in räumlichen Datensätzen, insbesondere in Situationen, in denen unbeaufsichtigte Methoden vor Herausforderungen stehen.
Anschließend bewertete das Forschungsteam die Leistung von Pianno bei der Annotation verschiedener Formstrukturen in räumlichen Transkriptomdaten auf unterschiedlichen Plattformen und verglich sie mit STAGATE.
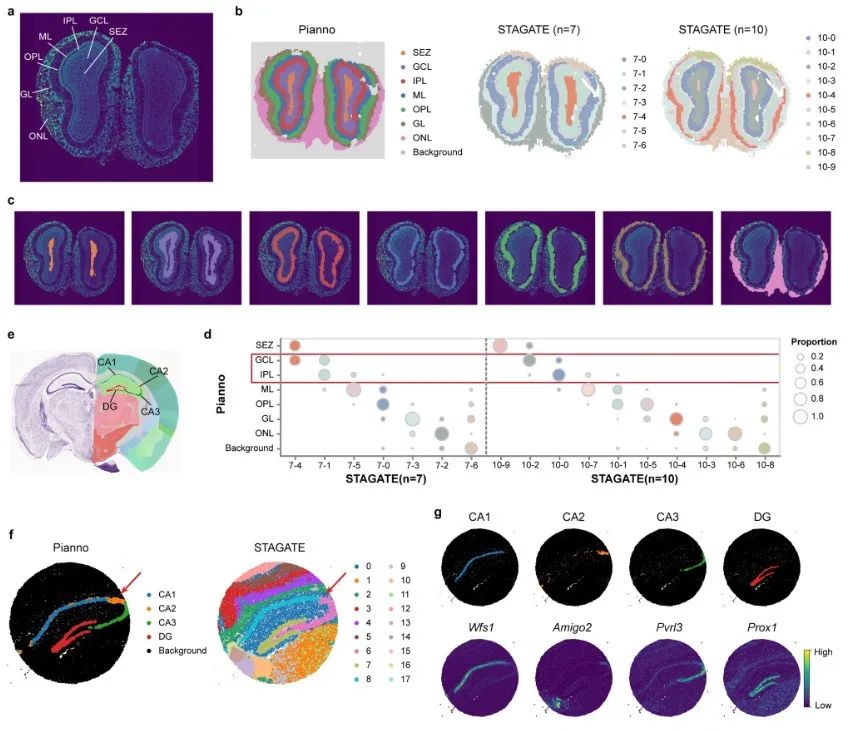
Das Forschungsteam verwendete Pianno, um die anatomischen Strukturen im Stereo-seq-Datensatz des Riechkolbens der Maus zu annotieren, der 10.747 räumliche Punkte enthält, die sowohl gewebebedeckte als auch Hintergrundbereiche abdecken.
Pianno konnte innerhalb weniger Minuten gleichzeitig Hintergrundsubtraktion und Strukturannotation durchführen. Wenn dagegen die Anzahl der Cluster auf die Anzahl der Strukturen eingestellt ist, kann STAGATE keine Cluster identifizieren, die allen anatomischen Strukturen entsprechen.
Das Forschungsteam bewertete außerdem Piannos Leistung bei der Annotation komplexer und verstreuter Strukturorganisationen im Hinblick auf die hohe Heterogenität der Tumormikroumgebung. In dieser Testrunde wurde die Mikroumgebung von zwei Proben menschlichen Pankreasgangadenokarzinoms und zwei Brustkrebsproben analysiert.
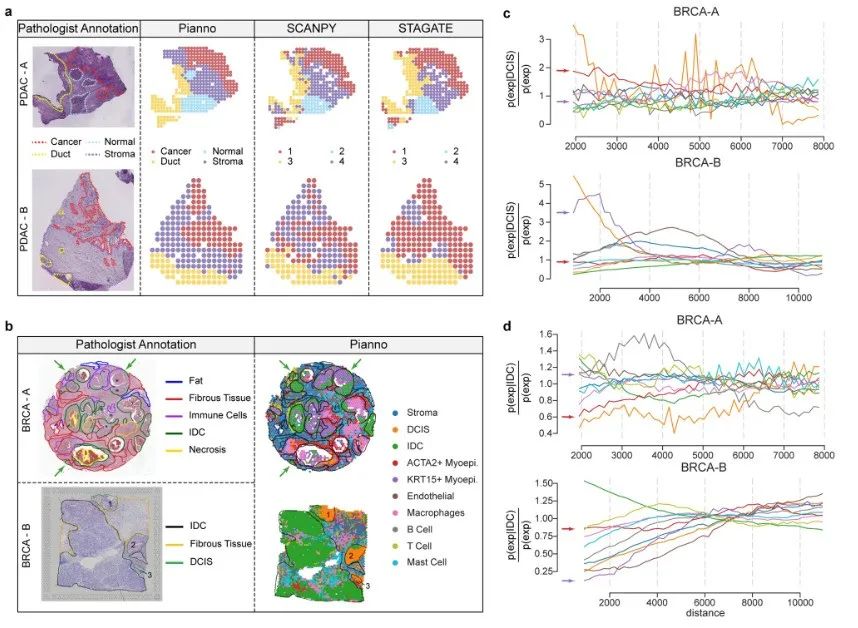
Gesamt,Pianno zeigte ein gewisses Maß an Übereinstimmung mit der manuellen Annotation durch professionelle Pathologen und demonstrierte sein großes Potenzial bei der Annotation unregelmäßiger und komplexer Strukturen, insbesondere in heterogenen Tumormikroumgebungen.Dies stellt für Pathologen eine wertvolle Hilfe beim Verständnis der Komplexität der Tumorbiologie dar und dürfte neue Ideen für die Bereitstellung personalisierter Behandlungsstrategien liefern.
Die Kombination künstlicher Intelligenz mit komplexer Biologie birgt großes Potenzial
Laut dem Institut für Gehirnforschung der Universität Fudan wurde das Forschungsprojekt vom Schlüsselprojekt des Nationalen Schlüssel-F&E-Programms „Biologische und Informationsintegration (BT- und IT-Integration)“, dem Großprojekt „Gehirnforschung und gehirnähnliche Forschung“ der Wissenschafts- und Technologieinnovation 2030, der Nationalen Naturwissenschaftsstiftung, dem Shanghaier Wissenschafts- und Technologie-Großprojekt und dem Zhangjiang-Labor finanziert.
Das Institut für Gehirnforschung der Universität Fudan wurde im April 2006 gegründet. Es handelt sich um eine universitätsweite neurowissenschaftliche Forschungseinrichtung der Universität Fudan und eine der wichtigsten wissenschaftlichen und technologischen Innovationsplattformen, die in der zweiten Phase des „Projekts 985“ des Bildungsministeriums aufgebaut wurden. Es handelt sich um ein „Zwei-in-Eins“-Bauprojekt mit dem National Key Laboratory of Medical Neurobiology.
Seit seiner Gründung hat das Institut für Gehirnforschung der Universität Fudan fruchtbare Ergebnisse erzielt. Das Institut hat wiederholt auf große internationale und nationale Bedürfnisse reagiert, große wissenschaftliche Forschungsprojekte durchgeführt und wichtige Forschungsergebnisse hervorgebracht. Der offiziellen Website des Instituts zufolge haben Forscher des Instituts eine Reihe bedeutender wissenschaftlicher Forschungsprojekte geleitet und daran teilgenommen, darunter das „973-Programm“ und das „863-Programm“ des Ministeriums für Wissenschaft und Technologie, „Gehirnforschung und gehirnähnliche Forschung“ im Rahmen von Science and Technology Innovation 2030, das National Key R&D Program und das National Major Science and Technology Special Project „Major New Drug Creation“ usw.
Tatsächlich haben neben dem Institut für Gehirnforschung der Universität Fudan auch viele Labore und Unternehmen begonnen, der räumlichen Transkriptomtechnologie Aufmerksamkeit zu schenken.
Zum Beispiel,Das Team von Zhang Shihua vom Institut für Mathematik und Systemwissenschaften der Chinesischen Akademie der Wissenschaften hat die STA-Toolreihe entwickelt. Im Jahr 2022 veröffentlichte das Team STAGATE, ein künstliches Intelligenztool zur Identifizierung räumlicher Substrukturen biologischer Gewebe, das an verschiedene räumliche Transkriptomtechnologien und verschiedene biologische Gewebe anpassbar ist. Seit Beginn des Jahres 2023 hat das Team eine Reihe von Ergebnissen rund um die räumliche Transkriptomtechnologie veröffentlicht: * Entwicklung eines neuen integrierten Analysetools namens STAligner für mehrschichtige räumliche Transkriptomdaten biologischer Gewebe aus verschiedenen Technologien, zu verschiedenen Entwicklungszeitpunkten und bei verschiedenen Krankheitszuständen. * STAMarker, eine räumlich domänenspezifische Methode zur Identifizierung variabler Gene, die auf Deep-Learning-Saliency-Maps basiert, realisiert gleichzeitig die räumliche Domänenidentifizierung und die entsprechende räumliche Identifizierung variabler Gene und soll eine effektive Methode zur detaillierten Analyse räumlicher Transkriptomdaten bieten. * In Zusammenarbeit mit dem Team von Yang Yungui und Cai Jun vom Beijing Genomics Institute der Chinesischen Akademie der Wissenschaften (Nationales Zentrum für Bioinformatik) haben wir die dreidimensionale räumliche Transkriptomkarte STAPR des Mittelmeerwurms während der Regeneration kartiert und systematisch mehrere wichtige regulatorische Faktoren für die Regeneration identifiziert.

Die Forschungsgruppe von Professor Zhang Xiaofei an der Fakultät für Mathematik und Statistik der Central China Normal University hat eine Rechenmethode namens ENGEP entwickelt.Mithilfe der k-Nearest-Neighbor-gewichteten Regression und Ensemble-Lernstrategien konnten wir die Expression nicht gemessener Gene im räumlichen Transkriptom genau vorhersagen. Darüber hinaus kann ENGEP die Expressionsmuster räumlich nicht gemessener Gene genau vorhersagen, was für die Verbesserung räumlicher Transkriptomikdaten von großer Bedeutung ist.
Es besteht kein Zweifel, dass die Möglichkeiten der KI in der räumlichen Transkriptomik und sogar in der Biologie nicht nur die Forschungseffizienz verbessert, sondern auch neue Lösungen für wissenschaftliche Forschungsschwierigkeiten bereitgestellt haben. Wie im Diskussionsteil des Artikels dargelegt, liegt der Wert von Pianno darin, dass es die bestehende arbeitsintensive manuelle Annotation ersetzen und auf automatisierte Weise effiziente, genaue und kostengünstige Formulare bereitstellen kann, um Änderungen in der räumlichen Transkriptomik herbeizuführen und neue Entwicklungen in der Biologie zu fördern.
Quellen:
1. https://news.fudan.edu.cn/2024/0407/c2474a139894/page.htm
2. https://bfse.cas.cn/sxyqyjc/kyjz/202311/t20231110_4985132.html
3. https://kjc.ccnu.edu.cn/info/1009/3744.htm








