Command Palette
Search for a command to run...
Universität Von Florida Nutzt Neuronale Netzwerke Zur Entschlüsselung Der Selektivität Der GPCR-G-Proteinkopplung
Inhalte im Überblick:G-Protein-gekoppelte Rezeptoren (GPCRs) sind Transmembranproteine, die Reize außerhalb der Zellmembran an die Zellmembran weiterleiten und in großem Umfang an physiologischen Aktivitäten des Menschen beteiligt sind. Kürzlich haben Forscher an der University of Florida die Bindungsselektivität von GPCRs und G-Proteinen bestimmt, einen Algorithmus zur Vorhersage der Selektivität beider entwickelt und die strukturellen Grundlagen dieser Selektivität untersucht.
Schlüsselwörter:GPCR neuronale Netzwerk-Medikamentenentwicklung
Autor | Xuecai
Herausgeber | Sanyang
G-Protein-gekoppelte Rezeptoren (GPCRs) sind Transmembranproteine, die Reize von außerhalb der Zellmembran ins Innere der Zellmembran übertragen. Durch die Aktivierung von G-Proteinen in der Zellmembran und ihren nachgeschalteten Signalwegen können GPCRs in großem Umfang an wichtigen physiologischen Aktivitäten wie Entwicklung, Immunität, Hormonregulierung und neuronaler Aktivität beteiligt sein.
G-Proteine bestehen aus Gα-, Gβ- und Gγ-Untereinheiten und ihre Vielfalt bestimmt die Vielfalt der GPCR-Signalreaktionen. Das menschliche Genom kodiert 16 Gα-Untereinheiten, die in vier Unterfamilien unterteilt sind:E/A , GαQ , GαS und Gα12/13 . G-Proteine bestimmen nachgeschaltete Signalwege, die wiederum zelluläre Reaktionen bestimmen. Daher ist die selektive Bindung von GPCRs und G-Proteinen der Schlüssel zum Verständnis des Signalsystems von Organismen.
Früher glaubte man in der Wissenschaft, dass GPCRs nur an ein einziges G-Protein koppeln würden, und unterteilte GPCRs daher basierend auf ihren Funktionen in vier Typen. Forscher haben jedoch kürzlich entdeckt, dass die meisten GPCRs an mehrere G-Proteine koppeln, um komplexe zelluläre Reaktionen zu aktivieren. Das Eins-zu-eins-Klassifizierungsmodell reicht nicht mehr aus, um die Kopplungsbeziehung zwischen GPCRs und G-Proteinen zu beschreiben, aber der Selektivitätsmechanismus von GPCRs-G-Proteinen ist noch nicht klar.
zu diesem Zweck,Forscher an der University of Florida verwendeten kinetische Messungen und die Biolumineszenz-Resonanzenergietransfer-Technologie (BRET), um die Guaninnukleotid-Austauschfaktoren von GPCRs für G-Proteine zu messen und die selektive Bindung der beiden zu analysieren. Darauf aufbauend klassifizierten die Forscher GPCRs nach ihrer Präferenz für G-Proteine und erstellten ein grobkörniges Modell von 124 GPCRs aus verschiedenen Säugetieren. Anschließend wurde ein Algorithmus zur Vorhersage der GPCR-G-Proteinselektivität entwickelt und die strukturellen Grundlagen der Selektivität untersucht.Ähnliche Ergebnisse wurden veröffentlicht inZellberichte".

Dieses Ergebnis wurde in „Cell Reports“ veröffentlicht.
Link zum Artikel:
https://doi.org/10.1016/j.celrep.2023.113173
01 BRET: GPCRs-G-Protein selektive Quantifizierung
Um die GPCRs-G-Proteinselektivität zu quantifizieren, verwendeten die Forscher die BRET-Technik, um die G-Protein-Aktivität in lebenden Zellen zu messen.

BRET-Technologie zur Echtzeiterkennung der G-Protein-Aktivität
Diesen Befund überprüften die Forscher anschließend am Cholecystokinin-Typ-II-Rezeptor (CCKBR). Die Ergebnisse der Reaktionsamplitude zeigten, dass CCKBR Gα aktivieren kannE/A , GαQ , Gα15 und Gα12/13 Die G-Proteine der Familie haben ähnliche Aktivierungsniveaus, können aber Gα nicht aktivierenS Proteinfamilie.
Die auf der Aktivierungsrate basierenden Ergebnisse zeigen deutlich, dass CCKBR einen wichtigen Einfluss auf Gα hatQ Der Aktivierungseffekt der Familie ist am besten, gefolgt von GαE/A , Gα15 und Gα12/13, was zeigt, dass die auf der Aktivierungsrate basierende BRET-Technologie subtile Unterschiede zwischen den Aktivitäten verschiedener G-Proteine erfassen kann.

Amplitudenbasierte BRET-Ergebnisse (C) und aktivierungsratenbasierte BRET-Ergebnisse (D)
Entsprechend,Die Forscher maßen die Selektivität von 124 GPCRs und G-Proteinen als Datensatz für diese Studie.

Selektivitätsmessungen von GPCRs und G-Proteinen der Klasse B
02 Modellkonstruktion: Neuronales Netzwerk zur binären Klassifizierung
Die obigen Ergebnisse zeigen, dass die auf der Aktivierungsrate basierende BRET-Technik die Selektivität zwischen Hunderten von GPCRs und G-Proteinen unterscheiden kann. Basierend auf diesem Ergebnis,Die Forscher entwickelten einen auf maschinellem Lernen basierenden Algorithmus zur Vorhersage der Proteinselektivität von GPCRs der Klasse A-G.
Der Algorithmus hat zwei Aufgaben:
1. Bestimmen Sie hinsichtlich der Kopplung von GPCRs, ob ein bestimmter GPCR mit G-Protein koppeln kann, d. h. die Amplitude > 0%;
2. In Bezug auf die Selektivität von GPCRs wird festgestellt, dass ein bestimmtes GPCR-G-Proteinpaar schnell aktiviert werden kann, d. h. die Aktivierungsrate beträgt> 30%.
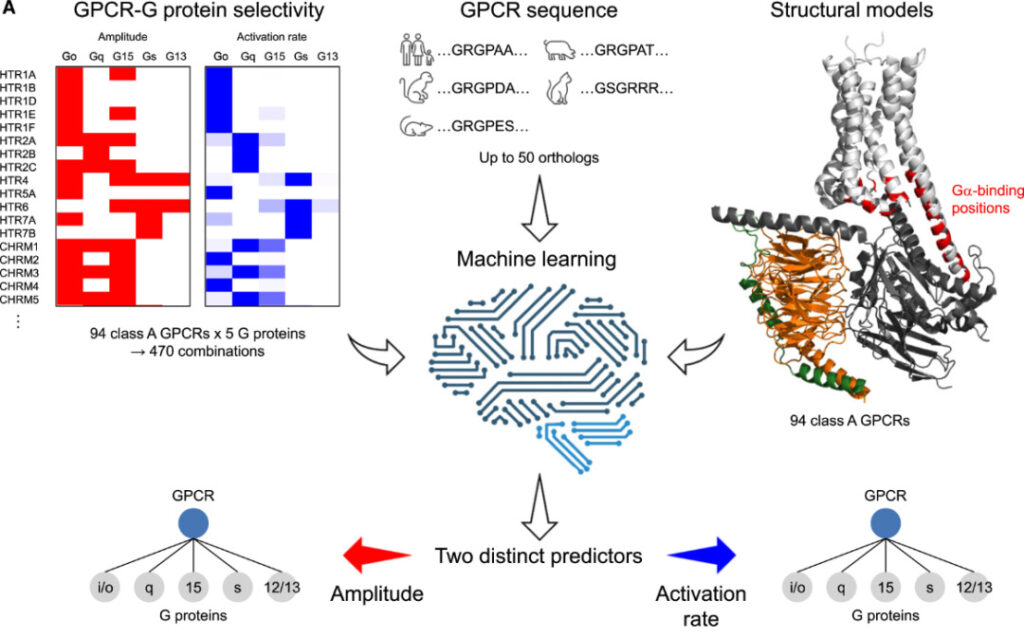
Konzeptdiagramm von Algorithmen für maschinelles Lernen
Die Kopplung zwischen jedem GPCR und einem G-Protein einer anderen Familie stellt ein Klassifizierungsproblem dar, sodass jede Aufgabe als 5 binäre Klassifizierungen konzipiert werden kann. Auf dieser Grundlage entwickelten die Forscher zehn neuronale Netzwerkklassifizierer zur Bewältigung dieser Aufgaben. Das neuronale Netzwerk besteht aus zwei vollständig verbundenen Schichten (mit 128 bzw. 16 Neuronen), einer Abflachungsschicht, drei vollständig verbundenen Schichten (mit 128, 32 bzw. 4 Neuronen) und einer Ausgabeschicht (1 Neuron). Die innere Schicht wird durch gleichgerichtete lineare Einheiten (ReLU) aktiviert und schließlich wird eine Batch-Normalisierung durchgeführt. Die Ausgabeschicht wird durch die Sigmoidfunktion aktiviert.
Aufgrund der begrenzten Datenmenge wurden jedem GPCR 50 homologe Sequenzen zur Datenerweiterung hinzugefügt, wobei davon ausgegangen wurde, dass die Sequenzen, die die G-Protein-Selektivität bestimmen, während der Evolution relativ konserviert bleiben. Im Modell wird ein Sequenzeinbettungsprotokoll eingesetzt, um die Eigenschaften von Proteinresten in einer bestimmten Umgebung mithilfe eines unüberwachten Deep-Learning-Modells zu beschreiben.
Die Eingabe für das neuronale Netzwerk ist ein Tensor der Größe B*30*1024. Die erste Dimension ist die Batchgröße (B=32), die zweite Dimension ist die Anzahl der Rückstände (30) und die dritte Dimension ist für jedenAminosäurerestDie Größe der vortrainierten Sequenzeinbettungen (1024).
Mittelung der Modellvorhersagen für Amplitude und Aktivierungsrate AUROC Beide liegen bei 0,85, was darauf hinweist, dass das Modell bei der Vorhersage beider Indikatoren gute Ergebnisse erzielt.Darunter für GS Familienproteine wurden mit AUROC-Werten von 0,89 bzw. 0,95 am besten vorhergesagt. Für Gα15 und Gα12/13 Proteinfamilie zeigte das Modell keine offensichtliche Lernfähigkeit.

ROC-Kurven zur Vorhersage von Amplitude (C) und Aktivierungsrate (D)
03 Entschlüsselung des Mechanismus der GPCRs-Gα-Proteinselektivität
BRET-Experimente und maschinelles Lernen bieten eine Lösung zur Entschlüsselung der strukturellen Grundlagen der GPCR-G-Proteinselektivität. Darauf aufbauend untersuchten die Forscher die verfügbaren GPCRs-G-Proteinkomplexe und analysierten 33 Rezeptoren der Klasse A, um die Struktur zu finden, die die Selektivität des GPCRs-Gα-Proteins der Klasse A bestimmt.
Die Forscher untersuchten das Restnetzwerk von GPCRs-Gα-Proteinen und stellten fest, dass alle zum Zytoplasma gerichteten Strukturen von GPCRs in unterschiedlichem Ausmaß an der Bindung an Gα-Proteine beteiligt waren. Ebenso gibt es 13 Strukturen im Gα-Protein, die mit der Bindung von GPCRs-Gα-Protein in Zusammenhang stehen, von denen die C-terminale α-Helix (H5) am stärksten beteiligt ist.

Wechselwirkungen zwischen verschiedenen Strukturelementen von GPCRs und Gα-Proteinen
Für die universelle GPCR-Gα-Kopplung verwenden GPCRs ICL2, H8 und die meisten TM-Reste zur Bindung an Gα-Proteine. Darunter war die überwiegende Mehrheit der Strukturen primär mit H5 verbunden, während ICL2 stärker verbunden war.
GPCRs und GαE/A und GαQ Die Proteinkopplungsmuster der Familien sind ähnlich, der einzige Unterschied besteht darin, dass die Bindung von GPCRs an erstere stark von TM6 abhängt, während dies bei der Bindung an letztere nicht der Fall ist. GPCRs und GαS Bei den Verbindungen wurde der Anteil von ICL2 und ICL3 stark reduziert und man verlässt sich stärker auf TM3 und TM5. Die obigen Ergebnisse zeigen, dass die Verbindung verschiedener Gα-Proteinfamilien mit GPCRs von unterschiedlichen Strukturen abhängt.
Darüber hinaus wurden die Auswirkungen spezifischer Strukturen auf Gα-Proteine verschiedener Familien durch Kombination der selektiven Sequenzen von GPCRs-G-Proteinen untersucht. Erstens, Richter und GαE/A Können die gebundenen GPCRs an Gα binden?15 Kombinieren und vergleichen Sie die Unterschiede in den Residuennetzwerken der beiden. Im Vergleich zu Ersterem trennte die Ligation von GPCRs an Letzteres die Verbindung zwischen ICL3 und H4, schwächte die Interaktion von ICL2-H5 und stärkte die Verbindung zwischen TM4-HN und ICL2-s2s3. Dies deutet darauf hin, dass die Verbindung zwischen ICL2 und anderen Resten mit Gα in Zusammenhang stehen könnte.E/A oder Gα15 Große Unterschiede bei den verknüpften GPCRs.

GPCRs interagieren nur mit GαE/A Mit GPCRs und Gα verbundenes Restnetzwerk15/GαE/A Verbundenes Residuennetzwerk (K)
In ähnlicher Weise sind GPCRs und GαS und GαE/A Nach der Kombination der Restnetzwerke zeigten die Ergebnisse, dass die Verbindung zwischen ICL1 und TM5 der Hauptunterschied zwischen den beiden war.
Die obigen Ergebnisse zeigen, dassBRET und maschinelles Lernen können das Proteinrestnetzwerk der GPCR-G-Protein-Bindung analysieren und die strukturelle Grundlage ihrer Selektivität finden, wodurch eine neue Methode für die Untersuchung von GPCRs bereitgestellt wird.
04 AI-GPCR: Unerforschte Regionen von 96.4%
Im letzten Jahrzehnt hat der Anteil von KI- und maschinellen Lernanwendungen im GPCR-Bereich stetig zugenommen. Im Jahr 2022 wurden in 3,61 TP3T der GPCR-bezogenen Artikel KI-bezogene Methoden erwähnt.
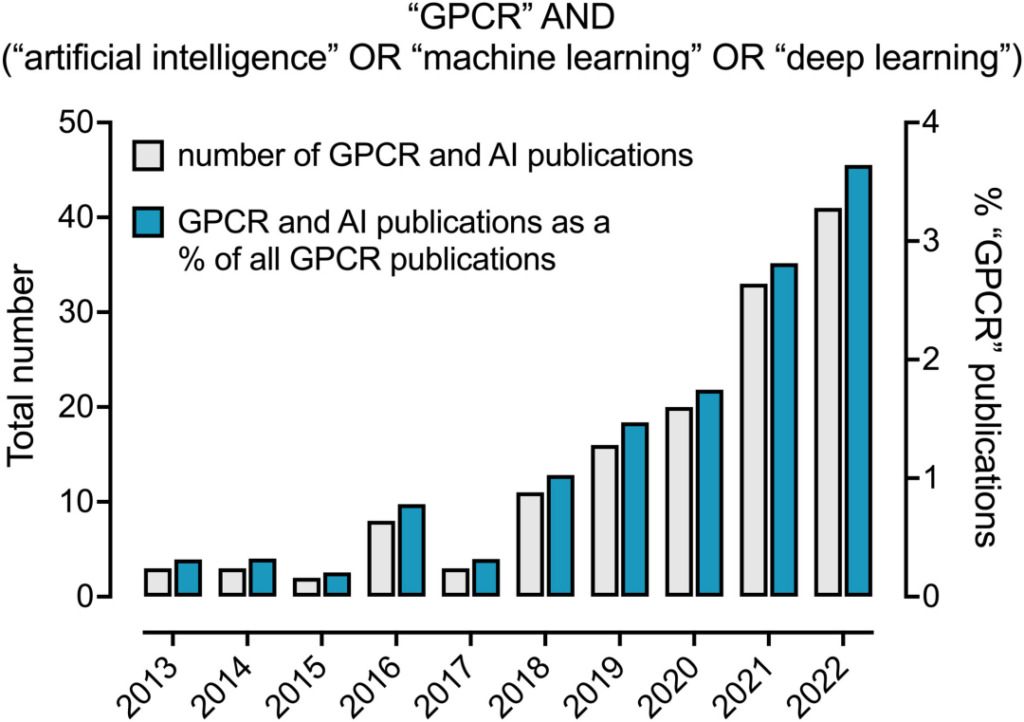
Der Anteil der in GPCR-bezogenen Artikeln erwähnten KI
Angesichts der zunehmenden Anwendung von KI in der GPCR-Medikamentenforschung werden auch entsprechende Algorithmen entwickelt. Bei Klassifizierungsproblemen werden am häufigsten Algorithmen des traditionellen maschinellen Lernens verwendet, beispielsweise solche aus der Scikit-Learn-Bibliothek, darunter Support Vector Machines (SVMs), Entscheidungsbäume, Gradient Boosting Machines und K-Nearest-Neighbor-Algorithmen.
Zur Lösung numerischer Ergebnisse, wie etwa der Affinität der Protein-Liganden-Bindung, werden häufig Regressionsalgorithmen wie multivariate lineare Regression, Support Vector Machines und Deep Learning-Netzwerke verwendet.
Aktuelle Ergebnisse nutzen zur Vorhersage hauptsächlich Deep-Learning-Algorithmen wie mehrschichtige Perzeptronen und Convolutional Neural Networks (CNN). Mit der Entwicklung generativer Deep-Learning-Algorithmen ist das Design von Proteinliganden und -strukturen effizienter und präziser geworden. Algorithmen wie Generative Adversarial Network, Recurrent Neural Network und Reinforcement Learning können die automatische Konstruktion und adaptive Messung des Vektorraums nutzen, um größere Generationsräume zu erkunden.

Die Rolle der KI in jeder Phase der GPCR-Medikamentenentwicklung
Daher können diese Algorithmen mehr Liganden mit gewünschten Funktionen erzeugen oder beispielsweise die Strukturen unbekannter Proteine genauer vorhersagen. AlphaFold2 . Obwohl Modelle wie AlphaFold2 nicht speziell für die Vorhersage der Struktur von GPCRs entwickelt wurden, können sie die Struktur von GPCRs dennoch effizient und genau vorhersagen. Darüber hinaus hält auch das unbeaufsichtigte oder selbstüberwachte Deep Learning Einzug in die Arzneimittelforschung.
Es ist ersichtlich, dass AI-GPCR eine neue Richtung für die zukünftige Arzneimittelentwicklung darstellen könnte, es lässt uns aber auch mit einem unbekannten Bereich von 96,4% zurück. Mithilfe effizienter Klassifizierung und präziser Vorhersagealgorithmen können die Kopplungsmechanismen von GPCRs besser verstanden und der Entwicklung der Biomedizin neue Impulse verliehen werden.
