Command Palette
Search for a command to run...
OBIA: Über 900 Patienten, Über 193 Bilder, Das Institut Für Genomik Der Chinesischen Akademie Der Wissenschaften Hat Die Erste Datenbank Zum Teilen Biologischer Bilder in Meinem Land Veröffentlicht

Es ist üblich, einen Arzt aufzusuchen und Röntgenaufnahmen zu machen. CT-, MRT-, Röntgen- und andere Bilddaten können auf nicht-invasive Weise durch den menschlichen Körper übertragen werden, wodurch der Zustand innerer Organe und Gewebe deutlich sichtbar wird und eine zuverlässige Grundlage für die klinische Diagnose und Behandlung von Krankheiten geschaffen wird.
Mit der weitverbreiteten Entwicklung der medizinischen Bildgebungstechnologie machen Bilddaten mittlerweile mehr als 801.000 t der inländischen medizinischen Daten aus.Schwachstellen wie der Mangel an Radiologen, Unterschiede bei den Diagnoseergebnissen zwischen Krankenhäusern auf allen Ebenen und die ungleiche Verteilung der medizinischen Ressourcen treten immer deutlicher hervor.
Bei der Kombination von KI und medizinischer Bildgebung gibt es viel Raum für Fantasie. Sensorische Wahrnehmung und Deep-Learning-Technologien verfügen bei der Ermittlung diagnostischer Ergebnisse medizinischer Bildgebungsverfahren über beispiellose Vorteile gegenüber dem Menschen. Sie können Ärzten dabei helfen, die Zahl der Fehldiagnosen zu senken und die Arbeitseffizienz zu verbessern.
Jedoch,Hochwertige KI-Algorithmen erfordern ausreichend große und repräsentative Bilddatensätze.Diese medizinischen Bilder enthalten häufig eine große Menge sensibler Datenschutzinformationen. Darüber hinaus gibt es zwischen Krankenhäusern auf allen Ebenen „Dateninseln“, und das unvollständige gemeinsame Nutzungssystem begrenzt die verfügbaren Ressourcen für KI in der medizinischen Bildgebung.
Autor | Turm
Herausgeber | Sanyang, Xuecai
Viele Länder auf der ganzen Welt haben verschiedene Datenbanken zum Austausch medizinischer Bilddaten aufgebaut. mein Land hinkt in diesem Bereich immer noch hinter der internationalen Gemeinschaft zurück. Um den Austausch hochwertiger medizinisch-biologischer Bilddaten zu fördern,Das Institut für Genomik der Chinesischen Akademie der Wissenschaften (Nationales Zentrum für Bioinformation, China) hat das Open Biomedical Imaging Archive (OBIA) eingerichtet.
Als erstes offenes Archiv für biomedizinische Bilddaten und damit verbundene klinische Daten in ChinaOBIA steht Medizinern und Wissenschaftlern weltweit kostenlos zur Verfügung. Die Vorabdruckversion der entsprechenden Ergebnisse wurde am 25. September 2023 auf „bioRxiv“ veröffentlicht.

Link zum Artikel:https://www.nature.com/articles/s42256-023-00704-7
Folgen Sie dem öffentlichen Konto „HyperAI Super Neural“ und antworten Sie mit „OBIA“, um das vollständige PDF des Dokuments zu erhalten.
Aufbau- und Implementierungsprozess der OBIA-Datenbank
Als zentrale Datenbankressource des China National Center for Bioinformation akzeptiert OBIA Bildeinsendungen aus der ganzen Welt und bietet kostenlosen offenen Zugriff auf alle öffentlichen Daten.Es unterstützt die De-Identifizierung, Verwaltung und Qualitätskontrolle von Bilddaten.Durch die Bereitstellung von Datendiensten wie Durchsuchen, Abrufen und Herunterladen kann die Wiederverwendung vorhandener Bilddaten und klinischer Daten gefördert werden.
OBIA verwendet fünf Arten von Datenobjekten (Sammlung, Einzelperson, Studie, Serie, Bild), um Daten zu organisieren.Akzeptiert die Einreichung von biomedizinischen Bildern verschiedener Modalitäten, mehrerer Organe und mehrerer Krankheiten.
Um Ihre Privatsphäre zu schützen,OBIA hat einen einheitlichen Prozess zur Deidentifizierung und Qualitätskontrolle entwickelt.Es bietet außerdem eine intuitive und benutzerfreundliche Weboberfläche zum Senden, Durchsuchen und Abrufen von Daten sowie zum Abrufen von Bildern. Insgesamt bietet OBIA eine zuverlässige Plattform für das nationale Management biomedizinischer Bilddaten und trägt so zur Unterstützung der globalen biomedizinischen Forschung bei.
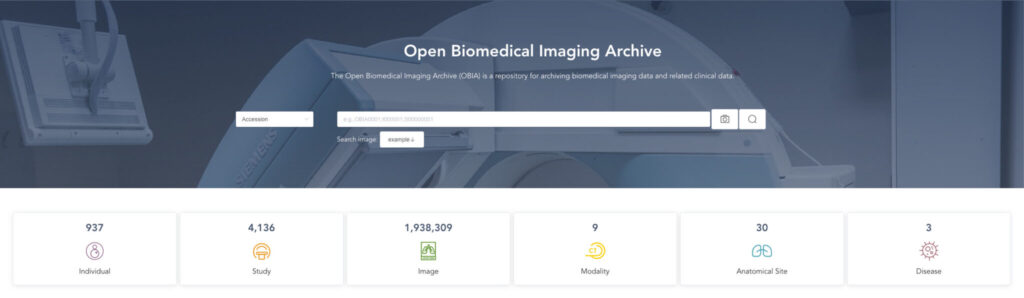
Abbildung 1: OBIA-Zugriffsschnittstelle
Besuchen Sie die URL:https://ngdc.cncb.ac.cn/obia
Implementierungsdetails——Bildabruf
Tiefe neuronale Netzwerke eignen sich gut zum Extrahieren vorteilhafter Merkmale.Damit können multimodale medizinische Bilder verschiedener Organe des menschlichen Körpers abgerufen und die Ranking-Leistung in kleinen Stichprobenfällen verbessert werden. Im Vergleich zu herkömmlichen Methoden können auf Deep Learning basierende Methoden wie Scale-Invariant Feature Transform (SIFT), Local Binary Patterns (LBP) und Histogram of Oriented Gradients (HOG) eine bessere Leistung aufweisen.
In OBIA verwendeten die Forscher EfficientNet als Merkmalsextraktor basierend auf multimodalen Krebsdaten aus der Krebsbilddatenbank TCIA, trainierten das Modell mithilfe eines Triplet-Netzwerks und eines Aufmerksamkeitsmoduls und komprimierten das Bild in diskrete Hashwerte (Abbildung 2). Um anschließend die Inferenzleistung zu beschleunigen und die Inferenzlatenz zu reduzieren, wird das trainierte Modell in das TensorRT-Format konvertiert und Faiss zum Speichern von Hashcodes verwendet.
Die Forscher berechneten die Bildähnlichkeit mithilfe der Hamming-Distanz und gaben das ähnlichste Bild zurück.Die Ergebnisse zeigen, dass der mittlere durchschnittliche Präzisionswert (MAP) des vorgeschlagenen Modells die Leistung bestehender erweiterter Bildabrufmodelle im TCIA-Datensatz übertrifft.
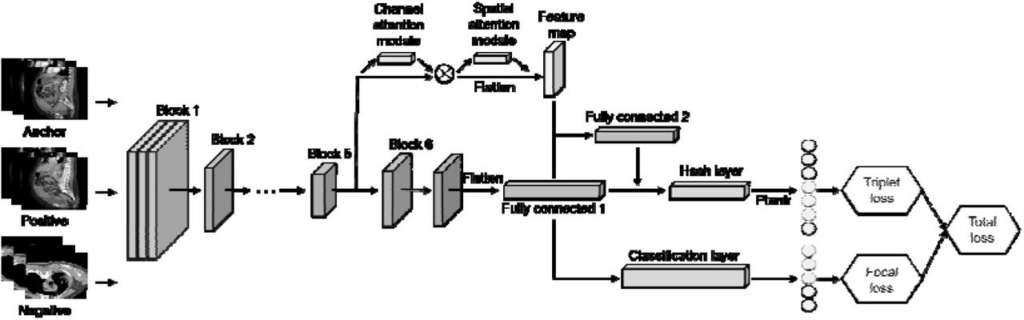
Abbildung 2: Deep Triplet Hashing basierend auf Attention- und Layer-Fusion-Modulen
Dieses Modell verwendet EfficientNet-B6 als Hauptnetzwerk und nutzt das CBAM-Aufmerksamkeitsmodul in Block5, um Feature-Maps zu erhalten. In den vollständig verbundenen Schichten wird die Schichtfusion angewendet, um Hashcodes und Klasseneinbettungen unter Verwendung von Fokalverlust und Triplettverlust zu generieren.
Notiz:
● CBAM: Faltungsblock-Aufmerksamkeitsmodul
● EfficientNet: Ein neuer Typ von CNN-Netzwerk, der 2019 von Google vorgeschlagen wurde. Es verfügt über eine extrem hohe Parametereffizienz und Geschwindigkeit und bietet gute Leistungen im Bereich der Bildklassifizierung.
● Faiss: Eine leistungsstarke Ähnlichkeitssuchbibliothek, die von Facebook AI Research entwickelt wurde und häufig im Deep Learning verwendet wird.
Datenbankinhalt und -verwendung – Datenmodell
Wie in Abbildung 3 dargestellt,Bilddaten in OBIA werden in fünf Objekttypen unterteilt:Sammlung, Einzelperson, Studie, Serie, Bild, jeweils beziehen sich auf:
• Sammlungen:Mit dem Präfix „OBIA“ versehen, um eine Gesamtbeschreibung der vollständigen Einreichung bereitzustellen;
• Person:Registrierungsnummern sind mit einem „I“ gekennzeichnet und definieren die Merkmale des menschlichen oder nicht-menschlichen Organismus, der Gesundheitsdienstleistungen erhält oder für den Erhalt dieser registriert ist.
• Studie:Die Zugangsnummer hat das Präfix „S“ und enthält beschreibende Informationen zur radiologischen Untersuchung der Person.
• Serie:Die Studie kann nach unterschiedlichen Logiken (z. B. Körperteil oder Richtung) in eine oder mehrere Serien unterteilt werden.
• Bild:Beschreibt die Pixeldaten einer einzelnen DICOM-Datei (Digital Imaging and Communications in Medicine). Ein Bild ist mit einer einzelnen Serie in einer einzelnen Studie verknüpft.
Hinweis: DICOM ist ein internationaler Standard, der im Bereich der medizinischen Bildgebung weit verbreitet ist. Es definiert eine Reihe von Spezifikationen und Protokollen zum Speichern, Übertragen, Teilen und Drucken medizinischer Bilddaten, sodass medizinische Geräte und Software verschiedener Hersteller kompatibel sind und miteinander kommunizieren können.
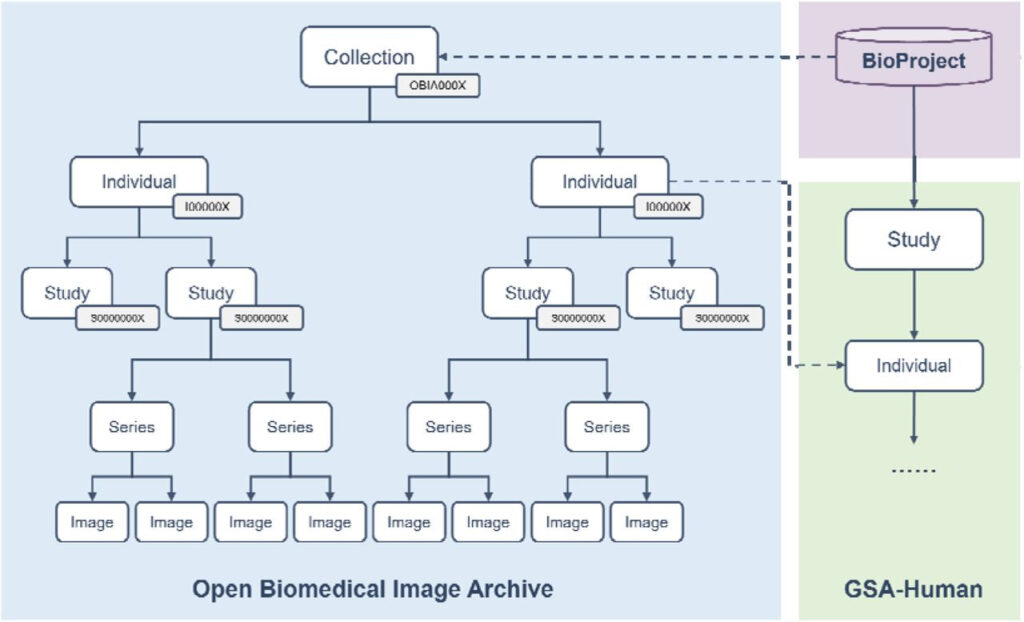
Abbildung 3: OBIA-Datenmodell
Basierend auf diesen standardisierten DatenobjektenOBIA verbindet die durch den DICOM-Standard definierte Bildstruktur mit tatsächlichen Forschungsprojekten.Datenfreigabe und -austausch werden realisiert.
Darüber hinaus ist jede Sammlung in OBIA mit BioProject verknüpft, um beschreibende Metadaten zum Forschungsprojekt bereitzustellen.
Sofern verfügbar, kann das Individuum von OBIA über die individuelle Zugangsnummer mit GSA-Human verknüpft werden, das Bilddaten mit Genomdaten verknüpft, damit Forscher Multi-Omics-Analysen durchführen können.
Bioprojekt-URL:
https://ngdc.cncb.ac.cn/bioproject/
GSA-Human-Linkadresse:
https://ngdc.cncb.ac.cn/gsa-human/
Datenbankinhalt und -nutzung——De-Identifizierung und Qualitätskontrolle
Biomedizinische Bilder können geschützte Gesundheitsinformationen (PHI) enthalten und müssen ordnungsgemäß verarbeitet werden, um das Risiko einer Verletzung der Privatsphäre zu minimieren. Um möglichst viele wertvolle wissenschaftliche Informationen zu erhalten und gleichzeitig geschützte Gesundheitsdaten zu löschen,OBIA bietet einen De-Identifizierungs- und Qualitätskontrollmechanismus, der dem DICOM-Standard entspricht (Abbildung 4).
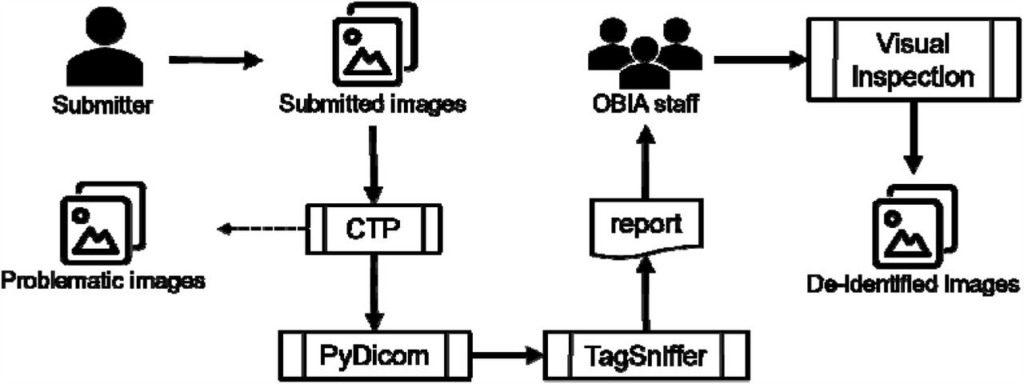
Abbildung 4: OBIA-Deidentifizierungs- und Qualitätskontrollmechanismen
OBIA verwendet den MIRC Clinical Trial Processor (CTP) der Radiological Society of North America (RSNA), um einen Großteil der Deidentifizierungsarbeit durchzuführen:
• Für Standard-Tags,Die Forscher erstellten ein CTP und entwickelten ein universelles Basis-Deidentifizierungsskript, um bestimmte Standardmarker, die PHI enthalten oder enthalten können, zu entfernen oder zu anonymisieren;
• Für private Tags,Verwenden Sie PyDicom zur Verarbeitung und bewahren Sie dabei seinen rein digitalen Charakter.
Nachdem der Deidentifizierungsprozess abgeschlossen ist, beginnt OBIA mit der Durchführung von Qualitätskontrollverfahren:
• Das fragliche Bild:Isolieren Sie Bilder, bei denen die Einsender relevante Informationen angeben können, um das Bild zu reparieren oder vollständig zu verwerfen (solche Bilder sind solche mit leeren Titeln oder fehlenden Patienten-IDs, beschädigt, mit anderen Patientenbildern vermischt usw.);
• Doppeltes Bild:Behalten Sie nur eines.
OBIA generiert dann mithilfe von TagSniffer einen Bericht für alle Bilder, in dem alle DICOM-Elemente sorgfältig überprüft werden, um sicherzustellen, dass sie keine PHI enthalten und dass bestimmte Werte (z. B. Patienten-ID, Studiendatum) wie erwartet geändert werden.
Auch,Die Mitarbeiter von OBIA führen auch visuelle Prüfungen der Bildpixel durch.um sicherzustellen, dass in den Pixelwerten keine PHI enthalten sind und das Bild sichtbar und unverfälscht ist.
Datenbankinhalt und -nutzung——Statistiken
Bis September 2023 hat OBIA 937 „Einzelpersonen“, 4.136 „Studien“, 24.701 „Serien“ und 1.938.309 „Bilder“ gesammelt, die 9 Modalitäten und 30 anatomische Teile abdecken.
Zu den repräsentativen Bildgebungsverfahren zählen die Computertomographie (CT), die Magnetresonanztomographie (MR) und die digitale Radiographie (DX). Zu den anatomischen Stellen zählen Bauch, Brustkorb, Thorax, Kopf, Leber, Becken usw.
Der erste an OBIA übermittelte Datensatz stammte vom Krankenhaus 301.Enthält Bilddaten für drei wichtige gynäkologische Tumore (Gebärmutterhalskrebs, Eierstockkrebs und Gebärmutterhalskrebs).
Wie in Tabelle 1 gezeigt, sind diese Daten in vier „Sammlungen“ unterteilt, wobei die Anzahl der „Einzelpersonen“, die Anzahl der „Studien“, die Anzahl der „Serien“ und die Anzahl der „Bilder“ aufgeführt sind. Auch,OBIA sammelt auch relevante klinische Metadaten.Wie zum Beispiel demografische Daten, Krankengeschichte, Familiengeschichte, Diagnose, Pathologietyp und Behandlungsmethode.

Tabelle 1: Erste an OBIA übermittelte Informationen
Datenbarrieren abbauen,Aufbau von Plattformen zum Austausch medizinischer Daten im In- und Ausland
Daten generieren nur dann einen Wert, wenn sie verbreitet werden. Um den Austausch biologischer Bilddaten zu verbessern,Viele Länder auf der ganzen Welt engagieren sich für den Aufbau offener medizinischer Datenbanken:
• Nationale Gesundheitsinstitute (NIH):Sponsor mehrerer Wissensdatenbanken, wie etwa MIDRC, eine Open-Access-Plattform für COVID-19-bezogene medizinische Bilder und Daten, IDA, NITRC-IR, FITBIR, OpenNeuro und NDA, die neuronale und Gehirnbilder sammeln, sowie TCIA und IDC, Krebsbilddatenbanken (TCIA stellt Bilder lokal bereit und IDC stellt Bilder in einer Cloud-Umgebung zum Austausch von Krebsforschungsdaten bereit);
• Krebsforschung UK:gesponserte OPTIMAM Mammography Image Database (OMI-DB);
• Universität Porto, Portugal:gesponsertes Breast Cancer Digital Repository (BCDR), das kommentierte Brustkrebsbilder und klinische Details bereitstellt;
In den oben genannten Repositorien, außer NITRC-IR und IDC,Die meisten anderen unterstützen die Anonymisierung und Qualitätskontrolle der Daten.Darüber hinaus stellen einige Universitäten oder Institutionen auch Open-Source-Datensätze zur Verfügung, wie etwa OASIS, EchoNet-Dynamic, CAMUS-Projekt usw.
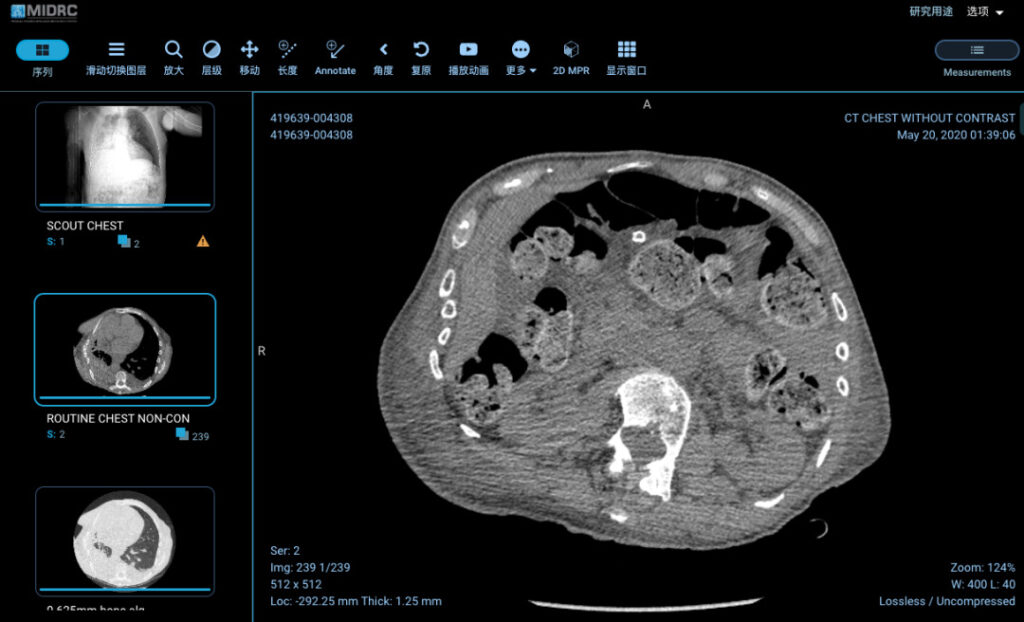
Abbildung 5: Thorax-CT eines 79-jährigen Patienten in der MIDRC-Datenbank
auf dem Land,Die Huazhong University of Science and Technology stellt offene Ressourcen mit integrierten CT-Bildern und CFs von COVID-19 bereit.Es umfasst CT-Bilder und klinische Merkmale von Patienten mit Lungenentzündung (einschließlich COVID-19), ist jedoch auf eine einzelne Krankheit beschränkt und die verfügbaren Forschungsressourcen sind begrenzt. Daher mangelt es in China noch immer an Datenbanken, die auf die Speicherung und Annahme von Einsendungen zu verschiedenen Krankheiten und Modalitätsdaten spezialisiert sind.
Das von der Chinesischen Akademie der Wissenschaften gegründete OBIA schließt die Lücke beim offenen Austausch inländischer biomedizinischer Bilddaten, das Forschern aus verschiedenen Institutionen den Austausch klinisch relevanter Bilddaten erleichtert und die Lücke im Bereich der biomedizinischen Bilddatenbanken in China wirksam schließen kann.
Die Forscher erklärten in dem Papier, dass sie die Infrastruktur von OBIA auch in Zukunft weiter verbessern und die Sicherheitsmaßnahmen verstärken würden. Sie werden außerdem mehr Arten biomedizinischer Bilddaten erfassen und die Datenquellen erweitern.Wir ergreifen zahlreiche Maßnahmen, um dem Ziel näher zu kommen, „so viele gültige Bildmetadaten wie möglich zu erhalten und der wissenschaftlichen Forschung qualitativ hochwertige Bilddaten bereitzustellen“.
-- über--








