Command Palette
Search for a command to run...
KI Bekämpft Superbakterien: McMaster University Nutzt Deep Learning Zur Entdeckung Eines Neuen Antibiotikums, Abaucin
Inhalte im Überblick:Acinetobacter baumannii ist ein häufiger, im Krankenhaus erworbener gramnegativer Erreger, der oft eine Multiresistenz gegen Medikamente aufweist. Mit herkömmlichen Methoden ist es schwierig, neue Antibiotika zu entdecken, die diese Bakterien hemmen. Doch mithilfe maschinellen Lernens lässt sich der chemische Raum rasch erforschen, was die Wahrscheinlichkeit erhöht, neue antimikrobielle Moleküle zu entdecken. Kürzlich wurde ein Forschungsergebnis in der internationalen Fachzeitschrift Nature Chemical Biology veröffentlicht. Forscher fanden heraus, dass Abaucin Acinetobacter baumannii wirksam hemmen kann.
Schlüsselwörter:Acinetobacter baumannii Abaucin Deep Learning
Dieser Artikel wurde zuerst auf der öffentlichen HyperAI WeChat-Plattform veröffentlicht~
Acinetobacter baumannii (wissenschaftlicher Name: Acinetobacter baumannii, allgemein bekannt als: AB-Bakterien) ist der häufigste gramnegative Bazillus der Gattung Acinetobacter. Es kommt in der Natur in großem Umfang in Wasser und Boden vor und ist auch in der normalen menschlichen Haut sowie in den Atemwegen, dem Verdauungstrakt und dem Urogenitaltrakt vorhanden. Dieses Bakterium kommt häufig bei Krankenhausinfektionen vor und ist auch ein Krankheitserreger bei Tieren in Aquakultur.Verursacht häufig Bakteriämie, Lungenentzündung, Meningitis, Peritonitis, Endokarditis sowie Harnwegs- und Hautinfektionen.
Acinetobacter baumannii ist sehr resistent gegen Feuchtigkeit, Hitze, UV-Strahlen und chemische Desinfektionsmittel und ist resistent gegen niedrige Temperaturen. Es kann länger als 25 Tage auf der Oberfläche trockener Gegenstände überleben. Herkömmliche Desinfektionsmittel können das Wachstum nur hemmen, es jedoch nicht abtöten. Außerdem ist es seifenbeständig. Es handelt sich um den am häufigsten isolierten gramnegativen Bazillus auf den Händen von medizinischem Personal, auf medizinischen Geräten und auf Objektoberflächen.Aufgrund des Missbrauchs von Antibiotika hat Acinetobacter baumannii eine multiple Arzneimittelresistenz entwickelt und wird von der Weltgesundheitsorganisation als eines der gefährlichsten antibiotikaresistenten Bakterien der Welt eingestuft.
Kürzlich nutzten Forscher, darunter Gary Liu von der McMaster University und Denise B. Catacutan vom Massachusetts Institute of Technology, Deep Learning, um etwa 7.500 Moleküle zu screenen und neue Antibiotika zu identifizieren, die Acinetobacter baumannii hemmen.Aktuell wurde die Forschungsarbeit in der Fachzeitschrift Nature Chemical Biology unter dem Titel „Deep learning-guided discovery of an antibiotic targeting Acinetobacter baumannii“ veröffentlicht.

Die Forschungsergebnisse wurden in der Fachzeitschrift Nature Chemical Biology veröffentlicht.
ArgumentAdresse:
https://www.nature.com/articles/s41589-023-01349-8#access-options
Experimentelle Verfahren
Datensatz
Die Forscher untersuchten 7.684 kleine Moleküle basierend auf dem Wachstum von Acinetobacter baumannii ATCC 17978 in LB-Medium.Zu den kleinen Molekülen gehörten 2.341 Generika und 5.343 synthetische Chemikalien, die alle aus der Hochdurchsatz-Screening-Bibliothek des Broad Institute stammten. Mithilfe der herkömmlichen Trefferquoten-Grenze (d. h. Moleküle mit einer Wachstumshemmungsrate von weniger als einer Standardabweichung vom Mittelwert des Datensatzes) identifizierten sie 480 Moleküle als „aktiv“ und 7.204 als „inaktiv“.
Modelltraining
Um die Modellstruktur zu verbessern, verknüpften die Forscher die molekulare Darstellung mit 200 zusätzlichen Merkmalen auf molekularer Ebene, die mit RDCit berechnet wurden. Um die Leistung des Modells weiter zu verbessern, verwendeten die Forscher außerdem ein Ensemble aus 10 Modellen, um das Modell anhand eines Trainingsdatensatzes von 7.684 Molekülen (mit ~6,2% gültigen Beispielen) zu erstellen und zu trainieren, und wendeten das Modell auf den aktualisierten Drug Repurposing Hub an.Die Forscher teilten den Datensatz zufällig in 80% Trainingsdaten, 10% Validierungsdaten und 10% Testdaten auf.
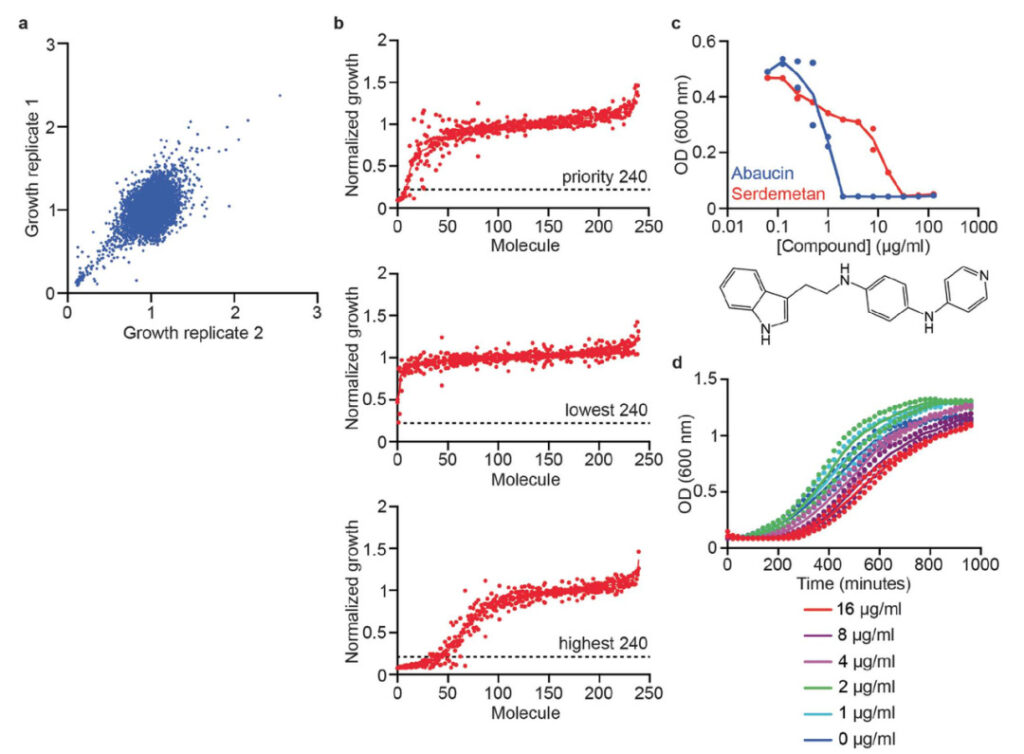
Abbildung 1: Trainingsdaten des Modells
A. Erste Screening-Daten für 7.684 kleine Moleküle, die das Wachstum von Acinetobacter baumannii ATCC 17978 in LB-Medium bei 50 μM hemmen.
B. Rangfolge der Daten zur Wachstumshemmung von 240 vorrangigen Molekülen, die zur Validierung aus dem Vorhersagesatz ausgewählt wurden (oben); Rangfolge der Wachstumshemmungsdaten von 240 vorhergesagten Molekülen mit den niedrigsten Vorhersagewerten (Mitte); Rangfolge der Wachstumshemmungsdaten von 240 vorhergesagten Molekülen mit den höchsten Vorhersagewerten, die nicht im Trainingsdatensatz gefunden wurden (unten). Die horizontale gestrichelte Linie stellt den strengen Treffergrenzwert von >80%-Wachstumshemmung bei 50 μM dar.
C. Das Wachstum von Acinetobacter baumannii im LB-Medium wurde durch Abaucin (blau) und Serdemetan (rot) gehemmt.
D. Wachstumskinetik von A. baumannii-Zellen nach Behandlung mit unterschiedlichen Konzentrationen von Abaucin für 6 Stunden.
Codeerfassung:
https://github.com/chemprop/chemprop
Experimente mit Mausmodellen
Um die Anzahl der Mäuse zu reduzieren, behandelten die Forscher 6- bis 8-wöchige weibliche C57BL/6N-Mäuse vier Tage und einen Tag vor Beginn des Experiments mit 150 mg/kg bzw. 100 mg/kg Cyclophosphamid, basierend auf dem Körpergewicht. Am Tag 0 wurden die Mäuse mit Isofluran anästhesiert und erhielten eine intraperitoneale Injektion von Buprenorphin in einer Dosis von 0,1 mg/kg als Analgetikum. Die Forscher verwendeten etwa 30–35 Stücke Hochdruckklebeband, um 2 cm2 große Abschürfungen auf dem Rücken von Mäusen zu erzeugen, zogen das Klebeband bis zur Basalschicht der Epidermis ab und infizierten die Mäuse dann mit etwa 6,5 x 106 KBE A. baumannii ATCC 17978.Mäuse (n = 5/6) wurden 1, 2, 3, 4, 6, 10, 21 und 24 Stunden nach der Infektion mit ~10–20 μl Glaxal Base mit Abaucin (Versuchsgruppe) oder DMSO (Kontrollgruppe) behandelt.
Experimentelle Ergebnisse
Mithilfe von maschinellem Lernen entdeckten Forscher eine antibakterielle Verbindung namens Abaucin.Es hat ein enges Wirkungsspektrum gegen Acinetobacter baumannii und kann den Transportprozess von Lipoproteinen stören, indem es den LolE-Mechanismus beeinflusst. Die folgende Abbildung zeigt den Prozess der Deep Learning-gesteuerten Entdeckung von Abaucin.
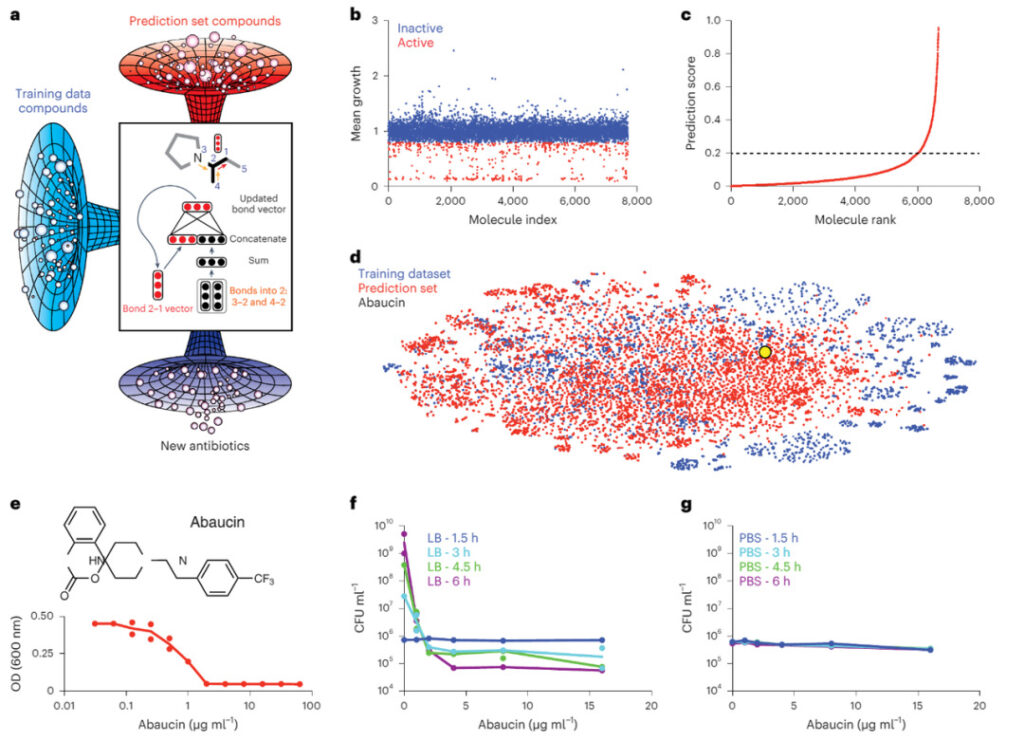
Abbildung 2: Maschinelles Lernen führte zur Entdeckung von Abaucin
A. ~7.500 gescreente Moleküle (blau); ein tiefes neuronales Netzwerk für gerichteten Informationstransfer, das mit diesem Datensatz zur Wachstumshemmung trainiert und im Drug Repurposing Hub (rot) vorhergesagt wurde; neue Moleküle, die Acinetobacter baumannii (lila) hemmen können.
B. Wachstumshemmung von Acinetobacter baumannii ATCC 17978 bei 50 μM durch eine Sammlung von 7.684 kleinen Molekülen.
C. Das trainierte Modell stuft die Vorhersagewerte von Molekülen im Drug Repurposing Hub ein. Moleküle mit Vorhersagewerten über 0,2 wurden als vorläufige Kandidatenmoleküle betrachtet.
D. Ein t-SNE-Diagramm zeigt die chemischen Beziehungen zwischen dem Trainingsdatensatz (blau), dem Vorhersagedatensatz (rot) und Abaucin (gelb).
e. Wachstumshemmung von Acinetobacter baumannii durch Abaucin in LB-Medium.
F. Die abtötende Wirkung von Abaucin auf Acinetobacter baumannii in verschiedenen Konzentrationen nach 1,5 Stunden (blau), 3 Stunden (cyan), 4,5 Stunden (grün) und 6 Stunden (violett) Inkubation unter nährstoffreichen Bedingungen.
G. Die abtötende Wirkung von Abaucin auf Acinetobacter baumannii nach 1,5 Stunden (blau), 3 Stunden (cyan), 4,5 Stunden (grün) und 6 Stunden (violett) Inkubation unter nährstoffarmen Bedingungen.
Wie in Abbildung 2 dargestellt, untersuchten die Forscher etwa 7.500 Moleküle, trainierten ein neuronales Netzwerk mithilfe dieses Datensatzes zur Wachstumshemmung und sagten neue Moleküle voraus, die die Aktivität von Acinetobacter baumannii hemmen könnten.Zusammenfassend deuten diese Daten darauf hin, dass Abaucin seine antimikrobielle Wirksamkeit durch die Hemmung eines biologischen Prozesses entfaltet, der während des Wachstums und der Teilung am aktivsten ist, was mit den meisten bekannten Antibiotika übereinstimmt.
Um die Wirksamkeit von Abaucin in vivo zu testen,Die Forscher stellten Wundinfektionen auf dem Rücken von Mäusen fest.
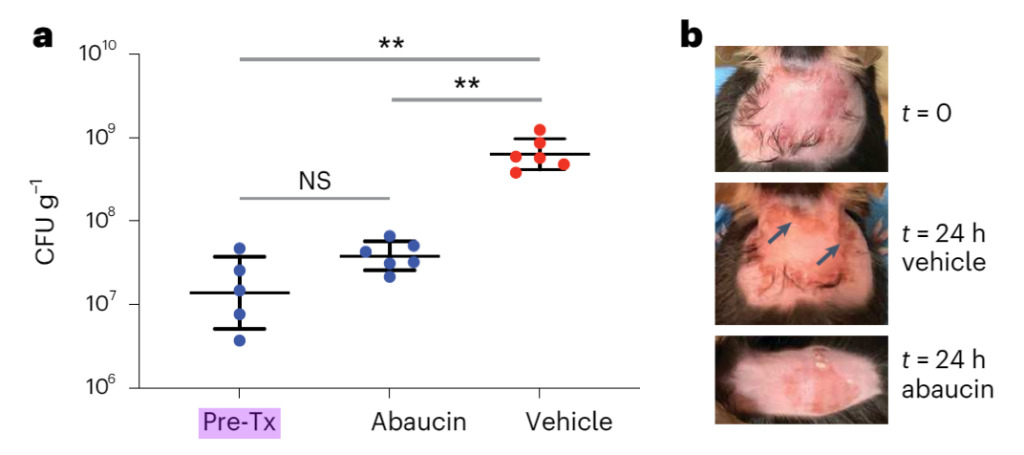
Abbildung 3: Abaucin kann eine Infektion mit Acinetobacter baumannii in einem Wundmodell hemmen
A. Im Modell der Rückenwundeninfektion wurden Mäuse mit Acinetobacter baumannii ATCC 17978 (~6,5 x 106 KBE) infiziert. Eine Stunde nach der Infektion wurden die Mäuse über 24 Stunden lang mit DMSO (rot; n = 6) oder 4%-Abaucin (blau; n = 6) behandelt. Die vertikale Achse stellt die Bakterienlast im Wundgewebe 25 Stunden nach der Infektion dar. Pre-Tx stellt die bakterielle Belastung zum Zeitpunkt der Erstbehandlung dar (n = 5). Schwarze Linien stellen Mittelwert ± Standardabweichung dar. NS bedeutet „statistisch nicht signifikant“; ** P < 0,005 bei Verwendung eines ungepaarten zweiseitigen t-Tests mit Welch-Korrektur (Pre-Tx vs. 4%-Abaucin, P = 0,0704; Pre-Tx vs. DMSO, P = 0,0034; DMSO vs. 4%-Abaucin, P = 0,0039).
ps: Im Allgemeinen steht ein Sternchen für einen p-Wert kleiner als 0,05, zwei Sternchen für einen p-Wert kleiner als 0,01 und drei Sterne für einen p-Wert kleiner als 0,001. Je kleiner der p-Wert, desto stärker die Signifikanz.
B. Bilder der Rückenoberfläche von Mäusen vor der Infektion (t = 0), nach 24 Stunden DMSO-Behandlung und nach 24 Stunden Abaucin-Behandlung. Die bei DMSO beobachtete Entzündung (Pfeile) war bei mit Abaucin behandelten Mäusen nicht vorhanden.
Abbildung 3 zeigt, dass die Bakterienlast in mit Abaucin behandelten Wunden deutlich geringer war als in der DMSO-Gruppe und die nach 24 Stunden DMSO-Behandlung noch vorhandene Wundentzündung bei Abaucin verschwand.Abaucin kann Infektionen, die durch Acinetobacter baumannii verursacht werden, wirksam bekämpfen.
Zusammenfassend haben die Forscher mithilfe von Deep Learning herausgefunden, dass Abaucin eine Infektion mit Acinetobacter baumannii wirksam kontrollieren kann.Diese Studie unterstreicht die Rolle des maschinellen Lernens bei der Entdeckung neuer Antibiotika und der Erforschung schwieriger gramnegativer Krankheitserreger.
KI verändert die traditionelle Art der Entdeckung neuer Medikamente
Es ist erwähnenswert, dass diese Forschung von Jon Stokes, einem Assistenzprofessor an der McMaster University, James J. Collins, einem Professor für Medizintechnik und -wissenschaft am MIT, und den Doktoranden der McMaster University Gary Liu und Denise Catacutan durchgeführt wurde. Jon Stokes, Gary Liu und Denise Catacutan sind alle Mitglieder des Stokes Laboratory.

Adresse des Stokes-Labors:
https://www.thestokeslab.com/projects
Das Labor entdeckt neue Antibiotika durch den Einsatz modernster Hochdurchsatzbiologie und moderner Ansätze der künstlichen Intelligenz.Konkret trainierten die Forscher Deep-Learning-Modelle, um neue Antibiotika anhand einer großen Computersimulationsdatenbank vorherzusagen. Darüber hinaus erstellten sie Modelle zur Vorhersage des Wirkungsmechanismus und der In-vivo-Eigenschaften neuer Antibiotika. Sie nutzen maschinelles Lernen, um die Entdeckung neuer Antibiotika zu beschleunigen und gleichzeitig die Forschungs- und Entwicklungskosten zu senken.
Professor Stokes sagte einmal in einem Interview, dass „künstliche Intelligenz die Art und Weise, wie neue Medikamente entdeckt werden, grundlegend verändern wird.“Die Ergebnisse deuten darauf hin, dass künstliche Intelligenz erhebliche Auswirkungen auf die Entdeckung neuer Antibiotika gegen eine Vielzahl schwieriger Krankheitserreger hat und dass dieser Ansatz auch bei der Suche nach anderen antimikrobiellen Therapien Potenzial birgt. Künstliche Intelligenz kann zwar nicht alle Probleme lösen, doch als leistungsstarkes Werkzeug kann sie uns dabei helfen, neue Medikamente zu finden und den Menschen Hoffnung zu geben.
Referenzartikel:
[1]http://ccm.dxy.cn/article/805579
[2]https://zh.wikipedia.org/wiki/
[3]https://www.ctvnews.ca/health/scientists-use-ai-to-discover-antibiotic-for-very-difficult-to-treat-bacteria-1.6411927
-- über--








