Command Palette
Search for a command to run...
VenusFactory 蛋白质工程设计平台

一、教程简介

VenusFactory 由上海交通大学、上海人工智能实验室、华东理工大学联合团队于 2025 年开发,相关论文成果为 VenusFactory: A Unified Platform for Protein Engineering Data Retrieval and Language Model Fine-Tuning 。
VenusFactory 是一个专为蛋白质工程领域设计的统一平台,旨在整合生物数据检索、标准化任务基准测试和预训练蛋白质语言模型(PLMs)的模块化微调功能。
该平台支持命令行执行和基于 Gradio 的无代码界面,集成了超过 40 个与蛋白质相关的数据集和超过 40 个流行的 PLMs,方便计算机科学和生物学领域的研究人员使用。
教程提供 7 个功能模块:
- Training:零代码训练模型,支持 40+ 大模型,使用私有数据集训练自己的模型。
- Evaluation:易操作的蛋白质模型全面性能评估工具。
- Prediction:使用训练好的模型对新的蛋白质序列进行功能预测。
- VenusAgent:蛋白质工程 Agent,搭配 DeepSeek 对话实现 AI 蛋白质计算。
- Quick Tools:轻松使用版,支持零样本突变预测(定向进化)、有监督预测(功能或性质预测)。
- Advanced Tools:进阶定制使用版,支持零样本突变预测(定向进化)、有监督预测(功能或性质预测)。
- Download:轻松链接蛋白质数据,支持多线程下载各大主流数据库(RCSB 、 UniProt…)。
该教程算力资源采用单卡 RTX 4090 。教程使用的模型保存在
/openbayes/input/input1目录下,所有数据均保存在/openbayes/home/VenusFactory目录下。
二、运行步骤
1. 启动容器
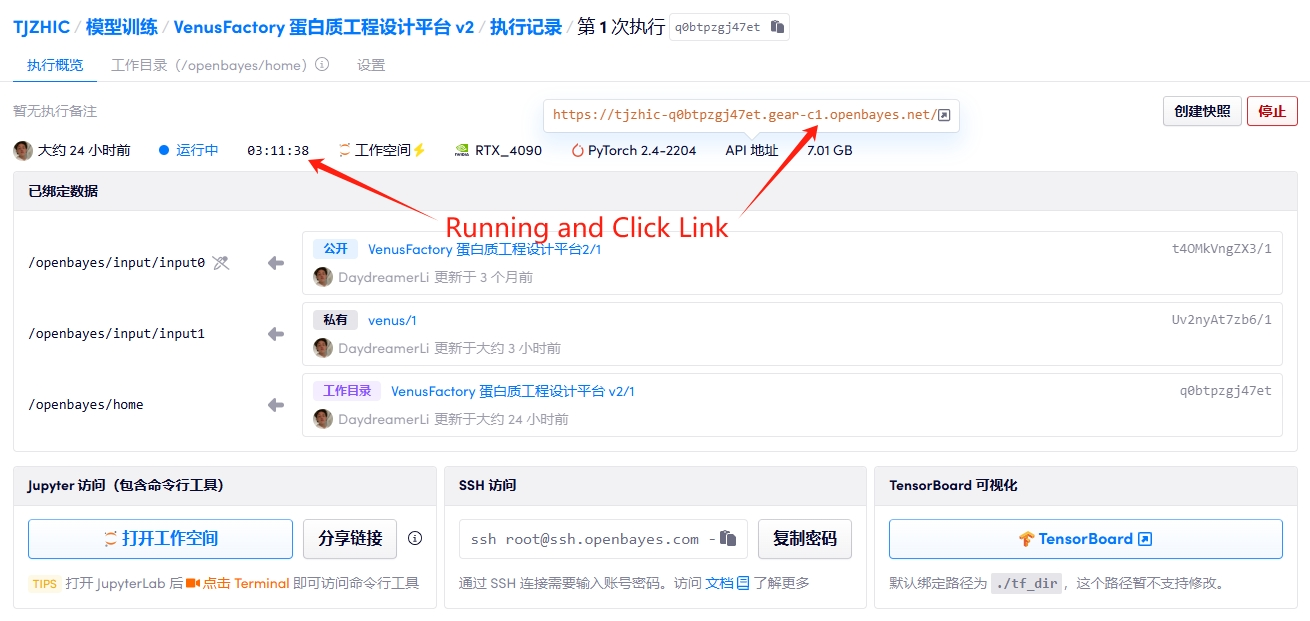
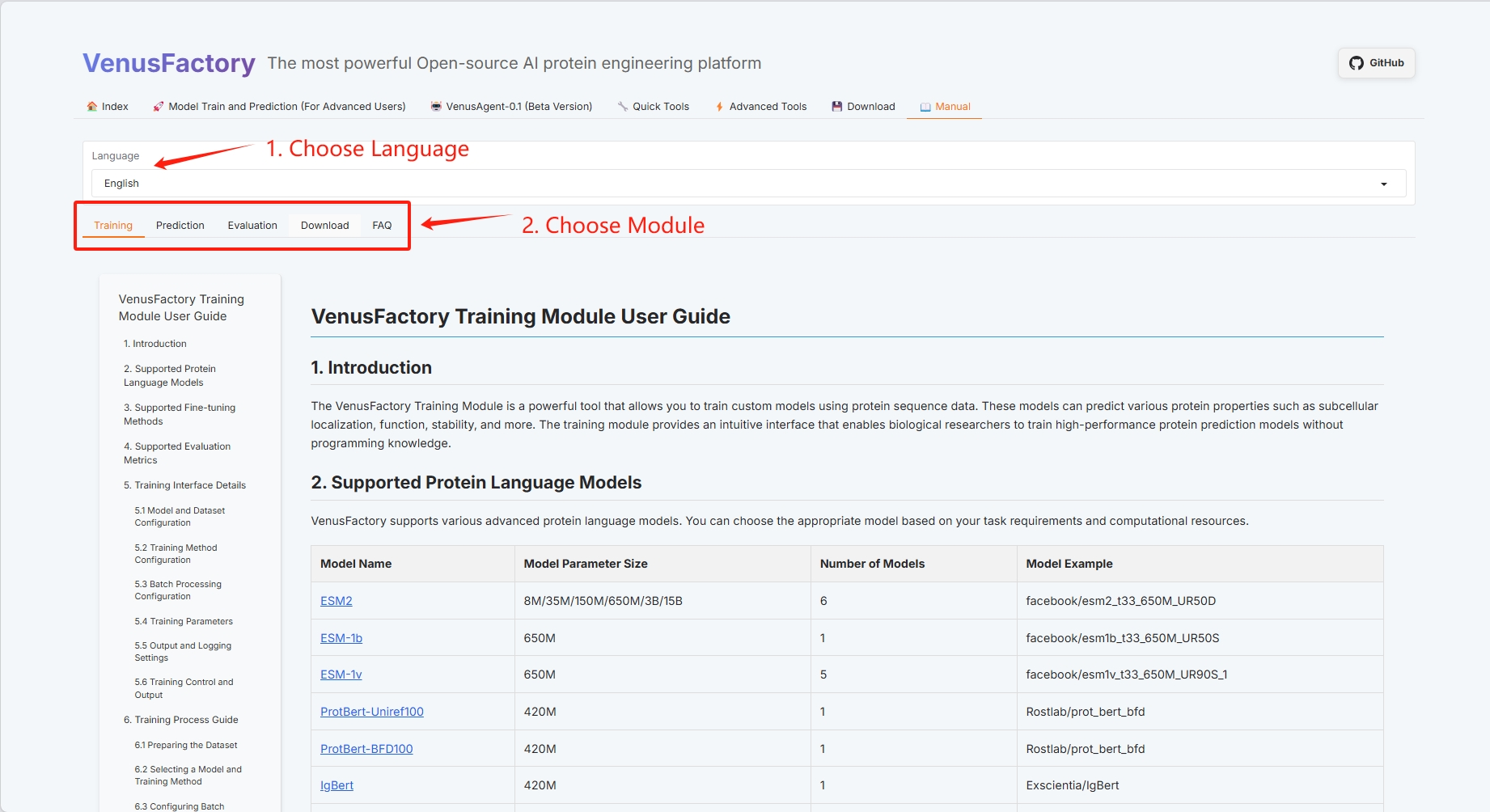
2. Manual 使用指南
若显示「Bad Gateway」,这表示项目正在初始化,请等待约 1-2 分钟后刷新页面。
VenusFactory 的「Manual」使用指南目前共包含 Training 、 Evaluation 、 Prediction 、 Download 四个模块。
三、具体功能演示
3.1 训练
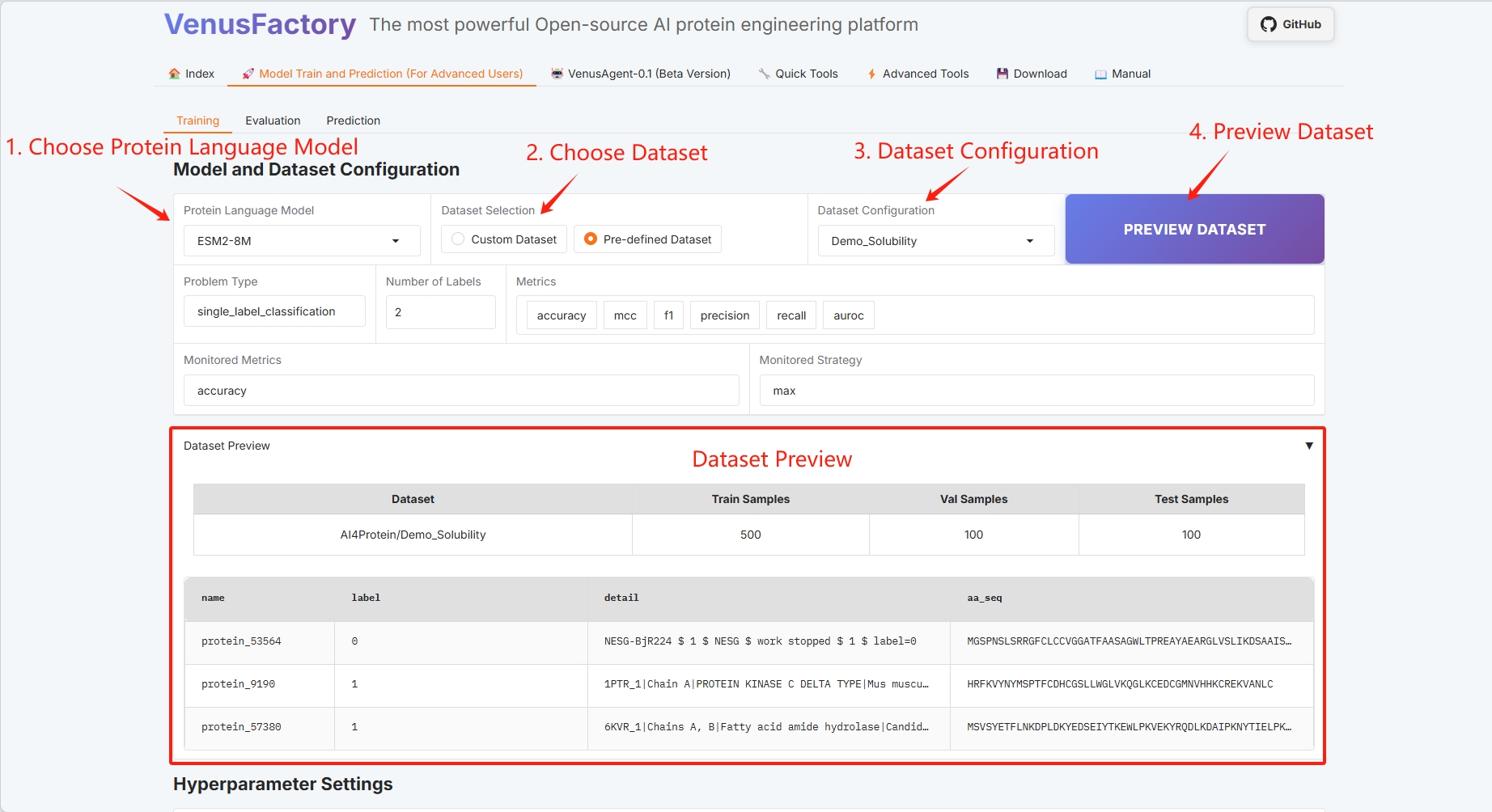
点击「Model Train and Prediction Training」模块中的「Training」模块
- 选择 Protein Language Model
- 数据集选择
- 数据集预览
- 训练方法配置(具体信息参考使用指南)
- 批处理配置(具体信息参考使用指南)
如果选择的模型参数较大,请更换更大的显卡设备。
设置训练模型保存路径,点击「START TRAINING」开始训练。
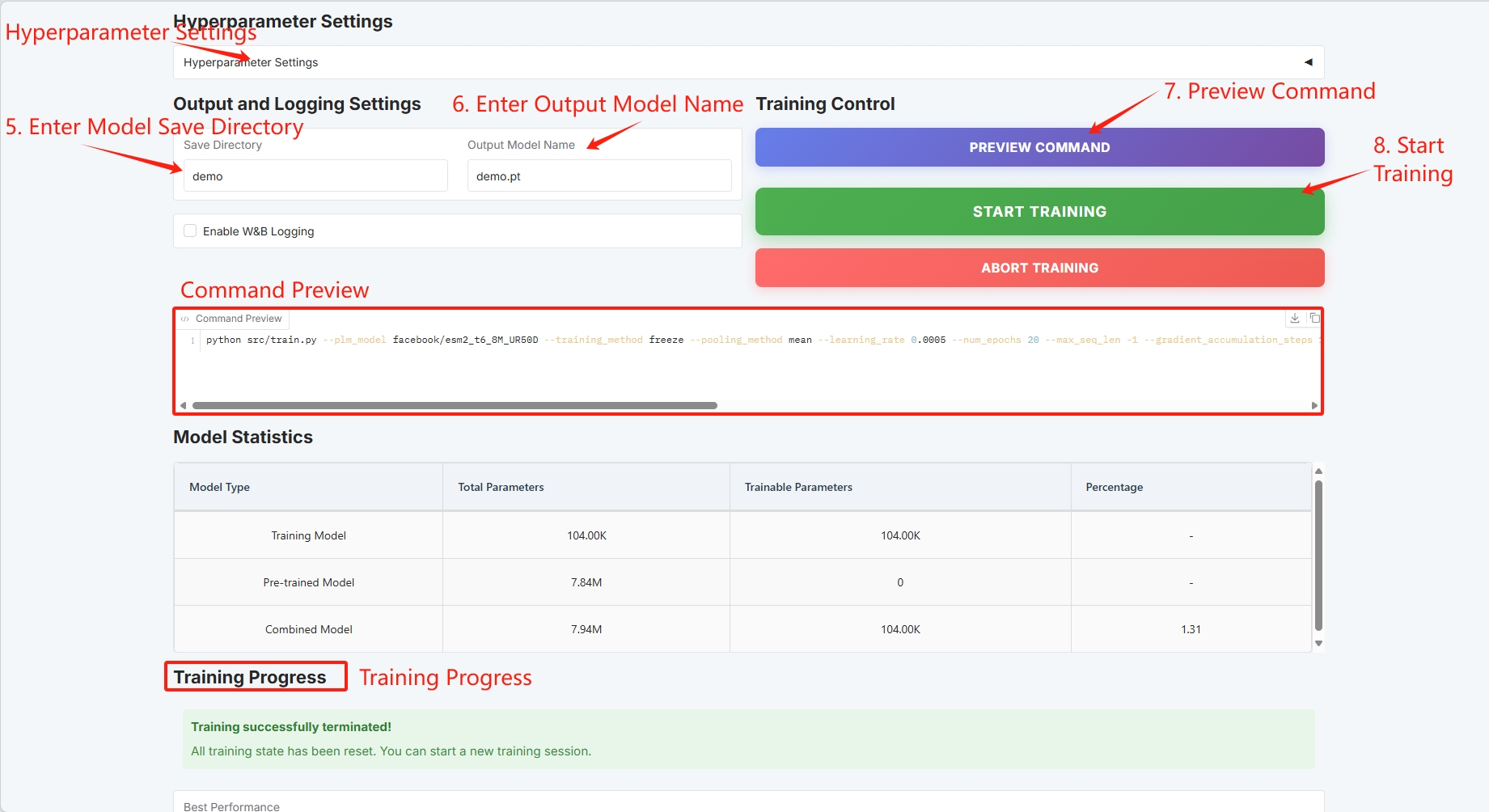
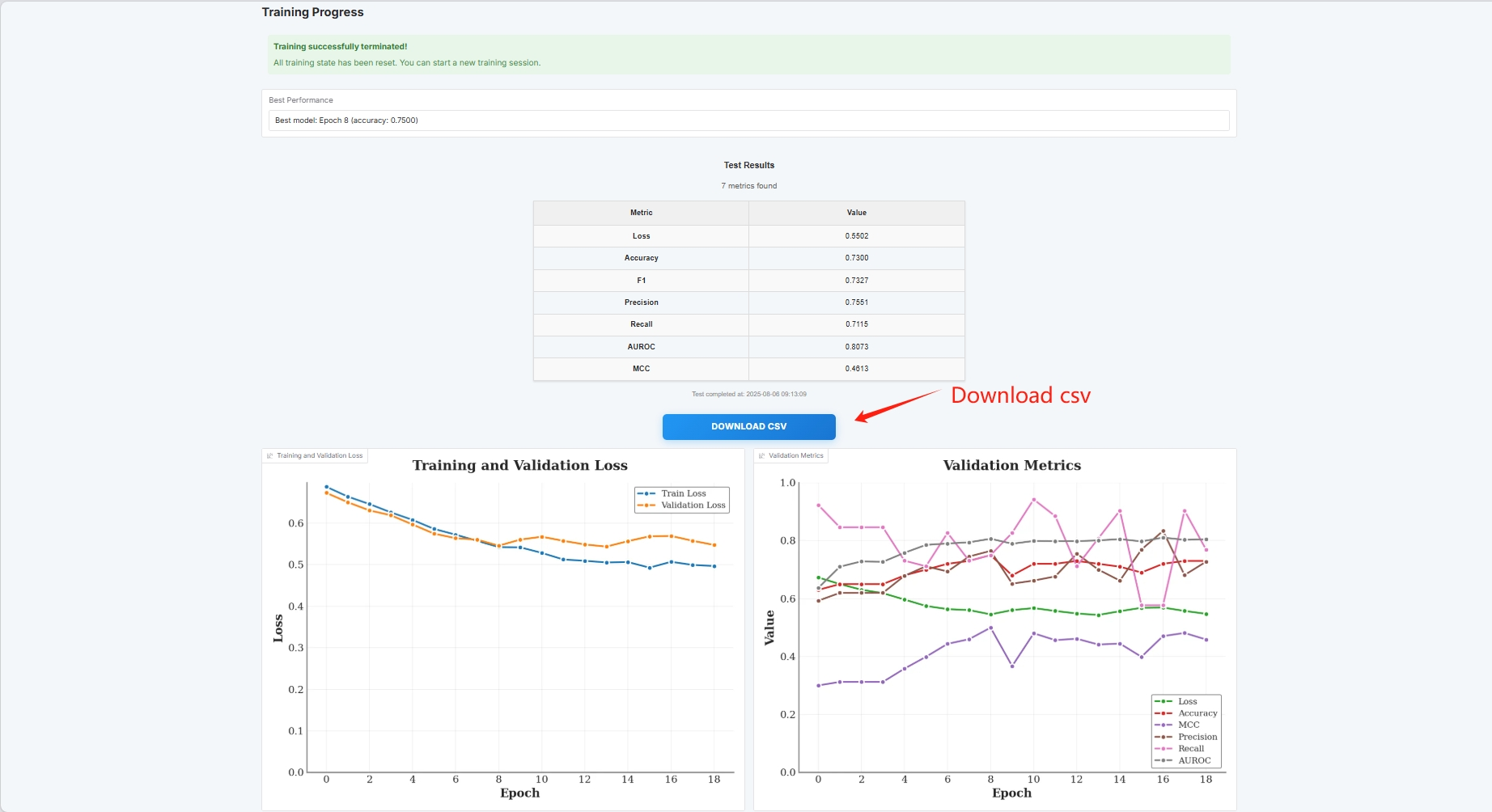
此时可以看到训练的参数量以及 loss 曲线图
如果想要使用自己的数据集可以通过 Custom Dataset 配置,只需要填写你的数据集路径即可(详情请参阅 Manual 使用文档)。
3.2 评估
点击「Model Train and Prediction Training」模块中的「Evaluation」模块
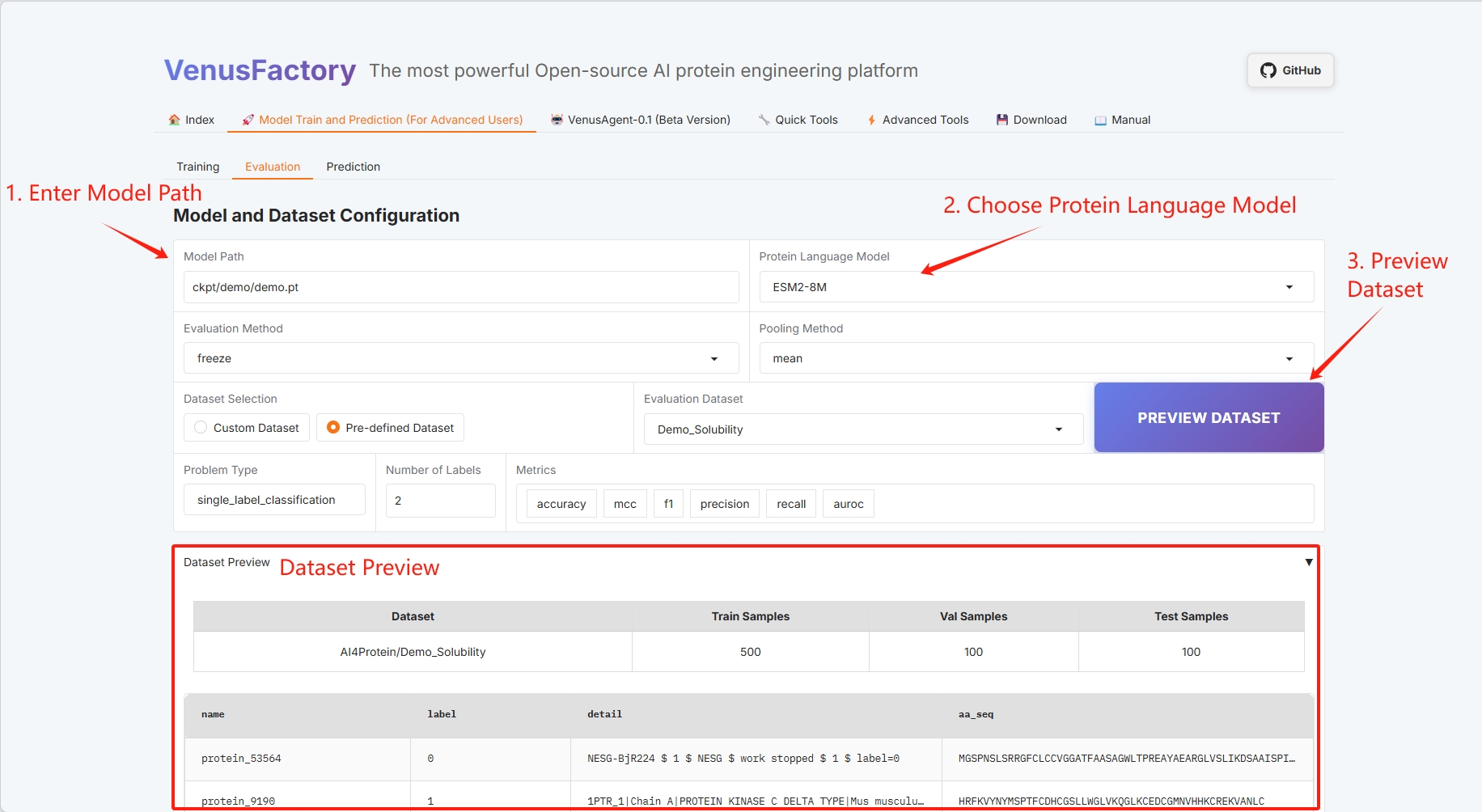
- 模型路径和蛋白质语言模型选择
- 评估方法和池化方法(具体信息参考使用指南)
- 数据集选择
- 数据集预览
- 问题类型和标签(具体信息参考使用指南)
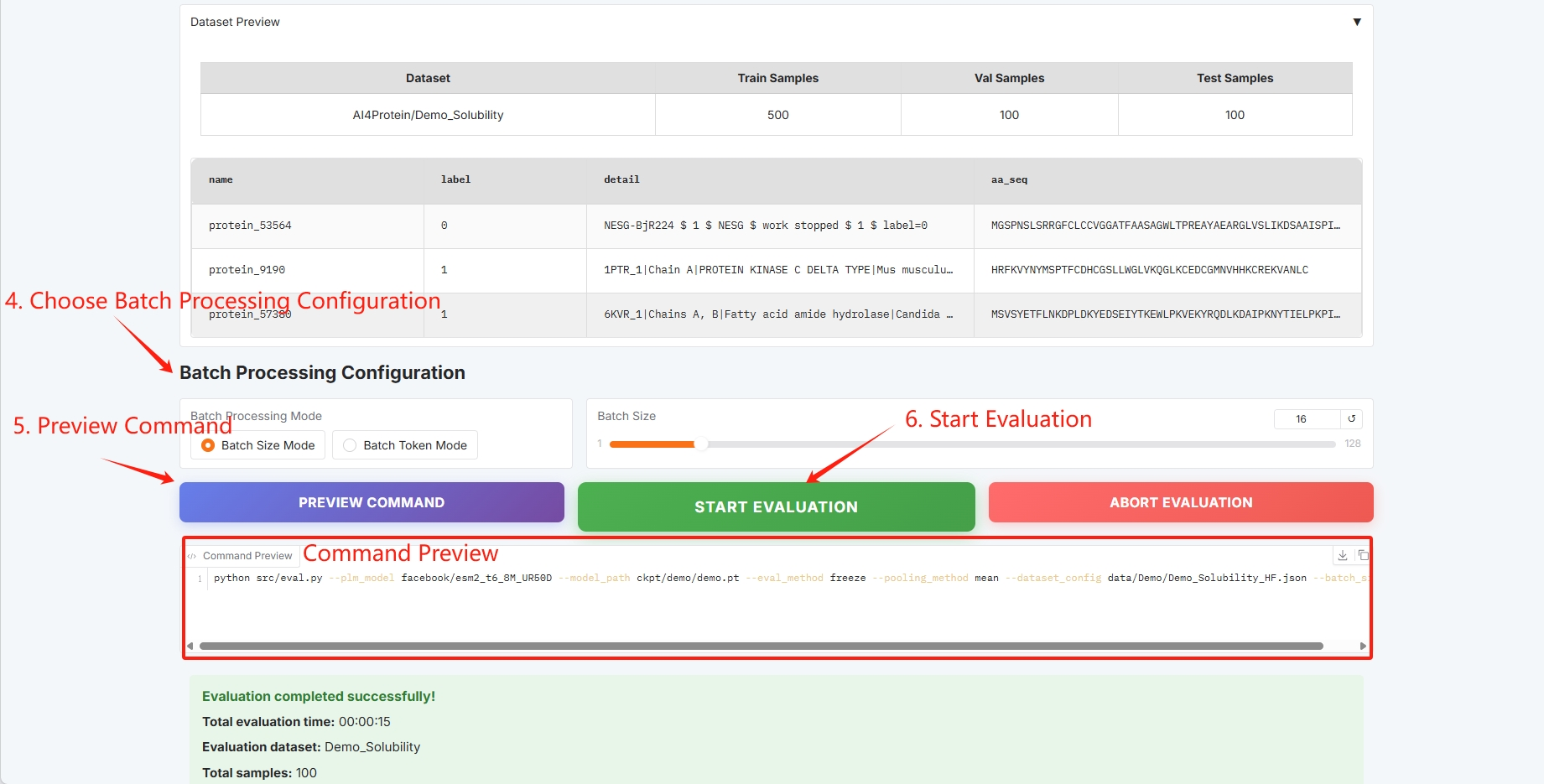
- 批处理配置(具体信息参考使用指南)
设置训练好的模型保存路径,选择蛋白质语言模型。
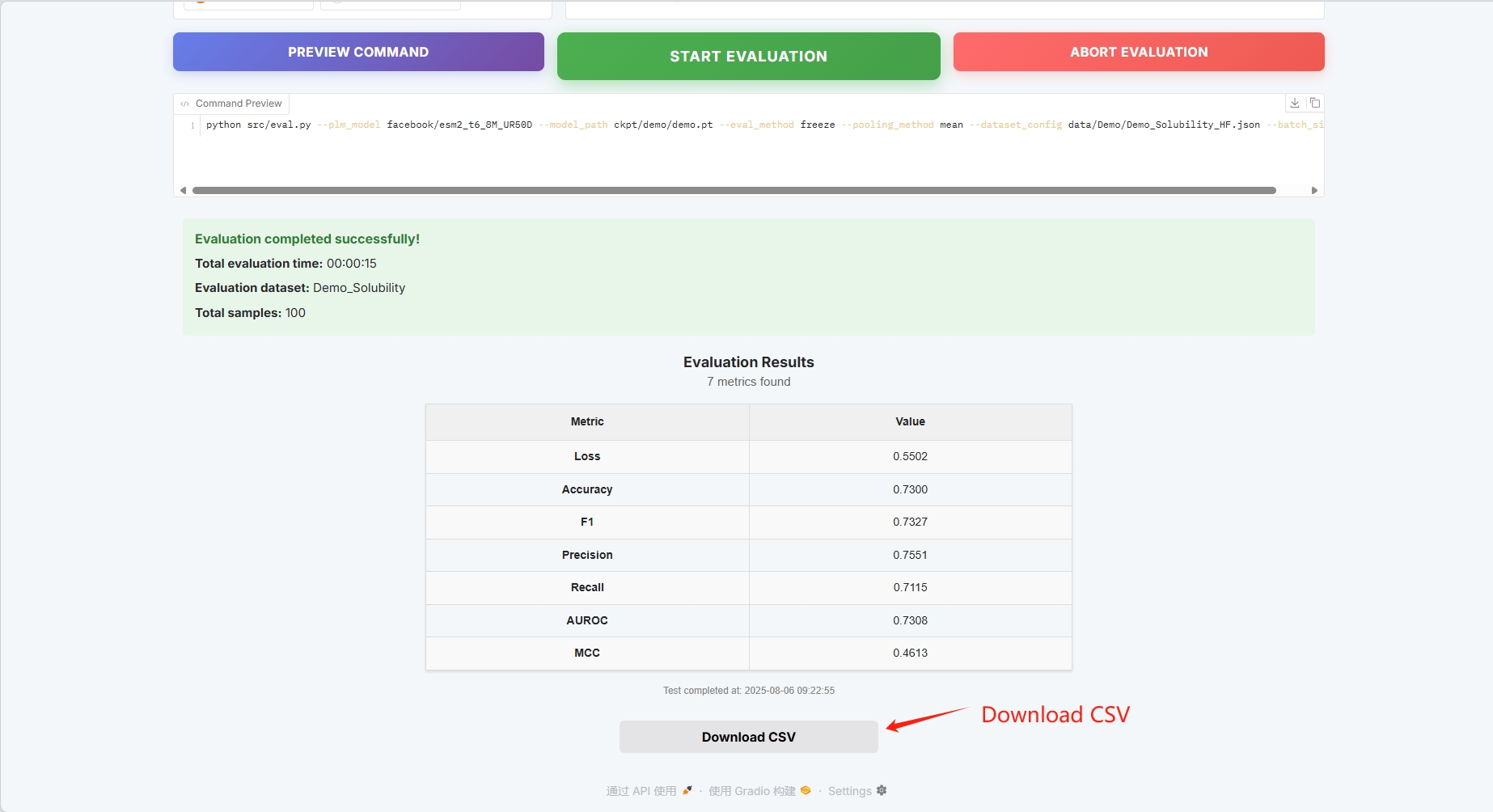
批处理配置,点击「START EVALUATION」开始训练。
评估结果如下,可以下载 CSV
如果想要使用自己的数据集可以通过 Custom Dataset 配置,只需要填写你的数据集路径即可(详情请参阅 Manual 使用文档)。
3.3 预测
点击「Model Train and Prediction Training」模块中的「Prediction」模块,可进行单序列预测和批量预测。
- 模型配置
- 选择预测模块(具体信息参考使用指南)
设置训练模型保存路径,选择蛋白质语言模型,点击「START PREDICTION」开始训练。
单序列预测
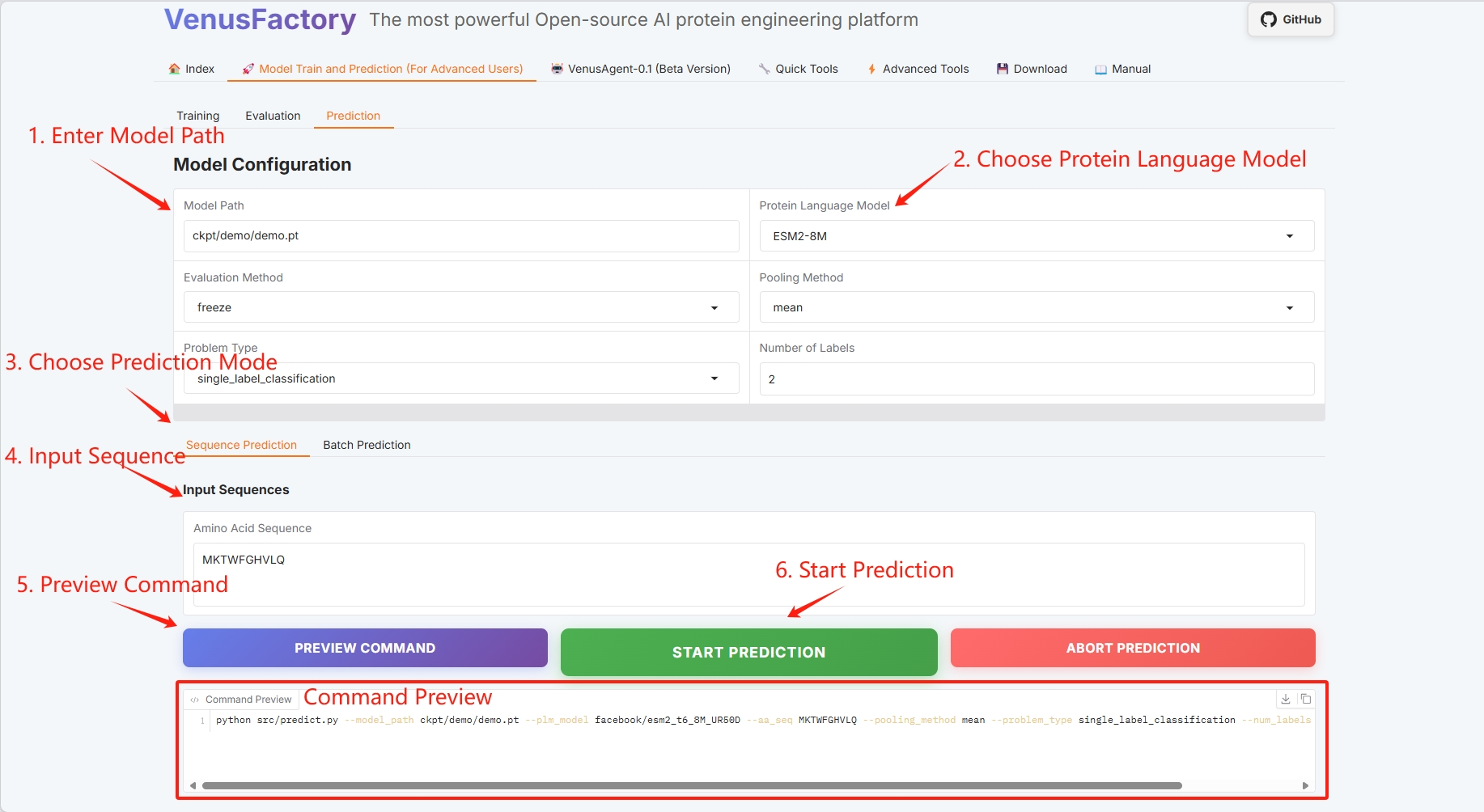
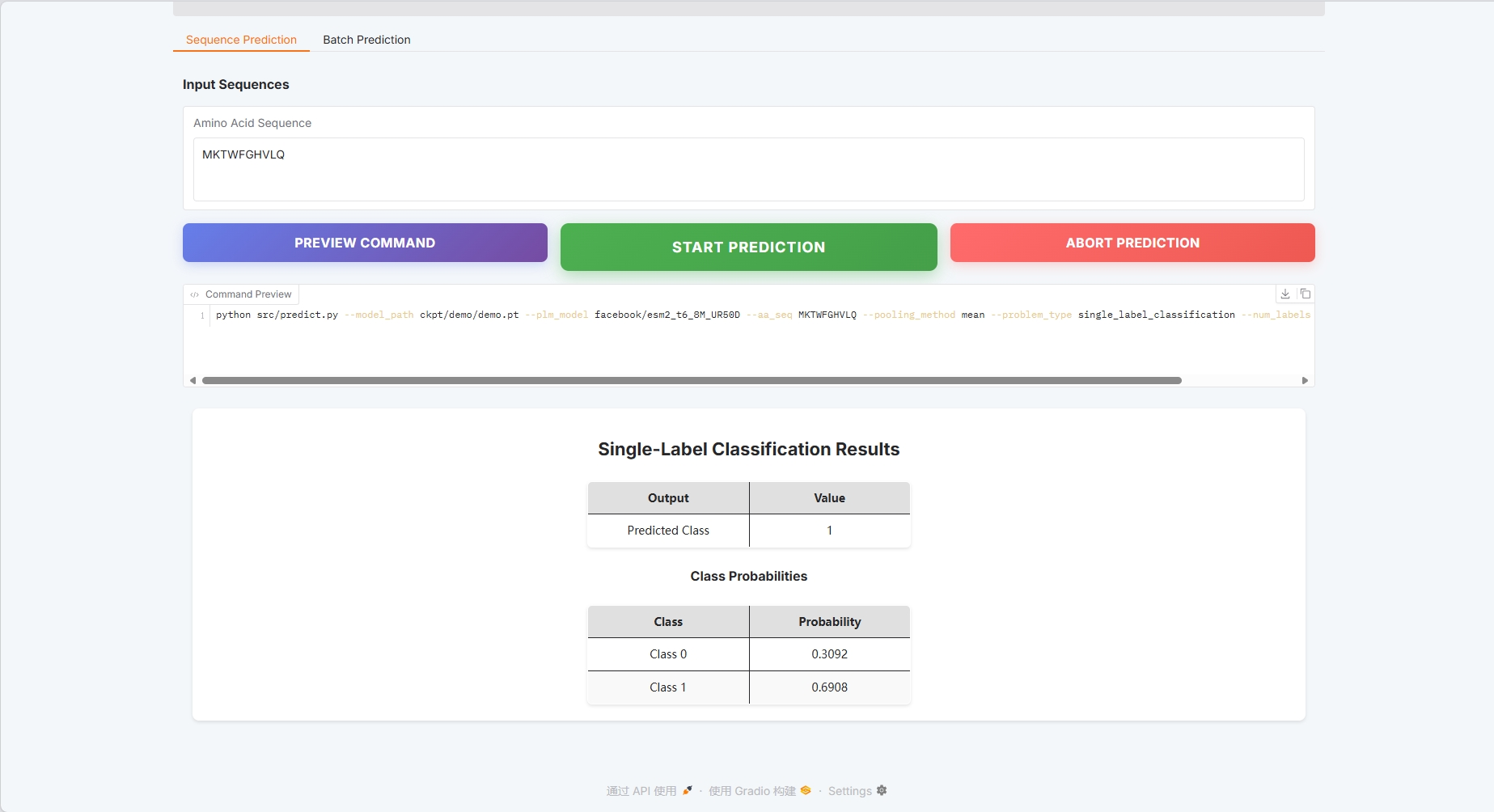
蛋白质序列示例:MKTWFGHVLQ
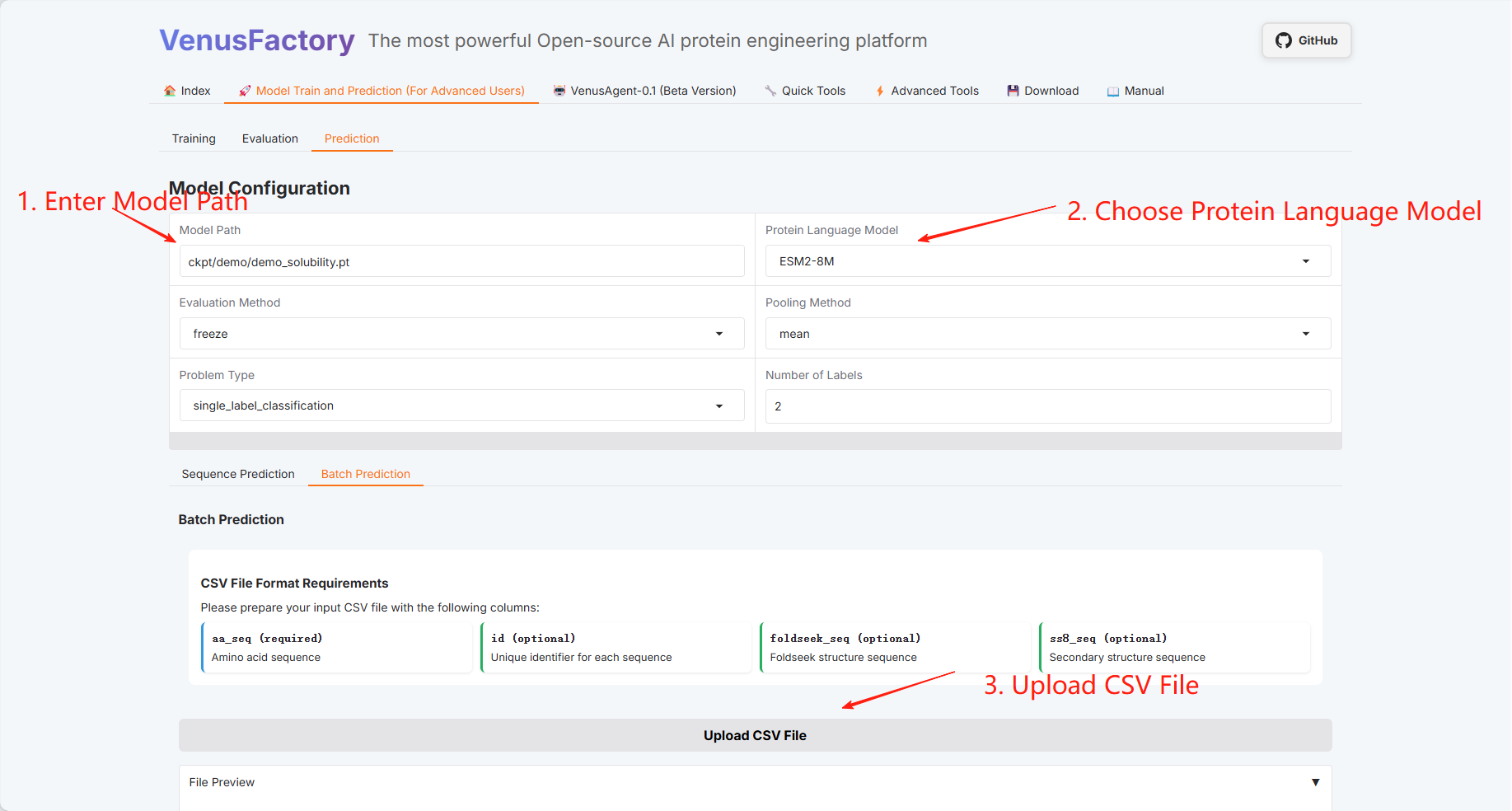
批量预测
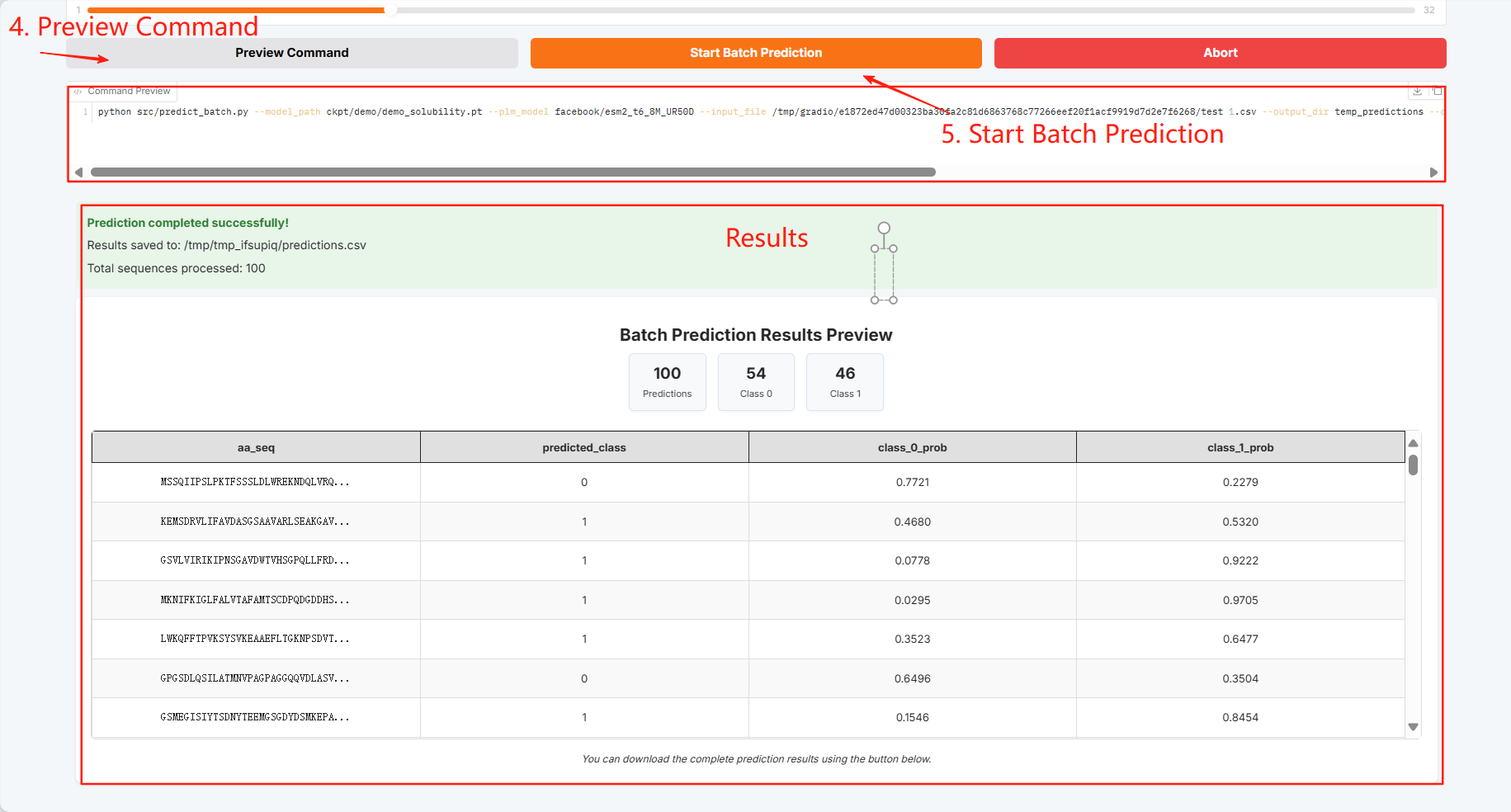
可以将批量预测结果下载保存
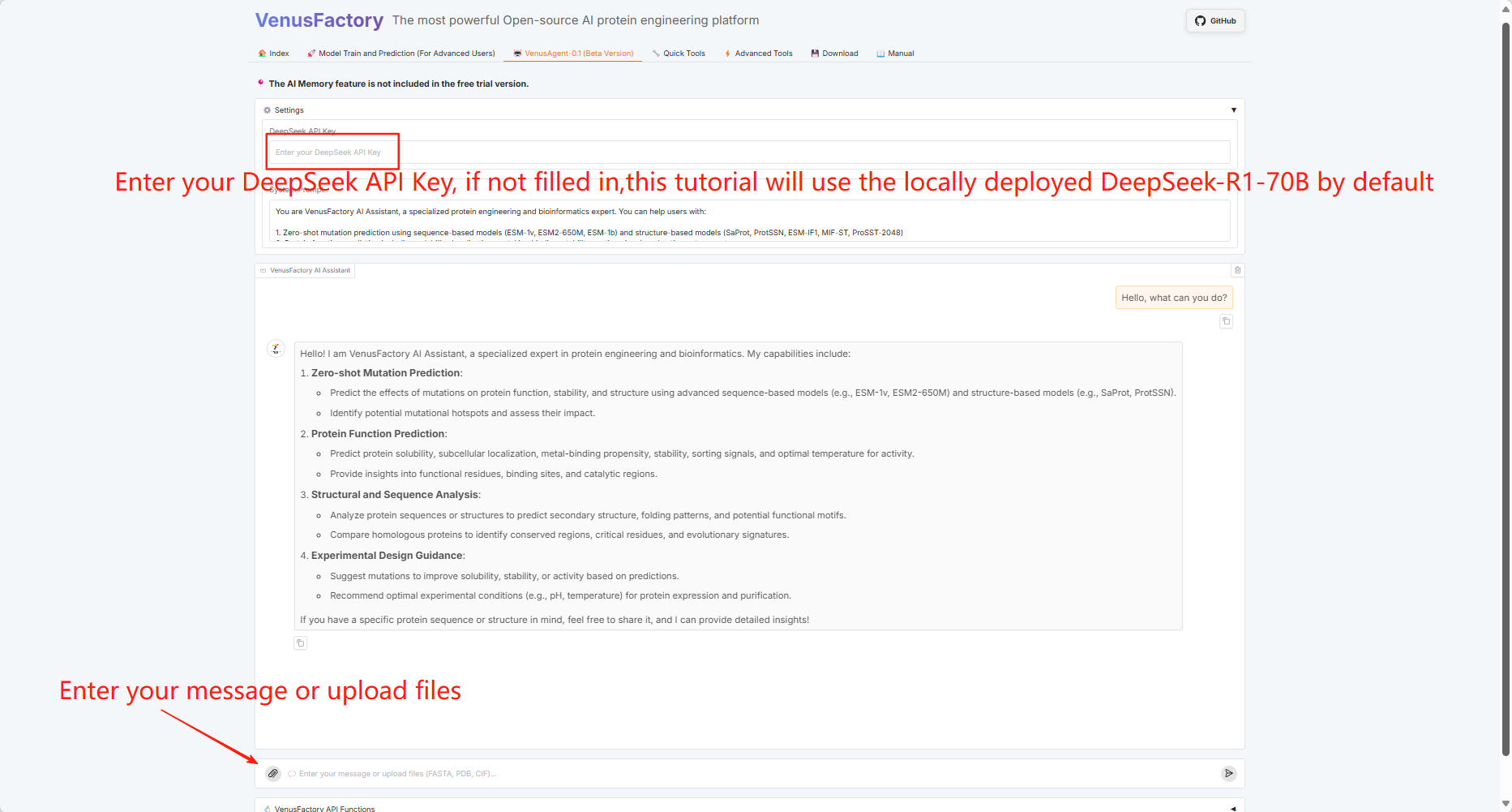
3.4 VenusAgent
点击「VenusAgent」模块
由于 VenusAgent 需要调用 DeepSeek 大模型,本教程提供 2 种调用方式:自行输入 API Key 或使用平台部署好的 DeepSeek-R1-70B 模型。
可以根据所需功能选用不同的显卡体验,选卡说明如下:
若使用单卡 RTX 4090,则不支持 VenusAgent 功能使用本地部署的大模型服务(使用 DeepSeek API Key 无限制)。
若使用双卡 RTX 4090,使用 VenusAgent 功能后不能立即(1-2 分钟后)使用其它功能(使用 DeepSeek API Key 无限制)。
若使用双卡 RTX A6000,VenusAgent 功能无限制。
该教程算力资源采用单卡 RTX 4090 。教程使用的模型保存在
/openbayes/input/input1目录下,所有数据均保存在/openbayes/home/VenusFactory目录下。
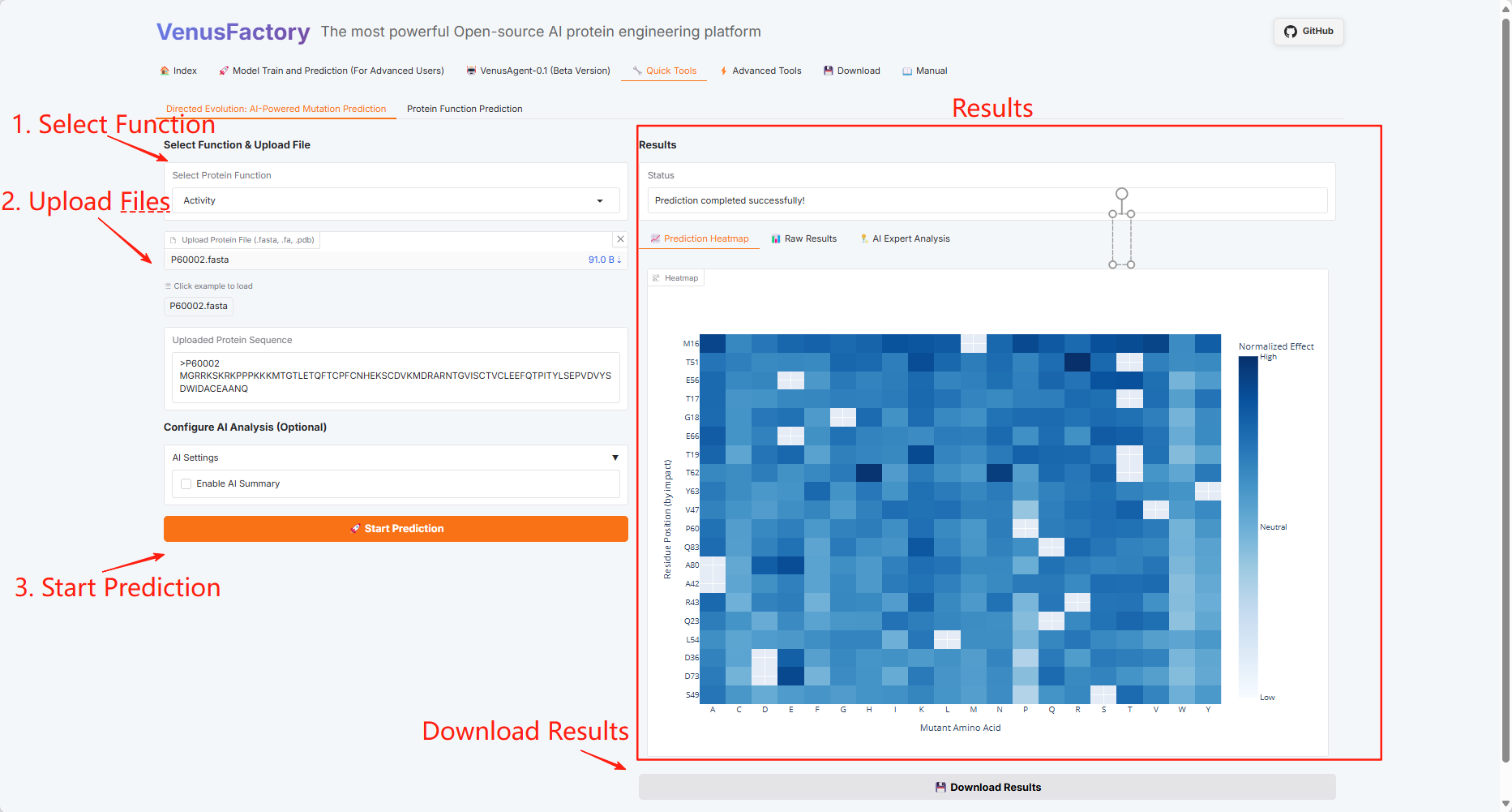
3.5 Quick Tools
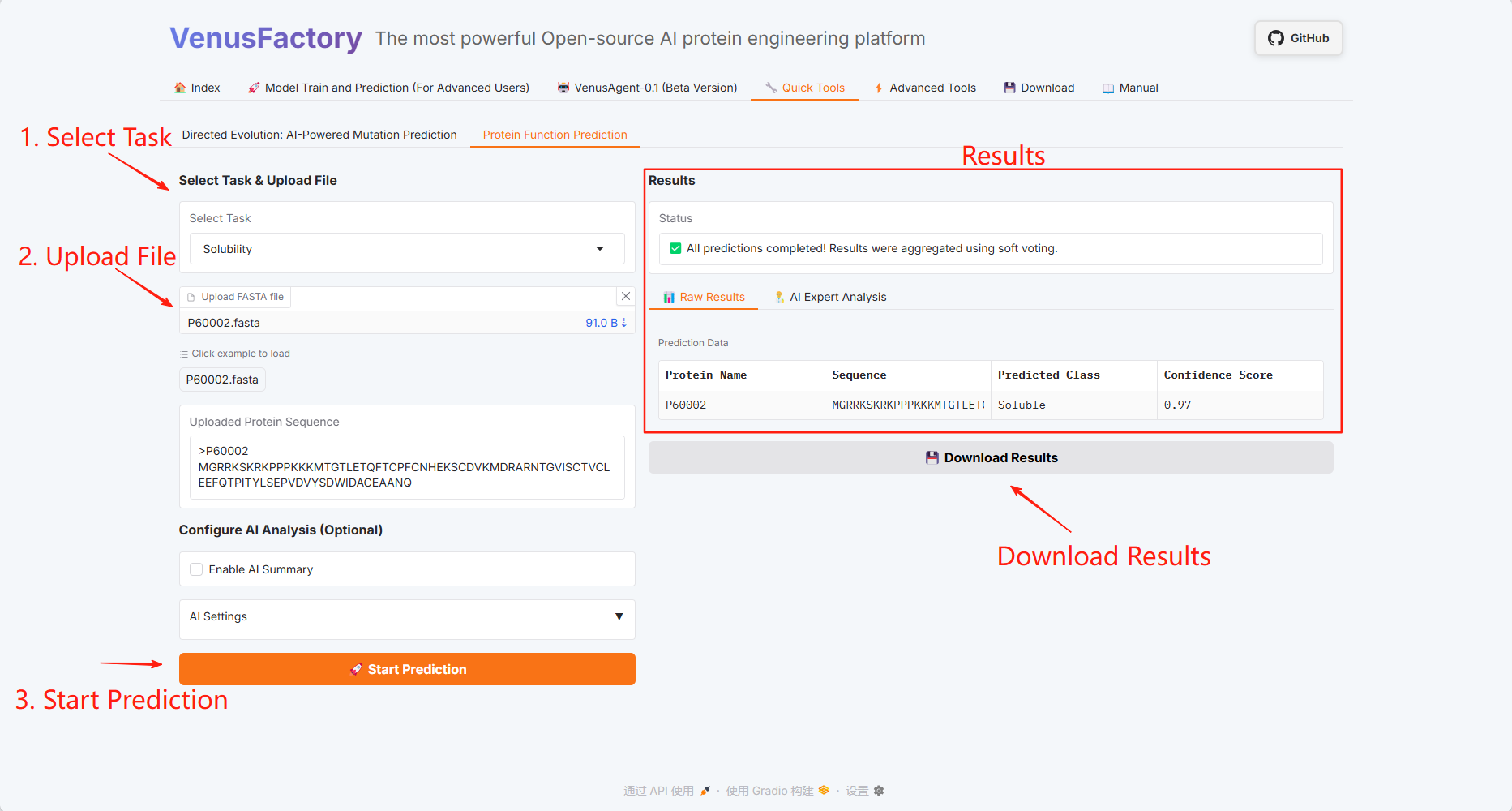
点击「Quick Tools」模块,此处包含 Directed Evolution: AI-Powered Mutation Prediction 和 Protein Function Prediction 两个功能。
Directed Evolution: AI-Powered Mutation Prediction
Protein Function Prediction
3.6 Advanced Tools
点击「Advanced Tools」模块,此处包含 Directed Evolution: AI-Powered Mutation Prediction 和 Protein Function Prediction 两个功能。
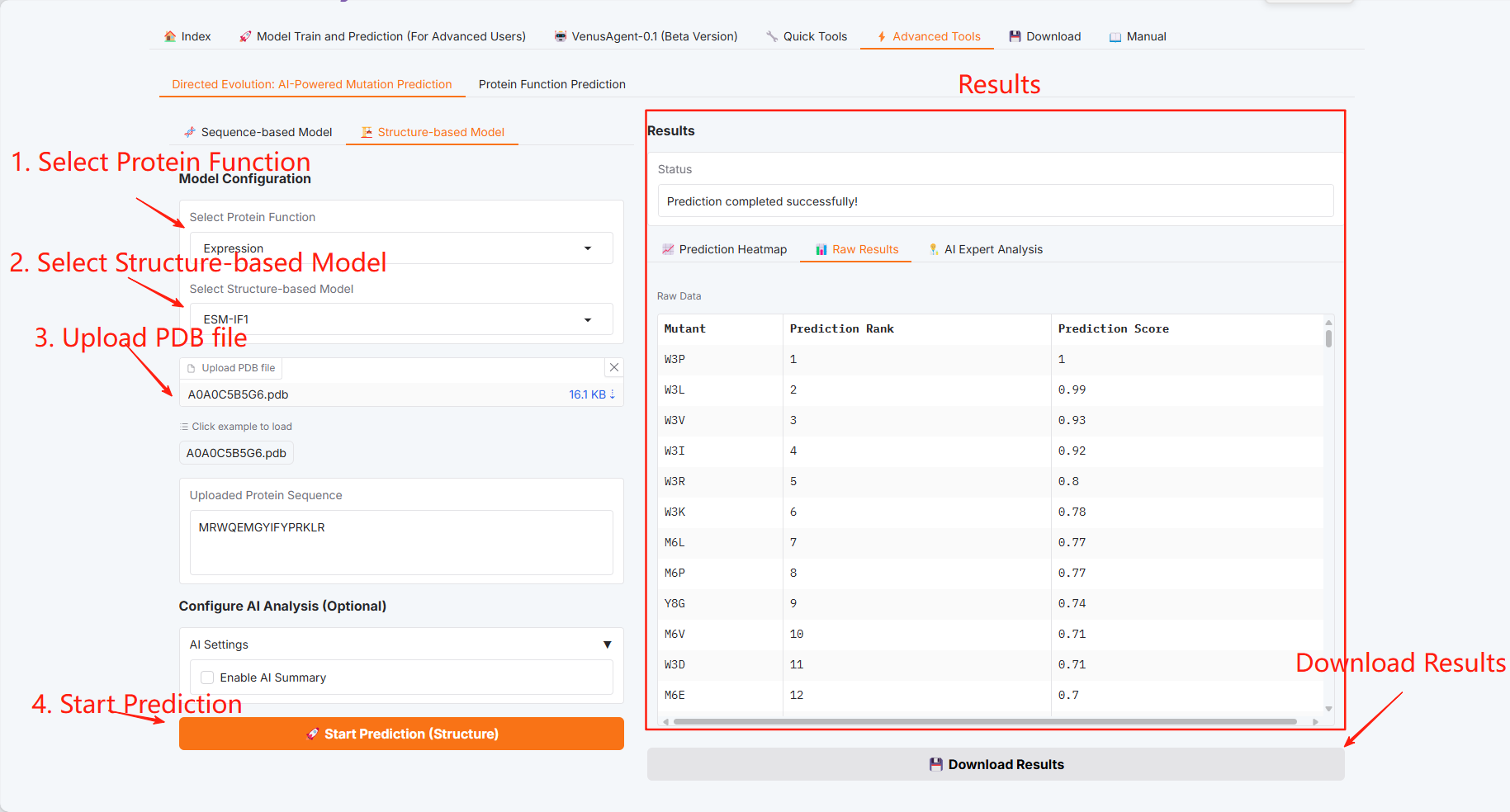
Directed Evolution: AI-Powered Mutation Prediction
Sequence-based Model
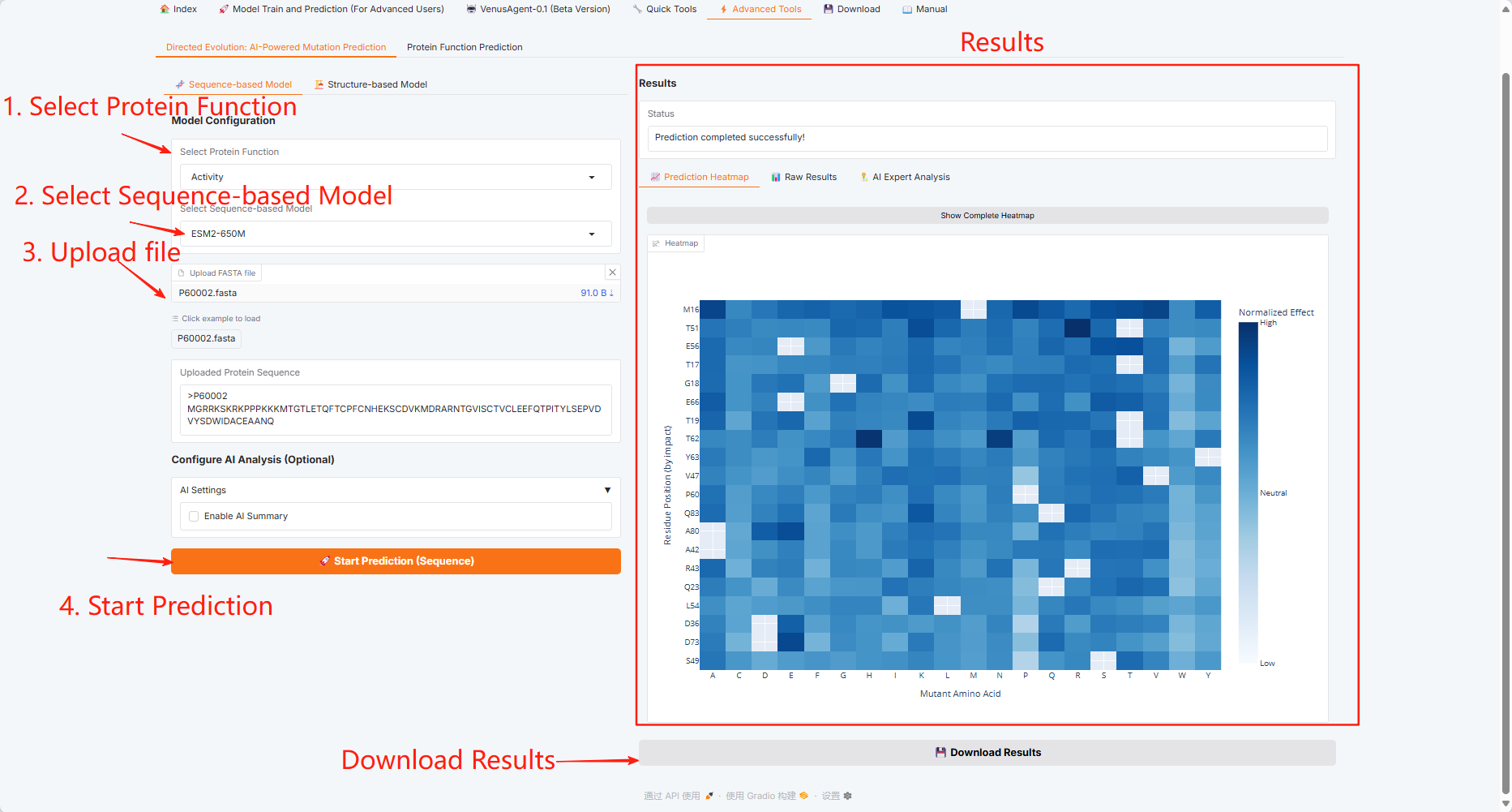
Structure-based Model
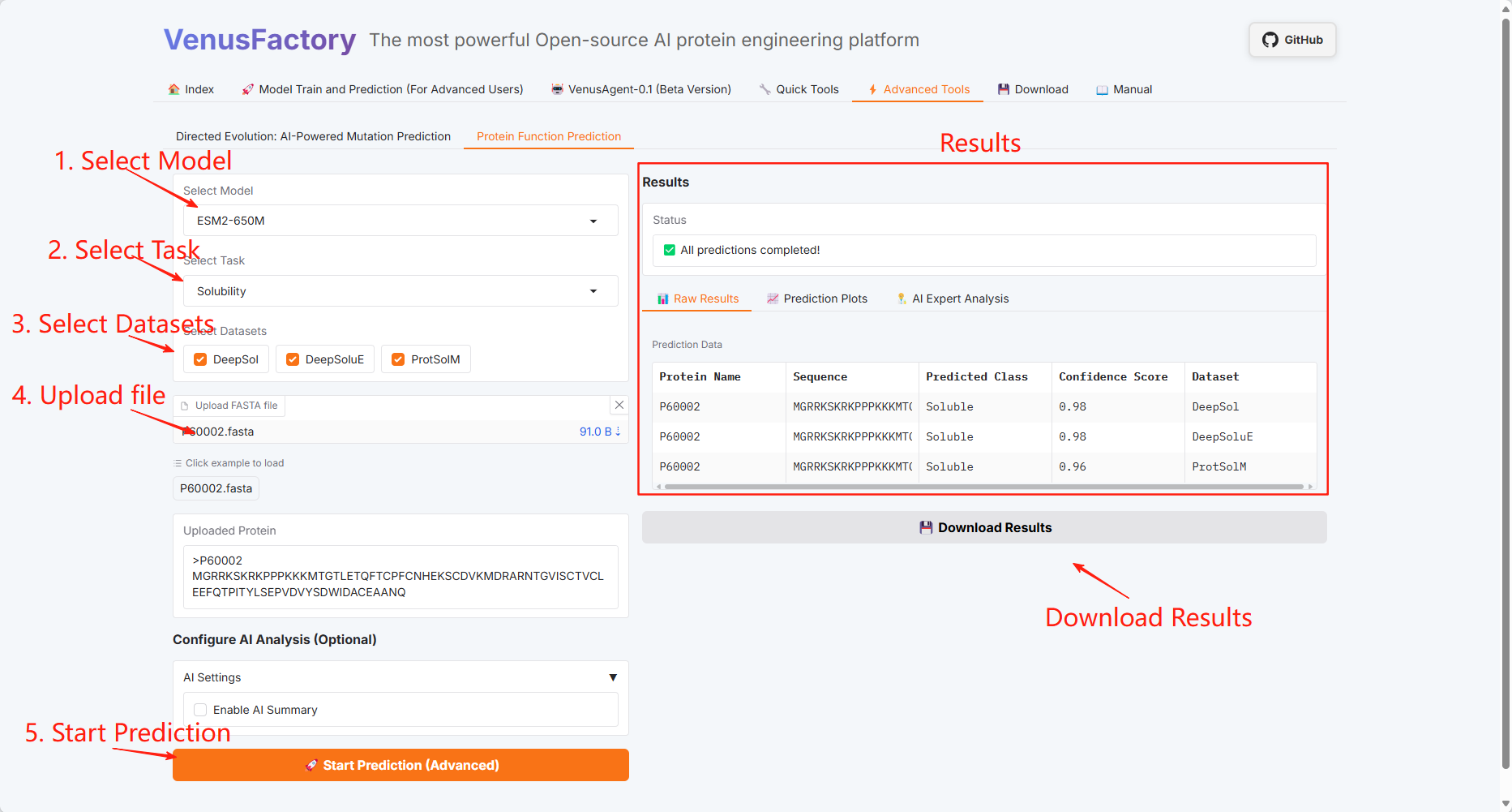
Protein Function Prediction
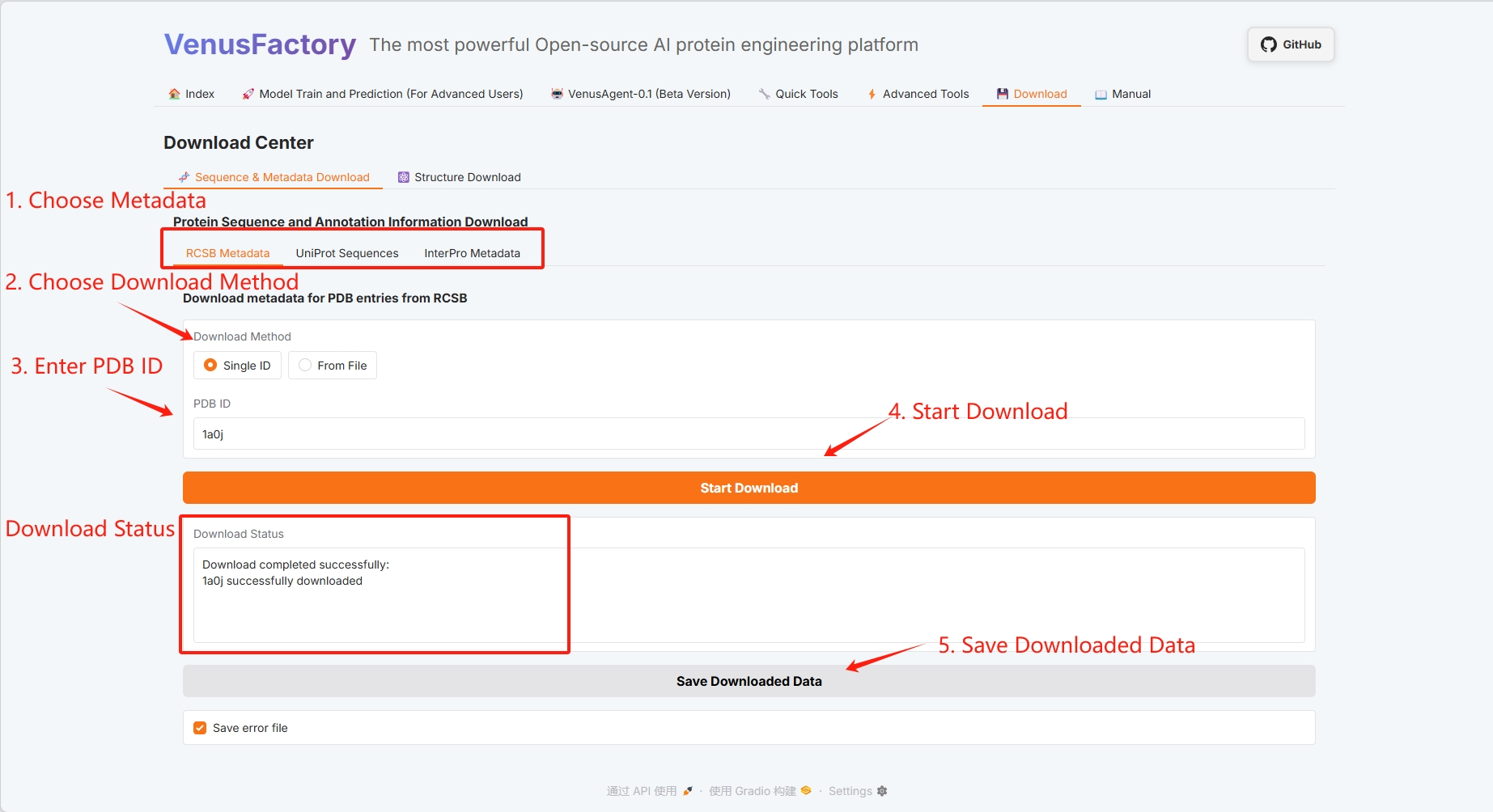
3.7 下载
点击 Download 模块,可以在该界面下载蛋白质数据。
三、交流探讨
🖌️ 如果大家看到优质项目,欢迎后台留言推荐!另外,我们还建立了 AI4S 交流群,欢迎小伙伴们扫码备注【AI4S】入群探讨各类技术问题、分享应用效果↓

引用信息
本项目引用信息如下:
@inproceedings{tan-etal-2025-venusfactory,
title = "{V}enus{F}actory: An Integrated System for Protein Engineering with Data Retrieval and Language Model Fine-Tuning",
author = "Tan, Yang and Liu, Chen and Gao, Jingyuan and Wu, Banghao and Li, Mingchen and Wang, Ruilin and Zhang, Lingrong and Yu, Huiqun and Fan, Guisheng and Hong, Liang and Zhou, Bingxin",
editor = "Mishra, Pushkar and Muresan, Smaranda and Yu, Tao",
booktitle = "Proceedings of the 63rd Annual Meeting of the Association for Computational Linguistics (Volume 3: System Demonstrations)",
month = jul,
year = "2025",
address = "Vienna, Austria",
publisher = "Association for Computational Linguistics",
url = "https://aclanthology.org/2025.acl-demo.23/",
doi = "10.18653/v1/2025.acl-demo.23",
pages = "230--241",
ISBN = "979-8-89176-253-4",
}