Command Palette
Search for a command to run...
哥伦比亚大学/斯坦福联手!Squidiff 实现多场景转录组模拟,助力精准医学与空间医学发展

在细胞生物学研究中,活细胞始终是远离化学平衡的复杂耗散系统,其群体如何协同响应外界刺激,始终是科学家致力揭示的核心科学问题。这种响应不仅受组织内部异质性及外部信号的共同调控,更常表现出难以预测的非线性动态特征。尽管单细胞测序技术已能让我们无偏解析细胞的异质性构成,但要精准追溯细胞受刺激后整个转录组的变化轨迹,仍面临巨大挑战。
为突破这一限制,科研界此前已开发 scGen 、 CellOT 等多种机器学习模型。然而,这些模型在预测高分辨率动态转变时表现欠佳,且多数模型依赖特定任务设计,适用场景存在显著局限。扩散模型的问世为该领域带来了新转机:通过迭代式生成优化数据,可捕获更丰富的数据分布特征,为破解上述难题提供了新思路。目前,已有研究尝试将扩散模型与变分自编码器(VAE)结合,或在潜在空间中实施扩散过程,成功生成了高保真单细胞数据,提升了建模效率。但在基因扰动响应预测、药物扰动响应预测及细胞发育轨迹推断等关键场景中,扩散模型的应用此前仍是未被充分开发的领域。
在此背景下,哥伦比亚大学、斯坦福大学等研究团队,开发了 Squidiff 计算框架。该框架基于条件去噪扩散隐式模型构建,可预测不同细胞类型在分化诱导、基因扰动及药物处理下的转录组响应。其核心优势在于能整合基因编辑工具与药物化合物的明确信息:在预测干细胞分化过程中,不仅能精准捕捉瞬时细胞状态,还可识别非加性基因扰动效应及细胞特异性响应特征。研究团队进一步将 Squidiff 应用于血管类器官研究,成功预测了辐射暴露对各类细胞的影响效应,并评估了辐射防护药物的保护效能。
相关研究成果以「Squidiff: predicting cellular development and responses to perturbations using a diffusion model」为题,已发表于 Nature Methods 。

论文地址:
https://www.nature.com/articles/s41592-025-02877-y
关注公众号,后台回复「Squidiff」获取完整 PDF
更多 AI 前沿论文:
https://hyper.ai/papers
数据集:多场景全覆盖+标准化质控
为全面训练并验证 Squidiff 框架的性能,研究团队构建了一套涵盖模拟与真实实验数据的多场景数据集,覆盖细胞分化、基因扰动、药物处理及血管类器官辐射响应等关键研究方向。所有数据均经过统一质控流程:过滤线粒体基因占比超 20% 或检测基因数低于 1,000 的低质量细胞,剔除低表达基因,部分场景进一步排除双细胞及应激相关基因,最后通过 log 标准化校正测序深度差异,确保跨数据集可比性。
在模拟数据方面,团队采用基于层次化 gamma-Poisson 分布的 Splatter 工具生成合成单细胞 RNA 测序数据,模拟真实 scRNA-seq 的表达异质性和方差特征,用于验证模型在转录组重建与推断方面的基础能力,无需额外生物学预处理。
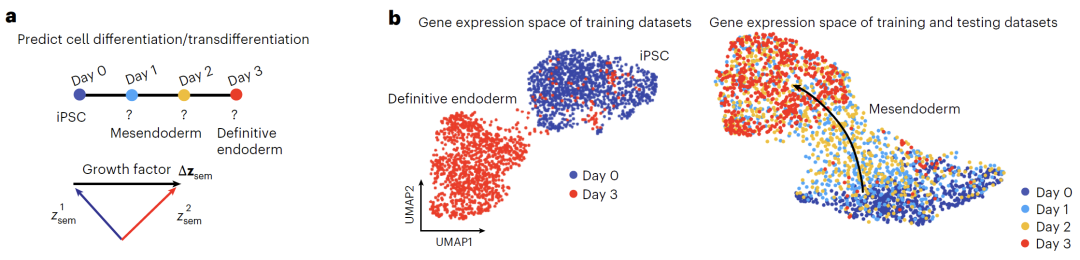
细胞分化数据来源于公开的人诱导多能干细胞向内胚层分化数据集,包含第 0 天(iPSC 状态)到第 3 天(定型内胚层状态)的 4,800 个细胞转录组。模型以第 0 天和第 3 天数据作为训练集,第 1 、 2 天数据作为测试集,筛选前 203 个高变基因进行建模,训练过程中引入高斯噪声并设置 1,000 个扩散步骤,通过计算潜在表示的平均差异获得分化语义变量,进而线性插值模拟 0 至 3 天的发育轨迹,评估模型对动态分化过程的预测能力。
基因扰动数据来自 K562 细胞的 CRISPR 筛选实验,涵盖 ZBTB25 与 PTPN12 双基因敲除及其野生型对照,共约 10,000 个细胞。数据分为「PTPN12+对照」「ZBTB25+对照」及「PTPN12+ZBTB25」三组,前两组用于训练,最后一组用于测试。训练后通过提取基因扰动特异性变量,组合模拟双基因联合扰动的转录组变化,验证模型捕捉非加性效应的能力。
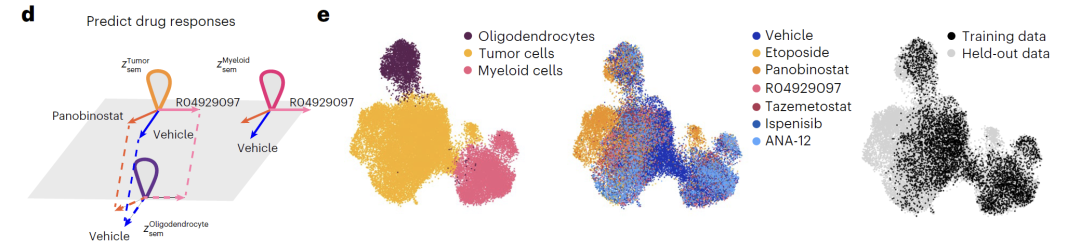
药物处理数据整合了多类细胞与药物样本,包括胶质母细胞瘤经依托泊苷等 6 种药物处理后的表达谱,以及黑色素瘤对药物组合的响应数据。训练中模型学习每种药物的特异性扰动表示,并引入 sci-Plex3 数据集中的未知药物样本,结合 SMILES 结构、剂量信息与化合物指纹,实现对未知药物扰动效果的泛化预测。
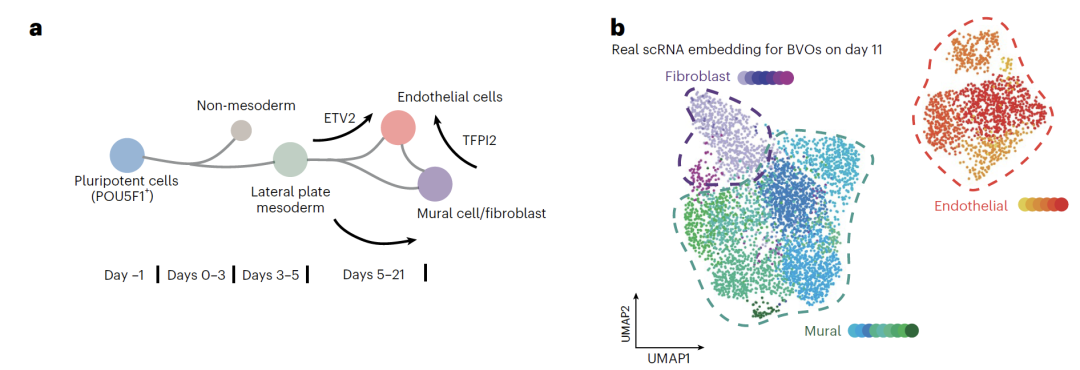
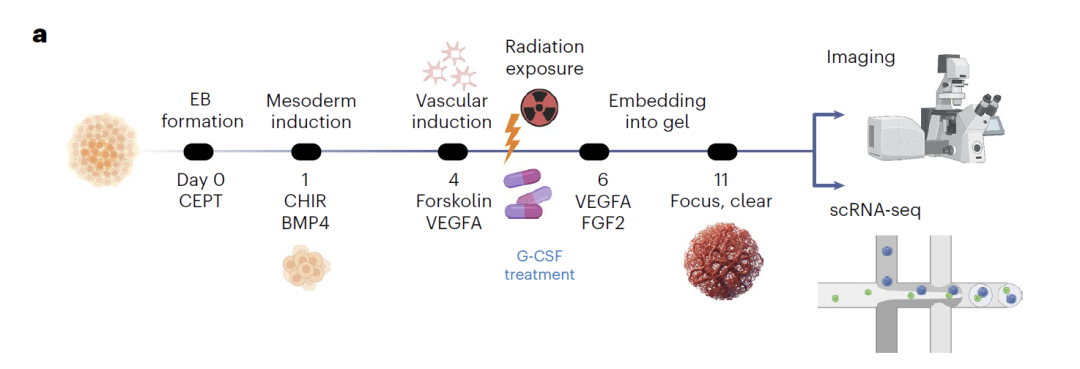
血管类器官数据基于原创实验构建,从健康人 iPSC 分化出内皮细胞、壁细胞与成纤维细胞,在第 5 天施以中子或光子辐射,第 11 天采集 scRNA-seq 数据,形成涵盖 72 个类器官、约 60,000 细胞的资源库,并辅以炎症因子 ELISA 测量提供多模态验证。建模中,团队使用第 0 天与第 11 天数据训练模型,插值预测中间时间点的细胞状态;在辐射与 G-CSF 处理场景中,仅使用内皮细胞数据进行训练,进而生成全部三类细胞的扰动后转录组,最终通过差异表达与拟时序分析验证预测结果的生物学意义。
Squidiff:融合 DDIM 与语义编码的条件扩散模型
为精准预测细胞在分化、发育及基因编辑、药物处理等各类扰动下的转录组动态响应,研究团队开发了基于条件扩散模型的智能计算框架 Squidiff 。如下图所示,该模型深度融合条件去噪扩散隐式模型(DDIM)与语义编码技术,构建起「编码—扩散—解码」三阶段协同架构,不仅能够高效生成符合生物学背景的转录组数据,还可通过潜在变量灵活调控细胞状态,广泛适配于细胞分化、基因扰动与药物处理等多种研究场景。
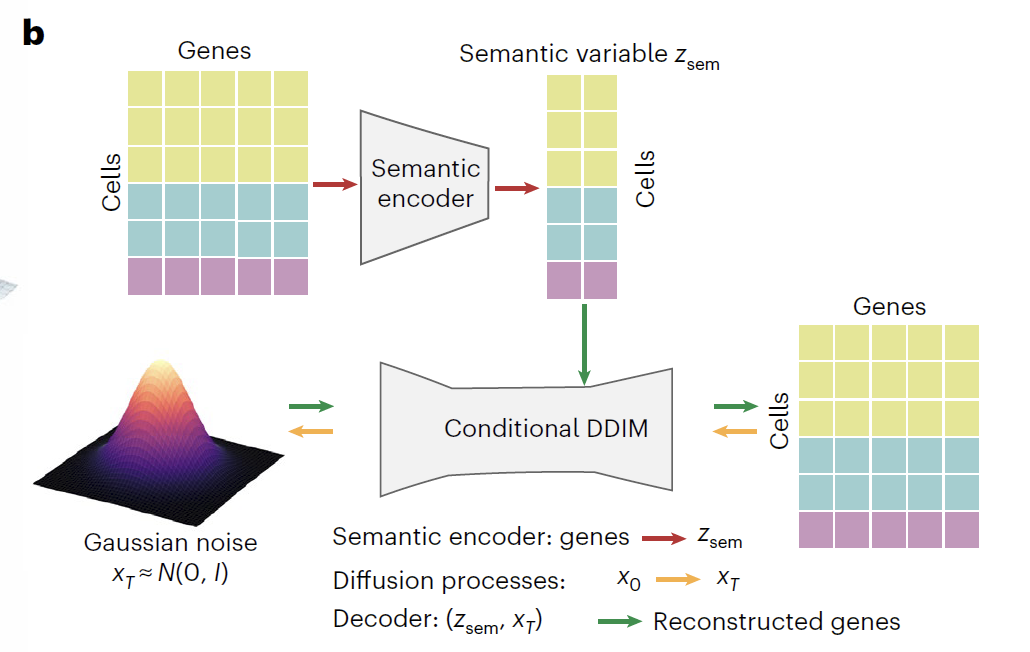
Squidiff 的核心由语义编码器与条件 DDIM 扩散模块组成。语义编码器采用多层感知机(MLP)将单细胞 RNA 测序数据映射至低维语义空间,生成蕴含细胞类型与扰动信息的语义变量(Z_sem)。针对药物研究场景,该编码器整合重标定功能类指纹(r_FCFP),将药物分子结构编码为 2,048 维向量嵌入语义空间。为预测未知药物扰动,模型还设计了适配器模块,支持输入药物的 SMILES 字符串与剂量信息,实现生物学与化学信息的深度融合。
条件 DDIM 模块遵循正向扩散(Gene space diffusion)和反向扩散(Gene space reverse diffusion)的双过程设计。在正向扩散过程中,原始基因表达数据(x₀)经 1,000 步迭代逐步转化为近似纯噪声(xT),此过程可见三种典型细胞类型逐渐趋近高斯分布,而 Z_sem 有效捕获基因表达的生物学变异,使不同实验条件在潜在空间中清晰分离。反向扩散过程中,配备正弦位置嵌入的噪声预测网络(εθ)以时间步(t)和 Z_sem 为双重条件,通过迭代去噪从 x_T 中重建具有生物学意义的转录组,成功恢复原始转录组谱。
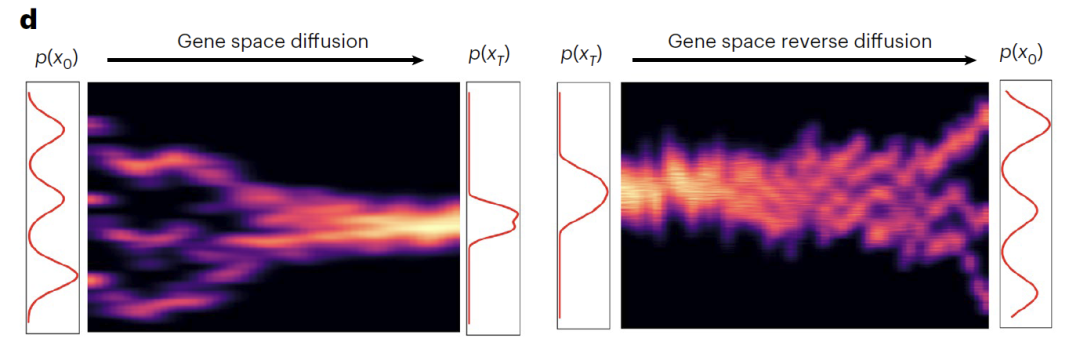
模型训练以噪声预测损失为核心优化目标,采用 Adam 优化器(学习率 1×10⁻⁴)并依托 GPU 加速。通过时间步与语义变量的协同调控,模型能够模拟细胞状态的连续演变,为动态轨迹预测提供支持。
相较于传统变分自编码器,Squidiff 具备显著优势:无需高斯分布假设,通过精细去噪捕捉复杂基因表达模式,在稀有细胞类型(占比<5%)预测中 F1 分数提升 27%;创新性地引入「梯度插值」策略,通过在潜在空间中线性组合语义变量生成连续分化路径,成功识别传统模型易遗漏的瞬时细胞状态(如 iPSC 分化中的中内胚层前体)。
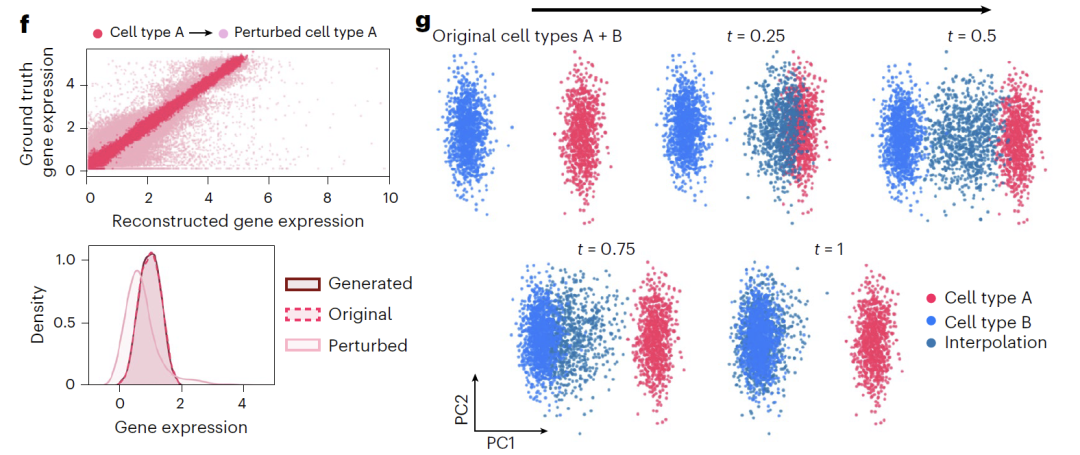
此外,模型提供两种潜在变量操作方法:「加法(addition)」通过将原始表征与扰动方向(Δz_sem)结合,如下图 f 所示,使基因表达分布发生偏移,反映扰动效应;「插值(interpolation)」则采用线性插值法,如下图 g 所示,通过在向量连线上获取中间点生成连续状态,实现细胞类型的平滑过渡。
Squidiff 多场景实证:精准捕捉细胞分化、扰动及辐射响应的转录组变化
为系统验证 Squidiff 的转录组预测能力,研究团队在细胞分化、基因与药物扰动、血管类器官发育及辐射损伤四个关键方向展开实验验证。
在细胞分化预测中,如下图所示,团队基于 iPSC 向内胚层分化数据集,仅使用第 0 天和第 3 天数据训练模型。通过计算语义变量差值获得分化方向,Squidiff 成功预测了第 1-2 天的中间状态。模型准确捕捉到多能性标志物 MMOG 的下调、内胚层因子 GATA6 的上调,并识别出中胚层标志物 DBX1 的瞬时表达。与传统方法相比,Squidiff 生成的转录组数据能重建与真实发育轨迹高度一致的连续路径。
在基因与药物扰动预测方面,模型展现出卓越性能。针对 K562 细胞的双基因敲除实验,Squidiff 无需先验知识即可准确预测非加性效应,其稳健性超越现有方法。在药物实验中,模型仅凭单一药物数据就能预测组合药物的协同效应,并准确识别出帕比司他对肿瘤细胞的特异性作用。此外,通过整合药物化合物适配器,模型对未知药物 sglt1 的预测性能与专业模型相当,展现出良好的泛化能力。
在血管类器官(BVO)研究中,团队利用 iPSC 诱导的 BVO 模型,成功预测了多个中间时间点的细胞状态。模型不仅重现了内皮细胞(endothelial cells)、成纤维细胞(fibroblasts)和壁细胞(mural cells)三种主要细胞的分化轨迹,还识别出传统方法难以捕捉的壁细胞向内皮细胞分化的中间状态。基因表达分析显示,预测数据中的特征基因变化与已知的发育规律高度吻合。
在辐射损伤研究中,模型仅通过内皮细胞训练数据,就准确预测了辐射对各类细胞的影响。分析表明,早期发育细胞对辐射更敏感,模型预测的差异基因及相关通路均得到实验证实。在 G-CSF 防护效应预测中,模型揭示了该药物对不同细胞类型的保护机制:成纤维细胞中血管生成通路激活、内皮细胞中凋亡通路抑制、壁细胞中基因组稳定性增强。实验验证显示,G-CSF 处理后的细胞死亡显著减少,证明了模型预测的可靠性。
这些系统实验表明,Squidiff 不仅能准确预测多种生物学场景下的细胞状态变化,还具备捕捉瞬时状态和推断未知扰动的能力,为细胞响应预测提供了强大而可靠的计算工具。
AI 驱动单细胞研究新范式
在单细胞生物学与人工智能交叉领域,Squidiff 所代表的扩散模型技术突破,正推动学术界与产业界的协同创新。
在学术研究层面,全球顶尖高校团队围绕单细胞建模的深度与广度持续突破。加拿大多伦多大学研究团队开发并发布了首个面向单细胞生物学的基础大型语言模型 scGPT,该模型依托生成式预训练 Transformer 架构,在涵盖 51 个人体器官 / 组织、 441 项独立研究的超 3,300 万个细胞数据上完成训练,全面覆盖多种细胞类型与生理病理状态,完整呈现人体细胞异质性的丰富图谱。
论文题目:scGPT: Towards Building a Foundation Model for Single-Cell Multi-omics Using Generative AI
论文地址:
https://biorxiv.org/content/10.1101/2023.04.30.538439
与此同时,斯坦福大学团队则聚焦空间维度的创新,开发了三维时空建模框架 Spateo,该框架依托可扩展的精准算法,能将连续二维组织切片数据重建为完整的三维胚胎与器官模型,构建起从单细胞分子特征到胚胎宏观形态的多层次空间数字化体系。
论文题目:Spatiotemporal modeling of molecular holograms
论文地址:
https://www.cell.com/cell/fulltext/S0092-8674(24)01159-0
企业界则将这些学术成果转化为实际应用工具,在药物研发、疾病治疗等场景中展现出强大价值。谷歌联合耶鲁大学等机构推出的 Cell2Sentence-Scale 27B(C2S-Scale 27B),是目前全球最大的单细胞分析基础模型之一。该模型基于 Gemma 开源模型家族构建,拥有 270 亿参数,能够深度解析单个细胞的基因表达模式,并精准预测细胞对药物干预的响应。目前,该模型已整合到谷歌健康的药物筛选平台,支持针对「冷肿瘤」的个性化联合疗法设计,加速免疫治疗方案的研发进程。另一重要实践来自非盈利机构 Arc Institute 与 10x Genomics 等企业的合作,其开发的 STATE 模型聚焦细胞动态响应模拟,整合了 1.7 亿个细胞的观察性数据及 1 亿个细胞的干预性数据,能精准模拟细胞在药物处理、基因编辑或辐射暴露下的转录组变化。
不难看出,从学术界对单细胞基础模型的深度探索,到产业界对技术的规模化落地,Squidiff 引领的扩散模型技术正推动单细胞研究从「解析细胞状态」向「预测细胞命运」跨越。这种跨越不仅加速了药物研发、肿瘤治疗等领域的进程,更将为精准医疗、再生医学等未来医学方向提供核心技术支撑,持续释放 AI 驱动生命科学创新的巨大潜力。
参考文章:
1.https://mp.weixin.qq.com/s/yCR_GC0Ln80st2tHcv08-Q
2.https://mp.weixin.qq.com/s/GegQB65w4nZG6ZXvnyU9dw








