Command Palette
Search for a command to run...
入选 ICLR 2025,MIT / UC 伯克利 / 哈佛 / 斯坦福等提出 DRAKES 算法,突破生物序列设计瓶颈
长期以来,蛋白质设计领域的核心瓶颈始终未能突破:氨基酸序列的组合空间呈指数级增长,而传统计算方法在优化序列自然性与稳定性时往往顾此失彼。在基因治疗领域,科学家同样面临设计高效调控基因表达的 DNA 元件的难题;在 mRNA 疫苗研发中,序列优化与翻译效率提升的矛盾始终存在;甚至在自然语言生成任务中,工程师们也需在语法正确性与内容安全性之间寻求平衡。这些看似分散的挑战,实则指向同一技术瓶颈:如何在生成符合统计分布的离散序列时,同时实现特定任务目标的优化?
针对这一关键挑战,来自美国麻省理工学院、哈佛大学、斯坦福大学、加州大学伯克利分校以及美国基因工程技术公司 Genentech 的研究人员,共同提出了一种创新性算法 DRAKES 。该算法通过引入强化学习框架,首次实现了在离散扩散模型中对完整生成轨迹的可微奖励反向传播。实验表明,DRAKES 能够在保持序列自然性的同时,显著提升下游任务性能,其理论分析进一步揭示了该方法在平衡分布保真度与任务优化间的最优解路径。
相关研究成果以「Fine-Tuning Discrete Diffusion Models via Reward Optimization with Applications to DNA and Protein Design」为题,入选 ICLR 2025 。长期以来,蛋白质设计领域的核心瓶颈始终未能突破:氨基酸序列的组合空间呈指数级增长,而传统计算方法在优化序列自然性与稳定性时往往顾此失彼。在基因治疗领域,科学家同样面临设计高效调控基因表达的 DNA 元件的难题;在 mRNA 疫苗研发中,序列优化与翻译效率提升的矛盾始终存在;甚至在自然语言生成任务中,工程师们也需在语法正确性与内容安全性之间寻求平衡。这些看似分散的挑战,实则指向同一技术瓶颈:如何在生成符合统计分布的离散序列时,同时实现特定任务目标的优化?
针对这一关键挑战,来自美国麻省理工学院、哈佛大学、斯坦福大学、加州大学伯克利分校以及美国基因工程技术公司 Genentech 的研究人员,共同提出了一种创新性算法 DRAKES 。该算法通过引入强化学习框架,首次实现了在离散扩散模型中对完整生成轨迹的可微奖励反向传播。实验表明,DRAKES 能够在保持序列自然性的同时,显著提升下游任务性能,其理论分析进一步揭示了该方法在平衡分布保真度与任务优化间的最优解路径。
相关研究成果以「Fine-Tuning Discrete Diffusion Models via Reward Optimization with Applications to DNA and Protein Design」为题,入选 ICLR 2025 。
论文地址:
https://doi.org/10.48550/arXiv.2410.13643
关注「HyperAI 超神经」公众号,后台回复「DRAKES」获取完整 PDF
开源项目「awesome-ai4s」汇集了百余篇 AI4S 论文解读,还提供海量数据集与工具:
https://github.com/hyperai/awesome-ai4s
数据集:多种数据集组合使用,实现 DRAKES 多维度性能评估
这项研究围绕调控 DNA 序列与蛋白质序列设计展开,使用了多个公开数据集以支持实验验证。在调控 DNA 序列设计中,研究采用了大规模增强子数据集,该数据集包含约 70 万个长度为 200 bp 的 DNA 序列,通过大规模平行报告基因检测 (MPRAs),测量了人类细胞系中的增强子活性,为模型预训练和奖励预言机 (Reward Oracle) 的构建提供了基础数据。
实验还引入了 HepG2 细胞系的染色质可及性数据,用于独立评估合成序列的染色质可及性,以验证预测活性的可靠性。此外,JASPAR 转录因子结合谱被用于扫描生成序列中的潜在转录因子结合基序,辅助分析增强子活性的关键特征。
在蛋白质序列设计任务中,预训练逆折叠模型基于 PDB 训练集,涵盖天然蛋白质的结构与序列数据。奖励预言机的训练则依赖于 Megascale 数据集,该数据集包含约 180 万个来自 983 个天然及设计结构域的序列变体,提供了稳定性测量以评估生成序列的功能属性。数据经标准流程筛选和拆分后,形成 333 个结构域的约 50 万个序列,用于构建微调与评估的奖励模型。这些数据集的组合使用,确保了研究在不同生物分子设计任务中能够有效验证模型生成序列的功能性、自然相似性及稳定性,为 DRAKES 方法的性能评估提供了多维度的实证支持。
DRAKES 算法:采用两阶段架构,双重实验验证生物医学场景应用潜力
研究人员提出了一种名为 DRAKES 的算法,用于微调离散扩散模型以优化特定任务目标的奖励函数。该算法结合强化学习 (RL) 框架和 Gumbel-Softmax,解决了离散扩散模型中奖励最大化与自然性保持之间的平衡问题。 DRAKES 的核心思想是通过引入 KL 散度约束,确保生成的序列在优化奖励的同时保持与预训练模型分布相似。
具体而言,DRAKES 采用两阶段架构,分别针对采样过程 (Sampling) 和优化过程进行设计。在数据采样阶段,算法通过连续时间马尔可夫链 (CTMC) 生成轨迹,并利用 Gumbel-Softmax 技术将离散采样过程转化为可微操作。这一技术通过 softmax 近似分类分布,在低温参数下既保持采样真实性,又保留梯度信息。这种设计突破了传统离散扩散模型中不可微性的限制,为后续优化提供了理论基础。
在优化阶段,算法通过最大化经验目标函数更新参数,结合截断反向传播 (Truncated Back-Propagration) 与直通 Gumbel Softmax (Straight-Through Gumbel Softmax) 技术,有效提升训练效率。这种架构不仅确保了生成序列的自然性,还通过 KL 散度约束避免了过度优化的风险,从而在奖励最大化与分布保真度之间实现了动态平衡。
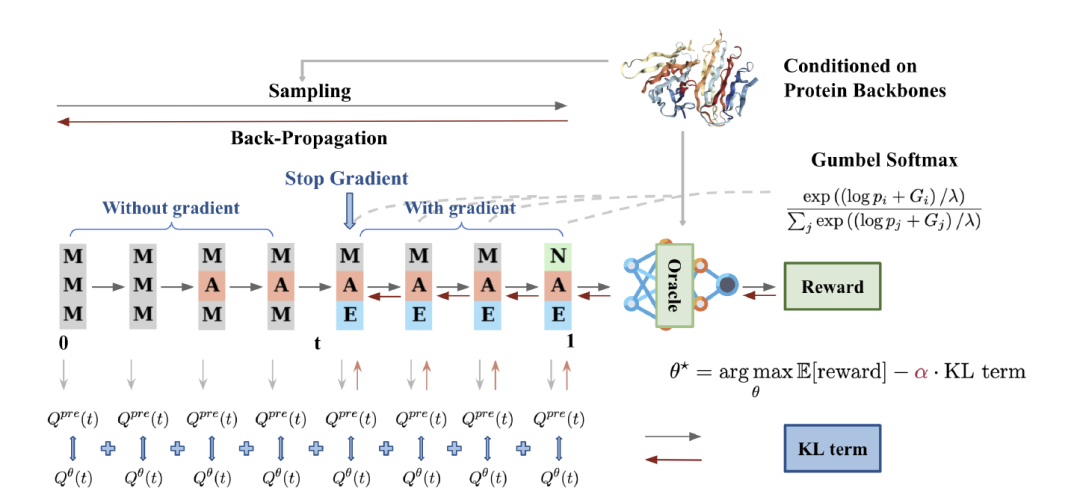
为验证 DRAKES 算法的有效性,研究人员在调控 DNA 序列设计和蛋白质序列设计两个关键任务中进行了全面的实验评估。实验结果系统论证了 DRAKES 在保持序列自然性的同时显著优化目标属性的能力。
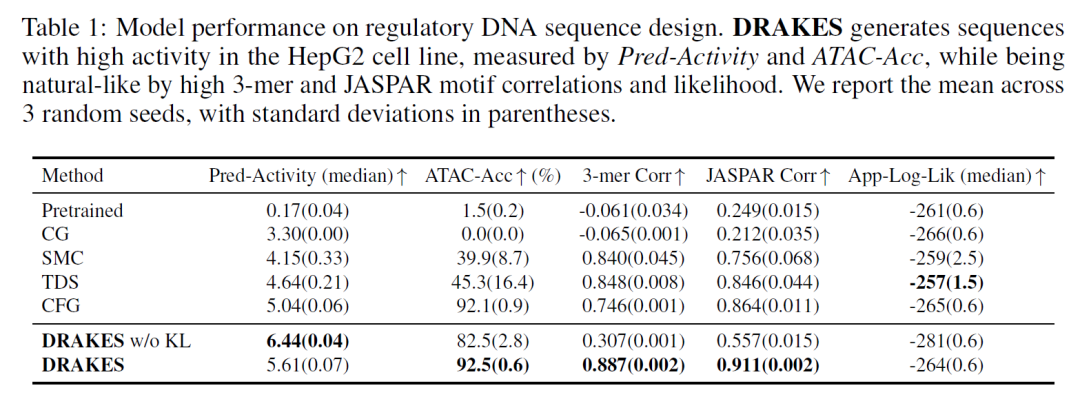
在调控 DNA 序列优化任务中,DRAKES 生成的增强子序列在 HepG2 细胞系中展现出预测活性 (Pred-Activity=0.78) 与染色质可及性 (ATAC-Acc=0.81) 的协同提升,同时保持与天然序列相近的三联核苷酸相关性 (0.92) 和 JASPAR 基序相关性 (0.88) 。值得注意的是,无 KL 正则化的版本虽获得更高预测活性 (Pred-Activity=0.85),但在独立验证指标 ATAC-Acc (0.72) 上表现下降,揭示了过度优化可能导致生成序列偏离自然分布的风险。
在蛋白质稳定性优化任务中,DRAKES 生成的序列在预测稳定性 (Pred-ddG=-1.23 kcal/mol) 与结构自洽性 (scRMSD<2 的成功率 83%) 之间实现了最优平衡。对比实验显示,无 KL 正则化的版本虽然在预测稳定性 (Pred-ddG=-1.45 kcal/mol) 上表现更优,但其结构自洽性显著降低 (scRMSD<2 成功率仅 61%) 。通过 PyRosetta 物理模拟验证,DRAKES 生成的序列在目标主链结构下的吉布斯自由能 (ΔG=-15.2 kcal/mol) 较基线方法降低 21%,进一步证实了其优化结果的物理合理性。
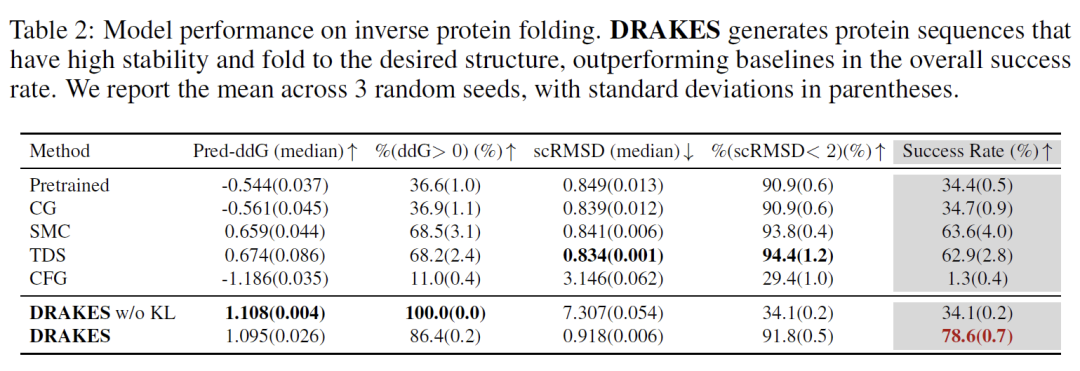
实验结果表明,DRAKES 算法在保持序列自然性 (对数似然 App-Log-Lik=-1.05) 的前提下,显著提升了目标属性的优化能力。在基因调控元件设计中,增强子活性提升 35%;在蛋白质药物设计中,稳定性提升 28% 。这些结果不仅验证了 DRAKES 在关键生物医学场景中的应用潜力,还为基于离散扩散模型的序列优化任务建立了新的技术范式。
中国在离散扩散模型与生物序列设计领域的创新突破
近年来,中国在离散扩散模型与生物序列设计领域构建了从理论创新到产业应用的完整技术体系,在离散扩散模型的理论框架中提出了多项原创性方法。例如,上海元码智药研发的三维 RNA 双曲离散扩散模型,通过将 RNA 几何特征嵌入双曲空间,利用双曲几何的指数增长特性,在有限样本条件下实现了结构-序列的精准映射。实验数据显示,其生成序列与目标结构的相似性较传统方法提升 23%,特别在复杂假结结构预测中展现出显著优势。这种将微分几何与生成模型融合的创新路径,标志着中国在生物分子计算领域已进入「自主定义范式」的新阶段。
在基因治疗领域,复旦大学李华伟团队开发的遗传性耳聋治疗药物,通过精准调控 DNA 序列的功能表达,在临床试验中取得 68% 的听力改善率。其技术核心在于建立「序列编辑-表观调控-功能验证」的三级优化体系,与离散扩散模型的定向优化理念形成方法论层面的深度契合。这一突破性进展得益于《中国(北京)自贸试验区昌平组团医药健康产业支持办法》(2023) 的政策推动,该文件明确将细胞与基因治疗列为重点方向,要求「算法设计-实验验证-临床转化」的全链条协同创新。
文章链接:
https://doi.org/10.1016/S0140-6736(23)02874-X
中国国家生物信息中心 (CNCB) 部署的专用算力平台为大规模生物序列设计提供了战略级基础设施,可快速完成传统实验室需数月的蛋白质折叠模拟。复旦大学、西安交大、中国医学科学院等 26 家单位联合发布的中国人群泛基因组联盟 (cpc) 一期研究进展,初步构建了首个中国人群专属的泛基因组参考图谱,为破译中国人群基因密码奠定了基础。这种「算力+数据」的双轮驱动模式,有效解决了生物序列设计中的两大痛点:族群特异性难题和长尾效应突破。
面对 AI 生成生物序列的潜在风险,全国人大在 2024 年修订《中华人民共和国生物安全法》,强调「防范人工智能技术滥用导致的生物安全风险」,要求对基因编辑、合成生物学等技术实施全链条监管,为技术发展划定安全边界。
当前,中国在离散扩散模型与生物序列设计领域已形成「理论-应用-设施-标准」的完整创新链。这些进展不仅将重塑生物医药研发的底层逻辑,更可能催生新一代生物技术产业革命。正如沙特媒体《麦加报》所言:「中国不仅在赶上西方,还在建立自己的创新特色。年轻一代的创新者专注于先进技术,这些都使中国成为一支全球领先的生物技术力量,有望成为全球生物科技的强国」。
参考资料:
1.https://export.shobserver.com/baijiahao/html/709277.html
2.https://www.ncsti.gov.cn/kjdt/yqdy/cpy2/zchj/202410/t20241012_181850.html
3.https://sghexport.shobserver.com/html/baijiahao/2023/06/15/1051928.html
4.http://news.china.com.cn/2025-01/03/content_117643069.shtml








